观光木SSR-PCR反应体系优化及引物筛选
黄芳芳,何长青,闫丽君,汪灵丹,徐刚标
(中南林业科技大学 林学院 林木遗传育种实验室,湖南 长沙 410004)
观光木SSR-PCR反应体系优化及引物筛选
黄芳芳,何长青,闫丽君,汪灵丹,徐刚标
(中南林业科技大学 林学院 林木遗传育种实验室,湖南 长沙 410004)
在单因素试验基础上,采用正交试验优化影响观光木SSR-PCR扩增的5种主要影响因素,获得最佳反应体系(10 μL)为:Mg2+1.0 mmol,dNTP 0.20 mmol,引物0.25 μmol,TaqDNA聚合酶0.75 U,模板DNA 60 ng。利用优化SSR-PCR扩增体系,从12对引物中筛选出5对多态性高、重复性好的引物。这为进一步开展观光木种群遗传多样性研究奠定了前期实验基础。
观光木;SSR;体系优化;引物筛选
观光木Tsoongiodendron odorumChun,属木兰科Magnoliaceae植物,为古老的孑遗树种,是著名的观赏树种,被列为我国二级珍稀濒危重点保护植物。观光木零星分布我国福建、海南、广东、广西、江西南部、贵州南部、云南东南部、湖南南部以及越南等地海拔300~1 100 m山地常绿阔叶林中、林缘以及村旁、房前屋后[1-2]。目前,观光木的研究主要集中在生物量循环、种群生态生理、生殖生物学及系统发育等基础生物学等方面[3-5],近期开展的观光木种群遗传多样性研究主要采用RAPD、ISSR[5-8]标记。由于RAPD和ISSR标记为显性标记,估算的种群遗传学参数是基于种群遗传平衡假说,与真实情况存在偏差。共显性SSR标记是揭示濒危植物遗传多样性、基因流模式及小尺度空间遗传结构首选分子标记[9],因此,为了探讨观光木种群进化机制,预期其小种群未来命运,开展SSR标记分析显得尤为迫切。
本研究拟通过单因素试验和正交试验,旨在建立出一套适合观光木种群遗传多样性分析SSRPCR扩增反应优化体系,利用优化体系对近期开发的12对观光木特异SSR引物[10]进行筛选,以期为进一步基于SSR标记开展观光木种群遗传多样性研究奠定基础。
1 材料与方法
1.1 材 料
观光木新鲜叶片置于密封袋中硅胶干燥处理后,-4℃冰箱保存。参考文献[10]中SSR特异引物 TOD8(F:CCCCATTGAAGTCATTTGAAG,R:ATCCCATGCGATAACCGATA)进行SSR-PCR反应体系优化。
1.2 方 法
1.2.1 基因组DNA提取与检测
观光木基因组DNA提取、检测参考文献[11]。
1.2.2 SSR-PCR扩增
参照文献[9],SSR-PCR扩增初始反应体系(10 μL):50 ng DNA,1 μL 10×PCR buffer,0.2 µmol引物,1.5 mmol MgCl2,0.2 mmol dNTP,1 UTaqDNA聚合酶。PCR扩增程序:94℃预变性5 min,94℃变性45 s,57℃退火50 s,72℃延伸90 s,35个循环;72℃延伸5 min,4℃保存。
1.2.3 单因素试验设计
以SSR-PCR扩增初始反应体系为基础,对影响PCR扩增反应的Mg2+、dNTP、引物、TaqDNA聚合酶、模板DNA 5个主要因素分别进行优化。其中,Mg2+浓度范围为0.5~3.0 mmol,梯度为0.5 mmol; dNTP浓度范围为0.05~0.30 mmol,梯度为0.05 mmol;引物浓度范围为0.10~0.35 µmol梯度为0.05 µmol;Taq酶用量为0.25~1.50 U,梯度为0.25 U;模板DNA用量为15~90 ng,梯度为15 ng。
1.2.4 正交试验设计
在单因素试验确定的各因素最适浓度范围基础上,利用L16(44)正交试验设计(表1),确定适合观光木SSR-PCR扩增的最优反应体系。
1.2.5 退火温度确定
以由Tm值计算出的理论退火温度57℃为标准,进行梯度为1℃的6个退火温度(55℃,56℃,57℃,58℃,59℃,60℃)试验,确定最适退火温度。
1.2.6 扩增产物检测
[12]。
1.2.7 引物筛选
基于优化的观光木SSR-PCR反应体系,对文献[10]中12对引物进行筛选。
1.2.8 最佳反应体系验证
利用已优化的SSR-PCR反应体系,对17株样本进行扩增反应,验证该体系的稳定性。
2 结果与分析
2.1 SSR-PCR反应体系单因素试验
影响PCR反应体系的5个主要因子的6个梯度扩增结果见图1。Mg2+:第1到第3条带随着Mg2+浓度的升高,条带逐渐变亮且数量增多,但是Mg2+浓度再升高条带亮度反而变弱,Mg2+在1.0 mmol时条带清晰明亮且特异性高。dNTP:6个浓度下的dNTP均扩增出产物,浓度越高条带越亮,浓度在0.15 mmol之后亮度基本无变化,故dNTP浓度在0.15~0.20 mmol之间均可。引物:引物浓度在很低时,扩增的条带不清晰,随引物浓度升高,条带逐渐变清晰,浓度达到0.25 µmol时最清晰。TaqDNA聚合酶:不同浓度的Taq酶均可扩增出条带,浓度为0.75U时,扩增条带最清晰。模板DNA:不同模板用量对SSR-PCR扩增影响不明显,当模板DNA用量为60 ng时,条带最清晰。
2.2 SSR-PCR反应体系正交试验
如图2正交试验结果所示,16个不同处理均可扩增出条带,其中第3个处理组合条件下扩增出的条带比较清晰明亮,最终确定为本试验的最优组合,即10 µL体系中含Mg2+1.0 mmol,dNTP 0.20 mmol,引物0.25 µmol,TaqDNA聚合酶0.75 U。
2.3 退火温度
不同退火温度扩增条带见图3,6个温度均能扩增出清晰的条带,条带由亮变暗然后又变亮。其中,58℃时条带最暗,56℃时最亮,据此把引物TOD8最适退火温度确定为56℃。

图1 单因素对 SSR-PCR 扩增的影响Fig.1 Effects of each factor on SSR-PCR
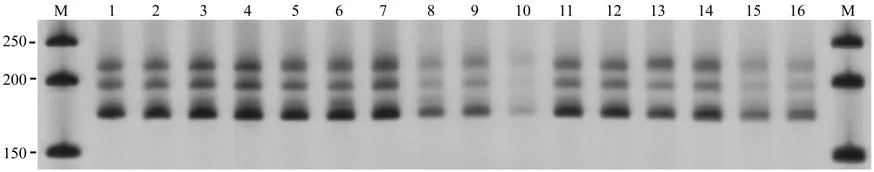
图2 观光木 SSR-PCR 正交试验扩增结果Fig.2 Results of SSR-PCR by orthogonal design of T. odorum
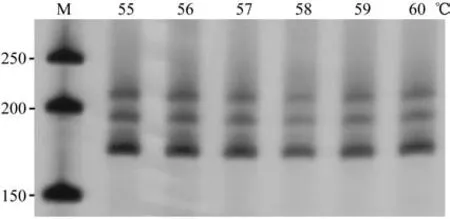
图3 退火温度对 SSR-PCR 的影响Fig.3 Effects of annealing temperature on SSR-PCR
2.4 引物筛选
利用确立的优化体系,筛选出5对条带清晰、重复性好、多态性高的SSR引物,见表2。
2.5 最佳反应体系验证
5对SSR引物对17株观光木样本进行最佳反应体系的验证结果,如图4所示。从图4可以看出,5对引物都能扩增出清晰的条带,说明优化的SSR-PCR反应体系比较稳定,可以满足对观光木SSR遗传多样性研究的需要。
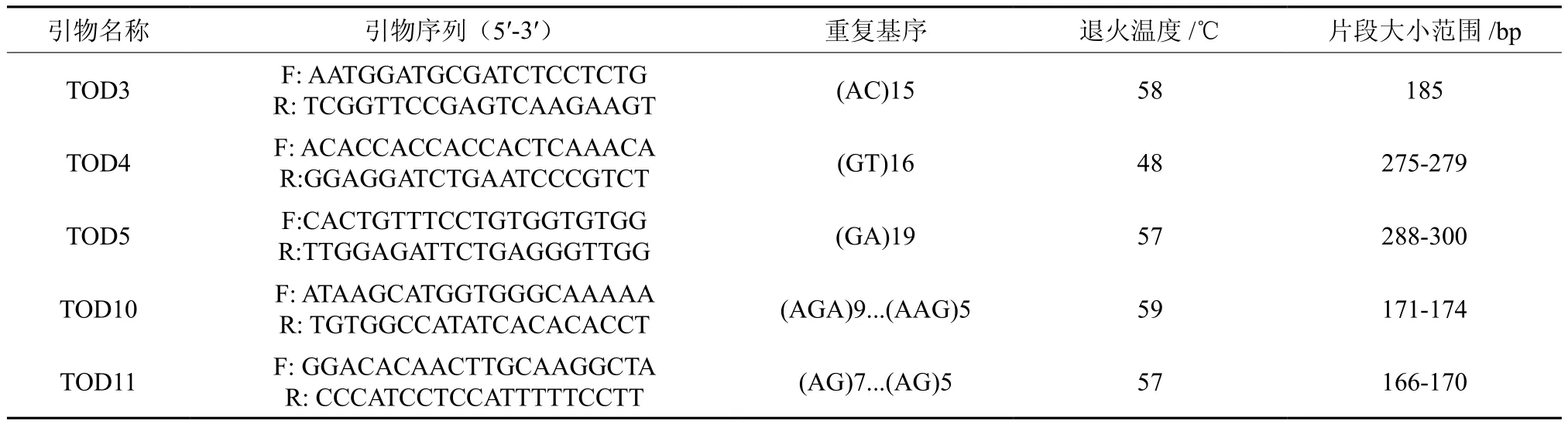
表2 筛选的5对引物Table 2 Screened 5 paris of primes
3 讨 论
SSR分子标记广泛存在于各种真核生物的基因组中,呈共显性遗传,具有丰富的多态性和信息量。SSR标记具有物种特异性强、模板DNA用量少、重复性和稳定性好、操作简单等优点,是目前植物种群遗传学分析和植物辅助育种最理想标记,已广泛应用于种群遗传多样性、分子辅助育种、基因定位、种质资源鉴定、遗传连锁图谱绘制等研究中[13-16]。
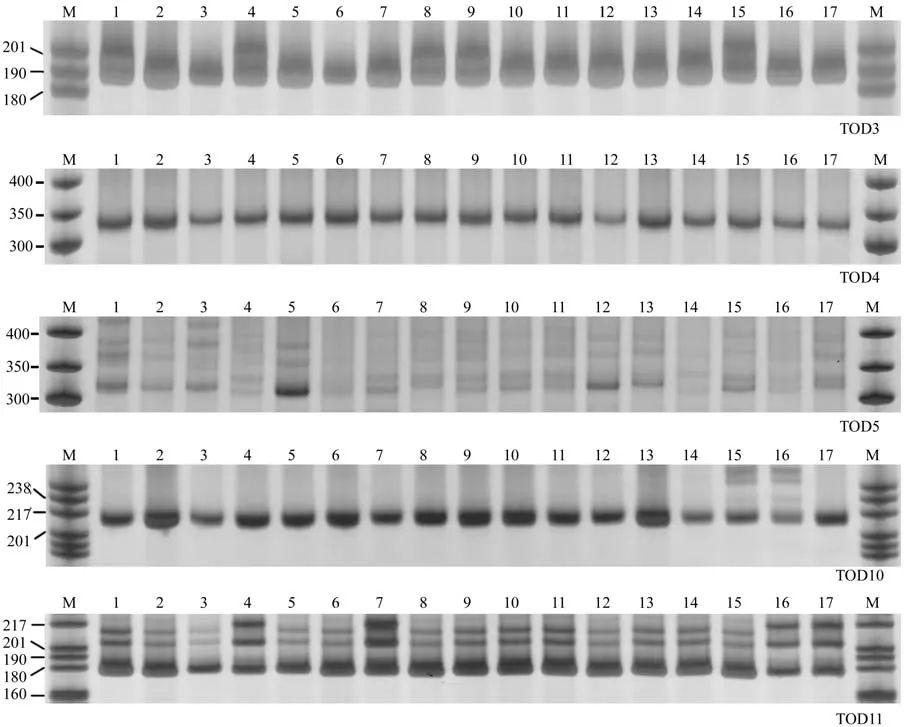
图4 优化的反应体系对17株观光木样本的SSR-PCR扩增结果Fig.4 SSR-PCR amplification results of 17 T. odorum samples by the optimal reaction system
稳定的SSR-PCR扩增反应优化体系,是进一步开展基因分型分析的前提条件[17]。PCR扩增反应受到Mg2+、dNTP、引物浓度,以及模板DNA、TaqDNA聚合酶用量等因素的交互作用影响[18]。PCR扩增反应中,Mg2+是TaqDNA聚合酶的激活剂,浓度过低降低Taq酶活力,过高导致Taq酶催化非特异扩增;dNTP是PCR扩增反应的底物,浓度过低影响扩增效率,过高降低Taq酶活性;引物是PCR特异性反应的关键,浓度过低扩增产物量少,过高促进非特异性扩增并增加引物二聚体的形成;模板是待扩增序列的核酸,浓度过低减少目的序列的扩增量,过高增加非特异性扩增产物;Taq酶是 PCR反应中的催化剂,用量过少靶序列产量将降低,过多导致非特异产物增加[19]。
退火温度是影响SSR引物与模板特异性结合,不同引物因其碱基序列不同,退火温度不同。退火温度太低,导致非特异的DNA片段扩增,特异性差;退火温度太高,引物不能与模板很好地结合,扩增产物很低[20]。本研究通过设计几个不同温度梯度,重复试验,获得了各SSR引物的适宜退火温度。
正交试验设计是针对多因素、多水平的试验设计,从中挑选出部分有代表性的点进行试验,具有高效率、快速、经济特点,能分析各因素水平组合的交互作用,使试验结果简便、科学。本研究在单因素试验基础上,通过正交试验设计优化影响观光木SSR-PCR反应体系各因素水平,建立了一套适用于观光木的最佳SSR-PCR扩增反应体系。利用优化的SSR-PCR反应体系,对来自不同种群的17株观光木样本经过重复试验,从12对引物中筛选出5对条带清晰、多态性丰富、重复性好的引物,为后期观光木遗传多样性、小尺度空间遗传结构、交配系统及基因流分析,以及进一步探讨观光木种群进化历史及预测其片断化小种群的未来命运,奠定了前期实验基础。
参考文献:
[1]Xia NH, Liu YH, Nooteboom HP.Flora of China[M].Beijing:Science Press, 2009: 80.
[2]李松海, 谢安德, 贲丽云, 等. 珍贵树种观光木研究现状及展望[J]. 南方农业学报, 2011, 42(8):968-971.
[3]邓贤兰, 吴 杨, 赖弥源, 等.江西中南部观光木种群及所在群落特征研究[J]. 广西植物, 2012, 32(2): 179-184.
[4]王 霞, 王 静, 蒋敬虎, 等. 观光木片段化居群的遗传多样性和交配系统[J]. 生物多样性, 2012, 20(6): 676-684.
[5]黄久香, 庄雪影. 华南三地观光木遗传多样性的RAPD分析[J].华南农业大学学报: 自然科学版, 2002, 23(2): 54-57.
[6]黄久香, 庄雪影. 观光木种群遗传多样性研究[J]. 植物生态学报 , 2002, 26(4): 413- 419.
[7]吴雪琴, 徐刚标, 梁 艳, 等. 南岭地区观光木自然和人工迁地保护种群的遗传多样性[J]. 生物多样性, 2013,21(1):71-79.
[8]徐刚标, 吴雪琴, 蒋桂雄, 等. 濒危植物观光木遗传多样性及遗传结构分析[J]. 植物遗传资源学报, 2014, (2): 255-261.
[9]Sampson JF, Byrne M, Yates CJ,et al. Contemporary pollenmediated gene immigration reflects the historical isolation of a rare, animal-pollinated shrub in a fragmented landscape.Heredity, 2014, 112: 172-181.
[10]Jiang J, Wang J, Kang M,et al.Isolation and characterization of microsatellite loci inTsoongiodendron odorum(Magnoliaceae)[J]. American Journal of Botany, 2011, 98(10): 284-286.
[11]吴雪琴, 徐刚标, 梁 艳, 等. 观光木ISSR-PCR反应体系的建立及优化[J]. 中南林业科技大学学报, 2012, 32(7): 76-79.
[12]闫丽君, 王红霞, 黄芳芳, 等. 伯乐树SSR-PCR反应体系的优化[J]. 中南林业科技大学学报, 2014, 34(12): 83-86.
[13]Corporation H P. SSR Locator: Tool for Simple Sequence Repeat Discovery Integrated with Primer Design and PCR Simulation.[J]. International Journal of Plant Genomics, 2008: 1-9.
[14]Cardle A L, Ramsy L, Milbourne D,et al.Computational and experimental characterization of physically clustered simple sequence repeats in plants. Genetics[J]. Genetics, 2000, 156(2):847-854.
[15]Duran C, Appleby N, Edwards D,et al. Molecular Genetic Markers: Discovery, Applications, Data Storage and Visualisation[J]. Current Bioinformatics, 2009, 4(1):16-27.
[16]Robinson AJ, Love CG, Batley J,et al.Simple sequence repeat marker loci discovery using SSR primer[J]. Bioinformatics,2004, 20(9):1475-476.
[17]文亚峰, Kentaro, Uchiyama,等. 微卫星标记中的无效等位基因 [J]. 生物多样性 , 2013, 21(1):15-15.
[18]许玉兰, 蔡年辉, 康向阳, 等. SSR-PCR反应体系建立与优化的研究概述[J]. 生物技术, 2012, 22(2): 73-76.
[19]俞志华, 王学德. 棉花细胞质雄性不育性状的RAPD标记及Taq酶用量[J]. 浙江大学学报:农业与生命科学版, 1999, (5):503-504.
[20]姜小凤, 高燕会, 童再康, 等. 石蒜属植物 SCoT-PCR 反应体系构建及优化[J].浙江农林大学学报, 2013,30(3):444-452
Optimization of SSR-PCR Reaction System and Prime Screening for Tsoongiodendron odorum
HUANG Fang-fang , HE Chang-qing, YAN Li-jun, WANG Líng-dan, XU Gang-biao
(College of Forestry, Central South University of Forestry and Technology, Changsha 410004, Hunan, China)
In this paper, the main factors affecting SSR-PCR system ofTsoongiodendron odorumwere optimized through orthogonal design with the samples ofT. odorum, which is based on single factor experiments. SSR-PCR amplytications were performed in a final volume of 10 μL containing 1.0 mmol Mg2+, 0.20 mmol dNTP, 0.25 μmol of each primers, 0.75 UTaqDNA polymerase, 60 ng of genomic DNA. Using the optimized SSR-PCR system 5 primes with high polymorphism and reproducible properties were selected from 12 primes. This paper establishs the foundation for further study of population genetic diversity inT. odorum.
Tsoongiodendron odorum; SSR; Reaction system optimization; Prime screening
S718.46
A
1673-923X(2015)10-0142-05
10.14067/j.cnki.1673-923x.2015.10.025
2015-06-23
国家林业公益性行业科研专项(201104033)
黄芳芳,硕士研究生
徐刚标,教授,博导;E-mail:gangbiaoxu@163.com
黄芳芳,何长青,闫丽君,等. 观光木SSR-PCR反应体系优化及引物筛选[J].中南林业科技大学学报,2015,35(10):142-146.
[本文编校:吴 彬]

