辐射松半胱氨酸蛋白酶OTUBAIN-like基因生物信息分析*
辐射松半胱氨酸蛋白酶OTUBAIN-like基因生物信息分析*
吴建忠
(黑龙江省农业科学院 经济作物研究所,哈尔滨黑龙江150086)
摘要:利用生物基因组学数据库,对辐射松半胱氨酸OTUBAIN-like基因进行生物信息学分析,预测该基因编码蛋白的理化性质、序列特征、蛋白质结构与生物学功能。结果表明,辐射松半胱氨酸OTUBAIN-like基因编码的蛋白质含294个氨基酸,具有非跨膜结构,含保守的PeptidaseC65结构域,预测其可能在蛋白质的翻译、合成及代谢中对胁迫应答和免疫应答等功能起关键性作用,本研究为辐射松半胱氨酸蛋白酶的OTUBAIN-like基因功能深入研究奠定基础。
关键词:辐射松;半胱氨酸蛋白酶;生物信息学
半胱氨酸蛋白酶是一种蛋白水解酶,一般通过特定的抑制剂来鉴定其活化位点,而硫醇化合物可使之活化[1]。植物中半胱氨酸蛋白酶大多属于papain(木瓜蛋白酶)(C1)、legumain(豆类天冬氨酸蛋白内切酶)(C13)、caspase(天冬氨酸特异性的半胱氨酸蛋白酶)(C14)和calpain(钙依赖半胱氨酸蛋白内切酶)(C2)家族[2]。研究发现了催化蛋白去泛素化的类蛋白酶和蛋白酶体[3~4]。泛素-蛋白酶体系统与蛋白质质量控制、细胞周期、DNA修复、转录及免疫应激等密切相关,也与许多种疾病的发生相关[5]。辐射松(Pinusradiata)体细胞胚胎发生时期对半胱氨酸蛋白酶的研究表明,一种新型的OTUBAIN-like基因优先表达,这是首次发现在高等植物中参与泛素途径的prOTUBAINS家族成员[6]。本研究基于NCBI上公布的辐射松半胱氨酸蛋白酶OTUBAIN-like基因(GenBank:EF405823),进行该基因及其编码蛋白质进行了一系列的生物信息学分析,以期为半胱氨酸蛋白酶的深入研究奠定基础。
1材料与方法
1.1序列来源
辐射松半胱氨酸蛋白酶OTUBAIN-like基因(EF405823)来源与NCBI网站的GenBank数据库,GI号为148362048。
1.2研究方法
氨基酸序列分析通过NCBI数据库找到辐射松半胱氨酸蛋白酶类泛素基因的mRNA完整编码序列,用在线分析数据库ORF Finder进行基因开放读码框与其编码氨基酸序列分析[7]。
蛋白质基本信息分析利用ProtParam工具分析蛋白质的基本信息[8],登录网站http://www.expasy.org/tools/pi_tool推测蛋白质的分子量和理论等电点,ProtScale工具进行蛋白质疏水性/亲水性分析[9],蛋白质保守结构域通过CDD分析获得[10],信号肽预测通过SignalP在线分析获得[11],跨膜区预测利用TMHMM方法在线分析[12],亚细胞定位通过TargetP分析[13],利用在线分析软件SOPMA预测蛋白的二级结构[14]。
蛋白功能预测在线软件ProtFun进行蛋白功能预测[15],采用BlastP在线软件进行蛋白的序列同源性分析[16]。
2结果与分析
2.1基因开放阅读框分析
开放阅读框(Open reading frame,ORF)是基因序列的一部分,可能是蛋白质编码序列的部分,包含一段可以编码蛋白的碱基序列,不能被终止子打断。ORF的识别是证明一个新的DNA序列为特定的蛋白质编码基因的部分或全部的先决条件[17]。
辐射松半胱氨酸蛋白酶OTUBAIN-like基因全长1 238 bp(base pairs)(图1),单链分子量为374 364.00 D(Daltons),双链分子量为751 223.00 D(Daltons),GC含量为43.38 %,AT含量为56.62 %,核苷酸数量及比例情况(表1)。该基因5’UTR长为125 bp,含15 bp的帽子结构,3’UTR长为227 bp,具有polyA尾,表明该基因结构完整。通过数据库ORF Finder分析发现,该基因起始密码子位于126 bp,终止密码子位于1 008 bp,编码294个氨基酸残基组成的蛋白质。

图1 辐射松半胱氨酸蛋白酶类泛素基因全长及编码蛋白质序列

核苷酸数量比例/%A35929.00C23218.74G30524.64T34227.63
2.2基因编码蛋白的理化性质
蛋白质的基本性质包括其相对分子质量、氨基酸组成、分子式和理论等电点等[18]。经BLAST序列比对,得到辐射松半胱氨酸蛋白酶OTUBAIN-like蛋白序列(Query ID:ABQ59604)(图1),利用ProtParam工具进行其基本理化性质分析,氨基酸和原子组成情况分别见表2和表3,氨基酸数目最多的是谷氨酸(Glu)和缬氨酸Val,分别占9.2 %和8.5 %,分子式为C1484H2299N383O458S12,294个氨基酸的相对分子量为33 218.59 D,消光系数在280 nm时为22 265,其不稳定指数为45.46,表明该蛋白稳定性一般,脂肪系数为91.77,总平均亲水性为-0.115,说明该蛋白的疏水性较弱。负电荷残基(Asp+Glu)总数为51,正电荷残基(Arg+Lys)总数为29,经等电点预测(http://cn.expasy.org/tools/pi_tool.html)显示其理论等电点为4.65。
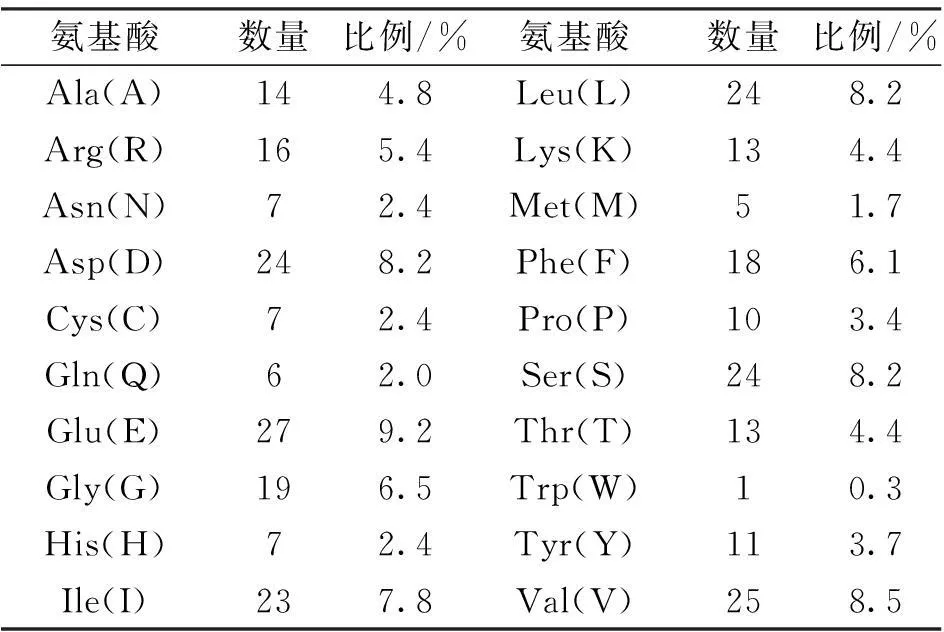
表2 氨基酸组成

表3 原子组成

图2 蛋白序列的ProtScale分析
2.3基因编码蛋白疏水性/亲水性预测及分析
氨基酸侧链的疏水性以各氨基酸减去甘氨酸疏水性之值来表示,蛋白质的疏水性在保持蛋白质三级结构的形成和稳定中起着重要作用[19]。利用在线分析软件ProtScale工具进行该OTUBAIN-like蛋白的疏水性预测分析(图2),说明该蛋白疏水区域和亲水区域均等,很难预测该蛋白的疏水性,因此结合其脂肪系数总平均亲水性,可以判定该蛋白疏水区域和亲水区域均等分布,进一步表明该蛋白稳定性一般。
2.4基因编码蛋白结构域预测及分析
通过CDD分析蛋白质保守结构域,发现从34到288氨基酸之间含有保守的Peptidase_C65结构域(图3),经Cn3D macromolecular structure viewer进行保守结构域模型分(图4),图中A、B、C和D分别为蛋白质Wire、Space Fill、Worms和Ball and Stick模型结构。将OTUBAIN-like蛋白用TMHMM 2.0分析得知,该蛋白不含跨膜结构域,表明该蛋白是非跨膜蛋白。

图3 蛋白的保守结构域

图4 蛋白质的保守结构域模型

图5 OTUBAIN-like蛋白的TargetP分析
2.5基因编码蛋白亚细胞定位
为确定OTUBAIN-like基因编码蛋白的亚细胞定位情况,采用在线软件TargetP分析结果(图5),表明该蛋白既不是叶绿体转运肽,也不是线粒体靶向肽,预测有可能是分泌通路信号肽和其他信号蛋白。利用在线软件SignalP 4.1 Server对该OTUBAIN-like蛋白进行信号肽结构预测分析,发现该蛋白不具有信号肽结构,推测其不是分泌通路信号肽。
2.6基因编码蛋白二级结构预测
对辐射松半胱氨酸蛋白酶OTUBAIN-like基因编码蛋白二级结构的分析将为进一步深入了解其生物学结构及功能奠定基础,蛋白质二级结构主要是指多肽链依赖氢键排列的具有周期性结构的构象,对其进行预测和分析将有助于认识蛋白的空间结构,采用SOPMA分析软件预测辐射松半胱氨酸蛋白酶OTUBAIN-like基因编码蛋白的二级结构(图6,表4),该蛋白由44.22 %α-螺旋(Helix),14.29 %延伸链(Strand),8.84 %β-折叠(Turn),32.65 %无规则卷曲(Coil)组成。由此可推测,α-螺旋和无规则卷曲是该蛋白主要的蛋白质二级结构元件,部分的延伸链和β-折叠则散布于整个蛋白质中。
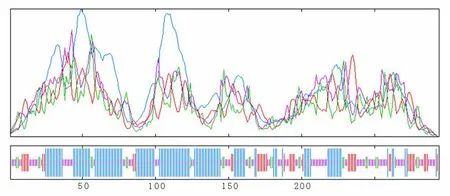
图6 蛋白质二级结构预测示意图

二级结构数量比例/%α-螺旋(Alphahelix-Hh)13044.22延伸链(Extendedstrand-Ee)4214.29β-折叠(Betaturn-Tt)268.84无规则卷曲(Randomcoil-Cc)9632.65
2.7基因编码蛋白功能预测与分析
利用在线软件ProtFun 2.2 server进行辐射松半胱氨酸蛋白酶OTUBAIN-like基因编码蛋白功能预测(表5),该蛋白具有蛋白质翻译、脂肪酸代谢、生物合成辅酶因子、嘌呤与嘧啶和能量代谢功能的可能性分别为3.254、2.690、2.144、1.601和1.561,因此预测该蛋白可能蛋白质翻译、生物合成及代谢过程中有重要作用,结合其在GO功能分析的结果(图7)显示,辐射松半胱氨酸蛋白酶OTUBAIN-like基因编码蛋白可能在蛋白质的翻译、合成及代谢中其胁迫应答和免疫应答功能起关键性作用。
2.8基因编码蛋白序列同源性分析
经BlastP在线进行蛋白质的序列同源比对(图8),辐射松半胱氨酸蛋白酶OTUBAIN-like基因编码蛋白(ABQ59604)与北美云杉(Piceaspp.)的一个未知功能蛋白(ABK21173)序列结构同源性最高,相似性高达98 %,与莲花(Nelumbonucifera)泛素硫酯酶OTUBAIN-like异构体X1(XP_010250517)及海枣(Phoenixdactylifera)泛素硫酯酶OTUBAIN-like同源性都较高,相似性分别达85 %和81 %。

表5 辐射松半胱氨酸蛋白酶OTUBAIN-like基因
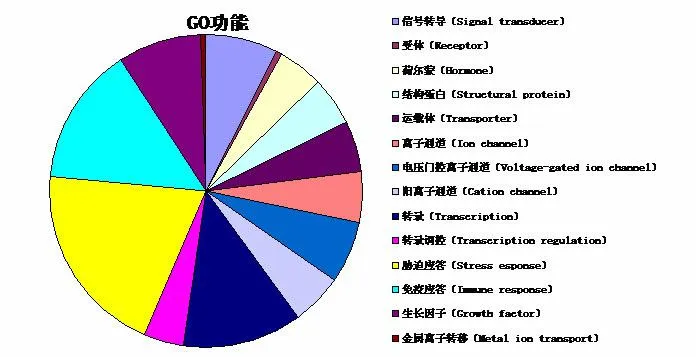
图7 辐射松半胱氨酸蛋白酶OTUBAIN-like基因编码蛋白GO功能分类

图8 氨基酸序列保守结构同源分析
3结论与讨论
半胱氨酸蛋白酶是植物中一类重要的蛋白酶家族。目前,在中华猕猴桃(Actinidiachinensis)果实[20]、大麦(Hordeumvulgare)种子[21]、豇豆(Vignaunguiculata)子叶[22]、豌豆(Pisumsatium)种子[23]及甘薯(Ipomoeabatatas)[24]等作物中均分离到了相关的半胱氨酸蛋白酶家族成员。研究显示半胱氨酸蛋白酶的富集不仅出现在干旱[25~26]、盐胁迫[23]等环境胁迫条件下,还与植物的细胞程序化死亡[27~28]、器官分化及木质部发生或分化[29]存在一定的联系,但半胱氨酸蛋白酶在植物响应非生物及生物胁迫、器官分化及衰老的过程中所发挥的功能和作用机制尚未得到详尽的简析。本研究通过辐射松半胱氨酸蛋白酶OTUBAIN-like基因生物信息学分析,该基因含1 238 bp,结构完整,编码由249个氨基酸残基组成的蛋白,含20种氨基酸,分子量为33 218.59 D,理论等电点PI为4.65,疏水性较弱,稳定性一般,分子式为C1484H2299N383O458S12,含有保守的Peptidase_C65结构域,不含跨膜结构,不具有叶绿体转运肽、线粒体靶向肽及分泌通路信号肽的亚细胞定位结构,经蛋白质二级结构预测分析,α-螺旋和无规则卷曲是该蛋白主要的蛋白质二级结构元件,部分的延伸链和β-转角则散布于整个蛋白质中,预测其可能在蛋白质的翻译、合成及代谢中其胁迫应答和免疫应答功能起关键性作用,因此,推断该蛋白酶可能作为一种转录因子调节相关基因的表达。本研究将为辐射松半胱氨酸蛋白酶OTUBAIN-like基因功能的深层次挖掘研究奠定基础。
参考文献:
[1]闫龙凤,杨青川,韩建国,等.植物半胱氨酸蛋白酶研究进展[J].草业学报,2005,14(5):11-19.
[2]Jinq M C,Neil D R,Richard A E,etal.Identification of the active site of legumain links it to caspases,clostripain and gingipains in a new clan of cysteine endopeptidases[J].FEBS Letters,1998,441(3):361-365.
[3]Vierstra R D.The ubiquitin/26S proteasome pathway,the complex last chapter in the life of many plant proteins[J].Trends in Plant Science,2003,8(3):135-142.
[4]Basset G,Raymond P,Malek L,etal.Changes in the expression and the enzymic properties of the 20S proteasome in sugarstarved maize roots,evidence for an in vivo oxidation of the proteasome[J].Plant Physiology,2002,128(3):1149-1162.
[5]李思滨,刘英,祖元刚.半胱氨酸蛋白酶在植物细胞程序性死亡中的作用[J].植物生理学通讯,2008,44(2):345-349.
[6]Gutierrez F,Medina C,Aquea F,etal.A novel Otubain-like cysteine protease gene is preferentially expressed during somatic embryogenesis in Pinus radiate[J].Mol Biol Rep,2008,35(4):567-573.
[7]Rombel I T,Sykes K F,Rayner S,etal.ORF-FINDER:a vector for high-throughput gene identification[J].Gene,2002,282(1):33-41.
[8]Stockinger H,Altenhoff A.M,Arnold K,etal.Fifteen years SIB Swiss Institute of Bioinformatics:life science databases,tools and support[J].Nucleic Acids Res,2014,42(W1):436-441.
[9]Linding R,Russell R B,Neduva V,etal.GlobPlot:exploring protein sequences for globularity and disorder[J].Nucleic acids research,2003,31(13):3701-3708.
[10]Marchler-Bauer A,Lu S,Anderson J B,etal.CDD:a Conserved Domain Database for the functional annotation of proteins[J].Nucleic acids research,2011,39(suppl 1):225-229.
[11]Petersen T N,Brunak S,von Heijne G,etal.SignalP 4.0:discriminating signal peptides from transmembrane regions[J].Nature methods,2011,8(10):785-786.
[12]Tusnady G E,Simon I.The HMMTOP transmembrane topology prediction server[J].Bioinformatics,2001,17(9):849-850.
[13]Emanuelsson O,Brunak S,von Heijne G,etal.Locating proteins in the cell using TargetP,SignalP and related tools[J].Nature protocols,2007,2(4):953-971.
[14]Geourjon C,Deleage G.SOPMA.significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J].Computer applications in the biosciences,CABIOS,1995,11(6):681-684.
[15]Sommer I,Rahnenführer J,Domingues F S,etal.Predicting protein structure classes from function predictions[J].Bioinformatics,2004,20(5):770-776.
[16]Lavigne R,Seto D,Mahadevan P,etal.Unifying classical and molecular taxonomic classification:analysis of the Podoviridae using BLASTP-based tools[J].Research in microbiology,2008,159(5):406-414.
[17]Vieira P,de Waal-Malefyt R,Dang M N,etal.Isolation and expression of human cytokine synthesis inhibitory factor cDNA clones:homology to Epstein-Barr virus open reading frame BCRFI [J].Proceedings of the National Academy of Sciences,1991,88(4):1172-1176.
[18]蒋彦.基础生物信息学及应用[M].北京:清华大学出版社,2003:150-160.
[19]黄曼,卞科.蛋白质疏水性测定方法研究进展[J].粮油食品科技,2004,12(02):31-32.
[20]Carne A,Moore C H.The amino acid sequence of the tryptic peptide from actinidin,a proteolytic enzyme from the fruit of Actinidia chinesis[J].Biochemical Journal,1978,173:73-83.
[21]Rogers J C,Dean D,Heck G R.Aleurain:a barley thiol protease closely related to mammalian cathepsin H[J].Proceedings of the National Academy of Science of the United States of America,1985,82(19):6512-6516.
[22]Akasofu H,Yamauchi D,Minamikawa T.Nucleotide sequence of the gene for the Vigna mungosulfhydryl-endopeptidase (SH-EP) [J].Nucleic Acids Research,1990,18 (7):1892.
[23]Jennifer T J,John E M.A salt-and dehydration-inducible pea gene,Cyp15a,encodes a cell-wall protein with sequence similarity to cysteine protease[J].Plant Molecular Biology,1995,28:1055-1065.
[下转第12页]
Bioinformatics Analysis of Cysteine Protease TUBAIN-like Gene
in Pinus radiata
WU Jian-zhong
(Institute of Industrial Crops,Heilongjiang Academy of Agricultural Sciences,Harbin Heilongjiang 150086 P.R.China)
Abstract:To forecast the physical and chemical properties, sequence characteristics,structure and biological function of the gene encoding protein,bioinformatics analysis of Cysteine Protease OTUBAIN-like gene in Pinus radiata with genomics database was conducted.The results showed that the protein encoded by the Pinus radiata Cysteine Protease OTUBAIN-like gene contains 294 amino acids,and it has the conservative PeptidaseC65 domain but no cross membrane structure.This study may play a critical role in protein synthesis and metabolism on stress response and immune response function.
Key words:Pinus radiata;OTUBAIN-like;bioinformatics
中图分类号:S 792.252
文献标识码:A
文章编号:1672-8246(2015)05-0001-08
作者简介:吴建忠(1983-),男,助理研究员,博士生,主要从事亚麻遗传育种及基因组学研究。E-mail:wujianzhong176@163.com
基金项目:哈尔滨市科技创新工程青年基金(2013RFQYJ010) 。
收稿日期:*2015-03-27
doi10.16473/j.cnki.xblykx1972.2015.05.001

