柽柳内生链霉菌CLR304抗MRSA次级代谢产物304A的研究
刘少伟,李舟,胡辛欣,游雪甫,孙承航
·论著·
柽柳内生链霉菌CLR304抗MRSA次级代谢产物304A的研究
刘少伟,李舟,胡辛欣,游雪甫,孙承航
目的 从沙生植物柽柳来源的内生链霉菌 CLR304 发酵液中分离鉴定具有强抗耐甲氧西林金黄色葡萄球菌(MRSA)活性的次级代谢产物。
方法 基于 16S rRNA 基因序列,对菌株 CLR304 进行初步分类鉴定;采用液体发酵、溶剂萃取、正相硅胶层析、薄层层析以及高效液相制备分离纯化 CLR304 的活性次级代谢产物 304A;根据紫外光谱、高分辨质谱和核磁共振波谱及文献比对,鉴定化合物 304A 的平面结构;采用琼脂稀释法测定化合物的抗菌活性。
结果 菌株 CLR304 为链霉菌,从其发酵液中分离获得抗MRSA 次级代谢产物 304A,证明为维吉尼霉素 M1。抑菌活性实验显示其对葡萄球菌、屎肠球菌的敏感株和耐药株均具有较强活性(MIC = 1~4 μg/ml)。
结论 首次从柽柳中获得产生维吉尼霉素 M1的内生链霉菌,表明沙生植物柽柳蕴含药用放线菌资源。
柽柳;内生放线菌;次级代谢产物;维吉尼霉素
植物内生菌(endophytes)是指在其生活史的全部或部分阶段生活于健康植物组织内部,不引发植物产生明显病症,与植物形成稳定共生关系的微生物菌群[1]。在长期的协同进化过程中,植物内生菌与宿主植物之间形成了一种动态平衡的内生关系,不仅参与植物次生代谢与成分的转化和合成,而且能够独立产生丰富的次级代谢产物,成为众多结构新颖天然产物的重要来源[2]。
柽柳属(Tamarix)植物为落叶灌木或小乔木,具有显著的耐高温、耐辐射、耐干旱、耐贫瘠、耐盐碱的特性,是荒漠、半荒漠生态系统的关键物种,主要分布在如新疆、甘肃、青海等我国西北干旱、半干旱地区以及华北滨海盐碱地区,在荒漠植被中占有重要地位[3]。对柽柳属植物的研究,目前主要集中在系统分类、形态特征、生理机制、植物化学、药理作用、生物防治等方面,对柽柳内生微生物的研究较少,尤其是对放线菌,这一具有重要药用价值的微生物的研究更加稀少[4-5]。为此,本课题组围绕柽柳根际和内生环境,在我国新疆地区展开了一系列药用放线菌资源勘探相关研究,并取得了一些研究结果[6-9]。另外根据报道[10],采集了北京房山地区野生柽柳,从根部分离到一株具有广谱抗菌活性的内生放线菌菌株 CLR304。本文报道该菌株的初步鉴定、该菌株产生的具有抗耐甲氧西林金黄色葡萄球菌(MRSA)次级代谢产物 304A 的分离纯化和结构鉴定及抗菌活性等相关的研究结果。
1 材料与方法
1.1 材料和仪器
1.1.1 菌株 菌株 CLR304 为从北京房山柽柳根部分离得到的内生放线菌;抑菌活性测试所用检定菌耐甲氧西林金黄色葡萄球菌(MRSA)、枯草芽孢杆菌、粪肠球菌、铜绿假单胞菌、大肠埃希菌、肺炎克雷伯菌由中国医学科学院医药生物技术研究所保存。
1.1.2 培养基
斜面培养基(高氏一号琼脂培养基):可溶性淀粉 20 g、KNO31 g、K2HPO40.5 g、MgSO4·7H2O 0.5 g、NaCl 0.5 g、FeSO4·7H2O 0.01 g、琼脂 20 g、蒸馏水 1000 ml,pH 7.4~7.6。
种子和发酵培养基(A2培养基):葡萄糖0.5%、酵母膏 0.5%、蛋白胨 0.5%、牛肉膏 0.5%、玉米浆 0.4%、可溶性淀粉 2%、黄豆饼粉 1%、CoCl20.02%、CaCO30.4%,pH 7.2。
检定菌培养基:Mueller Hinton(MH)液体培养基和 Mueller Hinton Agar(MHA)固体培养基为英国 Oxoid 公司产品。
1.1.3 主要试剂 PCR 常规操作所用试剂和 Taq酶均购自北京鼎国昌盛生物技术有限公司;High Fidelity PCR SuperMix、pEASY-T1 克隆试剂盒、EasyPure Quick Gel Extraction Kit、Trans-T1 感受态细胞均购自北京全氏金生物技术有限公司;乙酸乙酯、甲醇和丙酮等有机试剂均为分析纯,由北京化工厂生产;色谱级甲醇购于美国 Honeywell Burdick & Jackson 公司;蒸馏水为屈臣氏集团有限公司产品。
1.1.4 分离填料 层析硅胶采用青岛海洋化工厂生产的 100~200 目硅胶 G;薄层层析采用德国默克公司的 TLC Silica gel 60 F254玻璃薄层板;高效液相色谱柱为 Agilent ZORBAX SB-C18(250 mm× 9.4 mm,5.0 μm)。
1.1.5 主要仪器 Veriti 96 well fast thermal cycler PCR 扩增仪为美国 Applied Biosystems 公司产品;DYY-6C 型电泳仪为北京市六一仪器厂产品;OSB-2100 型旋转蒸发仪为日本东京理化公司产品;RVC2-18 型离心浓缩仪为德国 Christ 公司产品;LC-20AT 型高效液相色谱仪为日本岛津公司产品;ThermoScientific Q Exactive 高分辨质谱为美国Thermo 公司产品;Varian VNS-600 型核磁共振仪为瑞士瓦里安公司产品;MIT-P 细菌多点接种仪为日本 Sakuma 公司产品。
1.2 方法
1.2.1 菌株 CLR304 的分子分类 采用Chelex-100 法[11]提取菌株 CLR304基因组 DNA,并以此为模板进行 16S rRNA 基因 PCR 扩增,扩增引物为细菌通用引物:27f(5' AGAGTTTGATC CTGGCTCAG 3')和 1492r(5' TACGGCTACCTT GTTACGACTT 3'),50 μl PCR 反应体系中含有Taq SuperMix 25 μl、27f 引物 1.5 μl、1492r 引物1.5 μl、模板 2 μl、无菌水 20 μl。反应条件:95 ℃预变性 5 min;94 ℃ 变性 1 min,55 ℃ 退火 1 min,72 ℃ 延伸 2 min,共 35 个循环;最后 72 ℃ 后延伸 10 min。PCR 扩增产物由上海生工生物工程技术服务有限公司完成测序。根据测序结果,将16S rRNA 基因序列登录数据库 EzBioCloud 中进行相似性比对搜索,将相似性较高的有效发表种的16S rRNA 基因序列作为参比序列,用 Clustal[12]进行多序列比对,系统进化矩阵根据 Kimura 2-paramete 模型估计,采用 MEGA5.0[13]软件以邻近法(Neighbor-Joining)[14]进行聚类分析并构建系统发育树。系统发育树的拓扑结构稳定性采用自展值(bootstrap value)分析的方法并重复取样1000 次。
1.2.2 菌株 CLR304 的发酵 无菌操作下将新鲜生长于斜面的菌株 CLR304挖块接种于 250 ml三角瓶中(含 50 ml A2培养基),在 28 ℃、180 r/min条件下培养 48 h,作为一级种子;将一级种子按照5% 的接种量,接种于 5000 ml 立瓶中(含 1000 ml A2培养基),28 ℃、180 r/min 摇床上旋转振荡培养 72 h,最终收获发酵液约 20 L。
1.2.3 化合物 304A 的分离、纯化 20 L 发酵液经 4500 r/min,20 min 离心后,上清液用等体积乙酸乙酯萃取,有机相经无水 Na2SO4脱水处理、旋转蒸发干燥,获棕褐色黏稠状粗提物。粗提物用少量氯仿溶解后,湿法上样于硅胶柱,以氯仿-甲醇99∶1、49∶1、24∶1、9∶1 和甲醇各 400 ml 进行梯度洗脱,收集洗脱流份(20 ml/管)并以 MRSA 为检定菌,采用琼脂平板法测定抗菌活性。结果表明,活性流份集中在氯仿-甲醇 49∶1 部分和 24∶1 部分。将活性较大的氯仿-甲醇 24∶1 洗脱液部分合并,减压浓缩干燥获得棕黄色活性粗品。将粗品用少量甲醇溶解后,点样于 10 cm × 10 cm 硅胶层析板上,以展开剂甲醇-乙酸乙酯 1∶29 上行展开,将Rf值为 0.42 的活性条带刮下,装于小玻璃柱(15 cm × 1.5 cm),用适当体积的乙酸乙酯洗脱,经减压浓缩干燥后得到淡黄色的半纯品。半纯品溶于少量甲醇,经 0.22 μm 滤膜过滤后进行 HPLC 制备,HPLC 条件为:70% 甲醇-水溶液为流动相,流速为 2 ml/min;检测波长为 210、224 和 254 nm;按紫外吸收峰收集样品后测定活性,收集保留时间为 28 min 左右的活性峰(命名为 304A),旋转蒸发除去甲醇,冷冻干燥后,获得约 6.0 mg 纯品。
1.2.4 菌株 CLR304 酯提物的活性检测 1 L 发酵液的乙酸乙酯提取物旋转蒸发干燥后,用 1 ml甲醇溶解,制成浓缩 1000 倍的甲醇浓缩样品备用。以耐甲氧西林金黄色葡萄球菌、枯草芽孢杆菌、粪肠球菌、铜绿假单胞菌、大肠埃希菌、肺炎克雷伯菌为检定菌,采用琼脂扩散法对其进行活性检测。吸取 30 μl 甲醇溶解的乙酸乙酯提取物滴加于直径为 5 mm 的圆形无菌滤纸片上,待甲醇挥干后,贴于已制备好的检定菌平板上,同时以滴加 30 μl甲醇的空白纸片为阴性对照。37 ℃ 恒温培养箱培养 16~24 h 后,观察并测量抑菌圈直径。
1.2.5 化合物 304A 的结构鉴定 根据紫外光谱(UV)、电喷雾高分辨质谱(HR-ESI-MS)、氢谱(1H-NMR)、碳谱(13C-NMR)、无畸变极化转移增强谱(DEPT)、1H-1H 化学位移相关谱(1H-1HCOSY)、1H-13C 异核单量子相关谱(13C-1H COSY)和1H-13C 异核多键相关谱(HMBC)的数据,进行谱图解析并与相关文献数据比对,确定化合物304A 的平面结构。
1.2.6 化合物 304A 的抑菌谱活性测定 参照CLSI 推荐方法[15],采用琼脂稀释法和多点接种仪进行 MIC 测定。化合物 304A 经 DMSO 溶解后,用 MH肉汤二倍稀释成各种所需浓度,分别加适量到平皿中;MH 琼脂培养基融化后定量注入含304A 的平皿内混匀,使化合物的终浓度分别为0.25、0.5、1、2、4、8、16 μg/ml。检定菌用 MH 肉汤隔夜增菌,试验时制备成 0.5 麦氏浓度的菌悬液,1:10 稀释后以多点接种仪吸取菌液 1~2 μl 接种于琼脂平板表面,每点菌落数约为 104cfu。置37 ℃ 孵育 16~24 h 观察结果,以抑制细菌生长的最低药物浓度为 MIC 值。
2 结果
2.1 菌株 CLR304 的分子分类
菌株 CLR304 基于 16S rRNA 基因序列构建的 N-J 系统发育树见图1。菌株 CLR304 属于链霉菌属,与有效发表种的典型菌株 Streptomyces albogriseolus NRRL B-1305T(AJ494865)和Streptomyces viridodiastaticus NBRC 13106T(AB184317)同聚于一个进化分支并且与这两株典型菌株相似性最高,均为 99.92%。
2.2 菌株 CLR304 的抑菌活性
菌株 CLR304 的发酵液酯提物的甲醇浓缩样品的抑菌活性测定结果见图2。菌株 CLR304 的发酵产物具有广谱抑菌活性,对 MRSA、枯草芽孢杆菌及粪肠球菌活性最强(抑菌圈直径 > 17 mm),对铜绿假单胞菌和大肠埃希菌也具有较强的抑制作用(抑菌圈直径 > 12 mm),对肺炎克雷伯菌抑制活性略弱(抑菌圈直径约为 9 mm)。
2.3 化合物 304A 的理化性质及结构鉴定

图1 基于 16S rRNA 基因序列构建的 CLR304 及其相近菌株的 N-J 系统发育树(节点上的数据表示自举值,仅显示高于 50% 的数值)Figure 1 Neighbour-Joining tree based on 16S rRNA gene sequences of strain CLR304 and related strains (The numbers at the nodes indicate the bootstrap values based on analyses of 1000 resampled data sets)
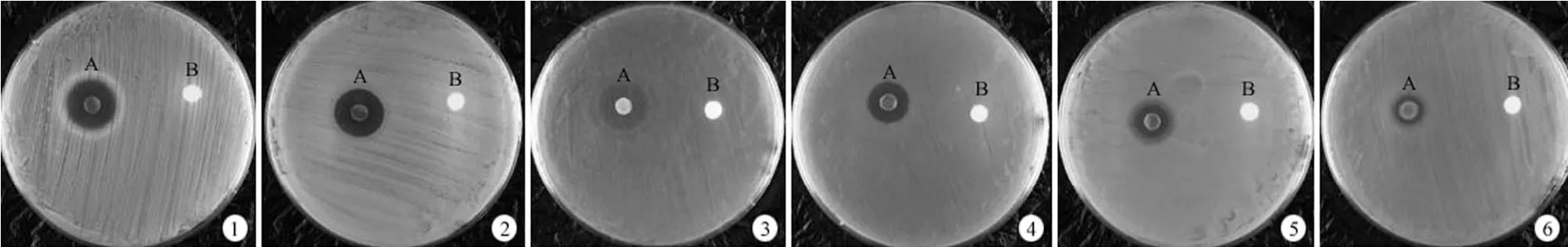
图2 菌株 CLR304 的发酵液酯提物的甲醇浓缩样品的抑菌活性(1:MRSA;2:枯草芽孢杆菌;3:粪肠球菌;4:铜绿假单胞菌;5:大肠埃希菌;6:肺炎克雷伯菌;A:发酵液酯提物的甲醇浓缩样品;B:空白甲醇样品为阴性对照)Figure 2 Antibacterial activity of concentrated sample of the ester extracts of fermentation broth produced by strain CLR304 (1:MRSA; 2: Bacillus subtilis; 3: Enterococcus faecalis; 4: Pseudomonas aeruginosa; 5: Escherichia coli; 6: Klebsiella pneumonia; A:Ester extracts of fermentation broth; B: Methanol as negative control)
化合物 304A 为淡黄色无定形粉末,易溶于DMSO、甲醇、乙醚、氯仿等有机溶剂,微溶于水。紫外光谱显示其在 225 nm 有最大紫外吸收。HR-ESI-MS 数据表明化合物 304A [M+H]+的测量值为 526.2550,理论值为 526.2548,分子量为525,分子式为 C28H35N3O7,不饱和度为 13。经1H-NMR、13C-NMR、DEPT、1H-1H COSY,13C-1H COSY 和 HMBC 解析及光谱数据的文献比对,确定化合物 304A 结构如图3 所示,为已知化合物维吉尼霉素 M1,结构解析具体过程如下。结合1H-NMR、13C-NMR、DEPT 和13C-1H COSY 的数据分析,确定化合物 304A 有 8 个季碳(δC:200.9,167.6,160.9,159.9,155.9,137.1,136.0,134.8)、11 个次甲基碳(δC:145.5,143.0,133.5,130.7,126.3,125.4,122.9,81.4,66.1,37.7,29.9),5 个亚甲基碳(δC:50.5,47.4,45.7,40.4,30.2)和4 个甲基碳(δC:19.6,18.9,12.7,12.2),共 28 个碳原子,其中 4 个为羰基碳信号(δC:200.9,167.6,160.9,159.9),11 个为烯碳信号(δC:155.9,145.5,143.0,137.1,136.0,134.8,133.5,130.7,126.3,125.4,122.9)。在1H-NMR 谱高场有 4 个甲基质子信号:33-CH3(1.60,s),32-CH3(1.11,d,J = 6.5),31-CH3(0.98,d,J = 7.5),30-CH3(0.97,d,J = 7.0),在1H-1H COSY 中 30-CH3和 31-CH3的两个甲基质子分别与 29-CH(2.02,m)的次甲基质子相关,表明化合物中存在异丙基结构CH3(30)-CH(29)-CH3(31);32-CH3的甲基质子信号与 4-CH(2.70,m)的次甲基质子信号相关,29-CH和 4-CH 两个次甲基质子信号同时与 3-CH(4.95,dd,J = 10.0,1.5)的次甲基质子相关,表明存在如下结构片段:CH3(32)-CH(4)-CH(3)-CH(29)-。 HMBC 中 32-CH3的甲基质子信号分别与 C-3(δ 81.4)相关,30-CH3的甲基质子信号分别与C-3 和 C-31(δ 19.6)相关,31-CH3的甲基质子信号分别与 C-3 和 C-30(δ 18.9)相关,确证了上述结构片段的存在。化合物 304A 结构中第一对烯碳信号 C-5 和 C-6 的化学位移分别为 143.0和 125.4,其在13C-1H COSY 谱中对应的质子信号5-H(6.59,dd,J = 16.0,7.0)和 6-H(6.01,d,J = 16.5)在1H-1H COSY 中相关,J = 16.5 Hz 表明两个烯氢质子信号处于反式,同时1H-1H COSY中 4-H 与 5-H 相关,HMBC 中 4-H 与 C-6相关,表明存在结构片段 -CH(4)-CH(5) = CH(6)-。第二对烯碳信号 C-10 和 C-11 的化学位移分别为 126.3 和 133.5,对应的质子信号 10-H(5.58,dt,J = 16.0,4.5)和 11-H(6.01,d,J = 16.5)在1H-1H COSY 中相关并处于反式。DEPT 显示为亚甲基的 C-9 化学位移为 40.4,对应的 9-CH2两个质子化学位移为 3.94(1H,m)和 4.22(1H,m),在1H-1H COSY 中 9-CH2的两个质子与 10-CH烯氢质子相关,与低场中化学位移为 7.58 的8-XH 质子相关;在 HMBC 中,8-XH 质子与化学位移为 167.6 的 7-C=O 相关,表明存在结构片段 -CO(7)-XH(8)-CH2(9)-CH(10) = CH(11)-。第三对烯碳信号 C-12 和 C-13 的化学位移分别为 134.8和 130.7,HMBC 中 10-CH 质子信号与 C-12,11-CH 质子信号与 C-33(δ 12.7)、C-13,33-CH3质子信号与 C-11、C-13 远程相关;1H-1H COSY 中13-H 与 14-H(4.81,m)相关,14-H 与 15-CH2亚甲基两个质子信号(2.76,dd,J = 14.0,3.5)和(3.16,dd,J = 13.5,9.5)相关,因此具有如下结构片段:-CH(10) = CH(11)-C(12) = CH(13)-CH(14)-CH2(15)-,其中 14-H 较低场的化学位移表明其与杂原子(O 或 N)相连,33-CH3与季碳 C-12 相连。在1H-NMR 中,化学位移为 3.5~4.0 的区域,明显可见 17-CH2的两质子信号(3.72,d,J = 13.0)、(3.93,d,J = 13.0)相互偶合,形成 AB 自旋系统,HMBC 中可见 17-CH2质子信号与 C-16(δ 200.9)、C-18(δ 155.9)及 C-15(δ 47.4)相关,15-CH2的质子信号与 C-16 相关,推测存在结构片段 -CH2(15)-C(16)-CH2(17)-C(18)-,超低场的化学位移表明 C-16 为一个酮羰基碳。另一个低场的C-7 羰基碳,在 HMBC 中显示与 5-H 和 6-H 相关,表明存在 -CH(5) = CH(6)-CO(7)-。根据以上结构解析,除杂原子外,基本确定了 C-3 到 C-18 的结构片段。

图3 304A 的平面结构(A)及 HMBC()和1H-1H COSY()相关图(B)Figure 3 Planar structure of 304A (A) and summary of HMBC () and1H-1H COSY() experiments of 304A in CDCl3(B)
化合物 304A 中存在由 -N(23)-CH2(24)-CH2(25)-CH(26)-CH(27)- 组成的二氢吡咯环结构,1H-1H COSY 中 24-CH2(4.35,m)的质子信号与25-CH2(2.71,m)(2.90,m)、26-CH(6.14,dd,J = 3.0,2.5)的质子信号依次相关,HMBC 中 24-H和 25-H 分别与化学位移为 137.1 的烯碳 C-27相关,构成片段 -CH2(24)-CH2(25)-CH(26) = C(27)-,24-H 的低场位移,证明 24-CH2与 C-27 通过一个氮相连,构成二氢吡咯环结构。至此,只剩在化学位移低场的 20-CH(7.88,s)尚未归属,其在13C-1H COSY 中对应的化学位移为 145.5,因此20 位为一个次甲基烯碳与杂原子相连。HMBC 中20-H 分别与 C-18、C-21(δ 136.0)相关,结合元素组成和不饱和度计算,证明化合物 304A 结构中还存在噁唑环。剩余两个未解析的羰基碳:C-1(δ 160.9)和 C-22(δ 159.9)分别为酯羰基碳和酰胺羰基碳,其中 HMBC 中 3-CH 质子信号与 C-1,24-CH2亚甲基质子信号与 C-22 存在远程相关。
综合以上结构片段及光谱数据与文献[16-17]的进一步比对,确定了结构中杂原子的位置,并确认化合物 304A 与维吉尼霉素 M1结构一致。化合物 304A 在 CDCl3溶剂中1H-NMR、13C-NMR、DEPT 数据如表1 所示。
2.4 化合物 304A 的抗菌活性测定结果
化合物 304A 抗细菌活性结果如表2 所示。304A 对革兰阳性菌有较强抑制活性,如对耐甲氧西林表皮葡萄球菌(MRSE)、耐甲氧西林金黄色葡萄球菌(MRSA)、万古霉素中度耐药金黄色葡萄球菌(VISA)、耐万古霉素屎肠球菌(VRE),MIC 值均在 1~4 μg/ml 范围内。化合物 304A 对测试的革兰阴性菌抗菌活性 MIC > 16 μg/ml,表现出弱抗菌活性或无抗菌活性。
3 讨论
维吉尼霉素是 1955年由比利时卢邦大学的戴索马教授从土壤筛选的一株弗吉尼亚链霉菌(Streptomyces virginiae)中分离得到[18],维吉尼霉素具有专一抗革兰阳性菌活性,但并不是单一组分,主要是由 M1和 S1两种抗菌因子组成,其中M1因子为大环内酯,分子式为 C28H35N3O7,占总量的 70%~80%;S1因子为环状多肽,分子式为C43H49N7O10,结构见图4,占总量的 20%~30%[19]。由于维吉尼霉素毒性低,在动物体内积累极少,生物降解性好,以及在肠道菌群中不易产生抗性突变株等优势,已成为一类功能卓越的兽用抗生素[20],广泛应用于畜禽配合饲料中。20 世纪90年代末,维吉尼霉素结构类似物奎奴普丁(quinupristin)和达福普汀(dalfopristin)两组分混合开发的注射用抗生素 synercid 已用于治疗耐万古霉素的屎肠球菌引起的重症感染[21]。2010年开发的口服复合物NXL-103,活性是 synercid 的 4 倍以上,现已进入了 II 期临床[22]。
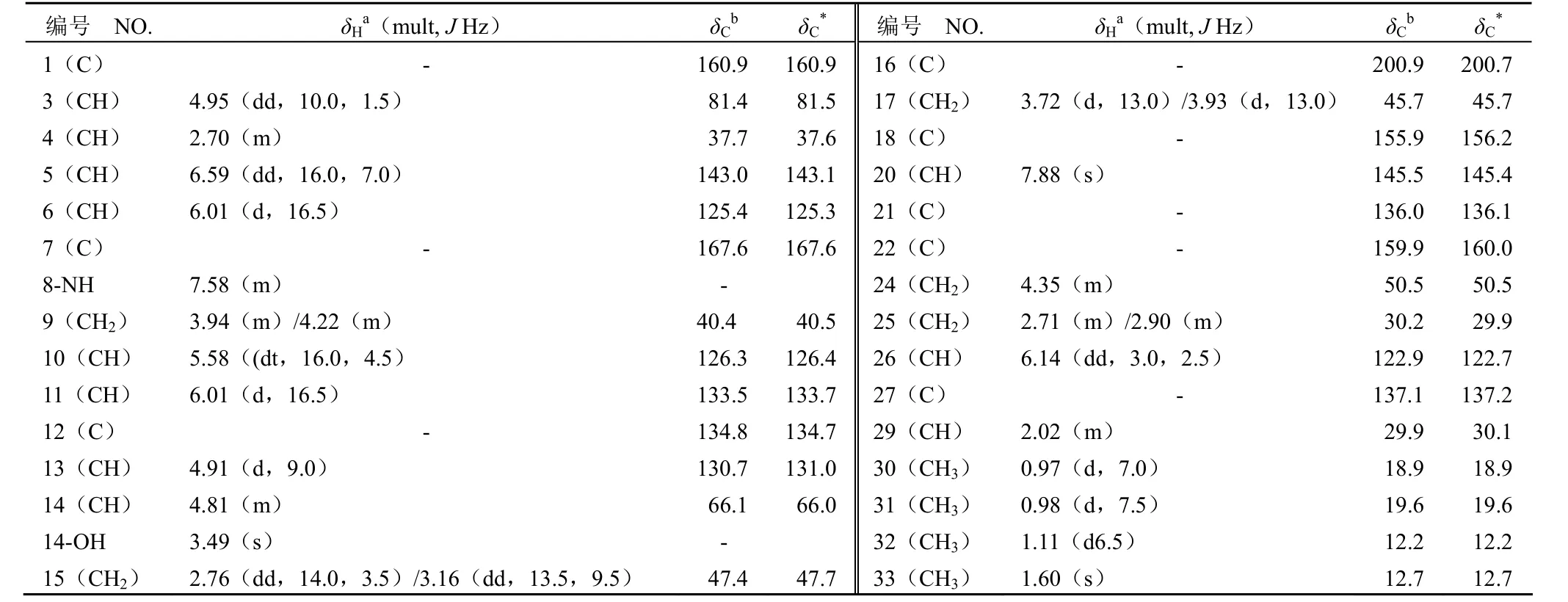
表1 化合物 304A 在 CDCl3中的 NMR 数据Table 1 The NMR data of compound 304A in CDCl3

表2 化合物 304A 的抗菌谱Table 2 Antibacterial spectra of compound 304A
综上所述,维吉尼霉素本身就是重要的药物或药物前体,其主要成分维吉尼霉素 M1由于产生菌不同而有不同名称,如产生菌为 Streptomyces mitakaensis 而命名为 mikamycin A,产生菌为Streptomyces pristinaespiralis 而命名为 pristinamycin IIA[23]等,这些产生菌多为分离自土壤的链霉菌。本研究首次从北京房山野生柽柳来源的内生链霉菌中分离到维吉尼霉素 M1,这一研究结果表明作为沙生植物关键和优势物种的柽柳是微生物药物的重要资源,同时也间接提示,我国种类多样的沙生植物内生放线菌作为微生物药物的重要来源值得挖掘和开发。
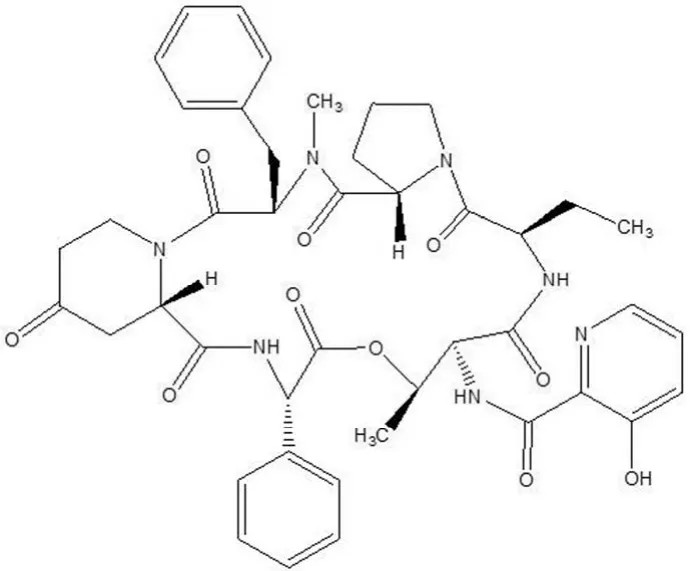
图4 维吉尼霉素 S1的化学结构Figure 4 Chemical structure of virginiamycin S1
[1] Huang XH, Li S, Tan ZJ, et al. Progress of study on endophytic actinomycetes in plant. Biotechnol Bull, 2008(1):42-46. (in Chinese)
黄晓辉, 李珊, 谭周进, 等. 植物内生放线菌研究进展. 生物技术通报, 2008(1):42-46.
[2] Huang BK, Qin LP. Biodiversity and active components of the endophytes in medicinal plants. J Pharm Pract, 2006, 24(4):193-196.(in Chinese)
黄宝康, 秦路平. 药用植物内生菌的生物多样性及活性成分. 药学实践杂志, 2006, 24(4):193-196.
[3] Zhang DY, Yin LK, Pan BR. Study on drought-resisting mechanism of Tamrix L. and assessing of its potential application. J Desert Res,2003, 23(3):252-256. (in Chinese)
张道远, 尹林克, 潘伯荣. 柽柳属植物抗旱性能研究及其应用潜力评价. 中国沙漠, 2003, 23(3):252-256.
[4] Huang SW, Liang JY. Progress of studies on Tamarix Linn. Strait Pharm J, 2007, 19(3):5-9. (in Chinese)
黄时伟, 梁敬钰. 柽柳属植物研究进展. 海峡药学, 2007, 19(3):5-9.[5] Chen M, Zhu JW, Sheng JD, et al. Study on soil enzyme activities and soil microorganisms under canopy of Tamarix spp. shrubs at Tarim Middle river. Southwest China J Agric Sci, 2008, 21(1):103-109. (in Chinese)
陈明, 朱建雯, 盛建东, 等. 塔河中游柽柳灌丛土壤酶活性及微生物数量变化的研究. 西南农业学报, 2008, 21(1):103-109.
[6] Tuo L, Habden X, Guo L, et al. Studies on diversity and bioactivity of rhizosphere actinomycetes from psammophyte in Lop Nor Region. Chin J Antibiot, 2012, 37(1):21-26. (in Chinese)
庹利, 旭格拉·哈布丁, 郭琳, 等. 罗布泊地区沙生植物根际放线菌多样性及生物活性的研究. 中国抗生素杂志, 2012, 37(1):21-26.[7] Guo L, Tuo L, Habden X, et al. Allosalinactinospora lopnorensis gen. nov., sp. nov., a new member of the family Nocardiopsaceae isolated from soil. Int J Syst Evol Microbiol, 2015, 65(Pt 1):206-213.
[8] Liu JM, Sun CH, Tuo L, et al. Study on diversity and bioactivity of
endophytic actinomycetes isolated from psammophyte in the south edge of the Taklamakan Desert//The thesis collection of the 5th National Conference for Microbial Resources and National Services Platform, Guangzhou, 2013:164. (in Chinese)
刘佳萌, 孙承航, 庹利, 等. 塔克拉玛干沙漠南麓沙生植物内生放线菌抗菌活性筛选//第五届全国微生物资源学术暨国家微生物资源平台运作服务研讨会论文集, 广州, 2013:164.
[9] Liu JM, Habden X, Guo L, et al. Prauserella endophytica sp. nov., an endophytic actinobacterium isolated from Tamarix Taklamakanensis. Antonie Van Leeuwenhoek, 2015, 107(6):1401-1409.
[10] Huang J. Beijing Fangshan find 2480 wild tamarisk. Beijing Evening News, 2009-02-02 [2015-07-12]. http://news.sina.com.cn/c/2009-02-02/132917133915.shtml. (in Chinese)
黄敬. 北京房山发现2480株野生柽柳. 北京晚报, 2009-02-02 [2015-07-12]. http://news.sina.com.cn/c/2009-02-02/132917133915.shtml.
[11] Zhang YQ, Li WJ, Chen GZ, et al. Comparison of five PCR template preparation methods for fast identification of Actinobacteria. Biotechnology, 2004, 14(5):37-39. (in Chinese)
张玉琴, 李文均, 陈国忠, 等. PCR法快速识别Actinobacteria的五种模板制备方法的比较. 生物技术, 2004, 14(5):37-39.
[12] Thompson JD, Higgins DG, Gibson TJ. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Res, 1994, 22(22):4673-4680.
[13] Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance, and maximum parsimony methods. Mol Biol Evol, 2011, 28(10):2731-2739.
[14] Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol Biol Evol, 1987, 4(4):406-425.
[15] Clinical and Laboratory Standards Institute. M7-A10 —— Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically; approved standard —— tenth edition. Clinical and Laboratory Standards Institute: Wayne, Pennsylvania USA, 2015. http://shop.clsi.org/c.1253739/site/Sample_pdf/M07A10_sample.pdf.
[16] Kingston DGI, Todd L, Williarns DH. Antibiotics of the ostreogrycin complex. 3. The structure of ostreogrycin A. Evidence based on nuclear magnetic double resonance experiments and high-resolution mass spectrometry. J Chem Soc Perkin 1, 1966, 19:1669-1676.
[17] Lefevre J, Glass T, Kolpa MX, et al. Biosynthesis of antibiotics of the Virginiamycin family, 2. Assignment of the 13C-NMR spectra of Virginiamycin M1 and antibiotic A2315A. J Nat Prod, 1983, 46(4):475-480.
[18] Yan M. Progress of studies on virginiamycin. Hubei J Anim Vet Sci,2014, 35(9):70-72. (in Chinese)
闫明. 维吉尼亚霉素的研究进展. 湖北畜牧兽医, 2014, 35(9):70-72.
[19] Zhao HJ. Study on the taxonomy of new virginiamycin-like compound X-435-C producing organism X-435. Beijing: Peking Union Medical College, 2000. (in Chinese)
赵洪娟. 维吉尼霉素类新抗生素X-435-C产生菌X-435的多相分类研究. 北京: 中国协和医科大学, 2000.
[20] Chen M, Wang XY, Zhang YQ. Studies on mutagenesis of virginiamycin producing strain X-435. Microbiology, 2007, 34(6):1090-1092. (in Chinese)
陈睦, 王选一, 张月琴. 维吉霉素产生菌株X-435的诱变育种. 微生物学通报, 2007, 34(6):1090-1092.
[21] Batts DH, Lavin BS, Eliopoulos GM. Quinupristin/dalfopristin and linezolid: spectrum of activity and potential roles in therapy--a status report. Curr Clin Top Infect Dis, 2001, 21:227-251.
[22] Politano AD, Sawyer RG. NXL-103, a combination of flopristin and linopristin, for the potential treatment of bacterial infections including community-acquired pneumonia and MRSA. Curr Opin Investig Drugs, 2010, 11(2):225-236.
[23] Ahmed F, Donaldson WA. Chemistry and biology of the streptogramin A antibiotics. Mini-Rev Org Chem, 2007, 4(2):159-181.
Methods Strain CLR304 was identified on the basis of 16S rRNA sequence analysis. The fermentation broth of strain CLR304 was extracted with EtOAc, and the EtOAc extract was isolated and purified by silica gel column chromatography, thin layer chromatography (TLC) and high performance liquid chromatography (HPLC). The chemical structure of compound 304A was elucidated by combinational analysis of data from UV, HR-ESI-MS, NMR spectra and comparison with those of published data. The antibacterial activity of compound 304A was tested by agar dilution method.
Results Producing strain CLR304 was identified as Streptomyces spp. A bioactive compound named as 304A was isolated from the fermentation broth of strain CLR304 and identified as virginiamycin M1. Compound 304A showed significant antibacterial activity against drug-resistant/sensitive staphylococcus and enterococcus faecium with the MIC values of 1-4 μg/ml.
Conclusion Virginiamycin M1is found from the fermentation broth of endophytic actinomycetes from Tamarix for the first time. The study reveals that psammophytes Tamarix represent new potentially valuable resources for pharmaceutical actinomycetes.
Study on 304A produced by an endophytic streptomyces CLR304 from Tamarix with anti-MRSA bioactivity
LIU Shao-wei, LI Zhou, HU Xin-xin, YOU Xue-fu, SUN Cheng-hang
Objective To discover and identify bioactive component against MRSA from the fermentation broth produced by an endophyticstreptomyces CLR304 from Tamarix.
TAMARIX CHINENSIS;Endophytic Streptomyces;Secondary metabolites;Virginiamycin
SUN Cheng-hang, Email: chenghangsun@hotmail.com
10.3969/cmba.j.issn.1673-713X.2015.06.010
国家自然科学基金(81172963、81373308)
100050 北京,中国医学科学院北京协和医学院医药生物技术研究所
孙承航,Email:chenghangsun@hotmail.com
2015-09-06
www.cmbp.net.cn 中国医药生物技术, 2015, 10(6):514-521
Author Affiliation: Institute of Medicinal Biotechnology, Chinese Academy of Medical Sciences & Peking Union Medical College,Beijing 100050, China
www.cmbp.net.cn Chin Med Biotechnol, 2015, 10(6):514-521

