依博素生物合成基因ste22的异源置换研究
郭连宏,张洋,鲍勇刚,白利平,姜蓉,李元
依博素生物合成基因ste22的异源置换研究
郭连宏*,张洋*,鲍勇刚,白利平,姜蓉,李元
目的 依博素是由链霉菌产生的一种新型胞外多糖,具有显著抗类风湿性关节炎的作用,我们研究已确定了该多糖的生物合成基因簇(ste)。为了对依博素结构进行改造以提高其生物活性,对其生物合成基因 ste22 进行了异源置换研究。方法 采用 PCR 扩增方法从乳酸菌获得了编码葡萄糖糖基转移酶的基因 gtf,通过基因同源重组双交换,从链霉菌中置换了 ste22 基因。采用白细胞介素-1 合成酶活性测定方法及小鼠巴豆油急性炎症模型,初步确定了基因置换株的依博素衍生物生物活性。
异源基因置换; 基因 gtf; 基因 ste22; 链霉菌; 乳酸菌
类风湿性关节炎(RA)是一种自身免疫性疾病,严重影响身体健康,目前尚缺乏有效治疗手段。本课题组从链霉菌 139(Streptomyces sp.139)的发酵液中成功分离获得了一种微生物新型胞外多糖,命名为依博素[1]。药效学证明其对大鼠 II 型胶原诱导的类风湿性关节炎模型和佐剂型类风湿性关节炎模型均有显著抑制作用[2]。作用机制研究证明其对白细胞介素-1β(interlukin-1β,IL-1β)等主要炎症细胞因子的生成及信号传导途径有明显抑制活性[3],依博素有可能发展为治疗 RA 的新药。
我们的研究已确定依博素的生物合成基因簇(ste)由 27 个开放阅读框架组成[4],对基因编码产物的生物功能已进行了较深入研究[5-7],其中基因ste22 编码产物被确认为鼠李糖糖基转移酶[8]。糖基转移酶在微生物胞外多糖(EPS)的生物合成过程中起重要作用,在多糖生物合成基因簇中引入异源糖基转移酶基因有可能改变多糖结构获得新衍生物,并可验证被置换基因功能及多糖构效关系。鉴于乳酸菌与链霉菌同属革兰阳性菌,且其多糖生物合成基因簇中葡萄糖糖基转移酶基因研究较深入,因此本文采用 PCR 扩增方法,从产生胞外多糖的乳酸菌基因组获得其多糖生物合成簇的葡萄糖糖基转移酶基因。以缺失基因 ste22(编码鼠李糖糖基转移酶)的依博素产生菌突变株为受体菌,通过基因同源重组双交换将葡萄糖糖基转移酶基因 gtf置换至依博素生物合成基因簇中,Southern 杂交验证获得了基因置换菌株 Streptomyces sp.139 (gtf-22),分离纯化了该菌株产生的胞外多糖EPS-22g,结果表明其单糖组分与依博素相比葡萄糖比例有显著提高,体内外活性研究结果初步证明该衍生物较依博素活性有所提高。
1 材料与方法
1.1 实验材料
1.1.1 菌株 Streptomyces sp.139(依博素产生菌)、ste 22 基因阻断株 Streptomyces sp.139 (ste22¯)、E.coli DH5α、E.coli ET12567[9]及 E. coli BL21(DE3)均为本实验室保存;Streptococcus thermophilus 购自中国工业微生物菌种中心,CICC编号 20370。
1.1.2 质粒 pKC1139[10]、pGEM-3zf-ErmE*、pGEM-T-tsr 和 pKC22 均为本实验室保存。
1.1.3 培养基 依博素产生菌 Streptomyces sp.139 及基因阻断株发酵培养基:葡萄糖 1.0%、淀粉 1.0%、黄豆饼粉 2.0%、胰蛋白胨 0.2%、牛肉膏 0.2%、酵母提取物 0.4%、K2HPO40.05%、CaCO30.3%,pH 7.2 ~ 7.4;乳酸菌(嗜热链球菌Streptococcus thermophilus)培养基(CM0005):蛋白胨 1%、麦芽提取物 1%、酵母提取物 0.5%、葡萄糖 0.5%、醋酸钠 0.5%、柠檬酸氢铵 0.2%、吐温 80 0.1%、K2HPO40.2%、MgSO4·7H2O 0.2%、MnSO4·H2O 0.005%、CaCO32%,pH 7.0。
1.1.4 动物 雄性昆明小鼠,体重 18 ~ 20 g,中国医学科学院实验动物研究所提供。合格证号:SCXK(京)2005-0013。动物饲养及实验过程遵循中国医学科学院实验动物管理规定。
1.1.5 试剂 限制性内切酶、T4 DNA 连接酶、Taq DNA 聚合酶、pfu 高保真 DNA 聚合酶、RNase、溶菌酶等分别购自 Promega、TaKaRa 公司;DNA 片段纯化试剂盒购自 SunBio;Southern杂交试剂盒购自 Amersham;重组 IL-1β 转化酶(ICE)及 ICE 酶底物 Ac-Tyr-Val-Ala-Asp-AMC为美国 Merck 公司产品。
1.1.6 仪器 Techgene gradient 型 PCR 仪为英国 Techne 公司产品;Victor X5 型微孔板荧光检测仪为美国 PerkinElmer 公司产品;HP5890 气相色谱分析仪为美国惠普公司产品;PRISM 377XL 测序仪为美国 ABI 公司产品。
1.2 方法
1.2.1 菌种培养 Streptomyces sp.139 及其变株在 28 ℃ 振荡培养(250 r/min);乳酸菌Streptococcus thermophilus 在 40 ℃ 以 250 r/min振荡培养。
1.2.2 DNA 分离和 Southern 杂交 乳酸菌振荡培养 48 h 后,按 Sambrook 和 Russell[11]所述方法分离总 DNA。链霉菌质粒及基因组 DNA 采用Kieser 等[12]提供的方法进行。Southern 杂交根据试剂盒生产商所述步骤进行。
1.2.3 乳酸菌 Streptococcus thermophiles 葡萄糖糖基转移酶基因 gtf 的克隆 采用 PCR 扩增法,以乳酸菌基因组 DNA 为模板进行。根据已知乳酸菌 Streptococcus thermophilus Sfi6 的糖基转移酶基因 epsI(GenBank:AAC44016.1)和 epsF (GenBank:AAC44013.1)序列,设计了引物P1-P2,P1:5' GC GAATTCTCTAGAATGGCGTGGC TAATTAA-AATG 3'(横线部分分别为 EcoR I 和 Xba I 酶切位点),P2:5' GCGAATTCTTAATCGCTT TCAATA 3'(横线部分为 Sal I 酶切位点);PCR 扩增条件:94 ℃ 变性 10 min;再行 94 ℃ 变性1 min,47 ℃ 退火1 min,72 ℃ 延伸 3 min,循环30 次;72 ℃ 延伸 10 min。扩增获得的 DNA 片段克隆至 EcoR I 和 Xba I 双酶切的质粒 pUC18,构建获得重组质粒 pUC-gtf,经测序证明 DNA 片段为葡萄糖糖基转移酶基因(gtf)。按分子克隆手册提供方法将重组质粒 pUC-gtf 转化至大肠杆菌E.coli DH5α 获得重组菌 E.coli DH5α(pUC-gtf),并制备重组质粒 pUC-gtf。
1.2.4 构建基因置换株 Streptomyces sp.139 (gtf-22)
1.2.4.1 硫链丝菌素抗性基因的克隆 以克隆有硫链丝菌素抗性基因的质粒 pGEM-T-tsr 为模板,采用 PCR 扩增方法获得该抗性基因(tsr)。采用引物 P3-P4,P3:5' GCTCTAGAAGGCGAATACTT CATATG 3'(横线部分为 Sal I 酶切位点),P4:5' G CGAATTCTCTAGATGATCATCACTGACGAAT 3'(横线部分为 Hind III 和 Xba I 酶切位点);PCR扩增条件:94 ℃ 变性 10 min;再行 94 ℃ 变性1 min,58 ℃ 退火 0.5 min;72 ℃ 延伸 2 min;循环 30 次;72 ℃ 延伸 10 min。经 Sal I 及 Hind III酶切验证后测序与已报道的硫链丝菌素抗性基因序列一致。扩增获得的 1.06 kb 基因片段(tsr)和Sal I-Hind III 酶切的重组质粒 pUC-gtf 连接,进一步经 Xba I 酶切后,从中分离获得 tsr-gtf 的 DNA片段。
1.2.4.2 乳酸菌葡萄糖糖基转移酶基因对依博素生物合成基因的置换 采用基因同源重组双交换方法进行[12]。在以往构建 ste22 基因阻断菌株Streptomyces sp.139(ste22¯)过程中,我们采用了构建的基因阻断质粒 pKC22[8],该质粒含有基因 ste22 上游片段 F1(1.1 kb)及下游片段 F2 (1.9 kb)。将 thior-gtf DNA 片段与 Xba I 酶切的pKC22 连接,获得基因置换质粒 pKC22-gtf,该质粒转化至缺失甲基化菌株 E.coli ET12567[9],制备质粒 pKC22-gtf。采用聚乙二醇(PEG)介导的原生质体转化方法[12],将质粒 pKC22-gtf 转化至ste22 基因阻断菌株 Streptomyces sp.139(ste22¯)。转化平板置于 28 ℃ 培养 16 ~ 20 h,然后覆盖一层含有硫链丝菌素(浓度为 50 μg/ml)的软琼脂(琼脂浓度为 0.6%),转化平板继续置于 28 ℃ 培养 2 d,待麻点状菌落出现后再于 37 ℃ 继续培养7 d,得到转化子。将生长出来的菌落同时影印到含有硫链丝菌素(50 μg/ml)和安普霉素(40 μg/ml)的 R2 培养基平板上,双交换突变株转化子菌落表现为 ThiorAms。在该菌株中,存在于 Streptomyces sp.139(ste22¯)染色体的卡那霉素抗性基因 kmr已被乳酸菌来源葡萄糖糖基转移酶基因 gtf 和硫链丝菌素抗性基因 tsr 片段 tsr-gtf 所置换。
随机挑选若干 ThiorAms菌落,提取Streptomyces sp.139(ste22¯)及基因置换株Streptomyces sp.139(gtf-22)的基因组 DNA[12],并分别以 Kpn I 酶切 DNA 以用于 Southern blot分析。
1.2.5 胞外多糖的分离 依博素产生菌Streptomyces sp.139 及基因置换变株 Streptomyces sp.139(gtf-22)分别在 28 ℃,250 r/min 振荡培养96 h,按照已建立的分离方法[1],分别从发酵滤液中分离获得依博素及其衍生物 EPS-22g。
1.2.6 胞外多糖的单糖分析 多糖 5 mg 用 5 ml (2 mol/L)三氟乙酸溶解,120 ℃ 水解 2 h。水解的糖残基分别加入 2 ml 氢氧化钠(0.05 mol/L)和40 ~ 50 mg 的硼氢化钠,室温放置过夜,滴入乙酸至无气泡为止。氮气吹干后,加入 1 ml 甲醇溶解再用氮气吹干。反复多次以除去硼酸根,残渣真空干燥过夜。加入吡啶 0.5 ml,乙酸酐 0.5 ml,在100 ℃ 下保温 30 min 后,用氮气除去反应试剂。加入 1 ml 的二氯甲烷溶解,过滤后收集二氯甲烷相,用氮气吹干后再用二氯甲烷定容。冷却后样品进行气相色谱分析。半乳糖醛酸分析按 Bitter 和Muir[13]报道方法进行。
1.2.7 IL-1β 转化酶的活性测定 ICE 酶是重要炎症细胞因子 IL-1β 的生物合成关键酶。酶活性测定根据 Koizumi 等[14]报道方法进行。以荧光标记的多肽 Ac-Tyr-Val-Ala-Asp-AMC 为底物,分别测定了依博素及其衍生物 EPS-22g 对重组 ICE 酶的抑制活性。其反应体系为:1 mmol/L 磷酸钠缓冲液(pH 6.8)、0.2% 牛血清白蛋白、重组 ICE酶(5 IU)、10 μmol/L ICE 底物 Ac-Tyr-Val-Ala-Asp -AMC 及不同浓度依博素及其衍生物 EPS-22g,总体积 100 μl,37 ℃ 避光反应 2 h,以只加重组酶的样品为对照,采用多标记微孔板检测仪在激发光380 nm、发射光 460 nm 条件下测定产物荧光强度。根据样品及对照的荧光强度值可以计算出对ICE 的抑制率。

1.2.8 巴豆油诱导的小鼠急性炎症模型 小鼠按体重分为模型组、依博素给药组及依博素衍生物EPS-22g 3 个实验组,每组 8 只小鼠。依博素及EPS-22g 分别溶于水,浓度为 100 mg/ml,模型组给水(10 ml/kg),灌胃给药。小鼠按体重给药 60 min后,左耳涂 2% 巴豆油液 50 μl。4 h 后处死小鼠,剪下双耳,用直径为 8 mm 的不锈钢铳子冲下左右耳片,分别称重,以左右耳片重量之差作为肿胀程度,计算对炎症的抑制率。
样品组抑制率(%)=(模型组平均肿胀程度 -样品组平均肿胀程度)/模型组平均肿胀程度 × 100%
1.3 统计学处理
2 结果
2.1 乳酸菌葡萄糖糖基转移酶基因 gtf 的克隆
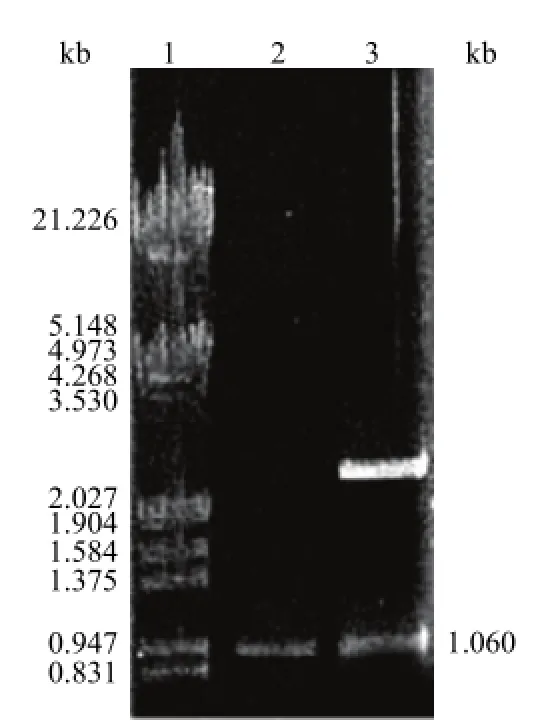
图1 重组质粒 pUC-gtf 酶切结果的琼脂糖凝胶电泳分析图Figure 1 Agrose gel electrophoresis of pUC-gtf digested with EcoR I-Sal I
以乳酸菌总 DNA 为模板,经 PCR 扩增获得DNA 片段,琼脂糖凝胶电泳分析(SDS-PAGE)结果显示其分子量为 0.94 kb(图 1)。将该片段克隆至 EcoR I 和 Xba I 双酶切的质粒 pUC18 构建获得重组质粒 pUC-gtf,经测序证明与已知乳酸菌Streptococcus thermophilus Sfi6 中葡萄糖糖基转移酶基因 eps I 一致。
2.2 硫链丝菌素抗性基因的克隆
以质粒 pGEM-T-tsr 为模板,利用引物 P3 和P4 扩增硫链丝菌素抗性基因,琼脂糖凝胶电泳显示扩增条带分子量为 1.060 kb,符合预期大小(图 2)。PCR 产物经 Sal I-Hind III 酶切后与用相同酶切的质粒 pUC-18 连接,得到重组质粒pUC-tsr,经 Sal I 及 Hind III 酶切验证后测序,结果与已报道的硫链丝菌素抗性基因序列一致。
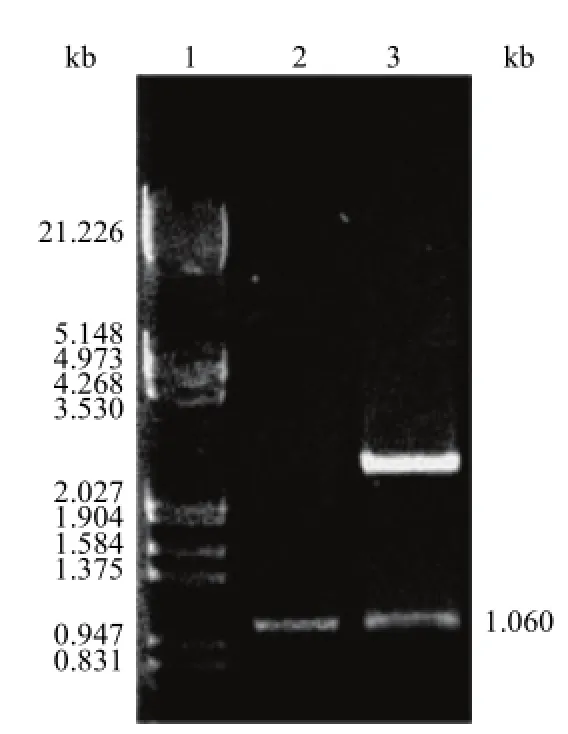
图2 重组质粒 pUC- tsr 的琼脂糖凝胶电泳图Figure 2 Agrose gel electrophoresis of pUC-tsr digested with Sal I/Hind III
2.3 异源基因置换株 Streptomyces sp.139 (gtf-22)构建的确认
为了证明 Streptomyces sp.139(ste22¯)染色体的卡那霉素抗性基因(kmr)已被乳酸菌来源葡萄糖糖基转移酶基因(gtf)和硫链丝菌素抗性基因(tsr)片段 tsr-gtf 所置换,分别提取异源置换菌株 Streptomyces sp.139(gtf-22)及链霉菌Streptomyces sp.139(ste22¯)DNA,以 Kpn I 酶切后进行 Southern 杂交实验。以荧光标记的 ste22基因上游片段 F1 为探针,结果显示 Streptomyces sp.139(ste22¯)DNA 处出现一条 2.35 kb 的阳性杂交信号条带,而 Streptomyces sp.139(gtf-22)总DNA 处出现一条 3.25 kb 的阳性杂交条带,结果与预期相同(图 3B)。实验结果表明,通过同源重组双交换,Streptomyces sp.139(ste22¯)的卡那霉素抗性基因 kmr确被乳酸菌来源葡萄糖糖基转移酶基因 gtf 和硫链丝菌素抗性基因片段 tsr-gtf 所置换,证明 Streptomyces sp.139(gtf-22)为异源基因置换株。图 3A 为本研究进行的基因同源重组双交换示意图。
2.4 胞外多糖的单糖分析

图3 ste22 基因的异源置换(A)及 Southern 杂交验证(B)Figure 3 Heterologous replacement diagram of ste22 gene with a double crossover via homologous (A) and Southern blot analysis (B)
气相色谱方法分析依博素及其衍生物EPS-22g 的单糖组分,图 4A,4B 显示,两种多糖均由相同种类单糖组成,分别为葡萄糖、甘露糖、阿拉伯糖、半乳糖、果糖、木糖、鼠李糖。但是EPS-22g 与依博素相比,葡萄糖比例从 2.14% 增至 48.64%,提高显著,同时 EPS-22g 较依博素其他单糖比例均不同程度降低(图 4C)。采用 Bitter和 Muir[13]报道方法也证明,两种胞外多糖均含有半乳糖醛酸。
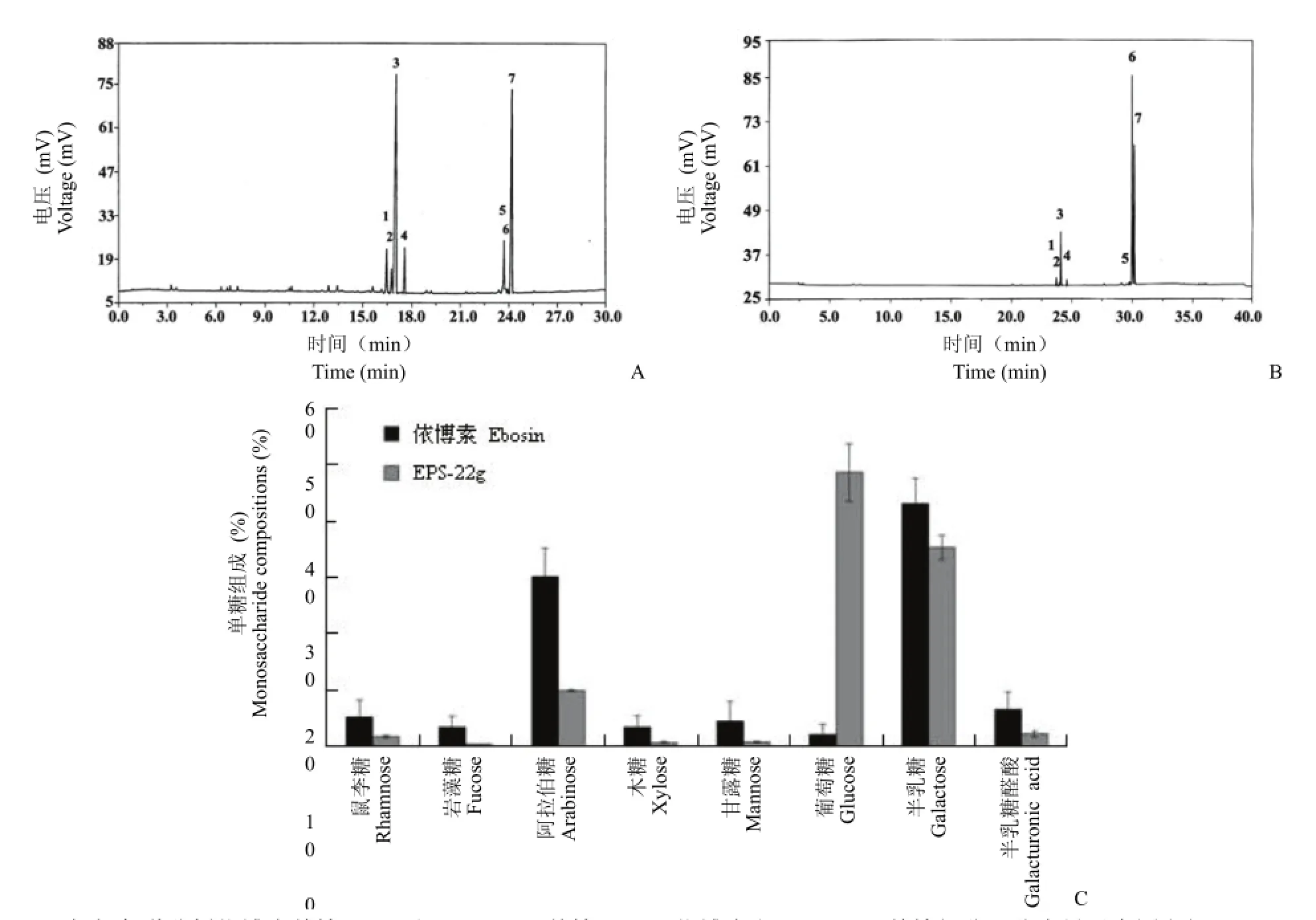
图4 气相色谱分析依博素单糖(A)和 EPS-22g 单糖(B);依博素和 EPS-22g 单糖组分百分含量示意图(C)Figure 4 GC chromatograms of sugar analysis of ebosin (A) and EPS-22g (B); Monosaccharide compositions of ebosin and EPS-22g (C)
2.5 依博素及其衍生物 EPS-22g 对 ICE 酶活性的抑制作用
实验结果显示(图 5),在 100 μl 反应液中,依博素及 EPS-22g 样品浓度分为 2.5 和 1.25 μg/μl的条件下,对 ICE 酶的活性的抑制率分别为:41.79%、55.3%(P < 0.05)和 9.85%、23.96%(P <0.05)。
上述结果表明 EPS-22g 较依博素对 ICE 酶的抑制作用强。鉴于 ICE 酶是重要炎症细胞因子IL-1β 的生物合成关键酶,因此可以说明 EPS-22g比依博素能更有效抑制 IL-1β 的表达水平。
2.6 依博素及其衍生物 EPS-22g 对巴豆油诱导的小鼠急性炎症抑制作用
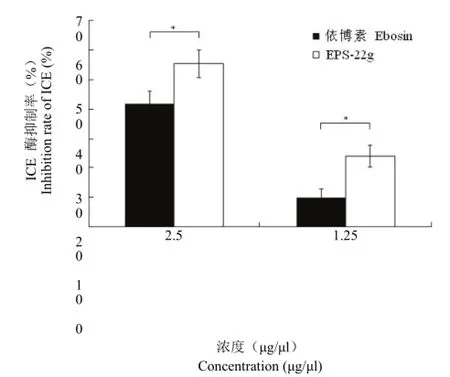
图5 依博素和 EPS-22g 对 ICE 酶的作用(*P < 0.05)Figure 5 Effect of ebosin and EPS-22g on ICE activity (*P <0.05)
为了确定依博素及其衍生物 EPS-22g 对巴豆油诱导的小鼠急性炎症的作用,在巴豆油诱导前1 h,分别对实验组小鼠灌胃依博素及 EPS-22g 100 mg/kg,和未给药急性炎症小鼠对照组比较,EPS-22g 对耳肿胀抑制率为 27.4%(P < 0.05),而依博素抑制率为 18.3%,结果表明,EPS-22g 对巴豆油诱导的小鼠急性炎症抑制作用略高于依博素(图 6)。
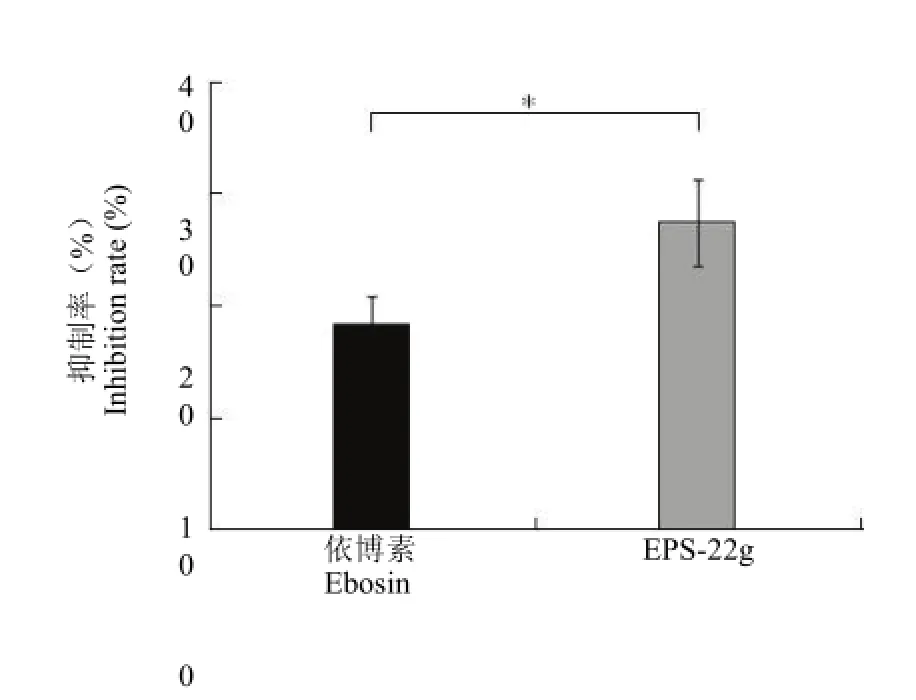
图6 100 mg/kg 依博素和 EPS-22g 对巴豆油诱导的急性炎症小鼠作用(*P < 0.05)Figure 6 Effect of ebosin and EPS-22g on the acute inflamed mice induced by croton oil (*P < 0.05)
3 讨论
微生物胞外多糖(EPS)是自然界微生物分泌至细胞表面的多聚物,不同的 EPSs 的重复单元各不相同,包括组成单糖的种类和数量不同,糖苷键连接方式的不同[15]。尽管这些 EPSs 表现出很少的共同特性,但由重复单元组成的多糖的生物合成途径基本相同,均通过糖基转移酶(GTF)将单糖从核苷酸糖顺序性转移而装配到脂类载体上形成重复单元,随后是这些重复单元的聚合和输出,形成细胞表面多糖[15]。
EPSs 在工业特别是乳制品业中已广为应用,近年来发现其可能具有药用前景,例如可增强免疫活性、降低胆固醇、抗糖尿病及抗癌等作用[16-18],一些 EPS 具有体内抗炎活性[19],Nowak 等[20]还报道了胞外多糖对类风湿性关节炎(RA)的抑制作用。依博素是一种由链霉菌产生的新型胞外多糖,药效学研究证明其对类风湿性关节炎有较好抑制作用。作用机制研究确定其对 IL-1β 等重要炎症细胞因子生物合成及信号传导途径均有显著抑制活性[20],有可能发展成为治疗 RA 的新药。
采用遗传工程可以改变 EPSs 生物合成,通过定向改变单糖组成和链长获得符合设计的多糖。如对编码黄原胶侧链合成的基因进行异源基因置换,获得了多种黄原胶衍生物,这些衍生物的区别在于侧链的乙酰化、丙酮酸化或截短链长,与黄原胶相比黏度发生了变化[21]。异源表达糖基转移酶基因可以在 EPS 生物合成不同阶段改变其单糖组成,从而使其性质发生变化,如将新的或已知的糖基转移酶基因导入乳酸菌可以有效控制新 EPS 衍生物的结构[22-23]。将乳酸菌 EPS 生物合成基因簇转入胞外多糖非产生菌,在异源宿主菌中已获得了成功表达[24]。
本课题组以往研究工作确定了依博素生物合成基因簇 ste[4],已对其生物合成基因性质及编码产物进行了较多研究。本研究拟采用异源基因置换方法获得活性较高的新结构依博素衍生物。乳酸菌是重要工业微生物,与链霉菌同属革兰阳性菌,其EPS 生物合成基因簇已有较多研究[25]。本文以乳酸菌葡萄糖糖基转移酶基因置换链霉菌编码鼠李糖糖基转移酶的基因 ste22,经 Southern 杂交证明,成功构建了基因置换菌 Streptomyces sp.139 (gtf-22)。研究结果表明置换株产生了依博素新衍生物 EPS-22g,其单糖组成比例与依博素相比有显著变化,特别是葡萄糖比例上升突出,从 2.14% 上升至 48.64%,这表明基因置换导致多糖由单糖组成的重复单元序列发生了结构变化。研究结果显示,EPS-22g 对 ICE 酶活性抑制率高于依博素,由于 ICE 酶是重要炎症细胞因子 IL-1β 生物合成关键酶,因此其对 IL-1β 生成有较强抑制作用。巴豆油诱导的小鼠急性炎症模型结果显示,EPS-22g对体内急性炎症抑制活性高于依博素。
上述结果表明,本研究采用异源基因置换获得了依博素新衍生物 EPS-22g,其体内外活性初步研究结果显示均高于依博素,推测葡萄糖比例的显著升高可能与活性提高相关。鉴于微生物胞外多糖结构复杂,因此有关依博素结构与生物活性的相关性尚需进行深入研究。根据本文初步结果可以推测,异源基因置换依博素生物合成基因可能是获得高活性多糖衍生物的一种重要途径。
[1]Jing C, Jianbo W, Yuan L, et al. A new IL-1 receptor inhibitor 139A:fermentation, isolation, physico-chemical properties and structure. J Antibiot (Tokyo), 2003, 56(2):87-90.
[2]Zhang Y, Wang LF, Li Y, et al. Anti-inflammatory effect of Ebosin on rat collagen-induced arthritis through suppression production of interferon-1β, interferon-6 and tumor necrosis factor-α. Eur J Inflamm,2013, 11(3):677-688.
[3]Zhang Y, Guo LH, Li Y. Effect of Ebosin on the inflammatory cytokines in THP-1 cells. Acta Pharma Sinica, 2013, 48(5):661-667. (in Chinese)
张洋, 郭连宏, 李元. 依博素对 THP-1细胞炎症细胞因子的作用.药学学报, 2013, 48(5):661-667.
[4]Wang LY, Li ST, Li Y. Identification and characterization of a newexopolysaccharide biosynthesis gene cluster from Streptomyces. FEMS Microbiol Lett, 2003, 220(1):21-27.
[5]Li X, Wang L, Li Y, et al. Cloning and characterization of a glucosyltransferase and a rhamnosyltransferase from Streptomyces sp. 139. J Appl Microbiol, 2010, 108(5):1544-1551.
[6]Qi XQ, Sun QL, Li Y, et al. Identification of alpha-D-glucose-1-phosphate cytidylyltransferase involved in Ebosin biosynthesis of Streptomyces sp. 139. Appl Microbiol Biotechnol,2009, 83(2):361-368.
[7]Zhang Y, Zhou J, Chang M, et al. Characterization and functional evidence for Ste27 of Streptomyces sp. 139 as a novel spermine/ spermidine acetyltransferase. Biochem J, 2012, 443(3):727-734.
[8]Zhang T, Wang L, Xu G, et al. Disruption of the ste22 gene encoding a glycosyltransferase and its function in buisynthesis of Ebosin in Streptomyces sp.139. Curr Microbiol, 2006, 52(1):55-59.
[9]MacNeil DJ, Gewain KM, Ruby CL, et al. Analysis of Streptomyces avermitilis genes required for avermecin biosynthesis utilizing a novel integration vector. Gene, 1992, 111(1):61-68.
[10]Bierman M, Logan R, O'Brien K, et al. Plasmid cloning vectors for conjugal transfer of DNA from Escherichia coli to Streptomyces spp. Gene, 1992, 116(1):43-69.
[11]Joseph S, David WR. Molecular cloning: a laboratory manual. 3rd ed. Cold spring Harbor. New York: Cold Spring Harbor Laboratory Press,2001.
[12]Kieser T, Bibb MJ, Butter MJ, et al. Practical Streptomyces Genetics. Norwich, England: The John Innes Foundation, 2000.
[13]Bitter T, Muir HM. A modified uronic acid carbazole reaction. Anal Biochem, 1962, 4:330-334.
[14]Koizumi F, Matsuda Y, Nakanishi S. EI-1941-1 and -2, novel interleukin-1beta converting enzyme inhibitors produced by Farrowia sp. E-1941. I. Biochemical characterization of EI-1941-1 and -2. J Antibiot (Tokyo), 2003, 56(5):464-469.
[15]van Kranenburg R, Boels IC, Kleerebezem M, et al. Genetics and engineering of microbial exopolysaccharides for food: approaches for the production of existing and novel polysaccharides. Curr Opin Biotechnol, 1999, 10(5):453-498.
[16]Hajime N, Yutaka S, Tetsuji H. Cholesterol lowering activity of ropy fermented milk. J Food Sci, 1992, 57(6):1327-1329.
[17]Jin M, Lu Z, Huang M, et al. Effects of Se-enriched polysaccharides produced by Enterobacter cloacae Z0206 on alloxan-induced diabetic mice. Int J Biol Macromol, 2012, 50(2):348-352.
[18]Fernandes MB, Habu S, de Lima MA, et al. Influence of drying methods over in vitro antitumoral effects of exopolysaccharides produced by Agaricus blazei LPB 03 on submerged fermentation. Bioprocess Biosyst Eng, 2011, 34(3):253-256.
[19]Joo JH, Yun JW. Structural and molecular characterization of extracellular polysaccharides produced by a new fungal strain,Trichoderma erinaceum DG-312. J Microbiol Biotechnol, 2005,15(6):1250-1257.
[20]Nowak B, Ciszek-Lenda M, Sróttek M, et al. Lactobacillus rhamnosus exopolysaccharide ameliorates arthritis induced by the systemic injection of collagen and lipopolysaccharide in DBA/1 mice. Arch Immunol Ther Exp (Warsz), 2012, 60(3):211-220.
[21]Hassler RA, Doherty DH. Genetic engineering of polysaccharide structure: production of variants of xanthan gum in Xanthomonas campestris. Biotechnol Prog, 1998, 6(3):182-186.
[22]Boels IC, Ramos A, Kleerebezem M, et al. Functional analysis of the Lactococcus lactis galU and galE genes and their impact on sugar nucleotide and exopolysaccharide biosynthesis. Appl Environ Microbiol, 2001, 67(7):3033-3040.
[23]Jolly L, Vincent SJ, Duboc P, et al. Exploiting exopolysaccharides from lactic acid bacteria. Antonie Van Leeuwenhoek, 2002, 82(1-4):367-374.
[24]Germond JE, Delley M, D'Amico N, et al. Heterologous expression and characterization of the exopolysaccharide from Streptococcus thermophilus Sfi39. Eur J Biochem, 2001, 268(19):5149-5156.
[25]Bolotin A, Quinquis B, Renault P, et al. Complete sequence and comparative genome analysis of the dairy bacterium Streptococcus thermophilus. Nature Biotech, 2004, 22(12):1554-1558.
Methods Gene ste22 has previously been demonstrated to code for a rhamnosyltransferase. With PCR amplification, a gene encoding glucosyltransferase was isolated from Streptococcus thermophilus. Using a double crossover via homologous recombination, the gene ste22 in Streptomyces sp.139 was replaced by the gene encoding glucsyltransferase originated from Streptococcus thermophilus. With the assay of IL-1β-converting enzyme (ICE) and acute inflamed mice model induced by croton oil,primary results of activity for novel ebosin derivative produced by the heterologous gene replacement strain was identified.
Results The heterologous gene replacement strain Streptomyces sp.139 (gtf-22) was constructed, which produced a novel ebosin derivative EPS-22g. This alteration resulted in EPS-22g with its monosaccharide composition dramatically changed, especially in the proportion of glucose, which increased from 2.14% (ebosin) to 48.64% (EPS-22g). Comparing with ebosin, EPS-22g exhibited a higher inhibitory effect on the activity of IL-1β-converting enzyme (ICE). Experiments with acute inflamed mice induced by crotonoil showed higher anti-inflammatory activity of EPS-22g than that of ebosin.
Conclusion To generate derivatives of ebosin for better activity, the strategy of ebosin biosynthesis gene replaced by heterologous gene may be more useful.
Study of ebosin biosynthesis gene ste22 replaced by heterologous gene
GUO Lian-hong, ZHANG Yang, BAO Yong-gang, BAI Li-ping, JIANG Rong, LI Yuan
Objective Ebosin is a novel exopolysaccharide produced by Streptomyces and evidenced to possess an anti-rheumatic arthritis activity. The ebosin biosynthesis gene cluster has been identified. Here, we aim to generate derivatives of ebosin for better activity,and replace ebosin biosynthesis gene ste22 by heterologous gene.
Heterologous gene replacement; Gene gtf; Gene ste22; Streptomyces; Streptococcus thermophilus
LI Yuan, Email: yuanwli@263.com
10.3969/cmba.j.issn.1673-713X.2015.03.007
国家自然科学基金重点项目(30530830);“重大新药创制”国家科技重大项目(2009ZX09301-003)
100050 北京,中国医学科学院北京协和医学院医药生物技术研究所卫生部抗生素生物工程重点实验室
李元,Email:yuanwli@263.net
2015-02-06
*同为第一作者
结果 获得了葡萄糖糖基转移酶基因置换株 Streptomyces sp.139(gtf-22),产生了依博素新衍生物 EPS-22g。研究表明该衍生物与依博素相比,葡萄糖比例从 2.14% 显著上升到 48.64%,体外活性实验显示,其对炎症细胞因子白细胞介素-1 合成关键酶 ICE 抑制率高于依博素,小鼠巴豆油急性炎症体内活性分析结果证明,该衍生物抑制活性亦高于依博素。
结论 采用异源基因置换生物合成基因的方法改造依博素产生菌,可能是获得生物活性较好新多糖衍生物的一种有效手段。
www.cmbp.net.cn 中国医药生物技术, 2015, 10(3):228-235
Author Affiliation: Key Laboratory of Biotechnology of Antibiotics of Ministry of Health, Institute of Medicinal Biotechnology,Chinese Academy of Medical Sciences & Peking Union Medical College, Beijing 100050, China
www.cmbp.net.cn Chin Med Biotechnol, 2015, 10(3):228-235

