食源性乳杆菌中耐药基因的转移研究
马沁沁,付 雨,孙 群,*
(1.四川大学生命科学学院,生物资源与生态环境教育部重点实验室,四川 成都 610064;2.四川师范大学生命科学学院,四川 成都 610101)
食源性乳杆菌中耐药基因的转移研究
马沁沁1,2,付雨1,孙群1,*
(1.四川大学生命科学学院,生物资源与生态环境教育部重点实验室,四川 成都610064;2.四川师范大学生命科学学院,四川 成都610101)
为评估食品中乳杆菌(Lactobacillus spp.)耐药基因转移所引起的食品安全风险,对耐药乳杆菌菌株进行了红霉素、万古霉素和氟喹诺酮类药物耐药基因的聚合酶链式反应(polymerase chain reaction,PCR)检测,植物乳杆菌(Lactobacillus plantarum)、嗜酸乳杆菌(Lactobacillus acidophilus)和德氏乳杆菌(Lactobacillus delbrueckii)中检测到红霉素耐药基因msrC,万古霉素和氟喹诺酮类药物耐药基因检测结果为阴性。以供体菌和受体菌菌液体积比为10∶1,通过滤膜杂交法,进行乳杆菌耐药基因msrC对金黄色葡萄球菌(Staphylococcus aureus)的体外转移,但仅获得1 个接合子菌落;乳杆菌对粪肠球菌(Enterococcus faecalis)msrC基因体外转移频率约为2.2×10-2,与敏感菌相比,接合子生长速率减缓。在无特定病原体(specific pathogen free,SPF)裸鼠肠道内进行的L. delbrueckii对E. faecalis的msrC基因体内转移中未检测到接合子。结果表明,乳杆菌携带的可转移耐药基因在肠道内传递至致病菌或机会致病菌的机率很小,且接合子生长比敏感菌缓慢,在抗生素选择压降低或消失的条件下易被淘汰。因此,目前食源性乳杆菌中耐药基因可能引起的食品安全风险较小。
耐药性;乳杆菌;基因转移;接合子;食品安全
自抗生素临床应用以来,细菌耐药性的产生和传播就成为威胁公众健康的主要因素之一。细菌的抗生素耐药性分为固有耐药和获得性耐药。获得性耐药是通过细菌的突变或通过基因水平转移(horizontal genetransfer,HGT)获得外源耐药遗传因子而产生,可以在细菌间传播[1]。欧洲食品安全局(European Food Safety Authority,EFSA)提出具有固有耐药或由染色体突变获得耐药性的菌株可以用作饲料添加剂,而携带可转移耐药基因的菌株发生耐药基因横向转移的风险最大,不应被引入食物链[2]。
乳酸菌(lactic acid bacteria,LAB)被广泛用于食品生产,一直被认为是对人体没有危害的天然发酵剂和益生菌,能够平衡肠道菌群,对人体健康有益[3]。但最近发现LAB携带多种抗生素耐药基因,可能作为抗生素耐药基因的储存库[4-5],在一定条件下通过HGT转移至致病菌或机会致病菌。目前关于乳杆菌(Lactobacillus spp.)耐药基因转移的报道有限,且不同研究中,结果不尽相同。后基因组学的分析发现在人肠道的天然微生物群落和致病菌之间的基因转移可能存在某种屏障[6]。植物乳杆菌(Lactobacillus plantarum)携带的红霉素耐药基因ermB对粪肠球菌(Enterococcus faecalis)的转移频率在无菌小鼠体内比在体外提高了3~4 个数量级[7],而罗伊乳杆菌(Lactobacillus reuteri)的四环素耐药基因tetW在人肠道内并未与肠球菌(Enterococcus spp.)、双歧杆菌(Bifi dobacteria spp.)、乳杆菌发生水平转移[8]。
本实验室研究发现分离自四川地区人肠道及发酵食品的52 株乳杆菌中,60%以上的菌株分别对红霉素、万古霉素、诺氟沙星和环丙沙星耐药,且受试菌株对上述抗生素的最低抑菌浓度(minimum inhibition concentration,MIC)分布均呈现双峰或多峰分布,因此耐药菌株的耐药性很可能是获得性耐药[3,9]。本研究拟对耐药乳杆菌菌株是否携带可转移的耐药基因进行鉴定,并进行乳杆菌对肠道致病菌和机会致病菌的耐药基因体外及体内转移研究,为食品中乳杆菌耐药性可能引起的食品安全风险评估提供理论依据。
1 材料与方法
1.1材料与试剂
1.1.1菌株
四川大学公共卫生学院保存的分离自健康人肠道及四川大学生命科学学院实验室分离自酸奶及四川泡菜的乳杆菌菌株共52 株;对红霉素敏感的金黄色葡萄球菌(Staphylococcus aureus)ATCC 29213及E. faecalis ATCC 29212,购自北京中原公司。
1.1.2实验动物
无特定病原体(specific pathogen free,SPF)BALB/c裸鼠由四川大学实验动物中心提供,小鼠平均年龄2 个月。
1.1.3试剂
细菌基因组DNA小量提取试剂盒及2×Taq Plus PCR MasterMix北京天根生化科技有限公司;质粒DNA小量提取试剂盒美国Axygen公司;胶回收试剂盒美国Omega公司;PCR引物英潍捷基(上海)贸易有限公司合成;红霉素美国Sigma-Aldrich公司;MRS琼脂及MRS肉汤培养基广州环凯生物制品有限公司;粪肠球菌琼脂及甘露醇-氯化钠琼脂培养基青岛海博生物技术有限公司;滤膜及滤头上海新亚净化器件厂。
1.2仪器与设备
PTC-200型PCR仪、DYY-Ⅱ恒压恒流电泳仪、凝胶成像仪美国Bio-Rad公司;生物安全柜青岛海尔集团;隔水式恒温培养箱上海一恒科学仪器有限公司;恒温振荡培养箱上海智城分析仪器制造有限公司。
1.3方法
1.3.1耐药基因的聚合酶链式反应(polymerase chain reaction,PCR)检测
提取耐药乳杆菌菌株基因组DNA,PCR检测红霉素、万古霉素及氟喹诺酮类药物耐药基因。PCR扩增引物见表1。PCR反应条件:95 ℃ 5 min;95 ℃ 1 min,45~62 ℃ 1 min,72 ℃ 2 min,30 次循环;72 ℃ 10 min。对目的条带用胶回收试剂盒回收,送上海美吉生物科技有限公司测序。

表1 抗生素耐药基因扩增引物Table 1 Primers chosen for detection of antimicrobial resistance determinants

续表1
1.3.2质粒中红霉素耐药基因的检测及体外基因转移
提取红霉素耐药乳杆菌菌株质粒DNA,以此为模板,分别扩增mefA/E基因和msrC基因。
分离自酸奶的德式乳杆菌(Lactobacillus delbrueckii)M1号菌株为供体,对红霉素敏感的E. faecalis ATCC 29212和S. aureus ATCC 29213为受体,供体菌和受体菌菌液体积比为10∶1,以滤膜杂交法[3]进行耐药基因体外转移,重复3 次。
1.3.3接合子的鉴定
挑取ATCC 29213接合子(29213C)及3 个ATCC 29212接合子(29212C)单菌落接种于含有5 μg/mL红霉素的BHI肉汤培养基中,37 ℃培养48~72 h后提取质粒DNA,PCR检测msrC基因。
1.3.4接合子MIC的测定
采用美国临床实验室标准化协会(Clinical and Laboratory Standards Institute,CLSI)公布的琼脂稀释法测定接合子的MIC值[14]。
1.3.5体内基因转移
将小鼠分为实验组、对照组和空白组,每组3 只小鼠,均为雄性小鼠。
根据酸奶中乳杆菌的浓度[15]及泡菜汁中乳杆菌的浓度[16-17],以及实验用裸鼠与我国18~69 岁健康汉族成年人平均体质量比[18],确定M1和E. faecalis ATCC 29212的灌喂量[7,19]。
向实验组小鼠灌喂109CFU/d ATCC 29212,喂食3 d后向实验组小鼠灌喂108CFU/d M1号菌株和109CFU/d ATCC 29212,喂食3 d。向对照组小鼠灌喂109CFU/d ATCC 29212,喂食6 d。向空白组小鼠灌喂灭菌生理盐水6 d。
取小鼠肠道剪碎,加入适量无菌生理盐水研磨,研磨后将研磨液转入10 mL离心管中,涡旋1 min。取研磨液进行10 倍梯度稀释,取实验组和对照组研磨液及稀释液100 μL分别涂布于含5 μg/mL红霉素的粪肠球菌琼脂和MRS琼脂,将空白组研磨液及稀释液涂布于不含抗生素的粪肠球菌琼脂,37 ℃培养24~72 h后进行菌落计数。
2 结果与分析
2.1乳杆菌耐药基因的扩增
在受试菌株中未得到12 个已知万古霉素耐药基因及10 个已知氟喹诺酮类药物耐药基因阳性片段。对红霉素耐药基因的扩增中,在8 株红霉素耐药的L. plantarum中检测到mefA/E基因;分别有2 株L. delbrueckii、1 株发酵乳杆菌(Lactobacillus fermentum)、1 株嗜酸乳杆菌(Lactobacillus acidophilus)以及22 株L. plantarum携带msrC基因(图1)。在红霉素耐药菌株的质粒中均检测到相应红霉素耐药基因。

图1 部分乳杆菌菌株质粒mefAmefA/E及msrCmsrC基因扩增结果Fig.1 PCR of mefA/E and msrC gene from plasmids in partial selected Lactobacillus strains
2.2M1与受体菌的耐药基因体外转移
经鉴定,L. delbrueckii M1号菌株质粒携带msrC基因,以M1为供体与S. aureus ATCC 29213杂交的3 次重复实验中,只有1 次实验一组平行的10-4稀释液于48 h后有一个可见菌落生长,直径约1 mm。其余样品及对照组均无菌落生长。M1与E. faecalis ATCC 29212的接合子在72 h后出现可见菌落,平均转移频率为2.2×10-2。敏感受体菌ATCC 29212和ATCC 29213在培养18~24 h后菌落可见。
2.3接合子的鉴定
通过PCR检测,接合子29212C和29213C的质粒DNA中扩增到msrC基因,而从阴性对照ATCC 29212和ATCC 29213的质粒DNA未扩增到阳性条带(图2),表明msrC基因通过滤膜杂交成功转移至受体菌ATCC 29213和ATCC 29212。
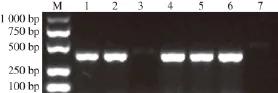
图2 接合子mmssrrCC基因的PCRR鉴定结果Fig.2 msrC gene in the exconjugants identified by PCR
2.4接合子MIC的测定结果
29212C和29213C对红霉素的MIC值为8 μg/mL,高于非接合子的正常MIC值(ATCC 29213为0.25~1 μg/mL,ATCC 29212为1~4 μg/mL),并达到CLSI提出的折点(8 μg/mL),说明ATCC 29212和ATCC 29213通过滤膜杂交,从M1获得红霉素耐药基因msrC,并产生了对红霉素的耐药性。
2.5体内基因转移
在SPF裸鼠体内进行的M1对E. faecalis ATCC 29212的耐药基因转移中,实验组未检出ATCC 29212接合子。对照组及空白组均无菌落生长。
3 讨 论
本研究在L. plantarum、L. acidophilus和L. delbrueckii中检测到msrC基因。Portillo[20]和Werner[21]等发现部分对红霉素敏感的E. faecalis菌株也携带msrC基因,而Singh等[22]却发现msrC基因的突变会导致E. faecalis菌株对红霉素的MIC明显降低。因此,msrC基因是否引起红霉素耐药,仍存争议。本研究中,敏感受体菌获得msrC基因后均产生了红霉素耐药性,表明msrC对红霉素耐药性确有贡献。受试乳杆菌中未检测到已知的万古霉素及氟喹诺酮类药物耐药基因,与其他研究结果相同[9-10],其耐药机制需进一步探讨。
可转移的抗生素耐药基因大都位于质粒和整合接合因子(integrative and conjugative elements,ICEs),而质粒和ICEs主要通过接合的方式发生HGT[23],因此目前耐药基因体外转移实验通常采用滤膜杂交法[3,7,24]。不同的供受体菌株的体外基因转移情况不同,四环素耐药基因能从屎肠球菌(Enterococcus faecium)水平转移至乳酸乳球菌(Lactococcus lactis)[25],从乳杆菌转移至L. lactis和E. faecalis,但从乳杆菌到S. aureus的体外转移却未能实现[24]。本研究中乳杆菌对S. aureus的红霉素耐药基因体外转移获得成功,但仅有1 个接合子生长,转移频率为1.1×10-9,与乳杆菌在红霉素选择压下产生获得性耐药的突变频率(<10-11~10-5)相当[26]。M1与E. faecalis ATCC 29212的滤膜杂交实验中,平均转移频率为2.2×10-2。Gevers等[24]得到乳杆菌四环素耐药基因tetM对E. faecalis的体外转移频率为10-4~10-6,而L. plantarum携带的红霉素耐药基因ermB对E. faecalis的体外转移频率为5.7×10-8[7]。供受体菌株的种类和接合子培养时间可能会对转移频率有影响。
值得注意的是,S. aureus接合子和E. faecalis接合子出现可见菌落的时间比敏感菌滞后约24~48 h,表明受体菌在获得外源红霉素耐药基因msrC后,付出了生长速率降低的适应性代价。若其在肠道内与敏感菌共存,当抗生素选择压降低或消失时,易在与敏感菌的竞争中被淘汰[27]。
已有关于乳杆菌耐药基因体内转移的研究结果不一,且乳杆菌和受体菌的灌喂量与我国成年人日常摄食的习惯不符[7-8]。本研究根据自然条件下我国成年人每天可能摄食乳杆菌的最大量,确定供体菌M1及受体菌E. faecalis ATCC 29212对裸鼠的灌喂量比例为1∶10,未检出接合子,而实际每日的乳杆菌摄入量还可能低于此剂量。在本研究基因体内转移的条件下,乳杆菌携带的可转移红霉素耐药基因msrC未转移至E. faecalis,据此可推断我国健康成年人通过日常摄入乳杆菌而将其携带的红霉素耐药基因转移给肠道致病菌和机会致病菌的风险很低。因此,本研究结果表明按照中国人食用乳杆菌的习惯,发酵食品中添加的乳杆菌即便携带耐药基因,在肠道内水平转移至肠道致病菌及机会致病菌的风险也很小,且获得外源耐药基因的菌株生长能力降低,竞争力减弱,易被淘汰。因此发酵食品中乳杆菌携带的耐药基因可能导致的食品安全风险较低。
[1]周宁, 张建新, 樊明涛, 等. 酸奶中保加利亚乳杆菌药物敏感性分析[J].食品科学, 2012, 33(21): 202-207.
[2]EFSA Panel on Additives and Products or Substances used in Animal Feed (FEEDAP). Guidance on the assessment of bacterial susceptibility to antimicrobials of human and veterinary importance[J]. EFSA Journal, 2012, 10(6): 2740-2749.
[3]NAWAZ M, WANG Juan, ZHOU Aiping, et al. Characterization and transfer of antibiotic resistance in lactic acid bacteria from fermented food products[J]. Current Microbiology, 2011, 62(3): 1081-1089.
[4]van REENEN C A, DICKS L M T. Horizontal gene transfer amongst probiotic lactic acid bacteria and other intestinal microbiota: what are the possibilities? A review[J]. Archives of Microbiology, 2011,193(3): 157-168.
[5]GAD G F M, ABDEL-HAMID A M, FARAG Z S H. Antibiotic resistance in lactic acid bacteria isolated from some pharmaceutical and dairy products[J]. Brazilian Journal of Microbiology, 2014, 45(1):25-33.
[6]SOMMER M O, DANTAS G, CHURCH G M. Functional characterization of the antibiotic resistance reservoir in the human microflora[J]. Science, 2009, 325: 1128-1131.
[7]FELD L, SCHJORRING S, HAMMER K, et al. Selective pressure affects transfer and establishment of a Lactobacillus plantarum resistance plasmid in the gastrointestinal environment[J]. Journal of Antimicrobial Chemotherapy, 2008, 61(4): 845-852.
[8]EGERVARN M, LINDMARK H, OLSSON J, et al. Transferability of a tetracycline resistance gene from probiotic Lactobacillus reuteri to bacteria in the gastrointestinal tract of humans[J]. Antonie Van Leeuwenhoek, 2010, 97(2): 189-200.
[9]CASADO MU.OZ MAR.A del C, BENOMAR N, LERMA L L, et al. Antibiotic resistance of Lactobacillus pentosus and Leuconostoc pseudomesenteroides isolated froSm naturally-fermented Alore.a table olives throughout fermentation process[J]. International Journal of Food Microbiology, 2014, 172(4): 110-118.
[10] TOOMEY N, BOLTON D, FANNING S. Characterisation and transferability of antibiotic resistance genes from lactic acid bacteria isolated from Irish pork and beef abattoirs[J]. Research in Microbiology, 2010, 161(2): 127-135.
[11] THUMU S C, HALAMI P M. Presence of erythromycin and tetracycline resistance genes in lactic acid bacteria from fermented foods of Indian origin[J]. Antonie Van Leeuwenhoek, 2012, 102(4):541-551.
[12] CHEN Xiang, ZHANG Weiqiu, PAN Weijuan, et al. Prevalence of qnr, aac(6')-Ib-cr, qepA, and oqxAB in Escherichia coli isolates from humans, animals, and the environment[J]. Antimicrobial Agents and Chemotherapy, 2012, 56(6): 3423-3427.
[13] ARSENE S, LECLERCQ R. Role of a qnr-like gene in the intrinsic resistance of Enterococcus faecalis to fluoroquinolones[J]. Antimicrobial Agents and Chemotherapy, 2007, 51(9): 3254-3258.
[14] Clinical Laboratory Standards Institute. Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically[M]. M07-A9, 9th ed. Wayne, PA: Clinical and Laboratory Standards Institute, 2012: 13-16.
[15] YU Jie, GAO Wa, QING Manjun, et al. Identification and characterization of lactic acid bacteria isolated from traditional pickles in Sichuan, China[J]. Journal of General and Applied Microbiology,2012, 58(3): 163-172.
[16] 罗珍兰, 谢继志. 酸乳制品中乳酸菌的分离培养与计数[J]. 食品科学, 1992, 13(11): 22-24.
[17] 石月锋, 杨海燕. 凝固型酸奶在储存过程中的特性研究[J]. 新疆农业大学学报, 2009, 32(5): 72-74.
[18] 张荣欣, 薛长勇, 王伟琴, 等. 988名汉族成年人人体测量调查研究[J].营养学报, 2000, 22(2): 179-183.
[19] 施新猷. 医用实验动物学[M]. 西安: 陕西科学技术出版社, 1989: 25.
[20] PORTILLO A, RUIZ-LARREA F, ZARAZAGA M, et al. Macrolide resistance genes in Enterococcus spp.[J]. Antimicrobial Agents and Chemotherapy, 2000, 44(4): 967-971.
[21] WERNER G, HILDEBRANDT B, WITTE W, et al. The newly described msrC gene is not equally distributed among all isolates of Enterococcus faecium[J]. Antimicrobial Agents and Chemotherapy,2001, 45(12): 3672-3673.
[22] SINGH K V, MALATHUM K, MURRAY B E. Disruption of an Enterococcus faecium species-specific gene, a homologue of acquired macrolide resistance genes of staphylococci, is associated with an increase in macrolide susceptibility[J]. Antimicrobial Agents and Chemotherapy, 2001, 45(1): 263-266.
[23] WOZNIAK R A F, WALDOR M K. Integrative and conjugative elements: mosaic mobile genetic elements enabling dynamic lateral gene flow[J]. Nature Reviews Microbiology, 2010, 8(8): 552-563.
[24] GEVERS D, HUYS G, SWINGS J. In vitro conjugal transfer of tetracycline resistance from Lactobacillus isolates to other Gram-positive bacteria[J]. FEMS Microbiology Letters, 2003, 225(1): 125-130.
[25] 杨埔, 孔文涛, 孙芝兰, 等. 食源性乳酸菌安全性的评价[J]. 食品科学, 2014, 35(19): 169-173. doi: 10.7506/spkx1002-6630-201419035.
[26] DRAGO L, MATTINA R, NICOLA L, et al. Macrolide resistance and in vitro selection of resistance to antibiotics in Lactobacillus isolates[J]. Journal of Microbiology, 2011, 49(4): 651-656.
[27] ANDERSSON D I, HUGHES D. Antibiotic resistance and its cost:is it possible to reverse resistance?[J]. Nature Reviews Microbiology,2010, 8(4): 260-271.
Antimicrobial Resistant Gene Transfer of Foodborne Lactobacilli
MA Qinqin1,2,FU Yu1, SUN Qun1,*
(1. Key Laboratory of Bio-resource and Eco-environment, Ministry of Education, College of Life Sciences, Sichuan University,Chengdu610064, China; 2. College of Life Sciences, Sichuan Normal University, Chengdu610101, China)
To assess the risk of antimicrobial resistant gene transfer from foodborne Lactobacillus strains to human pathogens or opportunistic pathogens, we performed the detection of the resistant genes of erythromycin, vancomycin and fluoroquinolones in resistant Lactobacillus strains. Erythromycin resistant gene msrC was shown in the plasmids of Lactobacillus plantarum, Lactobacillus acidophilus and Lactobacillus delbrueckii, while negative results were obtained for vancomycin and fluoroquinol ones. Filter mating was carried out at a donor/recipient ratio of 10:1 with Staphylococcus aureus and E. faecalis as the recipients and L. delbrueckii the donors, respectively. In L. delbrueckii and S. aureus mating pair, only one exconjugant was obtained, and the average gene transfer frequency of L. delbrueckii to E. faecalis was 2.2 × 10-2, indicating that the mobile resistant gene from lactobacilli could disseminate to pathogens. The growth rate of exconjugants was lower than the control, so exconjugants might be washed out more easily than their susceptible counterpart when antimicrobial selective pressure was absent. The transfer of msrC from L. delbrueckii to E. faecalis was not observed in gnotobiotic rats. Accordingly, lactobacilli widely used in foods now may not increase the antimicrobial resistance in pathogens, thus threatening public health severely.
antimicrobial resistance; Lactobacillus; gene transfer; exconjugant; food safety
Q939.99
A
1002-6630(2015)23-0167-05
10.7506/spkx1002-6630-201523031
2015-01-26
科技部国际科技合作专项(2011DFR31220;2015DFR31060);四川省科技支撑计划项目(14ZC1482);四川师范大学校级科研项目(10QNL02)
马沁沁(1978—),女,讲师,博士研究生,研究方向为应用微生物。E-mail:qqma@sicnu.edu.cn
孙群(1967—),女,教授,博士,研究方向为微生物学。E-mail:qunsun@scu.edu.cn

