一株拮抗链霉菌的鉴定及其多相分类特征
焦林培金 黄运红 李素珍 龙中儿
(江西师范大学生命科学学院,南昌 330022)
一株拮抗链霉菌的鉴定及其多相分类特征
焦林培金 黄运红 李素珍 龙中儿
(江西师范大学生命科学学院,南昌 330022)
通过16S rRNA基因序列分析一株拮抗放线菌JXNU03的系统发育关系,通过生理生化试验分析其生理生化特征,利用菌丝生长率法和杯碟法测定其拮抗活性。结果发现,放线菌JXNU03在高氏一号培养基上培养时,基内菌丝发达,分支多且不断裂,气生菌丝发育良好,孢子丝直形或螺旋状,孢子表面带刺,细胞水解液中检测到葡萄糖,未检测出特征性糖,糖类型属于C型,细胞壁氨基酸中含有L,L-DAP,属于细胞壁Ⅰ型,16S rRNA基因序列与灭癌素链霉菌的同源性高达98%,其发酵液对革兰氏阳性细菌、革兰氏阴性细菌、酵母菌和霉菌等都具有较强的拮抗作用。因此放线菌JXNU03被鉴定为灭癌素链霉菌,记为灭癌素链霉菌JXNU03(Streptomyes gancidicus JXNU03),是一株对细菌、酵母菌和霉菌均有较强拮抗活性的链霉菌,具有潜在开发价值。
灭癌素链霉菌;系统发育;多相分类
抗生素的发现及开发利用,对保障人类健康、延长人类寿命、防治动、植物病害等方面都做出了巨大的贡献,并对生物学等基础学科的发展产生重要的影响。然而,随着抗生素的广泛应用,耐药性问题也随之产生[1]。研究、开发具有全新结构或全新作用机制的新抗生素是克服细菌耐药性的重要途径之一,并受到各国政府及产、学、研各界关注,已成为研究的热点[2-4]。
国内外有关新抗生素的研发主要集中在三个方面,一是对已知抗生素的结构进行改造,通过对已知抗生素进行化学修饰,采用全合成的手段获得已知抗生素的结构类似物,甚至全新结构的抗生素,以克服细菌的耐药现象[5-7]。二是抗生素筛选模型的创新,构建基于抗生素作用机理及特定靶标,特别是作用于全新靶标的高通量筛选模型[8-13]。三是基于新的物种孕育新的基因,新的基因指导新型抗生素的生物合成,从各种特定环境中筛选新抗生素菌,不断拓展产抗生素的微生物来源[14-15]。
本实验室从江西龙虎山风景区的土壤样品中筛选得到一株对细菌、酵母菌、霉菌均有拮抗活性的放线菌JXNU03,研究该菌的多相分类特征,旨在为抗生素产生菌的开发和利用奠定基础。
1 材料与方法
1.1 材料
1.1.1 菌种
1.1.1.1 试验菌株 放线菌JXNU03,本实验室自行从龙虎山风景区土壤样品中分离获得,保藏于4℃的高氏一号琼脂斜面,中国典型培养物保藏中心保藏编号:CCTCC NO:M2014011。
1.1.1.2 拮抗靶菌 金黄色葡萄球菌(Staphylococcus aureus)、白葡萄球菌(Staphylococcus cremoris)、枯草芽孢杆菌(Bacillus subtilis)、苏云金杆菌(Bacillus thuringiensis)、大肠杆菌(Escherichia coli)、铜绿假单胞菌(Pseudomonas aeruginosa)、福氏志贺菌(Shigella flexneri)、藤黄八叠球菌(Sarcina lutea)、伤寒沙门菌(Salmonella typhi)、酿酒酵母(Saccharomy-ces cerevisiae)、白色念珠菌(Candida albicans)、红酵母(Rhodotorula glutinis)、桔青霉菌(Penicillium citrinum)、鲜绿青霉(Penicillium verrucosum)、黑曲霉(Aspergillus niger)、米根霉(Rhizopus oryzae)、高大毛霉(Mucor mucedo)、芝麻枯萎病菌(Fusarium oxysporum)、水稻纹枯病菌(Thanatephorus cucumeris)、小麦赤霉(Fusarium graminearumt)、辣椒根腐病菌(Fusarium vasinfectum)、油菜菌核菌(Sclerotinia libertiana)、小麦青枯菌(Ralstonia solanacearum)、疣孢青霉(Penicillium verrucosum),以上菌种均由江西师范大学微生物学实验室保藏于4℃的培养基斜面。
1.1.2 试剂 试验涉及的PCR材料购于大连宝生物有限公司,其他生化试剂均购于生工生物工程(上海)股份有限公司,其他化学试剂均为国产分析纯。
1.1.3 培养基 种子培养基(g/L):蔗糖22.5,黄豆饼粉12.5,NaCl 1,K2HPO40.2,Na2SO40.1,FeSO4· 7H2O 0.01,CaCO32,pH为7.2;发酵培养基(g/L):蔗糖45,黄豆饼粉25,NaCl 1,K2HPO40.2,Na2SO40.1,FeSO4·7H2O 0.01,CaCO33,pH为7.2;活性检测上层培养基(真菌为靶菌)(g/L):马铃薯200,葡萄糖20,琼脂20,pH自然;活性检测上层培养基(细菌为靶菌)(g/L):牛肉膏3,蛋白胨10,葡萄糖5,NaCl 5,琼脂15-20,pH7.0-7.2;活性检测下层培养基:20 g琼脂,加热溶解于水后以蒸馏水定容至1 000 mL,121℃灭菌20 min。
1.2 方法
1.2.1 形态特征观察 光学显微镜观察:采用插片法[16]观察菌株形态特征。即将菌株接种于高氏一号平板上,将灭菌的盖玻片斜插入平板,并于28℃培养3、5、7、10、14和21 d,分别取出盖玻片,用美蓝简单染色,在油镜下观察基内菌丝、气生菌丝、孢子丝形态特征。扫描电镜观察:将菌株接种到ISP2平板上插片培养14 d,取出插片,用HITACHI E-1045磁控溅射器喷金,然后用HITACHI S-3400N扫描电子显微镜扫描观察菌株形态特征。
1.2.2 细胞化学组分分析 分析细胞壁氨基酸组分及全细胞水解物糖型,具体按文献[18]的方法进行。
1.2.3 系统发育分析 利用优化SDS法提取菌体总DNA[17],1%琼脂糖凝胶电泳检测基因组DNA,采用细菌16S rRNA基因扩增通用引物正向Pf:5'-AGAGTTTGATCCTGGCTCAG-3';反向Pr:5'-GGCTACCTTGTTACGACTT-3'进行PCR扩增,PCR反应体系为25.0 μL,包括:5 U/μL TaKaRa LA Taq 0.5 μL,2×GC Buffer I 25 μL,2.5 mmol/L dNTP Mixt-ure各8 μL,10 μmol/L F27 2 μL,10 μmol/L R1492 2 μL,DNA模板1 μL,ddH2O 11.5 μL。PCR扩增条件:94℃ 1 min;94℃ 30 s,57℃ 30 s,72℃ 1.5 min,共30个循环;72℃ 5 min。将PCR产物送生工生物工程(上海)股份有限公司测序。根据测序结果,在NCBI网站上进行序列比对,使用MEGA5.0软件进行系统发育分析,用Neighbor-Joining的方法构建系统进化树,Bootstrap值参照。
1.2.4 培养特征分析 将JXNU03菌株分别接入ISP2-ISP5及高氏一号培养基中,分别于28℃恒温箱培养7、14及21 d后取出观察,记录气生菌丝、基内菌丝及可溶性色素(培养基颜色),具体参见文献[18,19]方法进行。
1.2.5 生理生化特征分析 将JXNU03菌株分别接入酪氨酸培养基、明胶液化培养基、淀粉水解培养基、Tresner琼脂培养基、硝酸盐还原培养液、滤纸条培养液、牛奶凝固与胨化培养基及不同碳源基础培养基中,检测其生理生化特征,具体参见文献[18,19]方法进行。
1.2.6 拮抗活性测定 用无菌打孔器在长有放线菌JXNU03单菌落的高氏一号营养琼脂平板上打4个菌饼(直径为5 mm)接种于发酵培养基中。200 r/min、28℃条件下发酵培养5 d。发酵液于8 000 r/min条件下离心10 min,取上清,重复离心一次后,上清液于4℃保存,杯碟法测定其抗细菌、酵母菌活性[20,21],菌丝生长速率抑制法测定其抗霉菌活性[22],抑制率的计算方法如下[23]:

2 结果
2.1 形态特征
放线菌JXNU03在高氏一号培养基中生长良好,菌落由最初的白色到4 d后逐渐变成灰色或灰白色,菌落之间差异不大,表面呈粉绒状突起,无明显褶皱;基内菌丝发达,分支多且不断裂,气生菌丝发育良好,孢子丝直形或螺旋状,孢子表面带刺,其形态见图1。根据其形态特征可初步确定放线菌JXNU03属于链霉菌属放线菌。

图1 放线菌JXNU03的孢子形态
2.2 细胞化学组分
对放线菌JXNU03全细胞糖型和氨基酸组分分析,结果表明,细胞水解液中检测到葡萄糖,未检测出特征性糖,糖类型属于C型,细胞壁氨基酸中含有L,L-DAP,属于细胞壁Ⅰ型。这一细胞化学成分特点进一步证实了放线菌JXNU03属于链霉菌属放线菌的判断。
2.3 系统发育分析
通过对菌株JXNU03的16S rRNA基因序列的扩增和测序,获得约1 434 bp的该基因序列(提交GenBank,获得登录号为KJ002642),该序列与GenBank中的序列对比,发现与其同源性较高的相关菌株均为链霉菌属(Streptomyces)放线菌。选取其中相似度较高的几株,利用MEGA5.0软件,采用N-J法(重复度1 000)构建系统发育树(图2)。系统发育分析结果表明,菌株JXNU03与灭癌素链霉菌NBRC 15412的16S rRNA基因序列同源性最高,达到98%。由此可将放线菌JXNU03鉴定为灭癌素链霉菌,记为灭癌素链霉菌JXNU03(Streptomyes gancidicus JXNU03)。
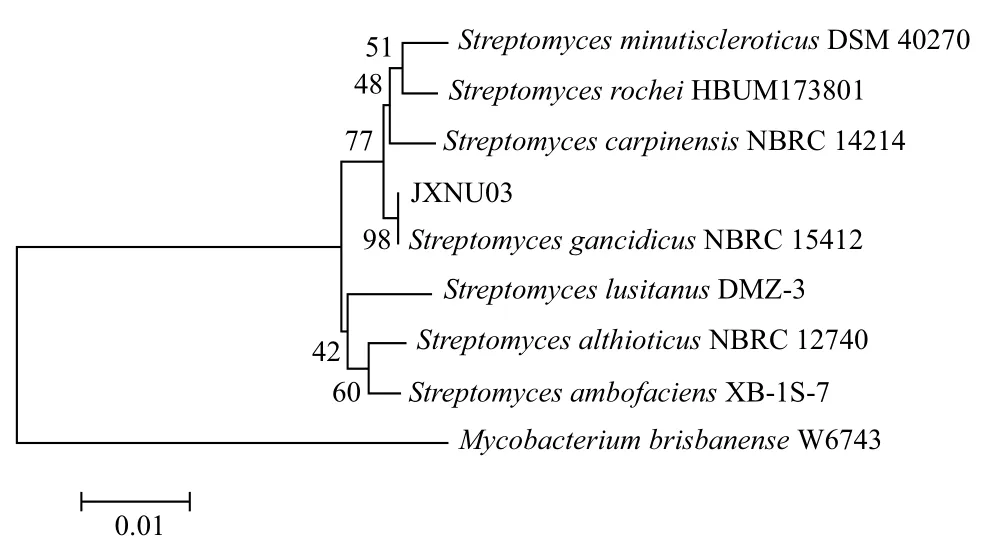
图2 基于16S rRNA基因序列同源性的放线菌JXNU03 系统发育树
2.4 培养特征
菌株JXNU03在5种培养基上,一定时间后观察其气生菌丝、基内菌丝的生长及颜色变化情况,详细培养特征见表1,颜色鉴定参考文献[19,24]。
2.5 生理生化特征
菌株JXNU03能利用葡萄糖、阿拉伯糖、木糖、果糖、鼠李糖、甘露醇、肌醇和蔗糖,但不能利用棉子糖;其过氧化氢酶、淀粉酶、明胶液化、纤维素水解、淀粉水解、牛奶凝固和胨化试验均呈阳性,氧化酶、硝酸还原、H2S产生及黑色素产生试验均呈阴性(表2)。

表1 放线株JXNU03的培养特征

表2 放线菌JXNU03 的生理生化特征
按照阮继生等[25]的放线菌分类教程中描述的灭癌素链霉菌不能利用蔗糖,硝酸盐还原呈阳性的特征,而放线菌JXNU03能利用蔗糖作为唯一碳源,硝酸盐还原为阴性。本研究报道的灭癌素链霉菌JXNU03与文献报道的灭癌素链霉菌(S. gancidicus)在生理生化特征方面也存在差异,如郑小伟等[26]获得一株抑藻活性的灭癌素链霉菌(S. gancidicus),其硝酸盐还原为阳性,但能利用蔗糖为唯一碳源。因此,将链霉菌JXNU03鉴定为灭癌素链霉菌,记为灭癌素链霉菌JXNU03(Streptomyces gancidicus JXNU03)。

表3 放线菌JXNU03的抗菌谱
2.6 拮抗活性及抗菌谱
放线菌JXNU03发酵液具有广谱的抗菌活性,抗菌结果(表3)表明,放线菌JXNU03对革兰氏阳性的金黄色葡萄球菌、白葡萄球菌、枯草芽孢杆菌、苏云金芽胞杆菌和革兰氏阴性的大肠埃希氏菌、铜绿假单胞菌、福氏志贺菌、伤寒沙门氏菌等细菌均具有抗菌活性,同时对红酵母、白色念珠菌、米根霉、鲜绿青霉、小麦青枯菌、小麦赤霉、油菜菌核菌、芝麻枯萎病菌和辣椒根腐病菌等真菌也表现出抗菌活性,而对酿酒酵母、黑曲霉、高大毛霉和疣孢青霉不具抗菌作用。放线菌JXNU03发酵液对金黄色葡萄球菌的抑菌圈直径为44 mm(图3),对米根霉的抑菌率达到57.68%(图4),均表现出较强的抗菌活性。

图3 抗金黄色葡萄球菌结果
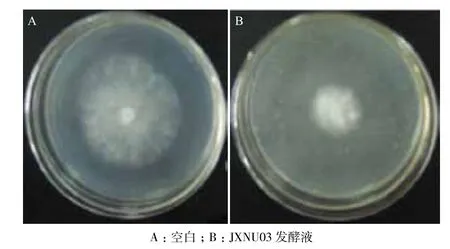
图4 抗米根霉结果
3 讨论
放线菌自发现至今已有百余年的历史,其分类学也已从经典分类或表观分类发展到化学分类,又由化学分类发展到分子分类,以及当今生物系统进化的多相分类。本研究按照阮继生等[25]的放线菌分类教程,根据放线菌的形态、细胞化学成分(细胞壁I 型,细胞水解液糖C型)确定放线菌JXNU03属于链霉菌属,然后依据其16S rRNA基因序列进化关系进一步确定其为链霉菌属放线菌。再由于放线菌JXNU03与灭癌素链霉菌NBRC 15412的16S rRNA基因序列具有最大的序列同源性(98%),二者形态特征相近,菌落颜色及个体形态在不同培养基上差异不大,表面均呈粉绒状凸起,孢子丝直至螺旋状,孢子表面带刺。在生理生化特性方面,放线菌JXNU03除了可以利用蔗糖作为唯一碳源,硝酸盐还原阴性不同之外,其它能利用葡萄糖、阿拉伯糖、木糖、果糖、鼠李糖、甘露醇和肌醇作为唯一碳源且不能利用棉子糖等方面相同。因此,将链霉菌JXNU03鉴定为灭癌素链霉菌,记为灭癌素链霉菌JXNU03(Streptomyces gancidicus JXNU03)。
据文献报道,灭癌素链霉菌(S. gancidicus)的代谢产物具有多种生物活性,如郑小伟等[26]筛选的一株灭癌素链霉菌(S. gancidicus)具有高效抑藻活性,陈杰等[27]筛选的一株灭癌素链霉菌则对马铃薯病原真菌具有较强抑菌作用,而本研究的灭癌素链霉菌JXNU03对G+细菌、G-细菌、酵母菌和丝状真菌都表现出较强的拮抗作用,预示其可能产生多种抗生素,是一种重要的抗生素产生菌,具有重要的开发前景。
4 结论
本实验室从江西龙虎山风景区的土壤样品中筛选获得一株对革兰氏阳性细菌、革兰氏阴性细菌、酵母菌和霉菌等都具有拮抗作用的链霉菌。该菌株在高氏一号培养基上培养时,基内菌丝发达,分支多且不断裂,气生菌丝发育良好,孢子丝直形或螺旋状,孢子表面带刺,细胞水解液中检测到葡萄糖,未检测出特征性糖,糖类型属于C型,细胞壁氨基酸中含有L,L-DAP,属于细胞壁Ⅰ型,16S rRNA基因序列与灭癌素链霉菌NBRC 15412的同源性高达98%。将放线菌JXNU03鉴定为灭癌素链霉菌,记为灭癌素链霉菌JXNU03(Streptomyces gancidicus JXNU03)。
[1] Byarugaba DK. Antimicrobial resistance in developing countries and responsible risk factors[J]. International Journal of Antimicrobial Agents, 2004, 24(2):105-110.
[2] Davies J. How to discover new antibiotics:harvesting the parvome[J]. Current Opinion in Chemical Biology, 2011, 15(1):5-10.
[3] Alanis AJ. Resistance to antibiotics:Are we in the post-antibiotic era?[J]. Archives of Medical Research, 2005, 36(6):697-705.
[4] Tan YT, Tillett DJ, McKay IA. Molecular strategies for overcoming antibiotic resistance in bacteria[J]. Molecular Medicine Today,2000, 6(8):309-314.
[5] Yan RB, Yuan M, Wu Y, et al. Rational design and synthesis of potent aminoglycoside antibiotics against resistant bacterial strains[J]. Bioorganic & Medicinal Chemistry, 2011, 19(1):30-40.
[6] Huigens-III RW, Reyes S, Reed CS, et al. The chemical synthesis and antibiotic activity of a diverse library of 2-aminobenzimidazole small molecules against MRSA and multidrug-resistant A. baumannii[J]. Bioorganic & Medicinal Chemistry, 2010, 18(2):663-674.
[7]Cheng K, Zheng QZ, Hou J, et al. Synthesis, molecular modeling and biological evaluation of PSB as targeted antibiotics[J]. Bioorganic & Medicinal Chemistry, 2010, 18(7):2447-2455.
[8] 谢练武, 李翔, 欧阳永长, 等.抗生素作用新靶点的发掘策略[J].中国抗生素杂志, 2007, 32(11):641-646, 668.
[9] Granieri L, Miller OJ, Griffiths AD, Merten CA. A competition-based assay for the screening of species-specific antibiotics[J]. Journal of Antimicrobial Chemotherapy, 2009, 64(1):62-68.
[10]Singh P, Panda D. FtsZ inhibition:a promising approach for antistaphylococcal therapy[J]. Drug News & Perspectives, 2010, 23(5):295-304.
[11]Kapoor S, Panda D. Targeting FtsZ for antibacterial therapy:a promising avenue[J]. Expert Opinion on Therapeutic Targets,2009, 13(9):1037-1051.
[12]Heath RJ, Rock CO. Fatty acid biosynthesis as a target for novel antibacterials[J]. Current Opinion in Investigational Drugs,2004, 5(2):146-153.
[13] Jain R, Chen D, White RJ, et al. Bacterial peptide Deformylase inhibitors:a new class of antibacterial agents[J]. Current Medicinal Chemistry, 2005, 12(14):1607-1621.
[14]Fiedler HP, Bruntner C, Bull AT, et al. Marine actinomycetes as a source of novel secondary metabolities[J]. Antonie van Leeuwenhock, 2005, 87(1):37-42.
[15]Peric-Concha N, Long PF. Mining the microbial metabolome:a new frontier for natural product lead discovery[J]. Drug Discovery Today, 2003, 8(23):1078-1084.
[16]周德庆.微生物实验教程[M].北京:高等教育出版社,2005:58-61.
[17]刘炳辉, 曹远银, 闫建芳, 等. 6 种链霉菌基因组DNA 提取方法比较[J].河南农业科学, 2008(10):86-89.
[18]阮继生, 黄英.放线菌快速鉴定与系统分类[M].北京:科学出版社, 2011:70-71.
[19]Shirling EB, Gottlieb D. Methods for characterization of Streptomyces[J]. International Journal of Systematic Bacteriology, 1966, 16(3):313-340.
[20]郝利民, 路福平.杯碟法检测抗生物质效价的几点讨论[J].四川食品与发酵, 1997, 4:1-3.
[21]沈萍, 范秀容, 李广武.微生物学实验[M].北京:高等教育出版社, 1995:173-177.
[22]田小卫, 龙建友, 白红进, 等.一株放线菌次生代谢产物抗菌活性的初步研究[J].植物保护, 2004, 30(2):51-54.
[23]Li Q, Jiang Y, Ning P, et al. Suppression of Magnaporthe oryzae by culture filtrates of Streptomyces globisporus JK-1[J]. Biological Control, 2011, 58(2):139-148.
[24] Kelly KL. Inter-society color council-national bureau of standards color name charts illustrated with centroid colors[M]. Washington,DC:US Government Printing Office, 1964.
[25]阮继生, 黄英.放线菌快速鉴定与系统分类[M].北京:科学出版社, 2011:172-188.
[26]郑小伟, 黄丽萍, 张帮周, 等.一株高效抑藻放线菌的分离筛选及鉴定[J].厦门大学学报, 2012, 51(5):923-928.
[27]陈杰, 郭天文, 谭雪莲, 等.马铃薯连作地健康株与病株根区土壤微生态特性比较[J].作物学报, 2013, 39(11):2055-2064.
(责任编辑 马鑫)
Identification and Polyphasic Taxonomy Character of a Strain of Streptomycete
Jiao Linpeijin Huang Yunhong Li Suzhen Long Zhonger
(College of Life Science,Jiangxi Normal University,Nanchang 330022)
To identify a strain of actinomycetes JXNU03 through the sequences of 16S rRNA gene. The physiological characteristics were acquired through physiological and biochemical experiments. The antagonistic activity was determined by plate assay and adopting mycelium growth rate method respectively. When the actinomycetes JXNU03 was cultured on Gause 1 medium, the vegetative hyphae were well developed and abundant without fracture, and the aerial hyphae were grown well with straight or spiral spore hypha. There were some thorns on the surface of spores. The glucose was detected in the whole cell hydrolysis, while not other dominant diagnostic sugars, and the L, L-DAP was detected in cell wall. The phylogenetic analysis showed that the level of 16S rRNA gene sequence similarity between the actinomycete JXNU03 and Streptomyes gancidicus NBRC 15412. The strain JXNU03 demonstrated a board spectrum of antagonistic activity against. The Gram-positive bacteria, the Gram-negative bacteria, yeasts and molds. The actinomycetes JXNU03 was identified as Streptomyes gancidicus, and termed Streptomyes gancidicus JXNU03. As its antagonistic activity against bacteria, yeast and filamentous fungi. The actinomycetes JXNU03 possessed an important prospect for antibiotics development.
Streptomyes gancidicus;phylogenesis;polyphasic taxonomy
10.13560/j.cnki.biotech.bull.1985.2015.01.021
2014-05-27
国家自然科学基金项目(31160029,31360018),江西省教育厅科技计划项目(GJJ12181)
焦林培金,女,硕士研究生,研究方向:微生物学;E-mail:523213792@qq.com
龙中儿,男,博士,教授,研究方向:基础和应用微生物学;E-mail:longzhonger@163.com

