海洋真菌提取物损伤大肠杆菌DNA活性的筛选及活性菌株研究
鲍海燕谷朋娟张翼穆军冯妍赵辰燕
(1.大连交通大学环境与化学工程学院,大连 116028;2.广东海洋大学食品与科技学院 广东省水产品加工与安全重点实验室 水产品深加工广东普通高等学校重点实验室,湛江 524088;3.浙江海洋学院海洋科学与技术学院,舟山 316022)
海洋真菌提取物损伤大肠杆菌DNA活性的筛选及活性菌株研究
鲍海燕1谷朋娟1张翼2穆军3冯妍2赵辰燕1
(1.大连交通大学环境与化学工程学院,大连116028;2.广东海洋大学食品与科技学院广东省水产品加工与安全重点实验室水产品深加工广东普通高等学校重点实验室,湛江524088;3.浙江海洋学院海洋科学与技术学院,舟山316022)
从海洋微生物中筛选抗菌、抗肿瘤物质是海洋药物研究的热点之一,而筛选模型在研究中起着重要作用。建立了可用于抗菌及潜在抗肿瘤活性物质筛选的大肠杆菌差异性DNA修复试验模型,并运用该模型对海洋共附生真菌的提取物进行了筛选。结果显示,5株海洋真菌的提取物具有较强的选择性抑制DNA损伤修复基因缺陷型大肠杆菌的活性,可能具有DNA损伤作用。对这5种活性真菌进行了分子生物学鉴定,而且薄层色谱(TLC)和高效液相色谱(HPLC)-活性追踪显示这些菌株可产生较多样化的活性物质。
海洋微生物;大肠杆菌差异性DNA修复试验模型;鉴定
海洋微生物药物是海洋药物与生物技术重要的交叉领域,海洋真菌作为海洋微生物的重要组成类群,具有生物活性代谢产物丰富且相对易于培养的优点,从海洋真菌中筛选生物活性物质已成为海洋药物研究的热点,据不完全检索,仅天然产物数据库(Dictionary of Natural Products 2011)中收录的海洋真菌抗菌与抗肿瘤活性化合物就已达87个[1],新的报道仍不断涌现[2,3]。
众所周知,高效、灵敏、可靠的药物筛选模型对药物发现非常关键。就抗肿瘤药物筛选而言,最常用的细胞毒体外筛选模型也存在一些局限性,如对实验条件的要求和运行成本都比较高,不太适合大量粗提物样品的经济快速筛选。而从药物作用机理来看,很多抗肿瘤或抗菌药物的作用机制主要是作用于DNA,而多数对DNA 有损伤的物质虽有一定毒性但也有抗肿瘤或抗菌作用,其中毒副作用小的可望用做临床药物[4]。大肠杆菌(Escherichia coli)差异性DNA损伤修复试验(Differentiated DNA damage repair test,DDRT)是一种用于检测物质对DNA损伤能力的试验方法,它以一对大肠杆菌菌株(其中一个具有正常的DNA损伤修复能力,而另外一个存在相应的基因缺陷)经样品处理后生长受抑制程度的差异来判断样品是否具有损伤DNA的作用,该方法被用于环境毒物遗传毒性评价,也被用于抗菌与潜在抗肿瘤物质活性筛选,该方法用于筛选抗肿瘤药物,简单快速,与肿瘤细胞株模型的结果具有一定的相关性[5-7],还可兼顾抗菌活性,且适合于大量粗提物样品的快速经济筛选。本研究建立该方法并对本实验室保藏的分离自大连潮间带海洋生物的共附生真菌[8,9]发酵提取物进行活性筛选与相关研究,旨在为从海洋微生物中筛选抗菌抗肿瘤物质用于海洋药物研究提供参考。
1 材料与方法
1.1 材料
1.1.1 菌株 供筛选菌株:本实验室保藏的海洋真菌66株;指示菌株:来自耶鲁大学菌种库,包括野生型大肠杆菌和缺陷性大肠杆菌两种,编号分别为AB1157(+)、AB3027(-)。
1.1.2 培养基 海洋真菌培养基(1 L):蔗糖20 g、马铃薯汁500 mL、4%的人工海水500 mL、pH自然。如需固体培养基,则加入1.5%-2%的琼脂粉。营养琼脂培养基(1 L):蛋白胨10 g、牛肉膏3 g、NaCl 5 g、琼脂粉15 g、纯水1 000 mL、pH自然。均高压蒸汽灭菌。
1.2方法
1.2.1 指示菌悬液的制备 指示菌E. coli AB1157(+)和AB3027(-)的斜面经37℃两次活化培养后,在超净台中加入少量无菌生理盐水洗涤菌落摇匀,分别倒入无菌试管,并均稀释至0.5麦氏浊度(OD600= 0.136)。
1.2.2 大肠杆菌差异性DNA损伤修复模型的验证 采用平皿滤纸片法检测博莱霉素、丝裂霉素、阿霉素、5-氟尿嘧啶、氨苄青霉素、紫杉醇6种药物各自对这两株大肠杆菌指示菌的抑制差异性。
将100 μL指示菌[AB1157(+)或AB3027(-)]悬液均匀涂布在营养琼脂平板表面,静置待表面稍干,用镊子贴上滴加有5 μg和2 μg药物(溶剂为25 μL纯水或甲醇)并晾干的无菌滤纸片,每个指示菌种、每种药物、每个剂量做3个平行,以滴加25 L甲醇并晾干的滤纸片为阴性对照。每个平皿均匀贴7片滤纸片,背面做好标记。将平皿于4℃倒置扩散过夜后,取出于37℃倒置培养12-16 h,取出、测量记录抑菌圈直径,并用抑菌圈直径数据计算药物对这两株指示菌的抑菌圈直径比[AB3027(-)/AB1157(+)]以及该比值的平均值和标准差,并采用t检验分析其比值与1之间的差异显著性(n=9)。
1.2.3 海洋真菌的发酵与提取 将在固体培养基上培养3 d活化的待筛菌种,接种到每个含100 mL海洋真菌液体培养基的500 mL锥形瓶中,无菌透气膜封口后28℃静置培养20 d。培养结束后,加入少量乙酸乙酯过夜杀菌,抽滤分离菌丝体与发酵液。菌丝体用100 mL甲醇浸泡过夜后抽滤,得滤液;发酵液用1/2体积乙酸乙酯萃取3次,收集乙酸乙酯层;将菌丝体提取物与发酵液提取物合并后旋转蒸发仪45℃浓缩至干,用少量甲醇∶二氯甲烷(1∶1)多次溶解洗涤后转入样品瓶,定容至3 mL,冷藏备用。
1.2.4 活性物质的筛选
1.2.4.1 初筛 将每种样品一个滤纸片(滴加25 μL提取物)晾干后贴到涂有100 μL指示菌AB3027(-)的营养琼脂平皿上,使用模型验证相同的方法培养,并观察、测量抑菌圈直径。对于抑菌圈较明显(抑菌圈大于8 mm)的样品,使用同样的方法重复筛选1次(二次初筛)。
1.2.4.2 复筛 将初筛得到的样品用两种指示菌缺陷型AB3027(-)和野生型AB1157(+)共同实验,进行复筛。配制两种指示菌的新鲜菌悬液,每种指示菌、每个样品做3个平行。在培养箱中37℃培养12-16 h后取出,测量记录不同样品分别对两种指示菌抑菌圈的直径,并进行数据分析,选出具有强DNA损伤活性的样品。
1.2.5 强活性菌株的分子生物学鉴定及薄层层析指纹图谱
1.2.5.1 分子生物学鉴定 对复筛得到的菌株进行分子生物学鉴定,包括DNA提取、PCR扩增、电泳以及测序[8]。测序引物为真菌通用引物ITS1和ITS4,将测序得到的核糖体DNA内转录间隔区(ITS rDNA)全序列提交GenBank进行BLAST检索,与数据库中已有菌株的ITS rDNA序列进行相似性比较。并用Clustal W进行序列比对后,采用Mega 4.0软件构建系统发育进化邻接树[10]。
1.2.5.2 薄层层析(TLC) 用毛细管吸取活性样品各约5 μL点样于GF254硅胶薄层层析板,置层析缸中以二氯甲烷∶甲醇(V/V=20∶1)为展开剂展开后取出晾干,置于254 nm及365 nm的紫外灯下观察并拍摄图像,再喷茴香醛-硫酸显色剂后加热显色,用扫描仪记录图像。
1.2.6 强活性样品的高效液相色谱-活性追踪
1.2.6.1 液相分离与96孔板收集 活性样品经3 000 r/min、20 min离心后,分别取20 μL进行依利特P230型高效液相色谱分析,色谱柱为4.6×250 mm,5 μm粒径Hypersil填料的C18柱,流动相为甲醇-水系统(梯度洗脱),流速0.6 mL/min,将96孔板置于检测器后的流出口,从第3孔起每30 s一孔依次收集流分直至46.5 min(第96孔)。收集完毕后,每孔用甲醇补满,用多道移液枪移1/2到另一个空96孔板中,一一对应,即一个样品对应两个96孔板(分别用于培养两株大肠杆菌指示菌)做好标记。将96孔板置于超净台中,用无菌风吹至近干,再置于洁净的真空干燥箱中彻底干燥。
1.2.6.2 指示菌孵育与DNA损伤活性追踪 先将96孔板表面及边缘用酒精棉擦拭后,揭开盖子置于超净台紫外杀菌20 min。其后在每块96孔板的A行第1孔加180 μL培养基和20 μL的生理盐水(空白),第2孔加入180 μL的培养基和20 μL的菌悬液(对照),其余各孔均加入180 μL的培养基和20 μL的菌悬液,小心的水平晃动混匀。每个样品对应的两块96孔板中,一个加AB1157(+)的菌悬液,另一个加AB3027(-)的菌悬液。将96孔板于37℃培养16-18 h后,在各孔中加入25 μL的MTT显色剂(2 mg/mL),于37℃再培养30 min,拍照,并用酶标仪测540 nm波长下各孔光吸收值(A)。
1.2.6.3 数据处理与作图 使用Excel软件,根据公式[抑菌率(IR)= 100%×(A对照-A样品)/(A对照-A空白)]计算各孔抑菌率,然后计算每个样品每个时间点(ti)的对两种指示菌的抑菌率差值,即抑菌率差值=IRti,AB3027(-)-IRti,AB1157(+),作抑菌率差值-时间折线图,此即样品的选择性抗大肠杆菌[AB3029(-)与AB1157(+)]活性色谱图。通过活性色谱图与HPLC谱图的对照,可指认样品中的活性峰。
2 结果
2.1 筛选模型的验证
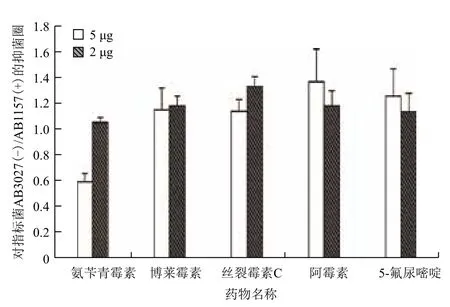
图1 五种抗菌或抗肿瘤药物对两种指示菌的抑菌圈直径之比
理论上讲,如果某药物对这一对指示菌存在无差别的抑制活性,则抑菌圈直径比值应等于1。如图1所示,博莱霉素、丝裂霉素C、阿霉素和5-氟尿嘧啶这4种抗肿瘤药物对这两株指示菌[AB3027(-)/AB1157(+)]的抑菌圈直径比值均大于1,且t值均大于t0.05(2.306),说明其对两种指示菌抑菌圈的比值与“1”之间都存在显著差异,其中以博莱霉素(2 μg)、丝裂霉素(5 μg、2 μg)和阿霉素(5 μg、2 μg)最为显著,其t值均大于甚至远大于t0.01(3.355)(如丝裂霉素2 μg对应的t值为12.38),说明它们都显示出了预期的DDRT差异活性(即对缺陷型菌株的抑制强于对野生型的抑制)。而氨苄青霉素在2 μg时t=3.75略大于t0.01,可认为与“1”之间具有显著差异,但差异不算太大,说明在该剂量下对两种指示菌活性差别较小;而在5 μg剂量下t=18.09,远大于t0.01,说明存在较显著差异,其具体机制有待研究。另外,筛选中也发现紫杉醇对两种指示菌均无抑制活性(即无抑菌圈)。
2.2 活性物质筛选结果
2.2.1 初筛结果 考虑到样品数量较多,先用指示菌AB3027(-)进行了初筛,从66株海洋真菌的提取物中发现了48株样品具有清晰的抑菌圈。在这些活性样品中有14个样品抑菌圈直径大于8 mm,如表1所示。
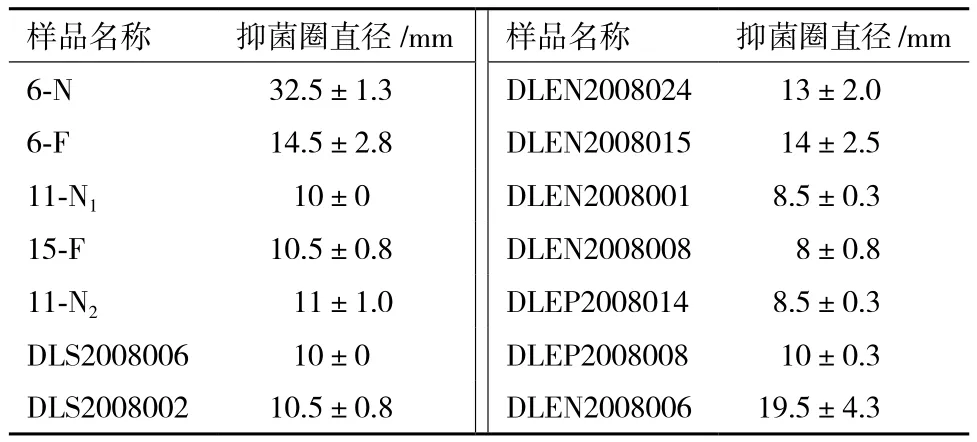
表1 纸片扩散法初筛中显示相对较强抑制E. coli AB3027(-)活性的样品
2.2.2 复筛的结果 为考察这些活性样品是否具有DNA损伤活性,从上述初筛结果中选取抑菌圈不小于10 mm的11个样品,进行了同时拮抗两株指示菌的复筛实验。结果(表2)表明,有5株真菌的提取物样品即6-N、6-F、DLEN2008006、15-F和DLEN2008024,表现出明显的选择性抑制缺陷型大肠杆菌的活性,可用于进一步的菌种鉴定及提取物成分研究。
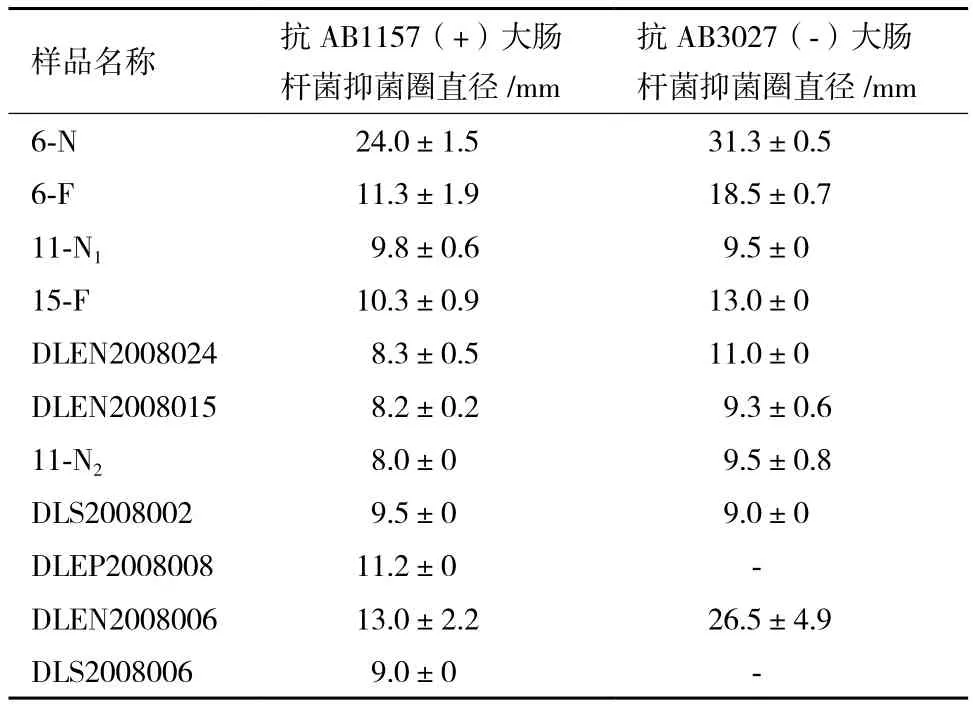
表2 同时使用E. coli AB1157(+)和AB3027(-)作为指示菌的纸片扩散法复筛结果
2.3 活性菌株的分子生物学鉴定及TLC指纹图谱

表3 五个活性菌株的测序和比对结果
通过真菌通用引物PCR扩增了该5株活性真菌的核糖体RNA基因内转录间隔区(ITS1-5.8S-ITS2 rDNA)序列,测得的序列用BLAST与GenBank中已知序列进行比对,显示它们与已知种属的相似度都达到了99%-100%(表3);而且,其序列与GenBank中收录的相同及相近种属的同源序列一起构建了系统发育树(图2),也显示菌株DLEN2008006、6-F和15-F分别与曲霉属的Aspergillus clavatus、A. sydowii和A. fumigatus亲缘关系较近,菌株6-N和DLEN2008024则分别与粪壳菌纲的Chaetomium globosum 和Glomerella acutata有较近亲缘关系。由此确定了这些菌株的种属,它们都属于子囊菌门中的盘菌亚门。
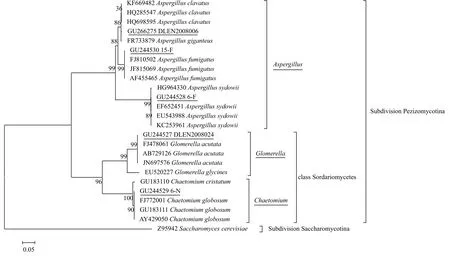
图2 根据ITS1-5.8S-ITS2 rRNA基因序列构建的5株活性真菌的系统发育邻接树
而且,薄层层析(图3)显示,在同一培养条件下,这5个菌株的次生代谢产物也有较大的差异,同属于曲霉属的6-F、15-F和DLEN2008006的次生代谢指纹差别显著。

图3 五种活性样品的薄层层析图像(254、365 nm双波长检测)
2.4 活性样品的HPLC-活性跟踪实验
通过HPLC-C18柱分离、96孔板收集及随后对两种指示菌的抑制活性测试与数据分析,可将各样品的HPLC谱图与其选择性抑制大肠杆菌[AB3027(-)/AB1157(+)]活性色谱图对应起来。结果(图4)显示,该5个样品的HPLC色谱图呈现较大的差异,这与分类学及TLC分析结果是一致的;而且这5个样品中的主要活性色谱峰也各不相同。
菌株6-N提取物中选择性抑制缺陷型大肠杆菌的活性主要集中在保留时间为8.75 min的显著色谱峰,其对缺陷型菌株及野生型菌株的抑制率差值达到100%;6-F提取物中选择性抑制缺陷型大肠杆菌的活性色谱峰的分布比较分散,有多个峰抑菌率差值介于50%-70%之间;DLEN2008006提取物中保留时间6.25 min的显著色谱峰显示了最强的选择活性,其对缺陷型大肠杆菌与野生型菌株的抑制率差值达到100%;15-F提取物中保留时间为4.75 min和41.25 min的色谱峰显示了相对较强的选择活性,其抑制率差值均在70%左右;而菌株DLEN2008024提取物中的色谱峰选择活性相对都比较弱,保留时间为5.25 min的色谱峰达到50%左右的抑菌率差值。
另外,上述样品中都存在一些色谱峰具有“负”的选择活性,即对缺陷型菌株及野生型菌株的抑制率差值为负值,特别是菌株6-F(保留时间2.75 min无紫外吸收的活性峰)和DLEN2008024(保留时间41.25 min的色谱峰)中都出现了抑菌率差值为-100%的色谱峰,这些色谱峰对应的物质也可用于非DNA损伤机制抗菌药物的研究。
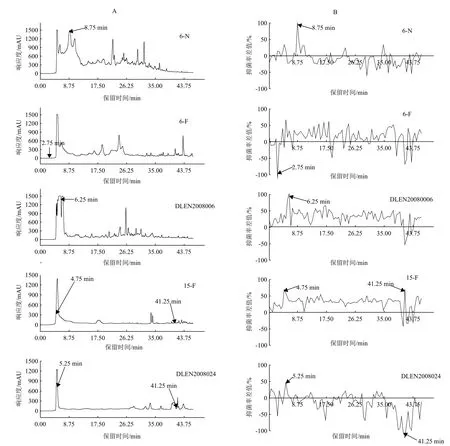
图4 五株活性真菌提取物的HPLC色谱图(254 nm,A)和选择性抗大肠杆菌[AB302F(-)/AB1157(+)]活性色谱图(B)(箭头标明主要活性色谱峰)
3 讨论
从海洋微生物中寻找抗肿瘤、抗菌活性物质是海洋药物研究的重要课题,而经济高效的筛选模型在研究工作中至关重要,一些采用特殊的微生物或其他低等生物作指示的方法常被用于对大量发酵粗提物的活性初筛,如大肠杆菌生化诱导分析(BIA)法、海虾致死法、稻瘟霉法等[11-13]。大肠杆菌差异性DNA 损伤修复试验法(DDRT)用于筛选基于DNA损伤机制的抗肿瘤抗菌药物,具有简单、快速、灵敏和经济的优点,可发现在不同位点、经各种途径诱导遗传损伤的样品,且能兼顾抗菌与抗肿瘤活性,适合于需要大量微生物发酵粗提物样品的筛选。大肠杆菌菌株对E. coli 343/591 和E. coli 343/636曾被引入国内用于该方法[14],本研究则采用了曾用于环境遗传毒理研究的大肠杆菌菌株对E. coli AB3027(-)(DNA损伤修复基因缺陷型)和E. coli AB1157(+)(具有正常DNA损伤修复功能的野生型)来建立该模型[7]。
在该模型对DNA损伤活性物质的响应能力验证实验中,博莱霉素、丝裂霉素C、阿霉素和5-氟尿嘧啶这4种抗肿瘤药物都显示出了预期的DDRT差异活性(即对缺陷型菌株的抑制强于对野生型的抑制),这可能与它们的作用机制均针对DNA有关[15-18];而前人药理学研究指出紫杉醇作用于微管,与DNA无关[19,20],本研究也显示它对这两株指示菌均无抑制。这些结果表明由这对大肠杆菌菌株组成的DDRT模型确可用于检测选择性损伤DNA的物质。当然该模型也存在一定局限性,在具体应用中最好能与肿瘤细胞株等模型结合使用,进行分级筛选,但作为一个经济的初筛模型仍然具有自身的价值。
使用该筛选模型,本研究发现5株海洋真菌显示出了较强的DDRT差异活性,很可能具有DNA损伤活性,它们均来自子囊菌门(Chaetomium、Aspergillus和Glomerella属)。目前已经报道发现的作用机制为损伤DNA的真菌天然产物并不多,检索表明天然产物词典(Dictionary of Natural Products on DVD)中收录的真菌产生的直接或间接损伤DNA活性的化合物仅9例报道,主要来自子囊菌(Paecilomyces、Alternaria、Fusarium和 Oidiodendron属)和担子菌(Marasmius、Curvularia和Lactarius属),化合物类型包括蒽醌或萘醌衍生物、二萜衍生物、生物碱以及含吡喃酮或呋喃酮结构的化合物等[1]。而本研究发现的菌株不同于这些已经报道的种属,且系统发育分析及TLC、HPLC-活性跟踪显示它们具有生物学和化学的多样性,因此从中发现新的DNA损伤活性物质可能性较大。
值得注意的是,这些菌株中弥散分布、含量较低但仍显示出中低选择活性(对两种指示菌的抑制率差值多数在40%-60%之间)的色谱峰多样性也很高,这为寻找多样性的DNA损伤活性物质提供了启示和深入研究的线索。其中有些活性峰可能是因为含量低,使得差别显现得还不够充分,如果加大进样量,可能会更加凸显。另外,也不排除其中有些是受到指示菌的生长状态不均衡、96孔板间移液操作不准确等实验误差的影响所造成的假阳性,采取更多的实验重复可能会排除这种影响。
当然,尽管对DNA损伤修复基因缺陷型大肠杆菌的选择性抑制表明这些样品可能作用于大肠杆菌的DNA,但其损伤效应还有待彗星实验的直观验证[21],其抗肿瘤潜力及对高等动物正常体细胞DNA是否具有损伤作用也有待深入研究。
4 结论
本研究建立了一种基于缺陷型/野生型大肠杆菌菌株对的DNA损伤活性筛选模型,该模型对于丝裂霉素C、阿霉素等抗肿瘤药物都有较好的响应,可用于抗菌及潜在抗肿瘤活性物质的筛选,因其成本低廉、操作简单的特点尤其适合海洋微生物等涉及大量粗提物的活性筛选。运用该模型,我们对实验室保藏的海洋真菌菌株进行了筛选,发现了5株活性海洋真菌,鉴定了其种属并对其TLC特征及活性成分进行了追踪分析,研究表明这些菌株具有较好的生物与化学多样性。
[1]Buckingham J. Dictionary of Natural Products on DVD. Chapman and Hall/CRC, London, UK, 2011.
[2] Blunt JW, Copp BR, Keyzers RA, et al. Marine Natural Products[J]. Natural Product Report, 2014, 31:160-258.
[3]Blunt JW, Copp BR, Keyzers RA, et al. Marine Natural Products[J]. Natural Product Report, 2013, 30:237-323.
[4]高传柱, 王天帅, 陈佳, 等. 铂类抗肿瘤药物作用机制研究进展[J]. 昆明理工大学学报, 2014, 39(4):83-92.
[5]李根, 林昱. DDRT法筛选抗肿瘤海洋微生物[J]. 中国抗生素杂志, 2004, 29:449-451.
[6]许文思, 彭剑, 王吉成, 等. 一种新的快速简便的抗肿瘤药物筛选模型[J]. 中国抗生素志, 2001, 26(1):15-18.
[7]Alam MZ, Ahmad S, Malik A. Genotoxic and mutagenic potential of agricultural soil irrigated with tannery effluents at Jajmau(Kanpur),India[J]. Arch Environ Contam Toxicol, 2009, 57:463-476.
[8]ZhangY, Mu J, Feng Y, et al. Broad-spectrum antimicrobial epiphytic and endophytic fungi from marine organisms:isolation, bioassay and taxonomy[J]. Marine Drugs, 2009, 7(2):97-112.
[9]苏煦, 张翼, 穆军, 等. 海洋真菌抗病原菌生物活性物质的研究[J]. 天然产物与开发, 2011, 23:1025-1030.
[10]Harrison CJ, Langdale JA. A step by step guide to phylogeny reconstruction[J]. The Plant Journal, 2006, 45:561-572.
[11]Singh MP, Janso JE, Brady SF. Cytoskyrins and cytosporones produced by Cytospora sp. CR200:taxonomy, fermentation and biological activities[J]. Marine Drugs, 2007, 5:71-84.
[12]Zhang Y, Han JY, Mu J, et al. Bioactivity and constituents of several common seaweeds[J]. Chinese Science Bulletin, 2013,58:2282-2289.
[13]郭文, 蒋继志. 利用稻瘟霉模型初级筛选抗肿瘤物质研究进展[J]. 生物学通报, 2010, 45(4):1-2.
[14]李根, 陈瑞川, 林昱. 具抗肿瘤活性的海洋微生物菌株的初步筛选[J]. 台湾海峡, 2002, 21(1):18-22.
[15]邹国林, 周晖. 博莱霉素与DNA的作用机理及其类酶性研究[J]. 武汉大学学报, 1998, 44(6):749-753.
[16]贾金海, 李勇, 张晓琳, 等. 丝裂霉素对体外胃癌细胞DNA损伤和增殖的影响[J]. 肿瘤防治研究, 2012, 39(9):1060-1063.
[17]冯硕, 李正平, 张淑红, 等. 阿霉素和DNA相互作用的研究初探[J]. 河北职工医学院学报, 2004, 21(2):43-44.
[18]刘永辉, 李公春, 崔娇娇. 5-氟尿嘧啶类抗肿瘤药物的研究进展[J]. 河北化工, 2008, 31(9):10-14.
[19]王世卿. 紫杉醇类化合物抗肿瘤作用机制研究[J]. 淮海医药,2008, 26(4):375-376.
[20]Oshige M, Takenouchi M, Kato Y, et al. Taxol derivatives are selective inhibitors of DNA polymerasea α[J]. Bioorganic & Medicinal Chemistry, 2004, 12(10):2596-2601.
[21]陈颖, 王磊, 王子健. 用彗星实验技术检测环境遗传毒性物质[J]. 土壤学报, 2006, 43(4):673-678.
(责任编辑 李楠)
Screening the Extracts of M arine Fungi for the Activity of Damaging the DNA of Escherichia coli and Researches on Active Strains
Bao Hanyan1Gu Pengjuan1Zhang Yi2Mu Jun3Feng Yan2Zhao Chenyan1
(1.College of Environmental and Chemical Engineering,Dalian Jiaotong University,Dalian116028;2.College of Food Science and Technology of Guangdong Ocean University,Guangdong Provincial Key Laboratory for Processing and Safety of Aquatic Products,Key Laboratory of Further Processing Aquatic Products of Guangdong Higher Education Institutions,Zhanjiang524088;3. College of Marine Science and Technology,Zhejiang Ocean University,Zhoushan316022)
Screening of antimicrobial and antitumor agents from marine microorganisms is one of the hot spots for the research of marine drugs, and screening models play an important role in the study. The authors set up an Escherichia coli differentiated DNA damage repair test model, which can be used for the screening of antimicrobial and potential antitumor substances. This model was further applied to screen the bioactivities of the extracts of marine symbiotic fungi. As the result, five marine fungal strains’ extracts showed strong selective inhibition to E. coli strains with DNA damage repairing genes-deficiency, i.e, the potential of damaging DNA. The five strains were identified by molecular biological methods. Furthermore, analyses by thin layer chromatography(TLC)and high performance liquid chromatography(HPLC)-bioactivity tracing revealed that these strains produced diverse bioactive substances.
marine microorganisms;Escherichia coli differentiated DNA damage repair test model;identification
10.13560/j.cnki.biotech.bull.1985.2015.08.020
2014-11-06
国家自然科学基金项目(20902009),中国博士后科学基金项目(2011M500051,2012T50258),广东省“扬帆计划”引进紧缺拔尖人才项目(201433009),广东海洋大学引进人才科研启动项目(E15155),广东海洋大学创新强校工程项目(GDOU2014030502,GDOU2014050203),广东海洋大学自然科学研究项目(C14519)
鲍海燕,女,硕士研究生,研究方向:海洋真菌的生物活性,E-mail:baohaiyanbhy@163.com;谷朋娟同为本文第一作者
张翼,男,硕士生导师,研究方向:海洋药用生物资源的研究与利用;E-mail:hubeizhangyi@163.com

