CBHⅡ和EGⅣ的重构基因在里氏木霉中的组成型表达
曾敏,邓丽瑜,汤新,刘刚,余少文,*
(1.深圳大学生命科学学院,深圳市微生物基因工程重点实验室,广东深圳518060;2.深圳大学生命科学学院,深圳市海洋生物资源与生态环境重点实验室,广东深圳518060)
CBHⅡ和EGⅣ的重构基因在里氏木霉中的组成型表达
曾敏1,2,邓丽瑜1,汤新1,刘刚1,余少文1,*
(1.深圳大学生命科学学院,深圳市微生物基因工程重点实验室,广东深圳518060;2.深圳大学生命科学学院,深圳市海洋生物资源与生态环境重点实验室,广东深圳518060)
重构基因cbh2-linker-CDeg4表达的融合蛋白同时获得具有外切葡聚糖酶CBH II和内切葡聚糖酶EG IV 2种催化活性,在纤维素降解等方面有着重要应用。本研究通过overlap PCR将里氏木霉丙酮酸脱羧酶(PDC)的启动子、纤维二糖水解酶cbh1的信号肽、重构基因cbh2-linker-CDeg4和pdc终止子依次连接,以质粒pPICZαA为基本骨架,构建表达载体pPIC-PCT。采用原生质体转化法将pPIC-PCT和含潮霉素抗性筛选标记的质粒pAN7-1共转里氏木霉。SDS-PAGE分析表明,重构基因cbh2-linker-CDeg4实现了在里氏木霉中的组成型表达,测得重组木霉发酵上清液的CMCNa酶活最高达到4.08 U/mL,是出发菌株的5.55倍,FPA酶活达到0.915 U/mL,较出发菌株提高了43.6%。酶学性质初步研究表明:粗酶液的最适pH为5.0,最适温度为50℃。
基因重构;外切葡聚糖酶Ⅱ;内切葡聚糖酶Ⅳ;丙酮酸脱羧酶启动子
纤维素酶能有效降解纤维素,将其水解成葡萄糖,广泛应用于纺织、食品加工、酿酒、造纸和饲料等领域,对解决环境污染和能源危机具有重大意义[1-2]。纤维素酶广泛存在于多种微生物中,其中里氏木霉(Trichoderma reesei)是目前研究最透彻的产纤维素酶模式菌株[3-4],它产生的纤维素酶主要由内切型葡聚糖酶(endo-glucanases,EG)、外切型葡聚糖酶(exo-cel-lobiohydrolase,CBH)以及β-葡萄糖苷酶(β-Glucosi dase,简称BG)组成[5-7],它们通过协同作用降解纤维素[8]。其中只有CBH可作用于纤维素结晶区域,从纤维素分子的非还原端进行水解,而自然界中的木质纤维素大都呈结晶状态,因此CBH在纤维素降解过程中的作用非常重要。纤维素酶有2个活性区域:催化结构域(catalytic domains,CD)和纤维素结合结构域构(celluose-binding domains,CBD)[9-10],其中CD是纤维素酶的“活性中心”,在水解过程中起着重要作用。
根据纤维素酶各组分间的协同作用研究,人们发现适当改变各组分的比例可提高纤维素酶的降解效率。近年来,从分子结构水平改造纤维素酶基因,提高纤维素酶的生物学活性已经取得了重大进展。Lemos[11]等在基因cbh2和eg中增加CBD基因,提高了这两种纤维素酶的活性。刘刚等[12]在eg4基因中增加一个其自身的CD基因,使其重组蛋白活性提高了4倍。本实验室在cbh2下游增加eg4的CD基因,得到含2个催化结构域的重构基因cbh2-linker-CDeg4。本研究将该基因与里氏木霉丙酮酸脱羧酶的组成型强启动子(含信号肽)融合,构建里氏木霉组成型表达转化系统,以期得到可以高效表达融合蛋白CBHⅡ-EGⅣ且具有较高酶活力的重组木霉。
1 材料与方法
1.1菌株及质粒
T.reesei QM9414、大肠杆菌E.coli Top10F’、质粒pPICZαA、pAN7-1由本实验室保藏。pMD18-T simple vector购自TaKaRa公司。质粒pPIC-cbh2,pPIC-eg4由本实验室前期构建。
1.2培养基
Mandels基础培养基:Mandels营养盐浓缩液100 mL/L,Mandels微量元素浓缩液1.0 mL/L,无水葡萄糖10 g/L,蛋白胨1.0 g/L,1 M的柠檬酸缓冲液(pH4.5)50 mL/L,吐温80 1.0 g/L~2.0 g/L。
筛选培养基:含有STC和潮霉素100 μg/mL的PDA培养基。其中,STC、葡萄糖和PDA培养基的其他成分分为3部分各溶于约1/3的水中。
产酶培养基:50×Mandels营养盐浓缩液20 mL/L,1 000×Mandels微量元素浓缩液1.0 mL/L,黄豆饼粉50 g/L,1 M柠檬酸缓冲液(pH 4.5)50 mL/L,吐温80 1.0 g/L~2.0 g/L,葡萄糖30 g/L。
1.3表达载体pPIC-PCT的构建
分别以实验室前期构建的质粒pPIC-eg4、pPIC-cbh2为模板,根据NCBI公布eg4、cbh2序列设计引物,经PCR获得基因片段linkereg4、CDeg4和cbh2。依次经双酶切连接至质粒pPICZαA,重构质粒pPIC-cbh2-linker-CDeg4。
以里氏木霉基因组DNA为模板,根据Genebank报道的序列设计引物,分别经PCR扩增获得纤维二糖水解酶I信号肽序列Scbh1、丙酮酸脱羧酶启动子Ppdc和终止子Tpdc序列。利用overlap PCR技术依次连接PSpdc、cbh2-linker-CDeg4、Tpdc,构建表达载体pPIC-PCT。
1.4里氏木霉的原生质体转化及筛选
表达载体pPIC-PCT经Xba I酶切纯化处理后,与含潮霉素筛选标记的质粒pAN7-1共转T.Reesei,主要参照Penttila等的方法进行[13]。取适量转化液涂布到含有100 μg/mL潮霉素的PDA平板上,28℃培养观察3 d~5 d至平板上长出菌丝,挑取单菌落菌丝转接至100 μg/mL潮霉素的PDA小平板上进行复筛,28℃培养2 d,再转接至新鲜无抗性PDA小板上培养7 d~10d。
1.5重组木霉的组成型表达
用无菌水刮取PDA培养平板上生长7 d的木霉转化子孢子,接种至含潮霉素抗性的Mandels基础培养基中,28℃,220 r/min振荡培养1.5 d~2 d。以5%的接种量转接至产酶培养基中,28℃,220 r/min,振荡培养。11 000 r/min,离心10 min,收集上清,即为粗酶液,每24 h取样一次检测粗酶液的酶活力。
1.6重组木霉的PCR鉴定
以重组木霉基因组DNA为模板,PCR扩增重构基因cbh2-linker-CDeg4,以木霉基因组DNA为对照,用1%琼脂糖凝胶电泳进行检测并测序。
1.7重组木霉表达产物的分析
SDS-PAGE电泳:取粗酶液进行SDS-PAGE,分析融合蛋白CBHⅡ-EGⅣ的表达情况。
CMCNa酶活的测定:采用DNS法测定内切葡聚糖酶的活性[14],酶活力单位(U)定义为1 min水解生成1 μmol葡萄糖所需的酶量。取适当稀释后的粗酶液0.5 mL与1%CMC-Na(用0.05 M柠檬酸缓冲液配制,pH4.8)于50℃水浴中反应30 min,立即加入3 mL DNS终止酶反应,充分混匀,沸水浴5 min,冷却,充分混匀后测OD540。每组实验做3个重复。
滤纸酶活(FPA)的测定:取0.5 mL适当稀释后的粗酶液、1 mL柠檬酸缓冲液(0.05 M,pH4.8)及50 mg的滤纸条(Whatman NO.1)在50℃水浴中反应30 min,立即加入3mLDNS终止酶反应,充分混匀,沸水浴5min,冷却,充分混匀后测OD540。每组实验做3个重复。
1.8重组木霉酶学性质的初步研究
酶反应的最适pH:分别配制pH为3.5、4.0、4.5、5.0、5.5、6.0、6.5、7.0的0.05 mol/L的柠檬酸缓冲液,在对应pH条件下进行CMCNa酶活测定。
酶反应的最适温度:分别在30、40、50、60、70、80℃下进CMCNa酶活测定。
2 结果与分析
2.1表达载体的构建
通过酶切连接法获得大小为2 205 bp的重构基因cbh2-linker-CDeg4,将该基因插入PSpdc和Tpdc之间,构建表达载体pPIC-PCT,大小约为7 000 bp,其测序结果与预期相符,可用于后续转化实验(如图1)。
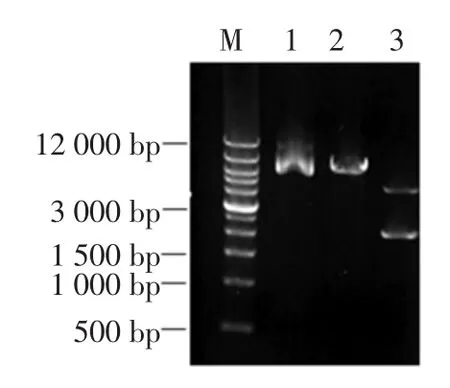
图1 表达载体pPIC-PCT的酶切鉴定Fig.1Enzyme digestion of the recombined plasmid pPIC-PCT
2.2重组木霉的PCR鉴定
如图2所示:可在大部分重组木霉的基因组中扩增到重构基因cbh2-linker-CDeg4,且测序结果与预期一致,表明重构基因cbh2-linker-CDeg4已成功整合到到里氏木霉基因组中。
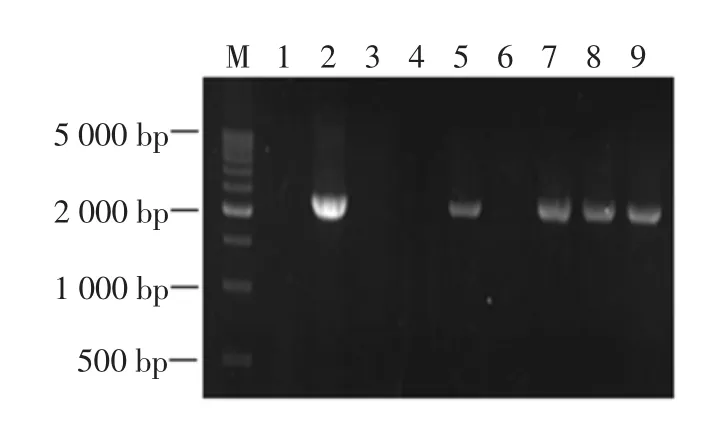
图2 重组木霉基因组的PCR鉴定Fig.2Identification of recombinant T.reesei by PCR
2.3SDS-PAGE分析
融合蛋白CBHⅡ-EGⅣ在pdc启动子的调控下,成功实现了在里氏木霉中的组成型表达。SDS-PAGE电泳分析结果表明(图3),融合蛋白CBHⅡ-EGⅣ的分子量约为100 KD,大于理论值80.56 KD,推测是重组蛋白在表达时发生了糖基化修饰。
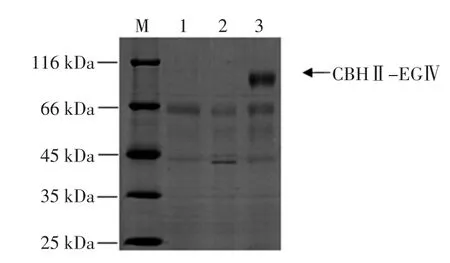
图3 重组木霉的SDS-PAGE分析Fig.3Analysis of of recombinant T.reesei by SDS-PAGE
2.4重组木霉菌株的酶活测定
在相同条件下对重组木霉菌株及出发菌株进行组成型产酶实验,测得重组木霉A4的CMCNa酶活最高,达到4.08 U/mL(图4),是出发菌株的5.55倍。重组木霉A4的FPA酶活为0.915 U/mL(图5),较出发菌株提高了43.6%。由于初始菌体浓度较低,前48 h内几乎检测不到A4的CMCNa酶活和FPA酶活,96、84 h其酶活分别达到最高。培养108 h时,CMCNa酶活和FPA酶活均下降,可能是由于融合蛋白被里氏木霉自身分泌的蛋白酶所降解。
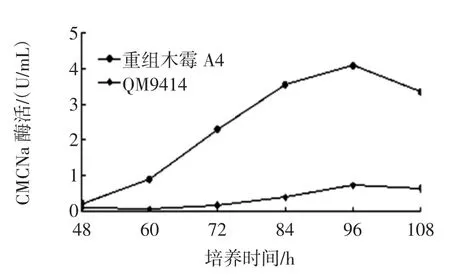
图4 重组木霉A4的CMCNa酶活Fig.4CMCNaactivity of recombinant T.reesei
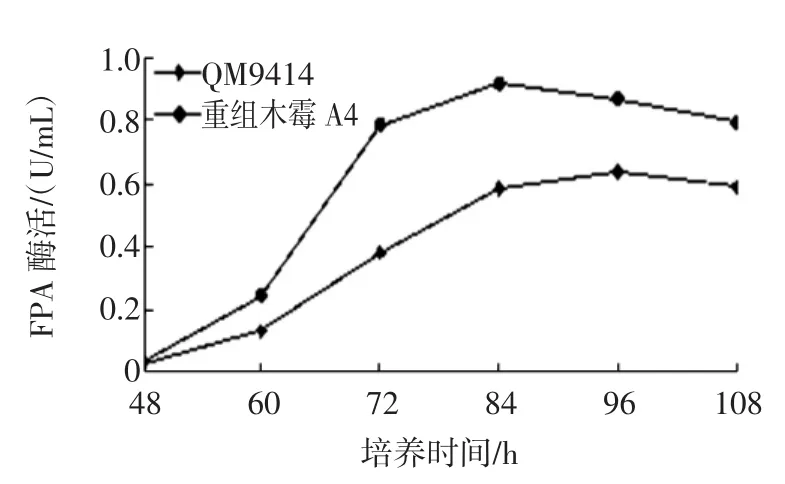
图5 重组木霉A4的FPA酶活Fig.5FPA activity of recombinant T.reesei
2.5酶学性质的初步研究
如图6所示,粗酶液的最适pH为5.0,在pH为4.0~5.5范围内相对酶活较高,pH过低或过高都对酶活有较大影响。
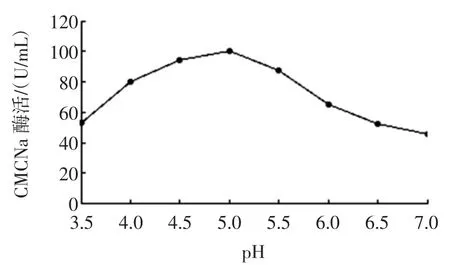
图6 粗酶液的最适pHFig.6The optimum pH of crude enzyme
如图7所示,粗酶液的最适反应温度为50℃,在50℃~60℃范围内相对酶活较高,在70℃时还能保持最高酶活的66.6%,当温度达到80℃时,酶活急剧下降。
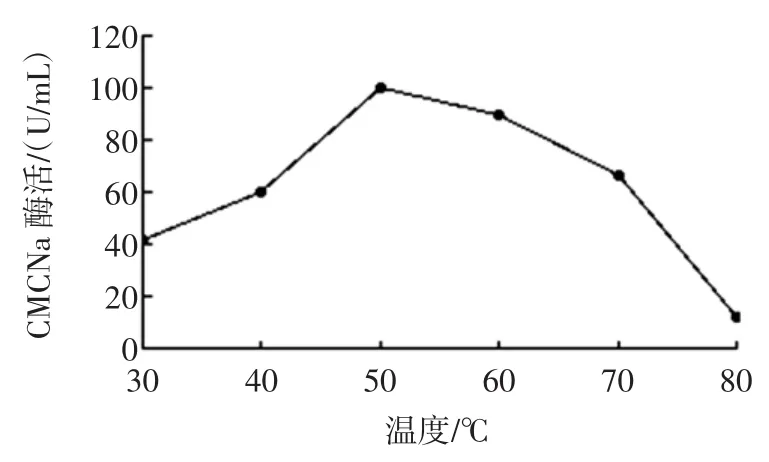
图7 粗酶液的最适温度Fig.7The optimum temperature of crude enzyme
3 结论与讨论
纤维素酶应用广泛,但因酶活低、成本高等因素不能进行大规模工业化生产和应用[15]。近年来,通过基因工程技术来获得高活性、高产量的纤维素酶逐渐受到重视。本研究对纤维素酶cbh2和eg4进行重构,获得具有2个催化结构域的重构基因,它表达的蛋白具有这2种纤维素酶组分的催化活性,在实际应用中具有更广阔的前景。
里氏木霉能高效合成和分泌蛋白质,是高产纤维素酶的代表菌种。它常使用cbh1启动子构建表达系统[16-18],实现目的基因的同源或异源表达,但cbh1启动子需要纤维素或乳糖的诱导才能调控目的基因的表达。此外,诱导物也能诱导里氏木霉其它纤维素酶的表达,加大了目的蛋白分离、纯化的难度。LI等[19]通过RT-qPCR技术从里氏木霉中筛选出可以使木聚糖酶Ⅱ基因(xyn2)高效表达的pdc启动子,且里氏木霉分泌的总蛋白中约83%是XYN2蛋白。WANG等[20]利用pdc启动子调控康氏木霉纤维素酶转录激活因子xyr1的表达,结果显著提高了纤维素酶的活性。pdc组成型启动子所构建的表达系统无需诱导物,能在富含葡萄糖的培养条件下启动基因的转录。里氏木霉自身分泌蛋白较少,能有效解决目的蛋白的分离及纯化问题。本研究利用pdc启动子实现了重构基因cbh2-linker-CDeg4在里氏木霉中的组成型表达,其CMCNa酶活达到4.08 U/mL,是出发菌株的5.55倍,FPA酶活达到0.915 U/mL,较出发菌株提高了43.6%,大大提高了酶活力。
综上所述,重构基因cbh2-linker-CDeg4在pdc启动子的调控下,实现了在里氏木霉中的组成型高效表达,且杂蛋白较少,容易提纯。酶学性质研究表明,粗酶液的最适pH为5.0,最适反应温度为50℃。cbh2-linker-CDeg4同时具有cbh2和eg4的催化活性,在食品、饲料、纺织等工业有着广泛应用前景。今后,我们将对产酶培养基进行优化,通过改变对酶活影响较大的3个因素(碳源、氮源、无机盐)来确定最优培养方案,进一步提高该融合蛋白的酶活。今后将对该蛋白进行纯化,并对其酶学性质做详细研究。
[1]邱雁临.纤维素酶的研究和应用前景[J].粮食与饲料工业,2001(8):30-31
[2]伯永科,崔海信.纤维素的循环经济效益[J].中国资源综合利用,2006,24(12):14-16
[3]Sun WC,Cheng CH,Lee WC.Protein expression and enzymatic activity of cellulases produced by Trichoderma reesei Rut C-30 on rice straw[J].Process Biochemistry,2008,43(10):1083-1087
[4]Martinez D,Berka RM,Henrissat B,et al.Genome sequencing and analysis of the biomass-degrading fungus Trichoderma reesei(syn Hypocrea jecorina)[J].Nat.Biotechnol,2008,26:553-560
[5]Wilson DB.Three microbial strategies for plant cell wall degradation[J].Ann.N.Y.Acad.Sci,2008,1125:289-297
[6]Foreman,P,Brown D,Dankmeyer L,et al..Transcriptional regulation of biomass-degrading enzymes in the filamentousfungus Trichoderma reesei[J].Biol.Chem,2003,278:31988-31997
[7]Saloheimo M,Kuja-Panula J,Ylösmäki E,et al.Enzymatic properties and intracellulase localization of the novel Trichoderma reesei b-glucosidase BGLII(Cel1A)[J].Appl.Environ.Microbiol,2002,68:4546-4553
[8]Singhania RR,Sukumara RK,Patel AK,et al.Advancement and comparative profiles in the production technologies using solidstate and submerged fermentation for microbial cellulases[J].Enzyme Microb Technol,2010,46:541-549
[9]Pentila M,Lehtovaara P,Nevalainen H,et al.Homolgy between cellulase genes of Trichoderma reesei:compete nucleotide sequence of endoglucanase I gene[J].Gene,1986,45:253-263
[10]Kauri T,Kushner DJ.Role of contat in bacterial degradation of cellulose[J].Fems Microbiol Ecol,1985,31:301-306
[11]the addition of cellulose binding domains derived from Trichodermareesei[J].Enzyme and Microbial Technology,2003,32:35-40
[12]LIU Gang,TANG Xing,TIAN Shengli,et al.Improvement of the cellulolytic activity of Trichoderma reesei endoglucanase IV with an additional catalytic domain[J].World J Microbiol Biotechnol,2006,22:1301-1305
[13]Penttil M,Nevalainen H,Ratto M,et al.A versatile transformation system for the celluloytic filamentous fungus Trichodema reesei[J]. Gene,1987,61(2):155-164
[14]乔宇,毛爱军,何永志,等.里氏木霉内切-β-葡聚糖酶Ⅱ基因在毕赤酵母中的表达及酶学性质研究[J].菌物学报,2004,23(3):388-396
[15]Himmel ME,Ding SY,Johnson DK,et al.Biomass recalcitrance:engineering plants and enzymes for biofuels production.[J].Science,2007,315(5813):804-807
[16]Zhang Jiwei,Zhong Yaohua,Zhao Xuena,et al.Development of the cellulolytic fungus Trichoderma reesei strain with enhanced β-glucosidase and filter paper activity using strong artifical cellobiohydrolase 1 promoter[J].Bioresource Technology,2010,101(24):9815-9818
[17]Zhong Yaohua,Liu Xuan,Xiao Peng,et al.Expression and Secretion of the Human Erythropoietin Using an Optimized cbh1Promoter and the Native CBH I Signal Sequence in the Industrial FungusTrichoderma reesei[J].Biochemistry and Biotechnology,2011,165(5/6):1169-1177
[18]Wang Bingbing,Xia Liming.High efficient expression of cellobiase gene from Aspergillus niger in the cells of Trichoderma reesei[J]. Bioresource Technology,2011,102(6):4568-4572
[19]Li Junxin,Wang Juan,Wang Shaowen,et al.Achieving efficient protein expression in Trichoderma reesei by using strong constitutive promoters[J].Microbial Cell Factories 2012,11:84
[20]Wang Shaowen,Liu Gang,Wang Juan,et al.Enhancing cellulase production in Trichoderma reesei RUT C30 through combined manipulation of activating and repressing Genes[J].Ind Microbiol Biotechnol,2013,40:633-641
Construction of CBH II and EG IV for Homologous Protein Expression with Constitutive Promoter in Trichoderma Reesei
ZENG Min1,2,Deng Li-yu1,TANG Xin1,LIU Gang1,YU Shao-wen1,*
(1.College of Life Science,Shenzhen Key Laboratory of Microbial Genetic Engineering,Shenzhen University,Shenzhen 518060,Guangdong,China;2.College of Life Science,Shenzhen Key Laboratory of Marine Bioresources and Ecology,Shenzhen 518060,Guangdong,China)
The reconstructed gene cbh2-linker-CDeg4gain two kinds of cellulase catalytic properties,which added the catalytic domain(CD)gene from eg4 to the 3'end of cbh2.The gene cbh2-linker-CDeg4was inserted between T.reesei QM9414 strong promoter PSpdc(including secreting signal peptide sequence)and terminator Tpdc,generating reconstructed plasmid pPIC-PCT.The plasmid pPIC-PCT and plasmid pAN7-1 were transformed into T.reesei via an improved protoplast transformation.SDS-PAGE analysis showed that the fusion protein CBHⅡ-EGⅣhad successfully expressed in T.reesei.The CMCNaactivity of the T.reesei recombinants could reach 4.08 U/mL,which is 5.55-fold as high as that of the host strain;the FPA cativity could reach 0.915 U/mL,which were increased by 43.6%.The cellulase characterization of recombinant showed that the optimum pH value was about 5.0,and the optimum reaction temperature was at 50℃.
gene recombination;cellobiohydrolaseⅡ;endo-β-1,4-glucanaseⅣ;pyruvate decarboxylase promoter
10.3969/j.issn.1005-6521.2015.13.032
2014-03-12
深圳市基础研究计划(JCYJ20120613115323982)
曾敏(1988—),女(汉),硕士研究生,研究方向:酶工程。

