鸡T细胞受体γ链基因位点重新测序和组装
马俊平,杨犀,律娜,刘飞,陈燕,3,朱宝利
1.中国科学院微生物研究所,北京 100101;
2.中国科学院大学,北京 100049;
3.中国农业科学院北京畜牧兽医研究所ASTIP-IAS03,北京 100193
鸡T细胞受体γ链基因位点重新测序和组装
马俊平1,2,杨犀1,2,律娜1,刘飞1,陈燕1,3,朱宝利1
1.中国科学院微生物研究所,北京 100101;
2.中国科学院大学,北京 100049;
3.中国农业科学院北京畜牧兽医研究所ASTIP-IAS03,北京 100193
动物T细胞受体(T cell receptor,TCR)基因由多个不同的高度同源的基因家族组成,通过全基因组测序很难获得准确的基因序列和排列位置。文章通过在NCBI中发布的鸡TCR的γ链(TCRγ或TRG)基因片段序列定位了鸡TRG基因所在区域,并确定了与鸡TRG基因位点对应的细菌人工染色体(BAC)克隆(CH261-174P24)。对该克隆进行高通量的重新测序和组装后,得到含有10个scaffolds的基因组草图,较完整地覆盖了鸡TRG基因位点及两侧区域。通过PCR扩增和测序证明了scaffold内部结构的正确性,校正了鸡参考基因组TRG基因位点一个可变基因和一个缺口序列(gap)附近各一处错误序列,以及可变基因区多处序列错误。文章通过校正鸡参考基因组TRG基因位点的序列,为鸡TRA/D和TRB基因位点的基因组序列分析提供了新方法。
鸡;T细胞受体(TCR);伽马链(TCRγ或TRG)基因位点;测序;组装
T细胞受体(T cell receptor,TCR)是T淋巴细胞表面特异性识别抗原的受体,在机体的免疫应答和免疫调节中发挥着重要作用。对TCR基因结构与功能的研究,是深入探索机体免疫应答机理的重要内容之一。
对人(Homo sapiens)和小鼠(Mus musculus)TCR的研究发现[1~4],TCR包括α、β、γ和δ 4种肽链,以两种形式存在,它们是由二硫键连接形成的α/β或γ/δ异二聚体蛋白链。在胚系中,编码TCR的基因序列是由多个可变的基因区域组成。TCRβ和TCRδ基因位点包括可变基因区(Variable gene region,V基因区)、多样基因区(Diversity gene region,D基因区)、结合基因区(Joining gene region,J基因区)和恒定基因区(Constant gene region,C基因区)等,TCRα和TCRγ基因位点由V基因区、J基因区和C基因区组成。但这些基因之间有较高同源性,且基因个数和排列顺序等较难确定,尤其是V基因区的基因数目众多且以基因簇的形式存在。这些都成为TCR研究的难点,使得TCR基因在染色体上很难进行准确定位。20世纪末期,随着以Sanger双脱氧链终止法为代表的第一代测序技术在研究领域的广泛应用,人和小鼠TCR基因位点的序列和结构才逐渐被解析清楚,其功能也得到了注释。
但到目前为止,鸡TCR基因的相关研究开展得较少,其序列和结构并不完全清晰。20世纪90年代以来,Cooper等相继发现了鸡TCR的α、β、γ、δ等4种亚型,并对胸腺、肝脏等器官中的部分TCR基因序列进行了测定和分析[5~13]。2004年,国际鸡基因组测序中心在《自然》杂志上公布了红原鸡(Gallus gallus)的参考基因组草图[14],采用鸟枪法对红原鸡的全基因组进行了测序和组装,并分别于2006年和2011年进行过两次补充。即便如此,NCBI数据库中鸡参考基因组上TCR基因位点仍然存在很多缺口序列。
本研究拟通过对鸡TCRγ(TRG)基因片段序列的分析,在红原鸡BAC克隆文库(CHORI-261)中挑选包含鸡TRG基因的BAC克隆,采用Illumina公司的Miseq高通量测序系统对这些克隆进行重新测序和组装,以期获得鸡TRG基因位点的草图,并对鸡参考基因组上TRG基因位点的错误信息进行校正。
1 材料和方法
1.1 鸡TCRγ链(TRG链)编码序列的定位
从NCBI数据库中下载鸡TRG链的编码序列。通过参考文献中报道的鸡TRG基因的一些特征[8~11],分别提取V区、J区和C区基因片段。然后与鸡参考基因组(WUGSC2.1/galGal3)进行比对。根据基因片段密集区域判断鸡TRG基因位点区间。
1.2 BAC克隆的确定
根据加州大学圣克鲁兹分校基因组网站上(UCSC Genome Browser)鸡参考基因组注释的BAC克隆信息(galGal3),参照上一步定位的鸡TRG基因位点区间,在鸡TRG基因位点上下游约50~100 kb范围内挑选出载有鸡全部TRG基因的1个BAC克隆,编号为CH261-174P24。
该克隆CHORI-261-174P24(以下简称克隆174P24)由奥克兰儿童医院及研究中心(Children's Hospital Oakland research Institute,CHORI)构建(网站:http: //bacpac.chori.org/library.php?id=120),所用载体为质粒pTARBAC2.1,此BAC克隆长度约为218 kb。
1.3 BAC DNA的提取
克隆174P24以半固体穿刺法保存,经过在含有氯霉素(终浓度为12.5 ug/mL)的固体LB平板上划线和活化培养,得到单克隆。挑取单克隆于液体LB培养基中,采用氯霉素做诱导,进行过夜培养。利用QIAGEN公司的质粒中量提取试剂盒(QIAGEN Plasmid Midi Kit)提取BAC DNA。
1.4 BAC DNA的初步鉴定
1.4.1 酶切鉴定
从NCBI网站下载克隆174P24的参考序列(鸡参考基因组版本为ICGSC Gallus_gallus-4.0/galGal4)。运用NEBcutter2.0软件预测参考序列的限制性内切酶酶切位点。在预测结果里筛选所用限制性内切酶,筛选标准为:(1)在参考序列中酶切位点较少,以便于识别产物条带;(2)需为常用限制性内切酶,以便购买和实验操作。经过筛选,限制性内切酶NotⅠ适合用于鉴定。经内切酶NotⅠ充分消化后,产物进行脉冲场凝胶电泳。根据酶切产物的条带初步判断所选克隆。
1.4.2 PCR鉴定
依据克隆 174P24的已知末端序列,运用Primer5.0软件设计引物。以克隆174P24的DNA为模板,进行PCR扩增。产物由北京六合华大基因科技股份有限公司测序。测序序列再与该克隆末端序列比对,证明所选克隆的正确性。
1.5 BAC克隆的高通量测序
克隆174P24的DNA经纯化后,采用美国柏尔生物技术公司的快速DNA测序试剂盒(Bioo Scientific NEXTflexTM Rapid DNA-Seq Kit)构建500~700 bp的测序文库。由北京诺禾致源生物信息科技有限公司进行双端250 bp的MiSeq测序。
1.6 BAC克隆的组装、分析和证明
运用HTQC软件[15]和Bowtie软件对测序的原始reads进行预处理。获得的高质量序列对,使用ABySS软件[16]和 Phrap软件[17]进行组装。组装得到的scaffolds使用NCBI BLAST软件与鸡参考基因组(galGal4)进行比对,并找出同源区域。比对结果及找到的同源区域使用自定义脚本进行提取与可视化。
针对scaffolds内部分段的连接处,运用Primer5.0软件分别设计引物(表1)。以克隆174P24的DNA为模板,通过PCR扩增、测序和BLAST比对进行证明。
2 结果与分析
2.1 鸡TCRγ链编码序列的定位
从NCBI数据库中下载鸡TRG链的编码序列,共有203条。从中提取V基因、J基因、C基因集合与鸡参考基因组(galGal3)的比对结果(图1)显示,鸡TRG基因定位于2号染色体的约49 150~49 230 kb区间内,其中C基因区、V基因区分别处于5′端和3′端,J基因区位于两者中间。TRG V基因平均间距约为1.5 kb。从现有鸡TRG链编码序列在鸡参考基因组上的比对位置发现,鸡TRG基因位点只含有一个TRG C基因,包含3个外显子。
2.2 BAC克隆的确定和初步鉴定
参照UCSC数据库中鸡参考基因组(galGal3)上注释的BAC克隆信息,根据编码序列比对结果定位的鸡TRG基因位点在染色体上的位置,在鸡TRG基因位点上下游约50~100 kb范围内选取了载有鸡全部TRG基因的一个克隆,编号为CH261-174P24 (图1)。该克隆位于鸡参考基因组(galGal3)2号染色体的约49 109~49 313 kb区间内。
利用内切酶NotⅠ充分消化克隆174P24的质粒DNA,并对产物进行脉冲场凝胶电泳。克隆174P24经酶切后在10 kb左右有载体DNA条带;该克隆的插入片段中仅有一个NotⅠ酶切位点,约220 kb的插入片段被酶切为约190 kb和约30 kb的两条带。酶切结果表明,该克隆含有插入片段,且大小与克隆174P24相符。此外,PCR结果表明,克隆174P24

表1 克隆174P24组装scaffolds内部连接处证明的引物信息

图1 鸡TRG链编码序列比对到鸡参考基因组的示意图及对应的BAC克隆位置示意图A:鸡TRG链已知V基因(紫色竖线)、J基因(黄色竖线)、C基因(绿色竖线)集合比对到鸡参考基因组的示意图;B:鸡TRG基因对应的BAC克隆及其在鸡参考基因组上的位置。
经过扩增得到约400 bp的单一产物,对产物的测序结果与已知末端序列完全一致。推测鸡TRG基因位点位于克隆174P24中。
2.3 BAC克隆的高通量测序
克隆174P24的测序数据量约为237 Mb,获得了473 548条reads。其中,宿主菌基因组的污染低于1/1000;两侧read质量均在29以上,即测序准确性在99%以上;测序深度为1331倍。
2.4 BAC克隆的组装、分析和证明
2.4.1 BAC克隆的组装和比对分析
本研究中鸡TRG链克隆174P24的测序数据已提交至NCBI/SRA数据库(登录号:SRR1615248),该克隆组装的scaffolds信息已提交至GenBank数据库(登录号:KP058483)。本文运用ABySS软件和Phrap软件对该克隆进行组装。组装过程中经过多次尝试(k-mer值:96~192)和优化,发现当k-mer值取192时组装获得的scaffolds最为完整。利用NCBI BLAST软件对得到的scaffolds与鸡参考基因组(galGal4)进行比对,并找出同源区域,结果使用自定义脚本进行提取和可视化,组装和比对结果见图2。
克隆174P24参考序列的GC含量分布图显示,在该克隆约178 kb区间内,GC含量普遍低于50%,总GC含量为39.13%;在鸡TRG基因集中分布的约49 670~49 730 kb(约60 kb)区间,GC含量为42.29%,较两侧序列高。参考序列6处gap中的5处也集中在这个区间内。从该克隆测序reads的丰度分布图可以看出,与两侧序列比较,TRG基因片段密集的约60 kb区间内的reads丰度稍低且分布不均匀。
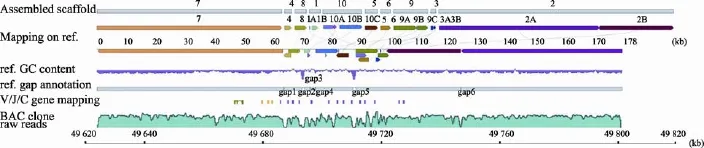
图2 鸡TRG链克隆174P24的组装草图和与参考序列的比对结果1:Assembled scaffold.(测序reads组装的scaffold(500 bp以上),上方数字表示scaffold编号);2:Mapping on ref.(scaffold与参考序列的比对情况);3:ref.GC content反应参考序列的GC含量分布,水平线代表GC含量为50%;4:ref.annotation展示参考序列的gap注释情况(gap1~gap6:100 bp、848 bp、100 bp、150 bp、958 bp、127 bp);5:V/J/C gene mapping(从鸡TRG链mRNA序列提取的V、J、C基因片段比对到参考序列的位置,其中绿色代表C基因,黄色代表J基因,紫色代表V基因);6:BAC clone raw reads展示测序reads的丰度分布。
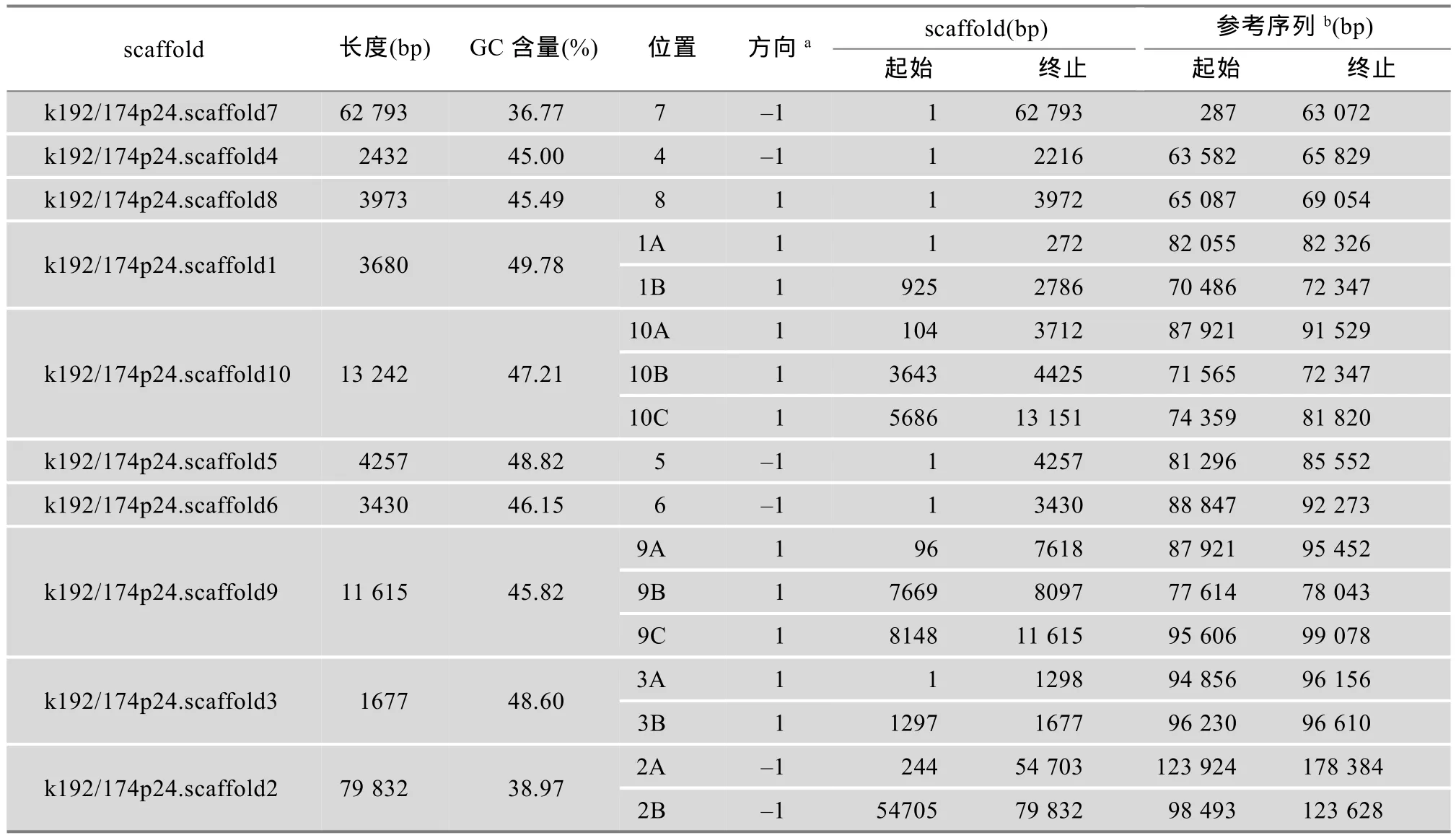
表2 鸡TRG基因位点克隆174P24组装的scaffold信息
经过组装本文得到了鸡TRG链克隆174P24的基因组草图,该草图包含10个长度介于1677~ 79 832 bp之间的scaffolds,与参考序列的比对结果见图2和表2。这10个scaffolds的总长度为18 6931 bp,与参考序列长度(约178 kb)接近,平均长度约为18 kb。经过与参考序列的比较发现,scaffold7长度为62 793 bp,位于TRG基因位点上游,scaffold2长度为79 832 bp,位于TRG基因位点下游,二者总长度共占10个scaffolds序列总长度的78%。这两个scaffolds组装得比较完整,并且位置与参考序列对应。而在TRG基因密集的60 kb区间内有以下8个长度在1677~13 242 bp间的scaffolds,包括scaffold1、scaffold3、scaffold4、scaffold5、scaffold6、scaffold8、scaffold9和scaffold10等(图2和表2),其平均长度约为5.5 kb。从图2可以看出,以上8个scaffolds在参考序列上出现多处重复比对现象(两个及以上scaffolds的内部区段比对到参考序列的相同或相近位置)以及同一个scaffold分段比对到参考序列不同位置的现象(图2和表2)。例如,scaffold6的全部、scaffold9和scaffold10的左端均比对到参考序列上90 kb附近,scaffold10分3段比对到参考序列上70 kb、80 kb、90 kb左右的3个不同区段。同时,本文统计了组装scaffolds的GC含量:总 GC含量为40.12%,TRG基因位点的8个scaffolds的GC含量均高于平均值,且远高于两侧scaffold7和scaffold2的GC含量(36.77%、39.77%)(表2)。
2.4.2 scaffolds内部连接的证明
克隆174P24组装的scaffolds与鸡参考基因组的比对结果表明,scaffold1、scaffold10、scaffold9、scaffold3、scaffold2等5个scaffolds的序列分2~3段分别比对到参考序列上相近或相距约10 kb的不同位置。针对这5个scaffolds内部区段连接处的PCR结果表明,本研究组装的这5个scaffolds内部区段的顺序、连接方向和序列正确:scaffold2内部PCR证明了参考序列上gap6和gap6两侧共约296 bp的序列(克隆174P24参考序列123 628~123 924 bp)不存在;scaffold9内部PCR结果表明9A和9C比对到参考序列上两个区段之间约 154 bp(95 452 bp~ 95 606 bp)的间隔序列应该为530 bp,且9A、9B和9C 3个区段相连;scaffold1、scaffold10和scaffold3的内部分段按图2中组装的顺序和方向相连,且序列与组装结果一致。
3 讨 论
目前,多数物种TCR基因位点的序列信息不完整、各基因片段的排列位置不清楚,对人和小鼠TCR近30多年的研究才得到了基本完整的TCR基因序列和基因片段的排列位置。TCR基因位点内的基因数目众多、排列位置难以确定,且每个家族内的基因片段同源性均很高,这些都导致TCR基因位点的测序和组装难度远远高于基因组上其他区段。以人TRB基因位点为例,位于7号染色体上约620 kb的区间内集中了68个TCRβ V基因片段、2个TCRβ D基因片段、14个TCRβ J基因片段和2个TCRβ C基因片段;TCRβ V区排列在染色体的5′端,TCRβ D区、TCRβ J区和TCRβ C区以D-J-C为重复单元位于染色体的3′端,两个重复单元的J基因片段个数分别为6个和8个,整体结构比较复杂[4,18~21]。与人和小鼠相似,鸡TCR基因位点的V基因片段数目众多、彼此间同源性很高,这使得测序和组装难度也很大。自21世纪以来,鸡TRG基因的研究报道较少。目前为止,已有研究结果表明,鸡TRG基因包括L区、V区、J区和C区;在T细胞分化成熟的过程中,在特异性重组酶作用下,TRG胚系基因发生重排,由分割的、无转录活性的V区、J区和C区基因片段连接形成一个完整的、有转录功能的TRG重排基因,它编码约含300个氨基酸的多肽[6,12]。但目前由全基因组鸟枪法测序得到的鸡参考基因组中TRG基因位点的序列不完整也不完全准确,并且基因个数和排列位置等数据依然缺乏。
为了获得更加完整和准确的鸡TRG基因位点的序列,本研究利用Illumina公司的MiSeq高通量测序平台对鸡TRG链的克隆174P24进行重新测序和组装。与常规的鸟枪法测序相比,单个BAC克隆的测序和组装有以下优势:(1)BAC克隆仅插入约200 kb的目的片段,并且插入片段经指纹图谱法在染色体上进行过准确定位。与全基因组测序相比,BAC克隆测序的基因序列短,且目的性强,准确性会更高;(2)排除BAC克隆区间外染色体上同源序列的干扰,降低了组装难度。综上,BAC克隆的测序和组装对基因片段数目众多、排列位置难以确定的TCR基因位点更具有优势。
本研究对鸡TRG链克隆174P24的测序和组装分析,既获得了新的信息,同时也发现了一些问题。组装scaffolds与鸡参考基因组的比对结果显示,鸡TRG基因位点定位于克隆174P24中间约60 kb的区间内,V区、J区和C区等基因片段均以基因簇的形式分布。在这个区间内,总GC含量(42.29%)略高于两侧区域(39.13%),并且分布不均一。而Illumina测序在高GC和高AT区域产生的reads数偏少,这可能也是导致TRG基因位点区间测序reads丰度比两侧区域偏低的原因。本文运用Celera软件、ABySS软件和Phrap软件等进行过多次尝试,并把在参考基因组上定位到克隆174P24区间内的fosmid文库的两侧末端序列用作辅助定位。结果发现,运用ABySS软件拼接获得叠连群,进一步采用Phrap软件进行融合,获得的scaffolds最为完整;本研究中fosmid文库末端序列对组装的帮助并不大。
经过多次优化后,本文获得了克隆174P24的基因组草图,该草图包含10个平均长度约为18 kb的scaffolds。与参考序列的比对结果显示,本文得到的10个scaffolds较完整地覆盖了鸡TRG基因位点及旁侧区域。旁侧区域包括scaffold7和scaffold2,其位置和序列与参考序列对应。而TRG基因位点包含在8个平均长度约为5.5 kb的scaffolds里,但它们的位置和序列与参考序列并不完全对应。这种不完全对应主要指多个scaffolds比对到参考序列的相近位置及一个scaffold分段对应到参考序列的不同位置。针对这些scaffolds的内部连接处的PCR结果显示,本研究组装的scaffold1、scaffold2、scaffold3、scaffold9、scaffold10等5个scaffolds内部结构正确。从图2中可以看出,scaffold2分两段比对到参考基因组约100 kb~178 kb的位置,且两区段连接处与参考序列的 gap6(长度为 127 bp)相互对应。但该scaffold内部PCR结果表明,这个gap和其两侧共约296 bp的序列(克隆174P24参考序列123 628~ 123 924 bp间)不存在。这表明由全基因组高通量测序组装得到的参考序列并不完全准确,组装时这178 kb的区间掺入了染色体上其他区间的序列,从而出现PCR扩增时参考序列并不存在或并不完全准确的情况。而scaffold9的9A、9B和9C 3段分别比对到参考序列上约80 kb、90 kb和100 kb的3个位置,但PCR结果表明这3个区段按图2中我们组装的顺序和方向连接形成一个完整的scaffold,而且9A和9C对应参考序列上相距约154 bp(95 452~95 606 bp)的间隔处应该为530 bp,这校正和补充了参考序列上不完全准确的序列信息。同样,scaffold10分成3段比对到参考序列上约70 kb、80 kb和90 kb附近的3个区域,而且前后顺序与组装结果不同。但该scaffold内部PCR结果表明,这3个区段连接在一起,而且顺序和方向与图中scaffold10一致。另外,经过PCR证明,scaffold1和scaffold3的内部区段按图2中组装的顺序和方向相互连接在一起。这表明鸡参考基因组TRG基因位点上V基因区(70~100 kb)内的序列信息不完全准确。这种情况可能是由于重复序列的存在、基因结构的复杂性和全基因组测序导致组装时出现了偏差。以上PCR结果表明单个BAC克隆的测序和组装能得到更加准确的序列信息,可以对现有参考基因组的序列信息进行校正。此外,这10个scaffolds间的顺序和方向虽然根据参考序列进行了初步排序,但还需要进一步的PCR证明。本研究虽然多次尝试过PCR扩增,但经常出现无扩增条带、非特异性扩增、测序时无反应及非特异结合等现象,而不能获得有用的序列信息。
综上所述,本研究通过对鸡TRG基因位点BAC克隆的重新测序和组装分析,得到了鸡TRG链克隆174P24的基因组草图,并校正了鸡参考基因组的多处序列错误,为鸡TRA/D、TRB基因位点及其他物种TCR基因位点等相关方面的研究提供科研思路和理论依据,为基因组复杂区域的研究提供参考和建议。
[1]Glusman G,Rowen L,Lee I,Boysen C,Roach JC,Smit AFA,Wang K,Koop BF,Hood L.Comparative genomics of the human and mouse T cell receptor loci.Immunity, 2001,15(3):337–349.
[2]Clark SP,Arden B,Kabelitz D,Mak TW.Comparison of human and mouse T-cell receptor variable gene segment subfamilies.Immunogenetics,1995,42(6):531–540.
[3]Mackelprang R,Carlson CS,Subrahmanyan L,Livingston RJ,Eberle MA,Nickerson DA.Sequence variation in the human T-cell receptor loci.Immunol Rev,2002,190(1): 26–39.
[4]李扬秋.T细胞受体的研究和应用.北京:人民卫生出版社,2009:53–55.
[5]Chen CL,Ager LL,Gartland GL,Cooper MD. Identification of a T3/T cell receptor complex in chickens. J Exp Med,1986,164(1):375–380.
[6]Sowder JT,Chen CL,Ager LL,Chan MM,Cooper MD.A large subpopulation of avian T cells express a homologue of the mammalian T gamma/delta receptor.J Exp Med, 1988,167(2):315–322.
[7]Vainio O,Lassila O,Cihak J,Lösch U,Houssaint E. Tissue distribution and appearance in ontogeny of α/β T cell receptor(TCR2)in chicken.Cell Immunol,1990, 125(1):254–260.
[8]Tjoelker LW,Carlson LM,Lee K,Lahti J,McCormack
WT,Leiden JM,Chen CL,Cooper MD,Thompson CB. Evolutionary conservation of antigen recognition:the chicken T-cell receptor beta chain.Proc Natl Acad Sci USA,1990, 87(20):7856–7860.
[9]McCormack WT,Tjoelker LW,Stella G,Postema CE, Thompson CB. Chicken T-cell receptor beta-chain diversity:an evolutionarily conserved D beta-encoded glycine turn within the hypervariable CDR3 domain.Proc Natl Acad Sci USA,1991,88(17):7699–7703.
[10]Cihak J,Hoffmann-Fezer G,Ziegler-Heibrock HW,Stein H,Kaspers B,Chen CH,Cooper MD,Lösch U.T cells expressing the V beta 1 T-cell receptor are required for IgA production in the chicken.Proc Natl Acad Sci USA, 1991,88(23):10951–10955.
[11]Göbel TW,Chen CL,Lahti J,Kubota T,Kuo CL, Aebersold R,Hood L,Cooper MD.Identification of T-cell receptor alpha-chain genes in the chicken.Proc Natl Acad Sci USA,1994,91(3):1094–1098.
[12]Six A,Rast JP,McCormack WT,Dunon D,Courtois D,Li Y,Chen CH,Cooper MD.Characterization of avian T-cell receptor γgenes.Proc Natl Acad Sci USA,1996,93(26): 15329–15334.
[13]Kubota T,Wang JY,Göbel TWF,Hockett RD,Cooper MD, Chen CH.Characterization of an avian(Gallus gallus domesticus)TCR αδ gene locus.J Immunol,1999,163(7): 3858–3866.
[14]International Chicken Genome Sequencing Consortium. Sequence and comparative analysis of the chicken genome provide unique perspectives on vertebrate evolution. Nature,2004,432(7018):695–716.
[15]Yang X,Liu D,Liu F,Wu J,Zou J,Xiao X,Zhao FQ,Zhu BL.HTQC:a fast quality control toolkit for Illumina sequencing data.BMC Bioinformatics,2013,14:33.
[16]Simpson JT,Wong K,Jackman SD,Schein JE,Jones SJM, Birol I.ABySS:a parallel assembler for short read sequence data.Genome Res,2009,19(6):1117–1123.
[17]Bonfield JK,Staden R.The application of numerical estimates of base calling accuracy to DNA sequencing projects.Nucleic Acids Res,1995,23(8):1406–1410.
[18]TillinghastJP,BehlkeMA,Loh DY.Structureand diversity of the human T-cell receptor beta-chain variable region genes.Science,1986,233(4766):879–883.
[19]Rowen L,Koop BF,Hood L.The complete 685-kilobase DNA sequence of the human β T cell receptor locus. Science,1996,272(5269):1755–1762.
[20]Ikuta K,Ogura T,Shimizu A,Honjo T.A joining-diversityjoining complex generated by inversion mechanism and a variable-diversity complex in the β-chain gene of the human T-cell receptor.Nucl Acids Res,1986,14(12): 4899–4909.
[21]Tunnacliffe A,Kefford R,Milstein C,Forster A,Rabbitts TH.Sequence and evolution of the human T-cell antigen receptor beta-chain genes.Proc Natl Acad Sci USA,1985, 82(15):5068–5072.
(责任编委:李辉)
Re-sequencing and assembly of chicken T cell receptor gamma locus
Junping Ma1,2,Xi Yang1,2,Na Lv1,Fei Liu1,Yan Chen1,3,Baoli Zhu1
1.Institute of Microbiology,Chinese Academy of Sciences,Beijing 100101,China;
2.University of Chinese Academy of Sciences,Beijing 100049,China;
3.Institute of Animal Sciences,Chinese Academy of Agricultural Sciences,ASTIP-IAS03,Beijing 100193,China
The genomic organization of the animal T cell receptor(TCR)loci is characterized by different gene families with high homology,and it is quite difficult to obtain accurate gene sequences and arrangements of these gene families.In this study,weidentified the location ofchicken TCR gammachain (TCRγ orTRG) genes by comparing those TRG gene sequences with the chicken reference genome,and the corresponding bacterial artificial chromosome(BAC)clone,CH261-174P24,was chosen for further high-throughput DNA re-sequencing and assembly.As a result,a draft genome assembly containing ten scaffolds was obtained,which almost covered the chicken TRG gene locus and the flanking regions.Subsequently,the internal structure of these scaffolds was confirmed by PCR amplification and Sanger sequencing.Our analysis corrected two errors in the sequence—one near a TRG variable gene and one close to a gap,respectively,and several errors in the TRG variable genes in the chicken reference genome.In conclusion,our work has partially corrected the erroneously assembled sequences of the TRG gene locus in the chicken reference genome and thus provides a new method for genome sequence analysis of chicken TRA/D and TRB gene loci.
chicken;T cell receptor(TCR);gamma gene locus;re-sequencing;assembling
2014-11-04;
2015-03-09
国家重点基础研究发展规划项目(编号:2015CB554204)资助
马俊平,硕士研究生,专业方向:遗传学和基因组学。E-mail:majp12@im.ac.cn
朱宝利,博士,研究员,研究方向:微生物与免疫基因组学。E-mail:zhubaoli@im.ac.cn
陈燕,博士,助理研究员,研究方向:动物遗传育种。E-mail:chenyan0204@163.com
10.16288/j.yczz.14-382
时间:2015-3-16 10:49:31
URL:http://www.cnki.net/kcms/detail/11.1913.R.20150316.1049.001.html

