基于esp基因和JCV标记的MST方法在五大流域典型水源地中的应用
王敬琦,孔维文,刘婷婷,陈南,杨柏云,何晓青
北京林业大学生物科学与技术学院,北京 100083
基于esp基因和JCV标记的MST方法在五大流域典型水源地中的应用
王敬琦,孔维文,刘婷婷,陈南,杨柏云,何晓青
北京林业大学生物科学与技术学院,北京 100083
非点源污染目前已经成为影响水体环境的重要污染。微生物源示踪技术(microbial source tracking, MST)是解决非点源污染的一项新技术,它可以确定污染的宿主来源。肠球菌esp基因和多瘤病毒JCV可以作为检测水体中人源粪便污染的分子标记,其灵敏度和特异性都很高。为分析五大流域水源地是否受到人源粪便的污染,对辽河、海河、淮河、长江和黄河五大流域典型水源地水样进行采集和检测,选取人源粪便特异病原微生物肠球菌的esp基因和多瘤病毒JCV建立了相应的MST分子检测方法。结果表明,五大流域的典型水源地采样点均有可能受到了人源粪便的污染,可为当地相关部门提供技术支持和数据参考。
肠球菌esp基因;多瘤病毒JCV;水源地;MST
非点源污染是指溶解的或固体污染物(地面的各种污染物质如城市垃圾、农村家畜粪便,农田中的化肥、农药、重金属及其他有毒或有机物),从非特定的地点随着暴雨产生的地面径流,进入受纳水体造成的污染[1-3]。与点源污染相比,非点源污染起源于分散、多样的地区,地理边界和发生位置难以识别和确定,随机性强、成因复杂潜伏周期长,因而防治十分困难,目前已成为非常重要的影响水体环境质量的污染源。只有准确的定位污染源,才能制定有效的治理方案解决污染危机。
传统的粪便污染监测方法仅能通过微生物的数量反映水体受粪便污染的程度,却不能识别其真正来源[4]。为了解决这一问题,1980年后,以美国为代表的国家开始了对微生物源追踪技术的研究。美国弗吉尼亚大学的环境学家Hagedorn和Wiggins提出微生物源示踪技术(microbial source tracking, MST)技术。MST是一种利用微生物在环境中的生化特性、遗传多样性及其特异性代谢产物确定其宿主来源的新技术,从而将非点源污染转换为点源污染[5-8]。其基本原理是:由于摄食、体内环境等不同,可以把包括人在内的不同动物的肠道(宿主)看作不同的生态环境,生活在不同宿主环境中的微生物群落,一方面由于对不同环境胁迫因子的适应和竞争能力的差异,导致微生物种群发生变化,进而形成一个区别于其他环境的微生物群,亦即具有宿主专一性的种群;另一方面,不同宿主肠道内同种微生物则为了适应所处的不同生态环境,在功能上产生了一系列特异环境适应性,并将这些特性遗传给子孙后代,从而使这类微生物都带有标志其宿主或生存环境的特异性指纹图谱(如:特殊代谢产物、遗传学特征等)。通过分析这些宿主特异性微生物或特异性指纹图谱,就能够达到识别粪便污染源的目的。
MST方案主要由源指示物的选择、源指示物的检测和源解析3部分组成。源指示物是一些用来标示不同粪便污染源的微生物或特殊的化学物质;源指示物的检测是指使用实验室分析方法检测源指示物及其特异性指纹图谱的方法;源解析是指将检测结果同已知粪便源的特异性指纹图谱进行比较,从而确定测试污染水样中的粪便污染源的过程[9]。
肠球菌(Enterococcus)主要存在于粪便、尿液中,寄存在动物肠道内,是一种常用的检测水体中粪便污染的指示微生物。传统检测肠球菌的方法为选择性培养,该方法操作复杂、耗时长、培养基成本高[10]。由于肠球菌的毒力因子中esp基因的检出率较高[11],且其基因结构非常特殊,本文利用esp基因指示肠球菌的存在的方法具有良好的可靠性。esp基因最初是在研究一株曾引起医院内暴发流行的多重耐药粪肠球菌(MMH594)时发现的(GenBank序列号为AF034779)。后来又在同一菌株中大小为153 kb的毒力岛中发现了该基因的存在。肠球菌表面蛋白基因esp位于肠球菌染色体上,基因结构非常独特,核心区由多个碱基序列组成。其中,5'端是1~3个A片段的核苷酸序列重复;3'端是占整个esp基因31%,高度保守的3~9个C片段的序列;2个B片段序列位于A和C片段的序列之后,基因水平的同源重组决定了重复片段核苷酸序列重复出现的次数。目前,肠球菌esp基因作为粪便污染指示的工作在国外常有报道[12-14],且检测人源粪便污染的特异性达到100%。
JC病毒(JCV)属乳多空病毒科多瘤病毒种人多瘤病毒分支[15],Padgett等[16]于1971年首次分离出JC病毒,研究表明,JC病毒与人类多瘤病毒SV40(恒河猴病毒)和BK病毒基因组的大小结构及序列非常类似,没有包膜,外被衣壳蛋白,内含一环状的小双链DNA。JC病毒基因组包括大功能区上游编码区下游编码区和非编码调节区上游编码区表达大T抗原和小t抗原,下游编码区表达蛋白(主要衣壳蛋白)、VP2蛋白、VP3蛋白和调节蛋白,非编码调节区包括启动子增强子和复制起始位点。美国调查表明半数以上儿童和大多数的成年人病毒抗体呈阳性[17],且由于病毒在肾脏组织中复制并通过尿液排出[18],故一般成年人尿液中可以检测到JC病毒,所以JC病毒可以作为分子marker识别水体中人源污染。近几年,JCV作为分子标记物来检测地表水中人源污染的工作在美国、澳大利亚等国家已经展开。
本文选取的重点流域典型水源地的主要类型主要是水库、河道、地下水,多用于供给城市生活用水、市政用水、工业用水、农业用水,同时兼顾发电、养殖、旅游等,与城市居民的生活密切相关。运用基于esp基因和JCV标记的MST技术对其进行检测,确定是否受到人源粪便的污染,为监测和防治水源地的水体污染提供数据支持,帮助识别非点源污染关键源区,制定合理的治理方案。
1 材料与方法 (Materials and methods)
1.1主要的试剂与设备
病毒基因组DNA/RNA提取试剂盒,细菌基因组提取试剂盒,2×Taq Platium PCR Mastermix,北京TIANGEN;LB培养基,北京拜尔迪生物技术有限公司;Ex-TaqDNA聚合酶,大连TAKARA。
安全操作柜(ESCO Class II,新加坡);电子天平(METTER,瑞士);H203程控金属浴(GingkoBio,北京);T-512基因扩增仪(Techne,英国);电泳仪(Bio-Rad,美国);凝胶成像仪(Bio-Rad,美国);5804 R冷冻离心机(Eppendorf,德国);90 min不锈钢换膜过滤器(MUlipore,美国);MF-Millipore滤膜 (Millipore,美国);15 mL 离心式过滤器 (Millipore,美国)。
1.2实验方法
1.2.1样品采集
于2012年7月到11月期间分别在辽河流域、海河流域、淮河流域、黄河流域和长江流域5个典型水源地,用灭菌的、干净的玻璃器皿采集水样后冷藏运回实验室,24 h内处理。采样点信息如表1所示。

表1 采样点信息
1.2.2多瘤病毒JCV检测方法
1.2.2.1样品处理方法
在500 mL水样中添加5 mL浓度为250 mmol·L-1的AlCl3,水样由蠕动泵传送用过滤器过滤。随后,将200 mL浓度为0.5 mmol·L-1的H2SO4溶液(pH=3)通过过滤膜,以去除Al3+离子和其他的阳性离子。然后,用10 mL浓度为1 mmol·L-1的NaOH溶液(pH=11)洗脱病毒,滤出液中添加50 μL浓度为100 mmol·L-1的H2SO4溶液(pH=1)以及100 μL的100×Tris-EDTA缓冲液(pH=8)中和。10 mL过滤液进一步用Amicon ultra15超滤管浓缩至700 μL左右,置于-20 ℃条件下保存。
1.2.2.2水样总DNA的提取
病毒基因组DNA/RNA提取试剂盒(DP315)购自北京天根生物技术有限公司,根据试剂盒说明书进行总DNA的提取。
1.2.2.3多瘤病毒(JCV)PCR检测方法
参照文献设计引物HPyV-F、HPyV-R[19],由上海生工生物工程股份有限公司合成,扩增片断为176 bp(JCV),详见表2。
PCR反应扩增体系如下:12.5 μL 2×Taq Platium PCR Mastermix、1 μL引物HPyV-F、1 μL引物HPyV-R、5.5 μL RNase-free ddH2O、5 μL DNA模版,反应体系共25 μL。以多瘤病毒质粒作为阳性对照,以RNase-free ddH2O作为空白对照。反应条件如下:94 ℃起始变性3 min后,94 ℃变性30 s,55 ℃复性30 s,72 ℃延伸50 s,并循环30次,最后72 ℃延伸5 min,反应结束后将扩增产物于4 ℃条件下保存备用。
取10 μL PCR产物用1.5%琼脂糖凝胶电泳进行检测,用Bio-Rad公司凝胶成像仪观察并拍照。
1.2.3屎肠球菌检测方法
1.2.3.1样品处理方法
取100 mL水样过膜(孔径0.45 μm,直径50 mm),将膜贴至mEI培养基上,41 ℃培养48 h。将膜转移至10 mL TSB培养液中,41 ℃温育3 h。
1.2.3.2DNA的提取
按天根细菌基因组DNA extract kit试剂盒说明书提取。
1.2.3.3屎肠球菌esp基因检测方法
参照文献设计引物esp2-F和esp2-R[20-22],由北京奥科公司合成,扩增片断为407 bp,详见表2。
PCR反应体系如下:5 μL 10×Ex-Taq buffer、1 μL 10 mmol·L-1dNTP、1 μL引物esp2-F、1 μL引物esp2-R、0.25 μL Ex-Taq DNA聚合酶、32.75 μL RNase-free ddH2O,反应体系共50 μL。以阳性菌作为阳性对照,以RNase-free ddH2O作为空白对照。反应条件如下:95 ℃起始变性15 min后,94 ℃变性30 s,52.0 ℃复性30 s,72 ℃延伸1 min,并循环30次,最后72 ℃延伸7 min,反应结束后保存在4 ℃条件下备用。
取10 μL PCR产物用1.5%琼脂糖凝胶电泳进行检测,用Bio-Rad公司凝胶成像仪观察并拍照。
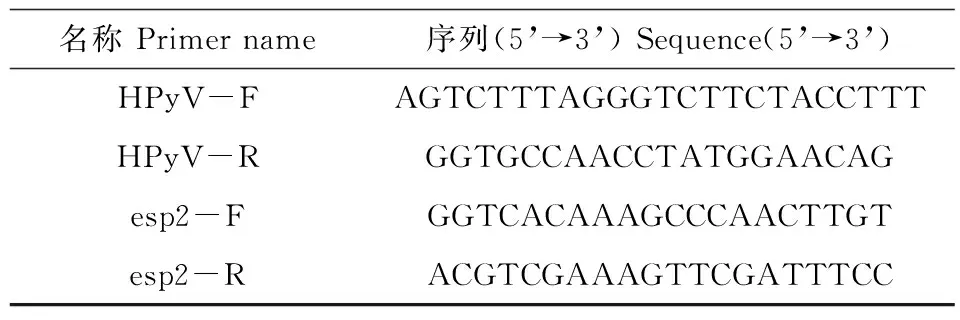
表2 引物序列
2 结果(Results)
五大流域典型水源地的指示人源粪便污染的病毒与细菌分子标记结果汇总如下(表3),“+”表示阳性结果,“-”表示阴性结果;各流域具体检测结果详述于如下文。
2.1辽河流域5个典型水源地检测结果
在辽河流域典型水源地采样点中多瘤病毒检测

图1 辽河流域典型水源地水中多瘤病毒(左)和屎肠球菌(右)检测结果注:M:Marker Ⅰ,至上到下条带依次为600 bp,500 bp,400 bp,300 bp,200 bp,100 bp;CK:空白对照;S:标准品;1:QH水库;2:GYG水库;3:HR水库;4:QH水库;5:DHF水库。Fig. 1 Detection of JCV and esp gene in Liaohe River basinNote: M: Marker I, 600 bp, 500 bp, 400 bp, 300 bp, 200 bp, 100 bp from top to bottom; CK: Control check; S: Salvicine; 1: QH reservoir; 2: GYG reservoir; 3: HR reservoir; 4: QH reservoir; 5: DHF reservoir.
5个点均呈阳性结果,肠球菌esp基因检测中采样点HR水库、QH水库、DHF水库呈阳性结果,表明这5个点有可能受到了人源粪便的污染,HR水库、QH水库和DHF水库受到人源粪便污染的可能性较大(图1)。
2.2海河流域4个典型水源地检测结果
海河流域典型水源地采样点中多瘤病毒检测4个点均呈阳性结果,肠球菌esp基因检测中4个点均呈阳性结果,所以这4个点受到人源粪便污染的可能性较大(图2)。
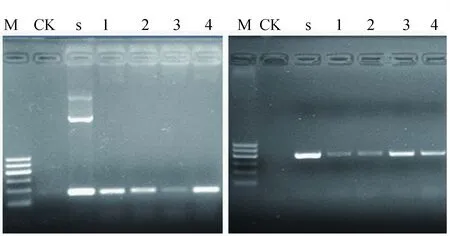
图2 海河流域典型水源地水中多瘤病毒(左)和屎肠球菌(右)检测结果注:M:Marker Ι,至上到下条带依次为600 bp,500 bp,400 bp,300 bp,200 bp,100 bp;CK:空白对照;S:标准品;1:DHT水库;2:PJK水库;3:DWS水库;4:YQ水库。Fig. 2 Detection of JCV and esp gene in Haihe River basinNote: M: Marker I, 600 bp, 500 bp, 400 bp, 300 bp, 200 bp, 100 bp from top to bottom; CK: Control check; S: Salvicine; 1: DHT reservoir; 2: PJK reservoir; 3: DWS reservoir; 4: YQ reservoir.
2.3淮河流域5个典型水源地检测结果
在淮河流域典型水源地采样点中多瘤病毒检测5个点均呈阴性结果,但是肠球菌esp基因检测中5个采样点均呈阳性结果,这说明5个采样点也有可能受到了人源粪便的污染(图3)。
2.4黄河流域5个典型水源地检测结果
在黄河流域典型水源地采样点中多瘤病毒检测采样点DK和采样点WJZ水库呈阳性结果,肠球菌esp基因检测中5个点均呈阳性结果,表明这5个点有可能受到了人源粪便污染的污染,其中DK和WJZ水库受到污染的可能性较大(图4)。
2.5长江流域5个典型水源地检测结果
在长江流域典型水源地采样点中多瘤病毒检测5个点均呈阴性结果,但是肠球菌esp基因检测中5个点均呈阳性结果,这说明5个采样点也有可能受到了人源粪便的污染(图5)。

表3 各流域病毒与细菌检测结果汇总
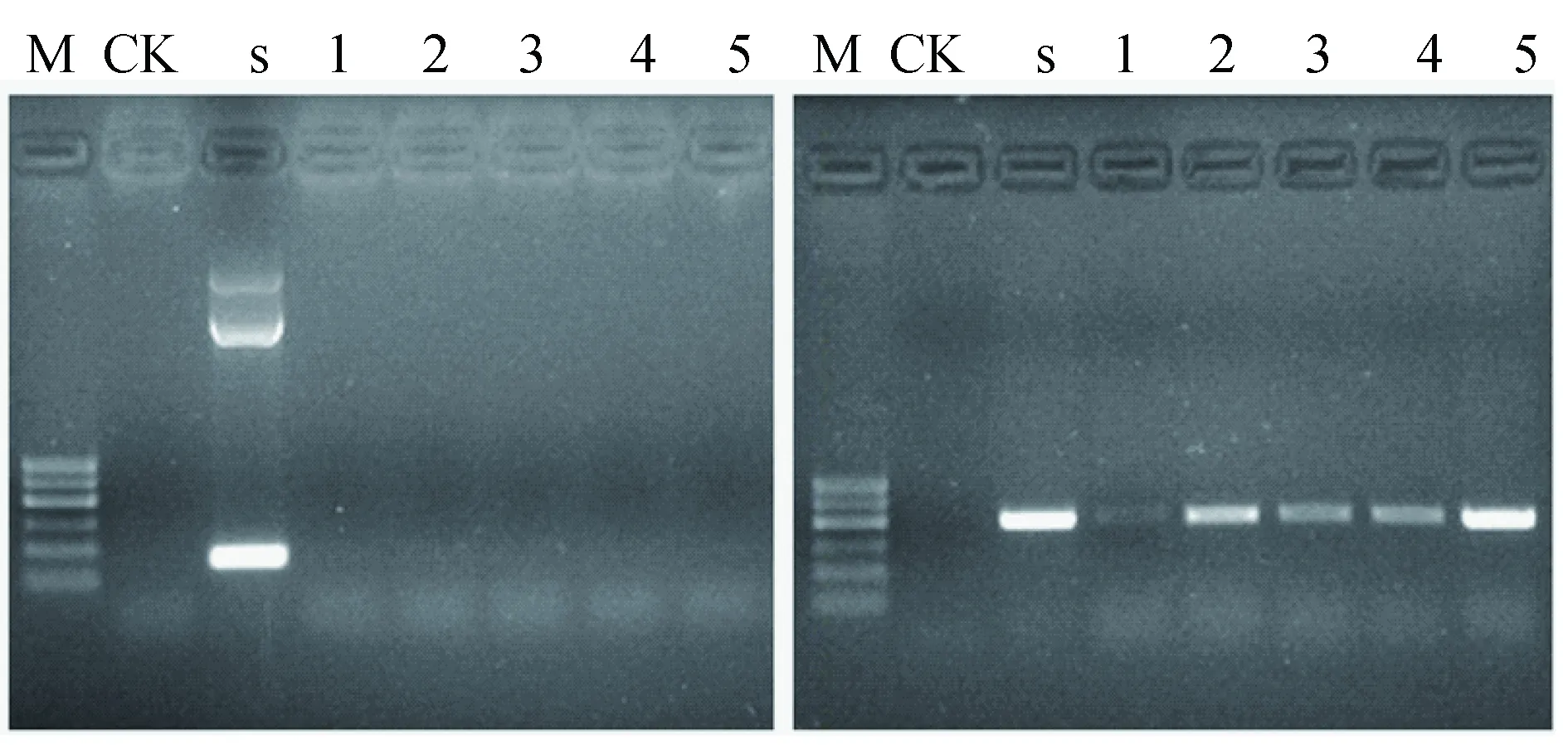
图3 淮河流域典型水源地水中多瘤病毒(左)和屎肠球菌(右)检测结果注:M:Marker Ι,至上到下条带依次为600 bp,500 bp,400 bp,300 bp,200 bp,100 bp;CK:空白对照;S:标准品;1:BGS水库;2:HH水源地;3:HZ水厂;4:DX水源地;5:BBZS水源地。Fig. 3 Detection of JCV and esp gene in Huaihe River basinNote: M: Marker I, 600 bp, 500 bp, 400 bp, 300 bp, 200 bp, 100 bp from top to bottom; CK: Control check; S: Salvicine; 1: BGS reservoir; 2: HH water source; 3: HZ water works; 4: DX water source; 5: BBZS water source.

图4 黄河流域典型水源地水中多瘤病毒(左)和屎肠球菌(右)检测结果注:M:Marker Ι,至上到下条带依次为600 bp,500 bp,400 bp,300 bp,200 bp,100 bp;CK:空白对照;S:标准品;1:DK;2:XCQ;3:HYK;4:WJZ水库;5:LJ水库。Fig. 4 Detection of JCV and esp gene in Yellow River basin Note: M: Marker I, 600 bp, 500 bp, 400 bp, 300 bp, 200 bp, 100 bp from top to bottom; CK: Control check; S: Salvicine; 1: DK; 2: XCQ; 3: HYK; 4: WJZ reservoir; 5: LJ reservoir.

图5 长江流域典型水源地水中多瘤病毒(左)和屎肠球菌(右)检测结果注:M:Marker Ι,至上到下条带依次为600 bp,500 bp,400 bp,300 bp,200 bp,100 bp;CK:空白对照;S:标准品;1:TK;2:S水厂;3:THX;4:WJ;5:WLM。Fig. 5 Detection of JCV and esp gene in typical water sources of Yangtze River basinNote: M: Marker I, 600 bp, 500 bp, 400 bp, 300 bp, 200 bp, 100 bp from top to bottom; CK: Control check; S: Salvicine; 1: TK; 2: S water works; 3: THX; 4: WJ; 5: WLM.
3 讨论(Discussion)
本研究以肠球菌和多瘤病毒作为MST中的指示微生物,选定肠球菌的esp基因和多瘤病毒基因为标记基因,通过对标记基因的检测和鉴定追踪选定微生物,最终达到了识别水体中人源粪便污染的目的。肠球菌的esp基因和多瘤病毒作为指示人源粪便污染的2种微生物指示物分子标记在国外已经被应用于水体的检测,文献报道它们的特异性和灵敏度都是100%。从结果可以看出,对某些采样点结果不是完全一致的,即2种标记不是同时呈阳性结果或阴性结果。如果某采样点受到了人源粪便的污染,理论上肠球菌和多瘤病毒JCV是都存在于该采样点水体中的,即对应标记基因是全部可以检测到的。这种阴阳结果不同的原因可能是,没有检测到的某种微生物在水体中的浓度低或短时间内的变化,抑或用来检测的水样由于受到取样点选择和风力等自然因素等原因,从而导致没有检测到。由此可见,为了更准确的追踪污染源,选用不同类型的微生物,甚至是选用不同的方法同时对水源地进行检测是很有必要的。到目前为止,任何一种方法都不能适用于各种环境条件下的粪便示踪,需要选取合适的方法,甚至多种不同的方法相结合,实现优缺互补将是MST的发展趋势[23]。本文建立的检测方法,操作简便、特异性高。根据本文结果可以判断,此方法在我国水源检测中具有应用可行性。
利用微生物源追踪分析,一方面可以确定非点源污染的来源,以采取针对性强的治理措施;另一方面可以为此类污染负荷控制提供有力的技术支持。美国、澳大利亚和欧盟部分国家已经开展了一些MST技术的相关工作。美国多条河流或海岸线都已采用该方法成功解决污染源头的问题,如在美国的维多利亚2处海滩Anderson和Hilton地区,其污染物主要来源于人,并针对此研究结果采取了行之有效的整治措施[24]。该方法已经被美国环境保护署(US EPA)列入水体监测的常规手段之一。我国目前主要通过检测细菌总数和粪大肠菌群值以及“三氮”(氨氮、亚硝酸盐氮和硝酸盐氮)含量对水体进行相关监测和评价,水中粪大肠菌群值及氨氮增高时,表示水体新近可能有粪便污染。但是这些方法都只能对水体污染做出卫生学评价,却无法查找水体中粪便污染来源。对于非点源污染源生物追踪技术研究,国内仍然是处于起步阶段。2005年,长江水资源保护科学研究所通过技术引进和合作,并在丹江口库区进行了MST技术应用试点。李伟伟等[25]应用建立的rep-PCR微生物源追踪方法区分水体中指示菌的不同来源,水体标本监测显示了桂五水库粪便污染来源种类多,其中人、鸡和牛相对较多,研究结果提示应对桂五水库地区加强人、家禽和家畜动物粪便的无害化管理。根据本研究的结果,建议五大水源地周边的居民区应该加大厕所改造力度,增进粪便无害化处理,并且对居民普及相关的水源卫生与健康知识。
MST是一个新兴的监测领域,尚处于不断探索发现和验证阶段,在未来的环境检测中,MST的目标绝不仅是定性,它的最终目标是定量。在定量检测中也建议同时用多种分子标记,这样做的好处是可以计算出不同宿主来源类便污染的相对值,比如人和某种动物。未来可以利用这项技术做成芯片[26],处理结果的方式可能往往是利用模型[27-28]。总之,MST技术还有很大的探索空间,该技术的不断完善必然会给水体检测带来更大的便利,给监测水体、控制污染源及制定相关环保条例提供更多可以利用的科学依据。
致谢:感谢中国科学院生态环境研究中心水生态毒理组和公共仪器组对本研究的支持。
通讯作者简介:何晓青(1977-),女,博士,副教授,从事水环境中微生物学的研究。
[1]Line D E, McLaughlin R A, Osmond D L, et al. Nonpoint Sources [M]. Water Environment Research, 1998: 895-912
[2]Parry R. Agricultural phosphorus and water quality: A US environmental protection agency perspective [J]. Journal of Environmental Quality, 1998, 27(2): 258-261
[3]贺缠生, 傅伯杰, 陈利顶. 非点源污染的管理与控制[J]. 环境科学, 1998, 19(5): 87-91
He C S, Fu B J, Chen L D. Non-point source pollution control and management [J]. Environmental Science, 1998, 19(5): 87-91 (in Chinese)
[4]Meays C L, Broersma K, Nordin R, et al. Source tracking fecal bacteria in water: A critical review of current methods [J]. Journal of Environmental Management, 2004, 73(1): 71-79
[5]Ruecker N J, Braithwaite S L, Topp E, et al. Tracking host sources of Cryptosporidium spp. in raw water for improved health risk assessment [J]. Applied and Environmental Microbiology, 2007, 73(12): 3945-3957
[6]Jiang S C, Chu W, Olson B H, et al. Microbial source tracking in a small southern California urban watershed indicates wild animals and growth as the source of fecal bacteria [J]. Applied Microbiology and Biotechnology, 2007, 76(4): 927-934
[7]Anderson M A, Whitlock J E, Harwood V J. Diversity and distribution of Escherichia coli genotypes and antibiotic resistance phenotypes in feces of humans, cattle, and horses [J]. Applied and Environmental Microbiology, 2006, 72(11): 6914-6922
[8]Whitlock J E, Jones D T, Harwood V J. Identification of the sources of fecal coliforms in an urban watershed using antibiotic resistance analysis [J]. Water Research, 2002, 36(17): 4273-4282
[9]Seurinck S, Verstraete W, Siciliano S D. Microbial source tracking for identification of fecal pollution [J]. Reviews in Environmental Science and Bio/Technology, 2005, 4(1-2): 19-37
[10]US EPA. Method 1600: Enterococci in water by membrane filtration using membrane Enterococcus Indoxyl-3-D-Glucoside Agar (mEI) [R]. Washington DC: US EPA, 2002
[11]吴敏. 粪肠球菌和屎肠球菌毒力基因, PA1相关基因及耐药性分析[D]. 广州: 南方医科大学, 2008
Wu M. Presence of virulence genes, PAI-associated genes and antibiotic resistance in E. faecalis and E. faecium [D]. Guangzhou: Southern Medical University, 2008 (in Chinese)
[12]Ahmed W, Powell D, Goonetilleke A, et al. Detection and source identification of fecal pollution in non-sewered catchment by means of host-specific molecular markers [J]. Water Science and Technology, 2008, 58(3): 579-586
[13]Abdelzaher A M, Wright M E, Ortega C, et al. Presence of pathogens and indicator microbes at a non-point source subtropical recreational marine beach [J]. Applied and Environmental Microbiology, 2010, 76(3): 724-732
[14]Kim S Y, Lee J E, Lee S, et al. Characterization of Enterococcus spp. from human and animal feces using 16S rRNA sequences, the esp gene, and PFGE for microbial source tracking in Korea [J]. Environmental Science and Technology, 2010, 44(9): 3423-3428
[15]Imperiale M J. The human polyomaviruses, BKV and JCV: Molecular pathogenesis of acute disease and potential role in cancer [J]. Virology, 2000, 267(1): 1-7
[16]Padgett B L, Zurhein G M, Walker D L, et al. Cultivation of papova-like virus from human brain with progressive multifocal leucoencephalopathy [J]. The Lancet, 1971, 297(7712): 1257-1260
[17]Major E O, Amemiya K, Tornatore C S, et al. Pathogenesis and molecular biology of progressive multifocal leukoencephalopathy, the JC virus-induced demyelinating disease of the human brain [J]. Clinical Microbiology Reviews, 1992, 5(1): 49-73
[18]Khalili K, Gordon J, White M K. The Polyomavirus, JCV, and its Involvement in Human Disease [M]// Polyomaviruses and Human Diseases. New York: Springer, 2006: 274-287
[19]Hammerum A M, Jensen L B. Prevalence of esp, encoding the enterococcal surface protein, in Enterococcus faecalis and Enterococcus faecium isolates from hospital patients, poultry, and pigs in Denmark [J]. Journal of Clinical Microbiology, 2002, 40(11): 4396-4396
[20]Allard A, Albinsson B O, Wadell G. Rapid typing of human adenoviruses by a general PCR combined with restriction endonuclease analysis [J]. Journal of Clinical Microbiology, 2001, 39(2): 498-505
[21]Harwood V J, Delahoya N C, Ulrich R M, et al. Molecular confirmation of Enterococcus faecalis and E. faecium from clinical, faecal and environmental sources [J]. Letters in Applied Microbiology, 2004, 38(6): 476-482
[22]Ahmed W, Stewart J, Powell D, et al. Evaluation of thehost-specificity and prevalence of enterococci surface protein marker in sewage and its application for sourcing human fecal pollution [J]. Journal of Environmental Quality, 2008, 37(4): 1583-1588
[23]段传人, 刘爱喜, 王贵学, 等. 应用肠道微生物示踪地表水粪便污染的研究进展[J]. 微生物学报, 2013, 40(12): 2319-2329
Duan C R, Liu A X, Wang G X, et al. Research progress of using intestinal microbes to track the sources of fecal pollution in surface water [J]. Microbiology China, 2013, 40(12): 2319-2329 (in Chinese)
[24]Dickerson J W, Hagedorn C, Hassall A. Detection and re-mediation of human-origin pollution at two public beaches in Virginia using multiple source tracking methods [J]. Water Research, 2007, 41(16): 3758-3770
[25]李伟伟, 陈晓东, 顾玲, 等. 应用rep-PCR微生物源方法追踪夏秋季桂五水库粪便污染来源[J]. 江苏预防医学, 2010, 21(3): 7-9
Li W W, Chen X D, Gu L, et al. Application of rep-PCR microbial source tracking to identify fecal source in Guiwu reservoir in the seasons of summer and fall [J]. Jiangsu Journal of Preventive Medicine, 2010, 21(3): 7-9 (in Chinese)
[26]Stapleton C M, Kay D, Wyer M D, et al. Evaluating the operational utility of a bacteroidales quantitative PCR-based MST approach in determining the source of faecal indicator organisms at a UK bathing water [J]. Water Research, 2009, 43(19): 4888-4899
[27]Roslev P, Bukh A S, Iversen L, et al. Application of mussels as bio samplers for characterization of fecal pollution in coastal recreational waters [J]. Water Science and Technology, 2010, 62(3): 586-593
[28]Soule M, Kuhn E, Loge F, et al. Using DNA microarrays to identify library-independent markers for bacterial source tracking [J]. Applied and Environmental Microbiology, 2006, 72(3): 1843-1851
◆
Detection ofEnterococcusespGene and Polyomavirus JCV and Its Application in Typical Water Sources of Five River Basins
Wang Jingqi, Kong Weiwen, Liu Tingting, Chen Nan, Yang Boyun, He Xiaoqing*
College of Biological Sciences and Technology, Beijing Forestry University, Beijing 100083, China
9 March 2015accepted 1 June 2015
Currently, non-point source pollution has become the major problem of the water environment. Microbial source tracking (MST) was a novel technique in solving non-point source pollution, which can determine the source of pollution. To study the effect of human fecal contamination in main water source, we selected specific pathogen Enterococcus esp gene and polyomavirus JCV as molecular markers to establish the molecular tracking methods. The markers exhibited high specificity and sensitivity. Five river basins including Liaohe River Basin, Haihe River Basin, Huaihe River Basin, Yangtze Basin and Yellow River Basin were analyzed by the methods. Our results suggested that all of the five typical water sources might have been polluted by human feces contamination. These data can provide important information and technical support for further research.
Enterococcus esp gene; polyomavirus JCV; water sources; MST
中央高校基本科研业务费专项资金资助(TD2012-03);水利部行业公益性行业科研专项(201201032);国家自然科学基金青年科学基金项目(51108029)
王敬琦(1990-),女,硕士研究生,研究方向为水环境中病原微生物,E-mail: wangjingqi90@126.com
Corresponding author), E-mail: lenahe@bjfu.edu.cn
10.7524/AJE.1673-5897.20150309013
2015-03-09录用日期:2015-06-01
1673-5897(2015)6-246-07
X171.5
A
王敬琦, 孔维文, 刘婷婷, 等. 基于esp基因和JCV标记的MST方法在五大流域典型水源地中的应用[J]. 生态毒理学报,2015, 10(6): 246-252
Wang J Q, Kong W W, Liu T T, et al. Detection of Enterococcus esp gene and polyomavirus JCV and its application in typical water sources of five river basins [J]. Asian Journal of Ecotoxicology, 2015, 10(6): 246-252 (in Chinese)

