洛伐他汀生物合成途径中酰基转移酶LovD的活性机制研究
于靖,曹绪芬,王佳旺,廉铮,赵晨
(1.河北省沧州市中心医院 心血管内一科,河北 沧州 061000;2.河北省人民医院 妇科,河北 石家庄 310014)
洛伐他汀生物合成途径中酰基转移酶LovD的活性机制研究
于靖1,曹绪芬1,王佳旺1,廉铮1,赵晨2
(1.河北省沧州市中心医院 心血管内一科,河北 沧州 061000;2.河北省人民医院 妇科,河北 石家庄 310014)
目的 将突变前后的蛋白进行分子动力学模拟,从而探讨酰基转移酶LovD活性提高的原因。方法 对酰基转移酶的平面结构进行拟合,通过底物通道NVR2.1计算软件得到LovD的主要底物,在酶活性测定过程中计算均方根偏差(root mean square deviation,RMSD)和势能、G0和G5的均方根波动(root mean square fluctuation,RMSF)数值、主要α螺旋区域I与通道周围其他螺旋区域的距离等指标。结果 RMSD和势能的计算结果表明,叠合后的蛋白结构组成并没有发生较大的变换;G0和G5的RMSD数值比较表明,G0和G5整体的运动没有发生较大的变化;G0和G5的RMSF数值比较表明,4个局部柔性峰值微弱变化区域和1个柔性变化较大的区域;通过底物通道计算软件得到LovD主要的4条底物通道,α螺旋区域I是形成通道的核心螺旋区域;比较G0和G5的主要通道的宽度、长度、弯曲率等数值,突变后G5的主要通道变宽、变短、弯曲率变小;G5的距离变大。结论 远距离的突变使得LovD活性提高是由于α螺旋I末端的柔性变化,导致α螺旋I的运动的变化,使得在α螺旋I附近底物通道变短、变宽、弯曲率减小。这些变化可能降低了底物进出主要通道的阻力,使得参与酶催化的底物在通过这些通道的时候的速率加快,从而使得催化效率提高。
酰基转移酶;LovD;洛伐他汀;酶活性;动力学模拟
自然界中他汀类化合物有洛伐他汀和康帕克汀,洛伐他汀主要由真菌土曲霉(aspergillus terreus)模式菌株产生,而康帕克汀由橘青霉(penicillium citrinum)菌株产生。在洛伐他汀的生物合成途径中,大部分的合成编码基因都比较清楚,而基因LovD 编码2-mehtylbutyryl/MJA(monacolin J acid,形成洛伐他汀的前体化合物)的转移酶催化最后一步,使得从LovF 过来的酰基部分与加载到MJA 上C8的位置,最后生物合成为洛伐他汀[1]。在这一过程中,LovD 和LovF 的酰基载体区域(acyl carrier protein,ACP)的蛋白-蛋白的相互作用使得这一最后一步反应在土曲霉中高效进行[2]。传统的他汀类化合物生产工艺是以洛伐他汀为原料,经化学合成得到。随着现代生物学的发展,洛伐他汀的生物合成受到越来越多的重视,酰基转移酶LovD是洛伐他汀的生物合成中一个重要酶,所以研究酰基转移酶LovD活性对提高洛伐他汀的生物合成具有重要的意义,底物通道中G0和G5是影响活性的重要通道。本研究将突变前后的蛋白进行分子动力学模拟,从而探讨酰基转移酶LovD活性提高的原因。
1 材料与方法
1.1 材料 LovD突变前后的酰基转移酶,购买于深圳市博谷网络科技有限公司,批号为ZL110619。
1.2 方法 对酰基转移酶的平面结构进行拟合,通过底物通道NVR2.1计算软件得到LovD主要的底物,在酶活性测定过程中计算RMSD和势能、G0和G5的RMSF数值(G0和G5通道中输入蛋白质结构的数目为a、b 是通道的平均半径[Å]、c是通道的最大位置处的半径[Å]、d为 通道的平均长度[Å]、e为通道平均弯曲率)、主要α螺旋区域I与通道周围其他螺旋区域的距离等指标。
1.3 洛伐他汀的生物合成途径,见图1[3]。
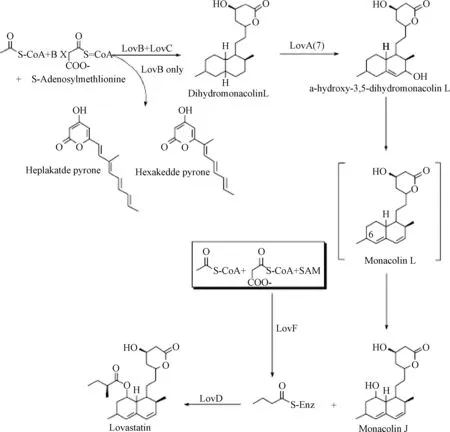
图1 洛伐他汀的生物合成途径Fig.1 Biosynthesis of lovastatin
2 结果
2.1 酰基转移酶LovD 分子动力学计算 在蛋白质I末端区域柔性发生了明显的变化:通过比较G0和G5的α原子均方根波动(root mean square fluctuation,RMSF)发现氨基酸区域的柔性变化位置并且柔性变化位置的氨基酸结构变化性较大。为了更加清楚RMSF变化区域所在的位置,将LovD主要α螺旋区域和β 折叠区域分别用大写英文字母A~N标记显示,如图2,每一段区域分别用一个字母所代替[3]。
在β折叠区域K(1)、loop区域K-B(2),β折叠区域L(3),G0 的波动稍大于G5,在β折叠区域M(4),G5的波动稍稍大于G0。而在α螺旋区域I 末端(5)有明显的波动变化,并且G5 的波动小于G0 的波动。6个突变的位点并不在有波动的区域,引起这样的结果可能是突变后,氨基酸残基的之间的相互作用发生变化。见图3。
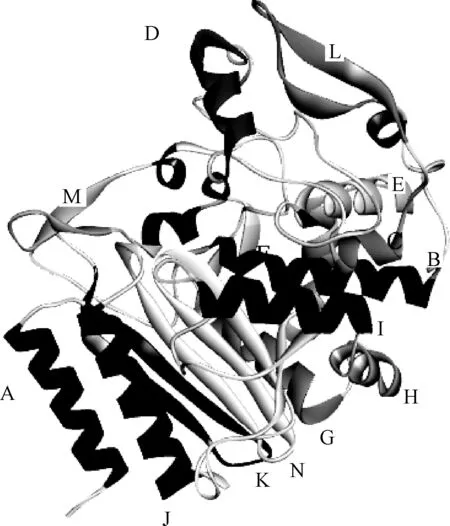
图2 LovD 主要α螺旋区域和β折叠区域的标记Fig.2 Main α-helix domain and LovD regional andβ folded tag
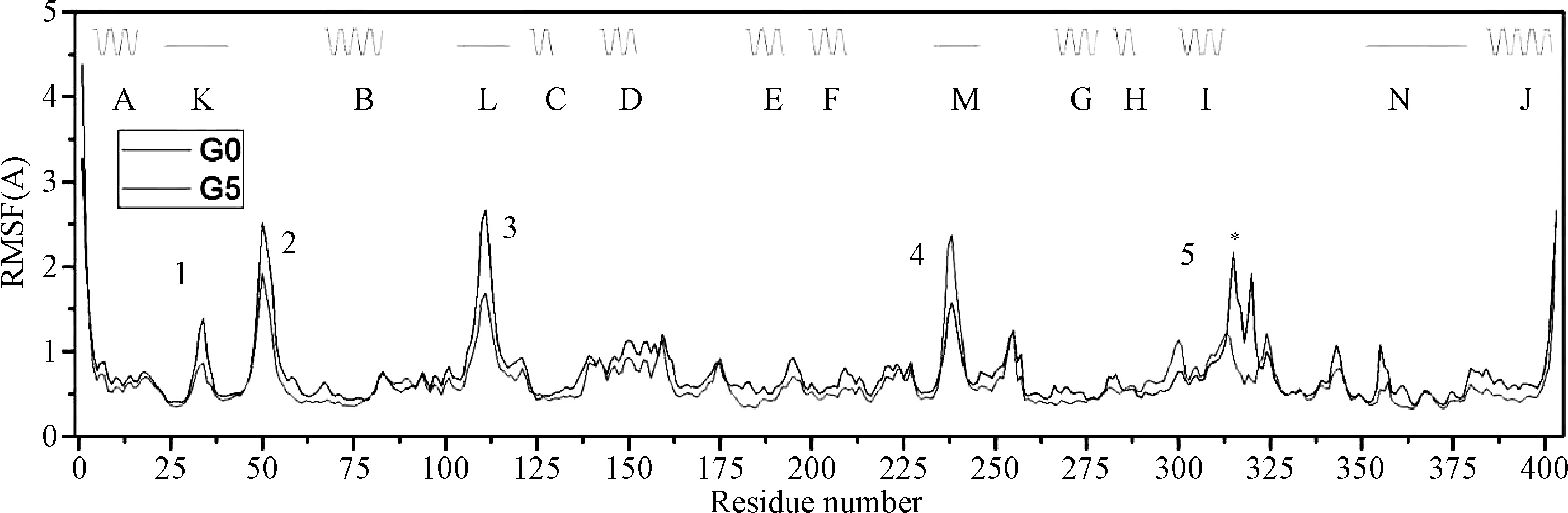
图3 G0和G5的α碳原子均方根波动(上线:G0;下线:G5)Fig.3 RMS fluctuation of alpha carbon atoms in G0 and G5
通过以上结构的分析和比较,发现这6个位点的突变并没有明显改变整体的蛋白结构,但是,G5 的活性口袋张开幅度减小,即活性口袋变小,口袋变小可能导致底物与酶的结合能力发生变化,可能是影响酶催化活性变化的一个原因,本研究认为这不是主要的原因。因此,将分析的重点放在在蛋白质I区域末端,可以发现G5的均方根波动(RMSF)明显降低,认为这个区域对LovD的酶催化活性有着很重要的影响,G5的活性提高与这个区域的氨基酸运动有很大关系。
G5 的柔性减小的区域I末端处在活性中心附近:通过前面的计算和分析发现,区域I末端对LovD有至关重要的影响,可能是引起催化活性变化的主要氨基酸位置,因此,为了更好地研究蛋白质区域I末端部分的氨基酸与底物的相互关系,本研究对原始晶体结构的底物蛋白相互作用进行分析。在α 螺旋区域I末端是一段loop区域,并且这段区域靠近活性中心,图3显示位于活性中心的底物MJA靠近柔性变化I末端区域。位于活性中心区域的氨基酸由于直接与底物发生相互作用,将直接影响酶催化的活性。
2.2 对LovD的底物通道的分析 LovD 突变后的主要通道的变化:为了进一步研究G0和G5的通道的特性,通过分析和比较G0和突变后G5主要通道的平均半径、最大半径、长度、弯曲率。表1显示,G0和G5的通道,除了3c,其他3个主要通道的平均半径(Avg_BR)与最大半径(Max_BR)增大了,在表格中箭头标示增大趋势,表明小分子底物进入反应活性中心的阻力将减小;主要通道的长度(Avg_L)也变短,并且通道的弯曲率(Avg_C)也减小,在表格中箭头标示下降趋势,表明长度越短小分子底物将在通道中的时间缩短,而弯曲率的降低表明小分子底物进入活性中心的阻力也将降低。
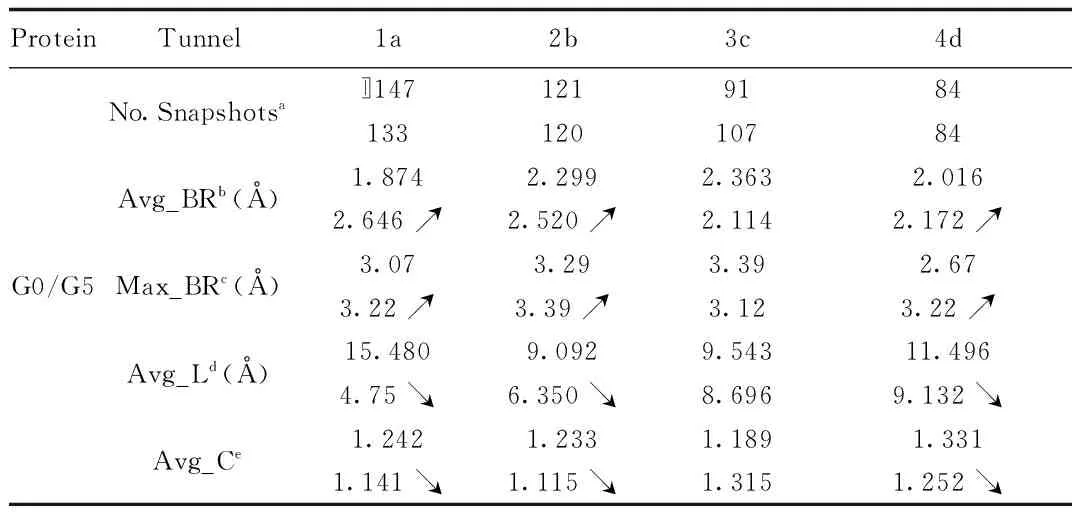
表1 G0 和G5 的通道特性的比较Tab.1 Comparison of the characteristics of G0 and G5 channel
注:a大于等于0.9Å通道的输入蛋白质结构的数目、b通道的平均半径[Å]、c通道的最大位置处的半径[Å]、d通道的平均长度[Å]、e通道平均弯曲率
从上面的分析可以看出,突变后的LovD(G5)主要通道变宽且变长,并且弯曲率也有所降低,由于这些通道特征值的改变,使得小分子在通过通道进入酶活性中心时与通道间的作用发生改变,小分子进入活性中心的阻力变小,小分子进入活性中心更加容易,这可能是G5产量提高的原因,而G0和G5的通道3c并没有发生上述变化,很有可能是与其他3个底物通道相区别的底物通道,即通过3c通道的底物与通过通道1a、2b、4d的底物是不同的。因此,提出假设,对于LovD本研究认为底物MJA是首先进入到活性中心的,然后参与反应。因此,在排除其他因素的条件下,通过计算出来并根据通道理论得出的结论是,影响反应速率的关键是DMB-SMMP进入到活性中心的速率。
LovD主要是α螺旋区域的运动导致了通道的变化:为了更进一步分析主要通道变化的过程,分别计算了G0和G5的主要α螺旋区I和B、I和J域随时间的距离变化,G0和G5I-B的距离的变化。G0的α螺旋区I和B的随时间的变化与G5有微弱差别,但是,α螺旋区域I和J随时间的变化有较大的差别,G5的α螺旋区I和J的距离比G0距离较大,而α螺旋区I和J是形成通道2b的主要螺旋区域,α螺旋区I和B是形成通道1a和4d(2条通道被一段loop区域隔开)的主要螺旋区域。因此,α螺旋区I、B和J区域是通道产生变化的直接原因。
3 讨论
本研究结合蛋白质的通道理论进行分子动力学模拟来解释酰基转移酶LovD突变引起酶催化效率的改变。通常酶底物通过蛋白质的表面或者内部的通道进入活性心,对酶催化效率也起着至关重要的作用[4-7]。通过以上计算和分析,本研究对一个全新的突变引起催化效率改变的原因并作出了合理的解释和阐明。首先,远离酶活性中心的氨基酸突变导致酶活性变化的解释本身是一个难点,其次,这样的突变引起的变化也是一个复杂的变化[8-10]。本文通过动力学的方法结合蛋白质通道分析,发现这样的突变结果并没有引起酰基转移酶LovD蛋白质结构和组成的变化,也并没有引起蛋白质整体运动的变化[11],但是,找到了远离活性中心的多位点的氨基酸突变导致活性变化的一个原因:突变后的LovD的某一局部柔性发生较大的变化[12],导致靠近这一区域的α螺旋区域的运动变化,使得以蛋白质α螺旋区域形成的主要通道的长度变短、平均半径变大、弯曲率降低等[13-16]。因此提出假设:这些通道特性的变化,降低了底物进出主要通道的阻力,使得参与酰基转移酶催化的底物在通过这些通道时的速率加快,使得催化效率提高。这一计算的研究发现,为解释酶催化效率的提高提供了新的可能性,也将为酶催化设计提供可靠的指导依据。。
[1] Organization WH.World health statistics 2012[J].World Health Organization,2012.
[2] Hubert HB,Feinleib M,McNamara PM,et al.Obesity as an independent risk factor for cardiovascular disease:a 26-year follow-up of participants in the Framingham Heart Study[J].Circulation,1983,67(5):968-977.
[3] John LS, Karine A, Jonathan K, et al.Transformations of cyclic nonaketides by Aspergillus terreus mutants blocked for lovastatin biosynthesis at the IovA and IovC genes[J].Ore Blomol Chem, 2003,23(1):50-59.
[4] Pan W,Pintar T,Anton J,et al.Statins are associated with a reduced incidence of perioperative mortality after coronary artery bypass graft surgery[J].Circulation,2004,110(11 Suppl 1):45-49.
[5] Moosmann B,Behl C.Selenoprotein synthesis and side-effects of statins[J].The Lancet,2004,363(9412):892-894.
[6] Calderon RM,Cubeddu LX,Goldberg RB,et al.In Statins in the treatment of dyslipidemia in the presence of elevated liver aminotransferase levels:a therapeutic dilemma[J].Mayo Clinic Proceedings,2010,85(4):349-356.
[7] 李为民,孔一慧.他汀类药物治疗慢性心力衰竭的利弊探讨[J].中华心血管病杂志,2006,34(9):774-775.
[8] Manzoni M,Rollini M.Biosynthesis and biotechnological production of statins by filamentous fungi and application of these cholesterol-lowering drugs[J].Appl Microbiol Biotechnol 2002,58(5):555-564.
[9] Xie X,Pashkov I,Gao X,et al.Rational improvement of simvastatin synthase solubility in Escherichia coli leads to higher whole-cell biocatalytic activity[J].Biotechnol Bioeng,2009,102(1):20-28.
[10] Gao X,Xie X,Pashkov I,et al.Directed evolution and structural characterization of a simvastatin synthase[J].Chem Biol 2009,16(10):1064-1074.
[11] Xie X,Wong WW,Tang Y.Improving simvastatin bioconversion in Escherichia coli by deletion of bioH[J].Metab Eng 2007,9(4):379-386.
[12] Valera HR,Gomes J,Lakshmi S,et al.Lovastatin production by solid state fermentation using Aspergillus flavipes[J].Enzyme and Microbial Technology,2005,37(5):521-526.
[13] Loncaric C,Merriweather E,Walker KD.Profiling a Taxol pathway 10beta-acetyltransferase:assessment of the specificity and the production of baccatin III by in vivo acetylation in Ecoli[J].Chem Biol 2006,13(3):309-317.
[14] Wagner UG,Petersen EI,Schwab H,et al.EstB from Burkholderia gladioli:A novel esterase with a β‐lactamase fold reveals steric factors to discriminate between esterolytic and β‐lactam cleaving activity[J].Protein science,2002,11(3):467-478.
[15] Xie X,Meehan MJ,Xu W,et al.Acyltransferase mediated polyketide release from a fungal megasynthase[J].J Am Chem Soc,2009,131(24):8388-8389.
[16] Xie X,Tang Y.Efficient synthesis of simvastatin by use of whole-cell biocatalysis[J].Appl Environ Microbiol,2007,73(7):2054-2060.
(编校:王冬梅)
Catalytic activity mechanism study of acyltransferase LovD in biosynthesis of lovastatin
YU Jing1,CAO XU-fen1,WANG Jia-wang1,LIAN Zheng1,ZHAO Chen2
(1.Cardiovascular Internal Medicine, Cangzhou Central Hospital, Cangzhou 061000, China; 2.Department of Gynaecology, Hebei General Hospital, Shijiazhuang 310014,China)
ObjectiveTo explore the reasons for the increase of acyltransferase activity of LovD by molecular dynamics simulation of before and after mutation protein.MethodsThe planar structure of acylase were fitted, the main substrate LovD through the substrate channel NVR2.1 calculation, calculation of RMS deviations(root mean square deviation, RMSD) and potential energy in the determination of enzyme activity, root mean square fluctuation (RMSF) value of G0 and G5, mainlyα helical region I and channel around other helical region distance index.ResultsThe results showed that the calculation of RMSD and potential energy, consisting of protein structure after superposition which there were no great transformation; G0 and G5 RMSD numerical comparison showed that the whole movement of G0 and G5 had not changed; G0 and G5 RMSF numerical comparison showed that the 4 local flexible peak weak change in area and 1 flexible change in the larger region; LovD 4 major channels were calculated by substrate channel calculation software, αhelical region I was the core of helical region formed channel; comparison of G0 and G5 values of the main channel of width, length, bending rate, the main channel of the G5 mutation become wider, shorter, bending rate become smaller; G5 distance enlarged.ConclusionConclusion Distant mutation which increases the LovD activity is due to the flexible change α helical I end, lead to changes in motion αhelical I, makes the α helix I near the substrate channel becomes short and wide, bending rate decreasing. These changes may reduce main channel resistance in and out the substrate, that participate in enzyme catalyzed substrate to accelerate rate through these channels, thereby improving catalytic efficiency.
acyltransferase; LovD; lovastatin; enzyme activity; dynamics simulation
浙江卫生厅(2012KYB010)
于靖,男,硕士,主治医师,研究方向:心血管内科方向,E-mail:yj12114@163.com。建议方法和结果也按照这个顺序写。
O64
A
1005-1678(2015)02-0096-04

