高效PiggyBac转座酶的构建与筛选
张 龙,梁明福,张智英
(西北农林科技大学 a 动物科技学院,b 生命科学学院,陕西 杨凌 712100)
高效PiggyBac转座酶的构建与筛选
张 龙a,梁明福b,张智英a
(西北农林科技大学 a 动物科技学院,b 生命科学学院,陕西 杨凌 712100)
【目的】 构建能够定点转座的高效PiggyBac转座酶系统。【方法】 以野生型PiggyBac转座酶为模板,通过易错PCR方法随机突变转座酶基因,构建转座酶突变库,并将其与能够靶向识别并结合DNA序列的锌指蛋白(ZFP)融合表达,构建ZFP-PiggyBac转座酶突变库系统。将转座酶载体突变库、转座子载体和报告载体共转到酿酒酵母JMY1中,通过Ura-5FOA酵母筛选系统筛选高效定点作用的转座酶。【结果】 成功构建ZFP-PiggyBac转座酶突变库系统,经过3轮系统筛选获得5个高效作用的突变转座酶。【结论】 新构建的ZFP-PiggyBac转座酶突变库系统具有在锌指蛋白ZFP靶向识别域Rosa26BS位点处进行高效转座的能力。
5FOA;转座酶;锌指蛋白
PiggyBac转座子最初由Fraser等[1]从甘蓝蠖度尺蛾基因组中获得,为Ⅱ型转座子,现在已经在转基因小鼠生产[2]、小鼠胚胎干细胞遗传学操作[3-5]、基因诱变等基因操作[6]、多能干细胞诱导[7-8]等领域得到应用。PiggyBac转座子转座时特异性地插入TTAA位点[9],由于其来源于高等生物昆虫,因而较其他转座子具有更高的活性[10],没有潜在的病毒遗传毒性,并且可改造性较强;PiggyBac转座子携带的外源基因片段较长,最长可以携带150 kb的目的片段[11];借助转座酶,PiggyBac转座子可以将目的基因从宿主精确切割敲除,且不会使宿主染色体发生重排。这些优势使得PiggyBac转座子在转基因操作中获得了广泛的应用。但是,由于PiggyBac转座子在转座时不易控制,易产生随机整合且转座效率较低,从而限制了PiggyBac转座子的进一步应用。
锌指蛋白(ZFP)存在于多种真核生物中,用于识别靶位点的DNA结合域,其能够以序列特异性的方式与DNA结合[12-13],通过其α螺旋特异性地插入到DNA双螺旋大沟并与DNA结合[14],对基因调控起重要作用。由于PiggyBac转座子本身不会自发转座,其作用机制是由PiggyBac转座酶的切割作用而完成转座[15],因此通过提高转座酶活性可以提高转座效率[16-17],解决其转座效率低及不易受控的缺点。本研究通过对野生型PiggyBac转座酶进行随机突变,同时在转座酶N端融合表达能够特异性识别DNA序列的ZFP,以期获得能够特异性识别靶位点的高效转座酶,为进一步提高外源基因在转基因操作中的位点特异性和安全性提供可能。
1 材料与方法
1.1 材 料
1.1.1 菌种与质粒 载体构建所需质粒JMB84-Ura、JMB405、JMB440、pst1374-trans、pst1374-ZFP、JMB440-ZFP-WTtrans、JMB1022-3TR-5TR及大肠杆菌DH5α感受态细胞、酿酒酵母菌株JMY1,均由西北农林科技大学动物科技学院基因组学实验室保存。
1.1.2 主要试剂 限制性内切酶、T4 DNA连接酶,购自NEB公司;pfu高保真酶、easyTaqDNA 聚合酶、dATP、dTTP、dGTP和dCTP,购自北京全式金生物技术有限公司;胶回收试剂盒,购自威格拉斯生物技术(北京)有限公司;质粒提取试剂盒、酵母提取试剂盒,购自Omega公司;核酸共沉剂,购自大连宝生物工程有限公司;5FOA,由宝鑫生物有限公司代购;其他试剂均为国产分析纯或更高级别试剂。引物由上海博尚生物技术公司合成。序列测定由南京金斯瑞生物科技有限公司完成。
1.2 转座子载体JMB405-3TR-5TR的构建
根据载体JMB1022-3TR-5TR上转座子序列,利用primer 5.0软件设计引物,上游引物3TR-F为:5′-GCGAGATCTTTAACCCTAGAAAGATA-ATCA-3′,下游引物5TR-R为:5′-GCGCTCGAG-TTAACCCTAGAAAGATAGTCT-3′ ,3TR-F和5TR-R中下划线部分分别为BglⅡ和XhoⅠ酶切位点。以JMB1022-3TR-5TR质粒为模板,采用降落式PCR扩增转座子序列,反应体系为:模板2.0 μL,引物3TR-F和5TR-R各0.5 μL,dNTPs 4.0 μL,10×Buffer 5.0 μL,easyTaqDNA聚合酶0.5 μL,ddH2O 37.5 μL。反应程序为:94 ℃预变性5 min;94 ℃反应30 s,68 ℃反应30 s,72 ℃反应30 s,16个循环,每个循环退火温度减低1 ℃;94 ℃反应30 s,52 ℃反应30 s,72 ℃反应30 s,25个循环;72 ℃延伸10 min。将PCR产物和JMB405载体分别经BamHⅠ和XhoⅠ双酶切并纯化回收后,用T4 DNA连接酶将其骨架与两端带有BglⅡ和XhoⅠ酶切位点的双链DNA片段于16 ℃连接过夜后,转化至大肠杆菌DH5α感受态细胞,涂布于含100 mg/L氨苄青霉素(Amp)的LB固体培养基上,37 ℃培养过夜,挑取单克隆,接种于2 mL含100 mg/L Amp的LB培养液中,37 ℃、250 r/min 培养10 h,提取质粒,经XhoⅠ和SmaⅠ双酶切鉴定,酶切产物用1%琼脂糖凝胶进行电泳检测,将构建正确的转座子载体命名为JMB405-3TR-5TR。
1.3 报告载体JMB84-Rosa26BS-Ura 的构建
Rosa26BS为锌指蛋白(ZFP)特异性识别位点,其碱基序列为:5′-GCAACTCCAGTCTTTCTTGGGCGGGAGTC-3′,根据该序列利用primer 5.0软件分别设计包含Rosa26BS锌指蛋白特异性识别位点的上游引物JMB84-F1、JMB84-F2和下游引物JMB84-R,JMB84-F1序列为:5′-GCAACTCCAGT-CTTTCTTGGGCGGGAGTCATGTCGAAAGCT-ACATATAAG-3′,JMB84-F2序列为:5′-AACCT-GCAGGAAACGAAGATAAATCATGTGCAAC-TCCAGTCTTTCTTGG-3′,JMB84-R序列为:5′- AGTGGATCCGAATTCGATATCAAGCTTATC-3′,JMB84-F2和JMB84-R序列中的下划线部分分别为PstⅠ和BamHⅠ酶切位点。通过2步PCR扩增Rosa26BS位点,以JMB84-Ura为模板,JMB84-F1、JMB84-R为引物进行第1步PCR扩增,PCR反应体系为:模板2.0 μL,引物各0.5 μL,dNTPs 4.0 μL,10× Buffer 5.0 μL,easyTaqDNA聚合酶0.5 μL,ddH2O 37.5 μL。PCR程序为:94 ℃预变性5 min;94 ℃反应30 s,68 ℃反应30 s,72 ℃反应2 min,16个循环,每个循环退火温度减低1 ℃;94 ℃反应30 s,52 ℃反应30 s,72 ℃反应2 min,25个循环;72 ℃延伸10 min。再以第1步PCR产物为模板,JMB84-F2、JMB84-R为引物进行第2步PCR扩增,反应体系和程序同第1步PCR。分别将第2步PCR产物和JMB84-Ura载体用PstⅠ和BamHⅠ进行酶切并连接、转化得到JMB84-Rosa26BS-Ura报告载体,使用PstⅠ和BamHⅠ双酶切JMB84-Rosa26BS-Ura报告载体对其进行鉴定,酶切产物用1%琼脂糖凝胶进行电泳检测。
1.4 JMB440-ZFP-trans 转座酶载体突变库的构建
1.4.1 JMB440-ZFP载体的构建 于37 ℃下用XbaⅠ和BamHⅠ双酶切Pst1374-ZFP和JMB440载体,酶切时间3 h,将得到的ZFP序列连接到骨架载体JMB440中,获得JMB440-ZFP载体,使用XbaⅠ和BamHⅠ双酶切JMB440-ZFP载体进行鉴定, 酶切产物用1%琼脂糖凝胶进行电泳检测。
1.4.2 易错PCR方法对突变转座酶的扩增 为增加转座酶突变库的多样性,通过改变PCR反应体系中Mg2+、Mn2+和dNTPs的浓度,使用易错PCR方法随机突变野生型PiggyBac转座酶基因序列,以期获得有活性的高效转座酶。根据野生型PiggyBac转座酶基因序列,利用primer 5.0软件设计引物PB-transF和PB-transR,PB-transF序列为:5′-GGTGGATCCATGGGTAGTTCTTTAG-3′,PB-transR序列为:5′-GCGCTCGAGGTCAGAAACAACTTTGGCAC-3′,引物PB-transF和 PB-transR下划线部分分别为BamHⅠ和XhoⅠ酶切位点。易错PCR反应体系如表1所示,PCR反应程序同1.2。用1%琼脂糖凝胶对PCR产物进行电泳检测。

表1 易错PCR反应体系的组成Table 1 Composition of error-prone PCR reaction system μL
注:A、B、C和D 4组中dATP、dTTP、dGTP和dCTP的体积分别为4,1,2,3;3,4,1,2;2,3,4,1;1,2,3,4 μL。
Note:The volumes of dATP,dTTP,dGTP and dCTP in A,B,C and D respectively are 4,1,2,3;3,4,1,2;2,3,4,1;1,2,3,4 μL,respectively.
用1%琼脂糖凝胶电泳对14个PCR反应产物进行检测,回收能够扩增出目的条带的PCR产物,用限制性内切酶XhoⅠ和BamHⅠ对胶回收后的PCR产物和JMB440-ZFP载体进行双酶切。根据PCR产物(trans)和JMB440-ZFP的浓度,设计不同的连接体系(表2),通过化学转化方法寻找最佳连接体系后(转化后对照组培养板中没有单克隆菌落或较少,而试验组培养板中单克隆菌落较多的连接体系为最佳连接体系),依据最佳连接体系进行大量连接。
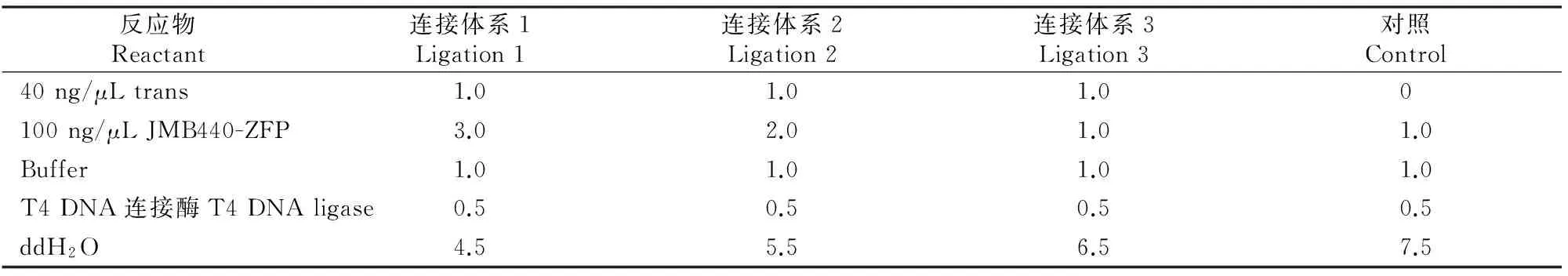
表2 基于化学转化方法寻找最佳连接体系的组成Table 2 Best ligation system composition by chemical transformation μL
1.4.3 连接产物的纯化 采用核酸共沉淀法纯化连接产物,去除盐离子,纯化方法如下:取200 μL连接产物,于65 ℃下灭活20 min;向连接体系中加4 μL DNAmate、1/10体积(20 μL)的CH3COONa和2.5倍体积的预冷无水乙醇;4 ℃、12 000 r/min 离心15 min;加500 μL 预冷的体积分数70%乙醇漂洗2遍,12 000 r/min 离心5 min;干燥,加入30 μL去离子水溶解。
1.4.4 纯化连接产物的电转化及突变库的获得 将纯化的连接产物电转入大肠杆菌DH5α感受态细胞中(电转化参数为2 500 V,200 Ω,25 μF(Bio-Rad Gene Pulser)),将电转化菌液涂布于含Amp (100 mg/L)的LB平板上,于次日将平板上长出的全部单克隆用含Amp (100 mg/L)的LB培养液冲洗干净,150 r/min、37 ℃摇菌4 h,提取质粒,得到JMB440-ZFP-trans转座酶突变库,使用XhoⅠ和BamHⅠ双酶切鉴定转座酶突变库,酶切产物用1%琼脂糖凝胶进行电泳检测。
1.5 酵母转化与高效转座酶的筛选
1.5.1 酵母转化 根据Gietz等[18]报道的LiAc酵母转化方法,将JMB440-ZFP-trans转座酶突变库(带Leu标签)、JMB405-3TR-5TR(带Trp标签)和JMB84-Rosa26BS-Ura(带Ura标签)以不同组合方式转化到酿酒酵母JMY1中,利用Ura-5FOA酵母筛选系统[19-20]进行转座酶筛选。
1.5.2 高效转座酶的筛选 由于Rosa26BS序列位于Ura基因上游,ZFP蛋白可以特异性地识别并结合在此位置,当ZFP与Rosa26BS序列结合后,与ZFP融合表达的PiggyBac转座酶的切割范围被限定于Rosa26BS位点附近的TTAA位点处。当PiggyBac转座子插入并阻断Ura基因的表达,Ura基因不表达时不会产生酶促反应,在含有0.5 mg/mL 5FOA培养基中不会发生毒性反应,酵母可以生长。当PiggyBac转座子未特异性地插入Ura基因序列中的TTAA位点时,Ura基因不会被阻断而表达,则会发生酶促反应,在含有0.5 mg/mL 5FOA培养基中会发生毒性反应,导致酵母死亡。
(1)第1次筛选。将JMB84-Rosa26BS-Ura载体作为阳性对照组,JMB84-Rosa26BS-Ura/JMB405-3TR-5TR作为阴性对照组,JMB84-Ro-sa26BS-Ura/JMB405-3TR-5TR/JMB440-ZFP-trans作为试验组,分别在含不同质量浓度(0,0.2,0.5,0.7 mg/mL) 5FOA培养基中进行筛选。筛选体系如表3所示。

表3 高效PiggyBac转座酶的第1次筛选体系Table 3 First round screening of hyperactive PiggyBac transposases
(2)第2次筛选。 根据第1次筛选结果,从筛选平板1-10、1-11中分别挑取10个克隆,分别标记为10-1~10-10和11-1~11-10,在SD(-Leu-Trp-Ura)酵母培养基中扩大培养至OD600=1,使用Omega公司酵母提取试剂盒提取酵母质粒。将提取的酵母质粒转化大肠杆菌感受态细胞DH5α,在含Amp (100 mg/L)抗性的LB固体平板上筛选。每个平板挑取10个阳性克隆,提取质粒,用XhoⅠ和BamHⅠ双酶切筛选JMB440-ZFP-trans载体,获得19个阳性质粒(10-3-5、11-8-6、10-1-8、10-2-7、10-4-3、10-5-6、10-6-5、10-7-7、10-8-2、10-9-2、10-10-8、11-1-8、11-2-1、11-3-5、11-10-4、11-5-1、11-6-4、11-7-8、11-9-2)。
分别将第1轮筛选出的19个JMB440-ZFP-trans转座酶载体与JMB405-3TR-5TR和JMB84-Rosa26BS-Ura共同转化到酿酒酵母JMY1中,在含 0.5 mg/mL 5FOA的培养基中筛选,转化组合如表4所示。

表4 高效PiggyBac转座酶的第2次筛选体系Table 4 Second round screening of hyperactive PiggyBac transposases

续表4 Continued table 4
(3)第3次筛选。根据第2次筛选结果,将筛选出最高效的11个JMB440-ZFP-trans转座酶载体(10-3-5、11-8-6、10-1-8、10-9-2、11-1-8、11-2-1、11-3-5、11-10-4、11-6-4、11-7-8和11-9-2)和JMB440-ZFP-WTtrans野生型转座酶载体进行第3次筛选,试验条件同第2次筛选。试验同时将JMB440-ZFP-WTtrans野生型转座酶载体作为对照。
2 结果与分析
2.1 转座子载体JMB405-3TR-5TR的鉴定
将构建的JMB405-3TR-5TR载体用XhoⅠ和SmaⅠ(该酶切位点位于JMB405载体上的BamHⅠ酶切位点之后)进行双酶切,结果获得了578 bp的条带(图1),与预期结果一致,表明JMB405-3TR-5TR载体构建成功。
2.2 报告载体JMB84-Rosa26BS-Ura 的鉴定
用PstⅠ和BamHⅠ双酶切报告载体JMB84-Rosa26BS-Ura,结果如图2所示。由图2可以看出,获得长度为 1 850 bp的条带,与预期的结果一致,表明报告载体JMB84-Rosa26BS-Ura构建成功。

图1 转座子载体JMB405-3TR-5TR的XhoⅠ和SmaⅠ双酶切鉴定M.2 000 Plus DNA Marker;1~3.酶切产物Fig.1 Double digestion of the recombinant vector JMB405-3TR-5TR with XhoⅠ and SmaⅠ M.2 000 Plus DNA Marker;1-3.Double digestion products
2.3 JMB440-ZFP-trans 转座酶载体突变库的构建
2.3.1 JMB440-ZFP载体的鉴定 将构建的JMB440-ZFP用XbaⅠ和BamHⅠ双酶切,结果获得了548 bp的条带(图3),与预期结果一致,表明JMB440-ZFP载体构建成功。

图3 载体JMB440-ZFP的XbaⅠ和BamHⅠ双酶切鉴定M.2 000 Plus DNA Marker;1.双酶切产物Fig.3 Double digestion of JMB440-ZFP with XbaⅠ and BamHⅠM.2 000 Plus DNA Marker;1. Double digestion products
2.3.2 易错PCR反应产物的电泳检测 由图4可知,在体系 1~6 和10~14中获得了1 700 bp的片段,结果与预期结果一致;而体系7~9中无PCR产物,其原因是体系中Mn2+浓度太高,导致PCR反应无法进行。
2.3.3 JMB440-ZFP-trans转座酶载体突变库的鉴定 根据1.4.2中寻找的最佳连接体系2进行大量连接,构建得到的JMB440-ZFP-trans转座酶载体突变库,用XhoⅠ和BamHⅠ双酶切,结果获得了1 792 bp的条带(图5),与预期结果一致,表明JMB440-ZFP-trans转座酶载体突变库构建成功。
2.4 高效PiggyBac转座酶的筛选
2.4.1 第1次筛选 由图6可知,阳性对照组中1-1号培养板上有单克隆菌落生长;而1-2和1-3号培养板上没有单克隆菌落生长,表明当5FOA质量浓度为0.2~0.5 mg/mL 时,Ura基因的表达杀死了酵母。阴性对照中1-4号培养板上有单克隆菌落存活,而1-5~1-7号培养板上无单克隆菌落存活,表明在仅含有转座子载体时无法完成转座,则转座子不能阻断Ura基因的表达,从而在5FOA质量浓度仅为0.2 mg/mL的培养板上Ura基因表达时即可杀死酵母。试验组培养板上均有单克隆菌落生长,表明在含有转座酶载体存在时,转座子成功阻断了Ura基因的表达,导致5FOA质量浓度为0.2~0.7 mg/mL时酵母仍可生长。酵母存活数量越多,表明转座酶将转座子成功转座到Ura基因序列的效率越高。
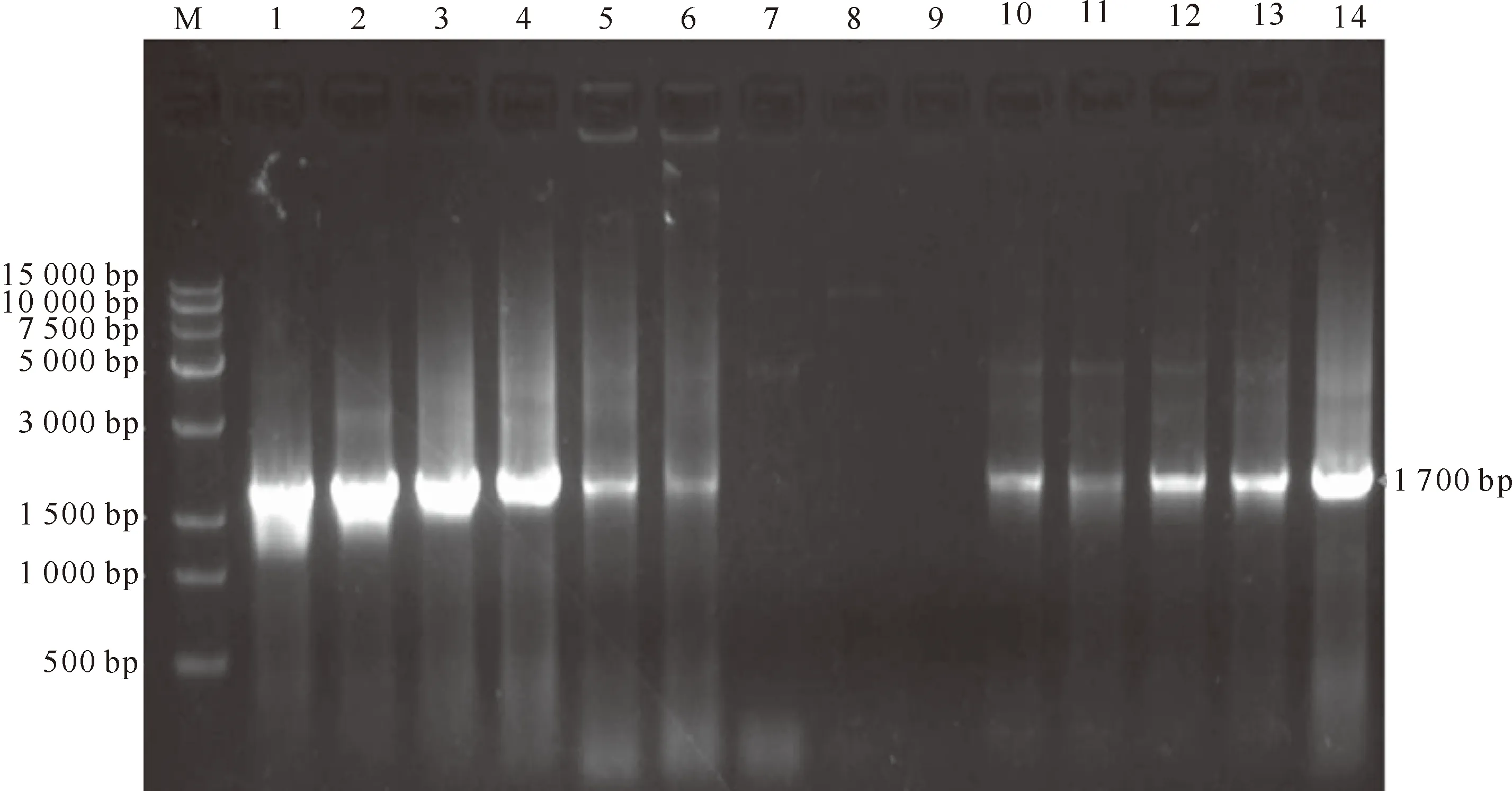
图4 易错PCR反应产物的检测
M.15 000 DNA Marker;1~14分别为表1中1~14号体系的易错PCR反应产物
Fig.4 Detection of error-prone PCR product
M.15 000 DNA Marker;1-14.Error-prone PCR products have the same numbers of 1-14 as Table 1
2.4.2 第2次筛选 由图7-A可知, 2-1、2-2、2-3、2-10、2-12、2-13、2-14、2-15、2-17、2-18和2-19号试验培养板中酵母菌落长势较好,表明这11组对应的转座酶载体(10-3-5、11-8-6、10-1-8、10-9-2、11-1-8、11-2-1、11-3-5、11-10-4、11-6-4、11-7-8和11-9-2)成功地将转座子转座到Ura基因中,并阻断了Ura基因的表达;2-4、2-5、2-6、2-7、2-8、2-9、2-11和2-16号培养板中酵母长势较差,表明其对应试验中的报告载体(10-2-7、10-4-3、10-5-6、10-6-5、10-7-7、10-8-2、10-10-8和11-5-1)没有或者仅有少量的Ura基因被阻断表达,转座酶活性较弱。
2.4.3 第3次筛选 由图7-B可知,10-9-2、11-1-8、11-10-4、11-7-8和11-9-2号培养板对应的单克隆菌落长势较好,表明其对应的转座酶具有较高的转座活性,阻断了Ura基因的表达。野生型对照组(WT)没有单克隆菌落生长,表明野生型转座酶在5FOA质量浓度为0.5 mg/mL时没有阻断Ura基因的表达。10-3-5、11-8-6、10-1-8、11-2-1、11-3-5和11-6-4号培养板中的酵母菌落长势较差,表明其对应的转座酶在转座过程中阻断Ura基因表达的效率较野生型转座酶(WT)效率高,但较10-9-2、11-1-8、11-10-4、11-7-8和11-9-2号培养板中的转座酶转座效率低。因此,经过3次0.5 mg/mL 5FOA筛选得到的10-9-2、11-1-8、11-10-4、11-7-8和11-9-2号培养板中5个转座酶即为高效转座酶。

图5 JMB440-ZFP-trans转座酶突变库的XhoⅠ和BamHⅠ双酶切鉴定M.2 000 Plus DNA Marker;1.转座酶突变库的双切酶切产物Fig.5 Double digestion of JMB440-ZFP-trans library with XhoⅠ and BamHⅠ M.2 000 Plus DNA Marker;1.Double digestion products

图6 高效PiggyBac转座酶的第1次筛选1-1~1-11与表3中的试验序号一致;1-1~1-3为阳性对照组;1-4~1-7为阴性对照组;1-8~1-11为试验组Fig.6 First round screening of hyperactive PiggyBac transposases 1-1-1-11 are consistent with Table 3;1-1-1-3 are positive controls;1-4-1-7 are negative controls;1-8-1-11 are experimental groups
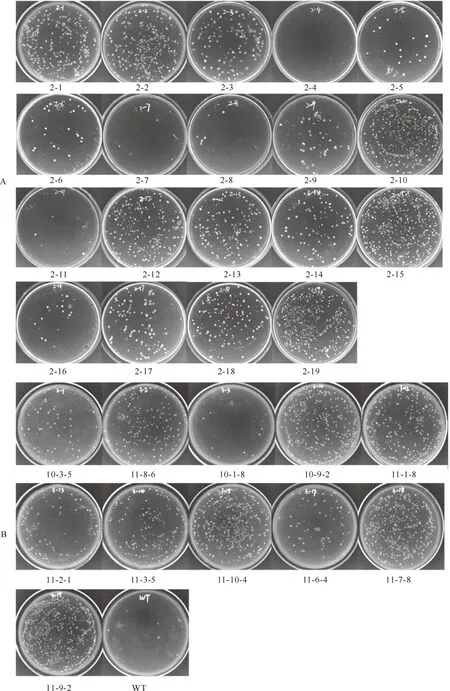
图7 高效PiggyBac转座酶的第2次(A)和第3次(B)筛选2-1~2-19与表4中的试验序号一致;10-3-5、11-8-6、10-1-8、10-9-2、11-1-8、11-2-1、11-3-5、11-10-4、11-6-4、11-7-8和11-9-2为载体编号;WT为JMB440-ZFP-WTtrans野生型转座酶载体Fig.7 Second round (A) and the third round (B) screening of hyperactive PiggyBac transposases 2-1-2-19 are consistent with Table 4;10-3-5,11-8-6,10-1-8,10-9-2,11-1-8,11-2-1,11-3-5,11-10-4,11-6-4,11-7-8 and 11-9-2 are the numbers of vectors;WT represents JMB440-ZFP-WTtrans wild type transposeses vector
3 讨 论
PiggyBac转座子属于离解型转座子,该转座子本身不能编码自己的转座酶,PiggyBac转座系统包括转座酶载体和转座子供体载体。转座子供体载体在转座酶载体协助下可携带外源基因在基因组中进行插入和转座,其转座效率依赖于转座酶的切割效率。因此,通过对PiggyBac转座系统中转座酶切割效率的优化,可以作为提高该系统转座效率的突破点。本研究通过易错PCR方法随机突变转座酶基因,构建转座酶突变库,获得高效的突变转座酶。为了提高转座子的定点识别能力,有效地克服其随机转座不易控制的缺点,本试验将突变的高效转座酶与ZFP具有靶向特异性识别的位点Rosa26BS融合表达,构建JMB440-ZFP-trans转座酶载体突变库。
本研究将JMB405-3TR-5TR转座子载体、JMB84-Rosa26BS-Ura报告载体、JMB440-ZFP-trans转座酶载体突变库共转化到酿酒酵母JMY1中,通过Ura-5FOA系统进行转座酶筛选。将ZFP与具有特异性转座能力的PiggyBac转座酶进行融合表达,具有位点特异性识别功能的ZFP靶向结合于Ura基因上游的Rosa26BS位点处,从而有效地控制了PiggyBac转座酶的切割范围。基于融合表达蛋白ZFP的限制,PiggyBac转座酶只切割ZFP识别位点Rosa26BS序列附近的TTAA位点,并阻断Ura基因的表达。Ura基因不表达,酿酒酵母JMY1可以在含有5FOA筛选培养基中生长。经过3轮有效筛选,笔者发现经过突变后的转座酶较野生型转座酶表现出更高的转座效率,并最终获得5个高效的能够定点转座的ZFP-PiggyBac转座酶。
高效ZFP-PiggyBac转座酶的建立可以实现对靶基因位点附近基因的定点敲除或者目的基因的定点引入。将筛选出的高效PiggyBac转座酶系统与根据特殊目的设计的转座子载体配合,形成高效的PiggyBac转座子系统,进一步提高外源基因在转基因过程中的整合效率,且能实现基因的可控切离,提高了转基因技术在转基因动物生产中的安全性。将高效PiggyBac转座酶与特异性识别DNA序列的锌指蛋白(ZFP)融合,构建ZFP-PiggyBac转座酶系统,在ZFP识别位点附近的TTAA处实现定点插入目的基因。通过与不同的ZFP进行融合,可以根据科研工作者的不同需求获得多种ZFP-PiggyBac转座酶系统,使得在动物基因组中感兴趣的位点插入目的基因成为可能。
[1] Fraser M,Smith G,Summers M.Acquisition of host cell DNA sequences by baculoviruses:Relationship between host DNA insertions and FP mutants of autographa californica and galleria mellonella nuclear polyhedrosis viruses [J].Journal of Virology,1983,47:287-300.
[2] Ding S,Wu X,Li G,et al.Efficient transposition of the PiggyBac (PB) transposon in mammalian cells and mice [J].Cell,2005,122:473-83.
[3] Wang W,Lin C,Lu D,et al.Chromosomal transposition of PiggyBac in mouse embryonic stem cells [J].Proceedings of the National Academy of Sciences of the United States of America,2008,105:9290-9295.
[4] Wang W,Bradley A,Huang Y.A PiggyBac transposon-based genome-wide library of insertionally mutated Blm-deficient murine ES cells [J].Genome Research,2009,19:667-673.
[5] Nakazawa Y,Huye L,Salsman V,et al.PiggyBac-mediated can-cer immunotherapy using EBV-specific cytotoxic T-cells expressing HER2-specific chimeric antigen receptor [J].Molecular Therapy,2011,19(12):2133-2143.
[6] Katter K,Geurts A,Hoffmann O,et al.Transposon-mediated transgenesis,transgenic rescue,and tissue-specific gene expression in rodents and rabbits [J].The FASEB Journal,2013,27:930-941.
[7] Woltjen K,Michael I,Mohseni P,et al.PiggyBac transposition reprograms fibroblasts to induced pluripotent stem cells [J].Nature,2009,458:766-770.
[8] Yusa K,Rad R,Takeda J,et al.Generation of transgene-free induced pluripotent mouse stem cells by the PiggyBac transposon [J].Nature Methods,2009,6:363-369.
[9] Fraser M,Ciszczon T,Elick T,et al.Precise excision of TTAA-specific lepidopteran transposons PiggyBac (IFP2) and tagalong (TFP3) from the baculovirus genome in cell lines from two species of lepidoptera [J].Insect Molecular Biology,1996,5:141-151.
[10] Wu S,Meir Y,Coates C,et al.PiggyBac is a flexible and highly active transposon as compared to sleeping beauty,Tol2,and Mos1 in mammalian cells [J].Proceedings of the National Academy of Sciences,2006,103:15008-15013.
[11] Rostovskaya M,Fu J,Obst M,et al.Transposon-mediated BAC transgenesis in human ES cells [J].Nucleic Acids Research,2012,40:e150.
[12] Sander J,Zaback P,Joung J,et al.An affinity-based scoring scheme for predicting DNA-binding activities of modularly assembled zinc-finger proteins [J].Nucleic Acids Research,2009,37:506-515.
[13] Desjarlais J,Berg J.Toward rules relating zinc finger protein sequences and DNA binding site preferences [J].Proceedings of the National Academy of Sciences of the United States of America,1992,89:7345-7349.
[14] Dreier B,Beerli R,Segal D,et al.Development of zinc finger domains for recognition of the 5′-ANN-3′ family of DNA sequences and their use in the construction of artificial transcription factors [J].Journal of Biological Chemistry,2001,276:29466-29478.
[15] Mossine V V,Waters J K,Hannink M,et al.PiggyBac transposon plus insulators overcome epigenetic silencing to provide for stable signaling pathway reporter cell lines [J].PLoS One,2013,8:e85494.
[17] Yusa K,Zhou L,Li M,et al.A hyperactive PiggyBac transposase for mammalian applications [J].Proceedings of the National Academy of Sciences of the United States of America,2011,108:1531-1536.
[18] Gietz R,Woods R.Yeast transformation by the LiAc/SS carrier DNA/PEG method [J].Methods in Molecular Biology,2006;313:107-120.
[19] Poon B P,Mekhail K.Effects of perinuclear chromosome tethers in the telomeric URA3/5FOA system reflect changes to gene silencing and not nucleotide metabolism [J].Frontiers in Genetics,2012,3:144.
[20] Yano T,Sanders C,Catalano J,et al.sacB-5-fluoroorotic acid-pyrE-based bidirectional selection for integration of unmarked alleles into the chromosome of rhodobacter capsulatus [J].Applied and Environmental Microbiology,2005,71(6):3014-3024.
Construction and screening of hyperactive PiggyBac transposases
ZHANG Longa,LIANG Ming-fub,ZHANG Zhi-yinga
(aCollegeofAnimalScienceandTechnology,bCollegeofLifeSciences,NorthwestA&FUniversity,Yangling,Shaanxi712100,China)
【Objective】 This study aimed to construct highly efficient and site-directed PiggyBac transposase system.【Method】 Wild-type PiggyBac transposase was used as template to construct mutant transposase library with random mutation transposase gene by error-prone PCR.Fusion expression with the ZFP protein that can recognize and bind the target DNA sequence was conducted to get ZFP-PiggyBac mutant transposase library system.Then the mutant transposase library,transposon and report vector were co-transformed into Saccharomyces cerevisiae JMY1.Highly efficient and site-specific transposases were screened by Ura-5FOA yeast screening system.【Result】 Highly efficient ZFP-PiggyBac mutant transposase library was successfully constructed.After three times of screening,five hyperactively mutant transposases were obtained.【Conclusion】 Newly constructed ZFP-PiggyBac mutant transposase library had the site-specific and efficient transposable ability at the ZFP protein recognition domain Rosa 26BS.
5FOA;transposase;zinc finger protein
2014-01-21
农业部“948”计划项目“高效PiggyBac转座酶引进及改造”(2013-Z17)
张 龙(1988-),男,安徽淮北人,硕士,主要从事转基因操作研究。E-mail:zhlong990@hotmail.com
张智英(1958-),男,陕西兴平人,教授,博士生导师,主要从事动物基因组学及转基因动物生产研究。 E-mail:zhangzhy@nwsuaf.edu.cn
时间:2015-06-30 13:47
10.13207/j.cnki.jnwafu.2015.08.008
Q78
A
1671-9387(2015)08-0001-09
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20150630.1347.008.html

