sh-Set7/9表达载体的构建及其在HepG2细胞中的功能
马克君,施星臣,李 平,李晓强,任 雯,秦 龙,施鑫鹤
(1.兰州大学第二医院中心实验室,甘肃兰州 730030;2.兰州市第二人民医院骨科,甘肃兰州 730030;3.兰州大学第二医院药剂科,甘肃兰州 730030)
◇基础研究◇
sh-Set7/9表达载体的构建及其在HepG2细胞中的功能
马克君1,施星臣2,李 平1,李晓强3,任 雯1,秦 龙3,施鑫鹤1
(1.兰州大学第二医院中心实验室,甘肃兰州 730030;2.兰州市第二人民医院骨科,甘肃兰州 730030;3.兰州大学第二医院药剂科,甘肃兰州 730030)
目的构建sh-Set7/9表达载体并筛选稳定转染HepG2细胞株,考察其干扰效果,为后续研究Set7/9基因的功能及其在肝癌细胞系中的作用提供实验基础。方法寻找靶向序列,设计siRNA片段,构建针对人Set7/9基因的sh RNA和对照载体,将干扰载体和载体稳定转染肝癌细胞HepG2,Real-time PCR检测细胞中Set7/9基因的表达水平;同时利用Western blot方法在蛋白质水平进行检测,确定该基因的干扰效果。结果成功构建载体Set7/9-sh RNA;Real-time PCR结果显示该基因表达水平明显被抑制(P<0.05),同样Western blot检测表明其蛋白表达也显著下调。此外,与其相关的Sirt1蛋白表达水平提高8.4倍,Suv39h1蛋白表达水平升高1.1倍。结论构建稳定转染株后,可以显著下调Set7/9基因的表达,同时影响与其相关的Sirt1和Suv39h1蛋白表达水平,提示对肝癌HepG2细胞活性产生影响。
Set7/9;Hep G2细胞;Western blot;甲基转移酶;载体构建;肝癌
肝癌(hepatocellular carcinoma,HCC)是指发生于肝脏的恶性肿瘤,包括原发性肝癌和转移性肝癌两种,在我国居恶性肿瘤死亡率第2位,严重危害群众的身体健康[1-2]。因此,对其形成机制的研究报道较多。随着表观遗传学的发展,从相关的甲基转移酶的角度对其成因和干预的研究也常有报道,但其中的具体关系和机制尚有待深入研究。
越来越多的研究涉及蛋白甲基转移酶Set7/ 9[3-4],它含有SET结构域,以S-腺苷甲硫氨酸(SAM/Ado Met)为底物,是蛋白赖氨酸甲基化转移酶(protein lysine methyltransferases,PLMTs或PKMTs)家族成员[5-6]。有研究表明,Set7/9介导Suv39h1甲基化,导致了异染色质松散和基因组不稳定[7];还可以影响Sirtuin 1(Sirt1)去乙酰化作用[8]。Suv39h1与白血病、胃癌等一些癌症的发生发展相关。而对于Set7/9在肝癌细胞中是否能调控其细胞活性、细胞周期的变化,以及如何影响Sirt1和Suv39h1蛋白的表达和功能的实现仍有待研究。
本研究以人Hep G2细胞株为靶细胞,构建了Set7/9-shRNA质粒,通过慢病毒包装,转染到HepG2细胞系中,并筛选到Set7/9沉默的稳定细胞株HepG2,用以研究在肝癌细胞系HepG2中该基因的功能,并初步探索与其相关的基因Sirt1和Suv39h1的表达变化情况,从而为进一步阐释Set7/ 9、Sirt1和Suv39h1在肝癌细胞中的功能及其对肝癌细胞活性及细胞周期的影响提供研究基础。
1 材料与方法
1.1 细胞株及主要试剂和仪器肝癌细胞株HepG2、大肠杆菌Top10感受态细胞由本实验室保存;质粒抽提试剂盒购自上海生工生物工程有限公司;DNA内切酶(Bam HⅠ、Eco RⅠ)、DNA连接酶、DNA marker购自宝生物工程(大连)有限公司;琼脂糖购自加拿大BBI公司;DMEM、胎牛血清FBS、双抗(青链霉素)均购自GIBCO公司;LipofectamineTM2000转染试剂购自Invitrogen公司;Polybrene购自Sigma公司;PBS购自AMEROSCO公司;SET7/9的单克隆抗体SET7/9(sc-56774)、Sirt1的多克隆抗体Sirt1(sc-15404)、Suv39h1的单克隆抗体Suv39h1(sc-377112)购自Santa Cruz公司;生物安全柜购自上海上净净化设备有限公司;荧光显微镜购自Motic公司;MX3000P实时荧光定量PCR仪购自Stratagene公司。
1.2 SETD7-shRNA载体质粒的构建和鉴定将SET7/9 mRNA序列输入在线软件http://genomics.jp/sidirect/,根据文献报道,SET7/9基因设计的干扰序列为GGGCACCTGGACGATGACGGA,阴性对照选择无意序列TTCTCCGAACGTGTCACGT。LV3-shRNA模板中的loop结构选用了TTCAAGAGA以避免形成终止信号。两端添加Bam HⅠ与Eco RⅠ酶切位点。将DNA oligo分别用TE(p H 8.0)溶解,浓度为100μmol/L。在PCR仪上按照如下程序进行退火处理:95℃、5 min;85℃、5 min;75℃、5 min;70℃、5 min;4℃保存。退火处理后得到浓度为10μmol/L的sh RNA模板。将所得模板溶液稀释50倍,终浓度为200 nmol/L,用于连接反应。用Bam HⅠ与Eco RⅠ酶切载体,琼脂糖电泳,使用Agarose Gel DNA Purification Kit Ver 2.0回收。之后进行连接反应,将连接产物转化至感受态的Top10菌种中,挑选阳性克隆,抽提质粒,单酶切再测序鉴定。
1.3 慢病毒包装取细胞状态良好,处于对数生长期的293FT细胞,细胞计数后,按照每个10 cm的培养皿5×106个细胞数接种于培养皿中,37℃、50 m L/L CO2的培养箱中培养过夜;次日加入5 m L新鲜的含100 mL/L血清DMEM培养液,DNA-Lipofectamine 2000复合物共转染293FT细胞,轻轻地前后摇晃培养皿以混匀复合物。放置37℃、50 m L/L CO2饱和湿度培养箱中过夜培养;转染后48 h收集培养上清进行浓缩;加入10 mL新鲜的培养液继续培养,转染后72 h再次收集浓缩,测定滴度;分装好的病毒放置-80℃保存。
1.4 重组慢病毒感染HepG2Hep G2在6 cm dish中培养至80%~90%融合时,倾去培养液,用3 mL D-Hank’s液洗涤细胞2次。加1 m L Trypsin-EDTA液,混匀后,小心吸去胰酶溶液,37℃放置3 min。再加入2 m L DMEM培养液,吹打使细胞形成单细胞悬液。血球计数板计数,按10×105细胞/孔的密度接种于6孔板,混匀后37℃、50 m L/L CO2培养24 h。将慢病毒原液200μL,用100 m L/L FBS的DMEM培液5倍稀释,并加入终浓度为0.5μg/m L的Polybrene。吸去6孔板中的培养液,每孔加入上述1 mL稀释的病毒液,同时设立空白对照组,于37℃、50 mL/L CO2培养24 h。吸弃6孔板中的稀释病毒液,每孔加入2 mL 100 mL/L FBS的DMEM培液于37℃、50 mL/L CO2继续培养至72、96 h分别收样。
1.5 SET7/9基因和蛋白表达效果的检测用puromycin进行选择,获得稳筛株,并扩增稳筛株细胞,分别收样,用于提取细胞总RNA,进行荧光定量PCR实验。GAPDH(内参)F primer:CATGAGAAGTATGACAACAGCCT,R primer:AGTCCTTCCACGATACCAAAGT;SET7/9(目的)F primer:CACAGATGGGAGACTGATCTTCAAG,R primer:TTACTTCTCCTACAAGGCTTCCTCC。实验扩增条件为:95℃、3 min预变性;95℃、30 s;62℃、40 s;40循环;Western blot检测干扰后SET7/9蛋白的表达情况。
1.6 统计学处理应用SPSS 18.0统计软件分析,数据以表示。进行方差齐性检验和正态性检验。两组以上的比较采用单因素方差分析。P<0.05为差异有统计学意义。
2 结 果
2.1 Set7/9-sh RNA载体质粒的鉴定和测序结果将p GLV3/H1/GFP/puro空载体进行酶切,回收,与插入子连接,转化Top10细菌株,挑取4个菌落抽提质粒。所得质粒用Eco RⅠ进行单酶切鉴定,电泳后可以看到约8 000 bp的线性载体,挑取1、3号克隆进行测序,证明SET7/9sh RNA载体构建成功(图1)。
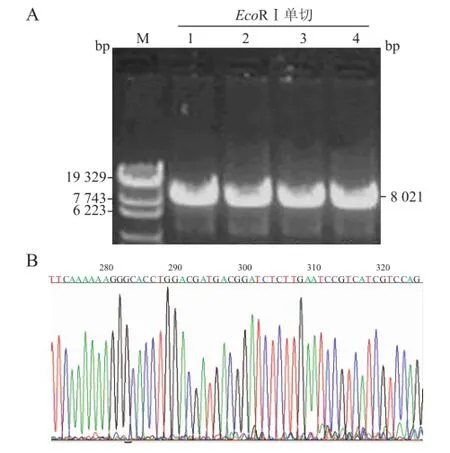
图1 pGLV3/H1/GFP/puro-Set7/9质粒克隆的鉴定Fig.1 Identification ofplasmid pGLV3/H1/GFP/puro-Set7/9
2.2 慢病毒载体的包装及侵染细胞提取质粒,将慢病毒包装系统质粒共转染293FT细胞,48 h后收集培养上清进行浓缩;转染后72 h再次收集浓缩。测定滴度为1×108TU/m L,重组后的病毒侵染HepG2细胞,并获得最适puromycin浓度,经过培养筛选获得稳筛株细胞(图2)。
2.3 Real-time PCR及Western blot实验证实SET7/9基因被沉默通过稳筛株细胞扩增,分别收样,用于Real-time PCR、Western blot检测。从基因和蛋白水平上对HepG2细胞中SET7/9基因的沉默效果进行检测,表明该基因表达水平明显被抑制(P<0.05);同样Western blot检测显示,其蛋白表达也显著下调。以GAPDH和β-actin为内参,检测转染72 h后SET7/9基因和蛋白表达情况(表1、图3)。
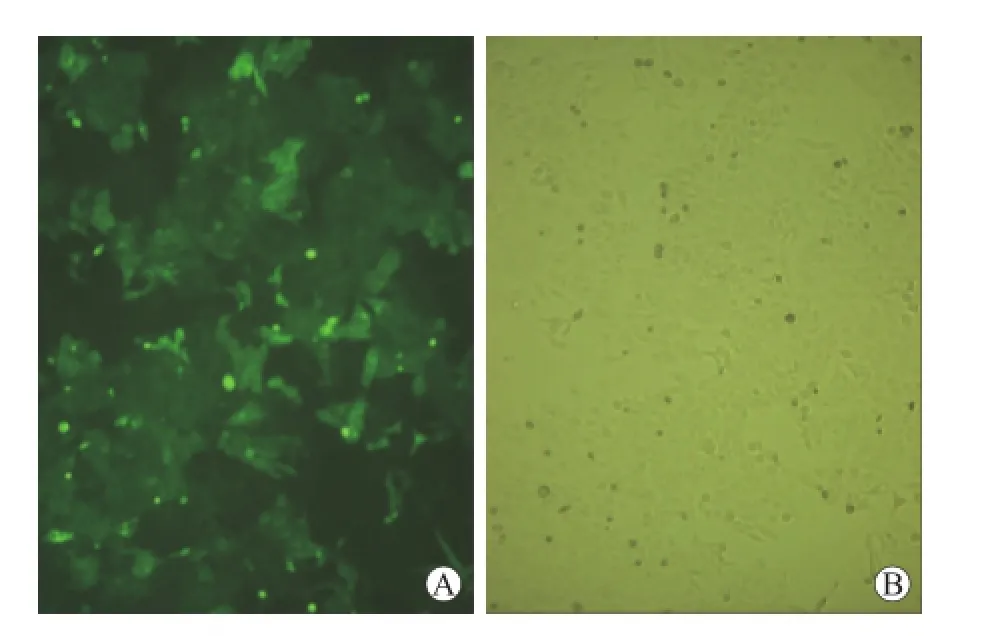
图2 病毒侵染HepG2细胞图Fig.2 Virus infected Hep G2(×100)
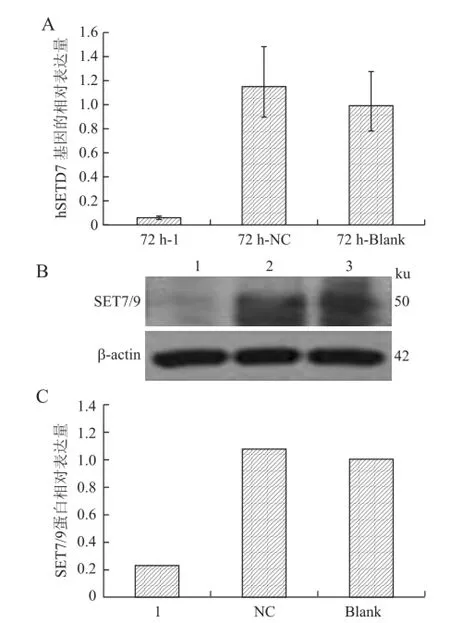
图3 SET7/9基因沉默后基因和蛋白的相对表达情况Fig.3 Relative expressions of gene and protein after SET7/ 9 knockdown
2.4 SET7/9基因影响Sirt1及Suv39h1的表达在SET7/9基因被成功沉默后,用Western blot实验分别对Sirt1及Suv39h1的蛋白表达水平进行检测,结果发现Sirt1蛋白表达水平显著增加8.4倍,Suv39h1蛋白表达水平也有上调1.1倍,但变化不明显(图4)。
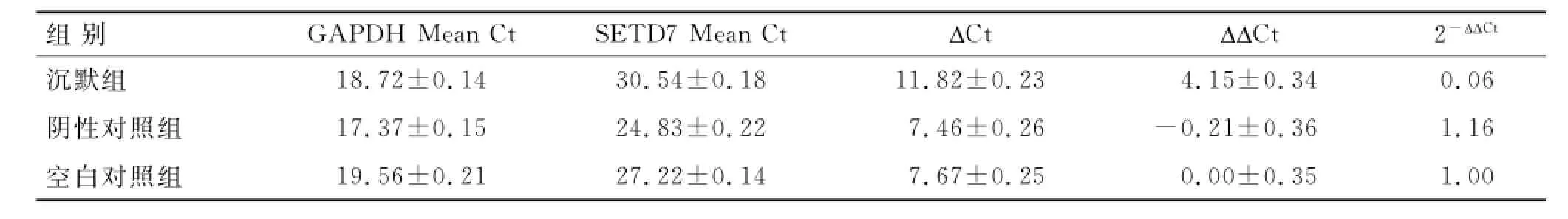
表1 Real-time PCR检测基因沉默后SET7/9表达Tab.1 Detection of SET7/9 expression after knockdown detected by Real-time PCR
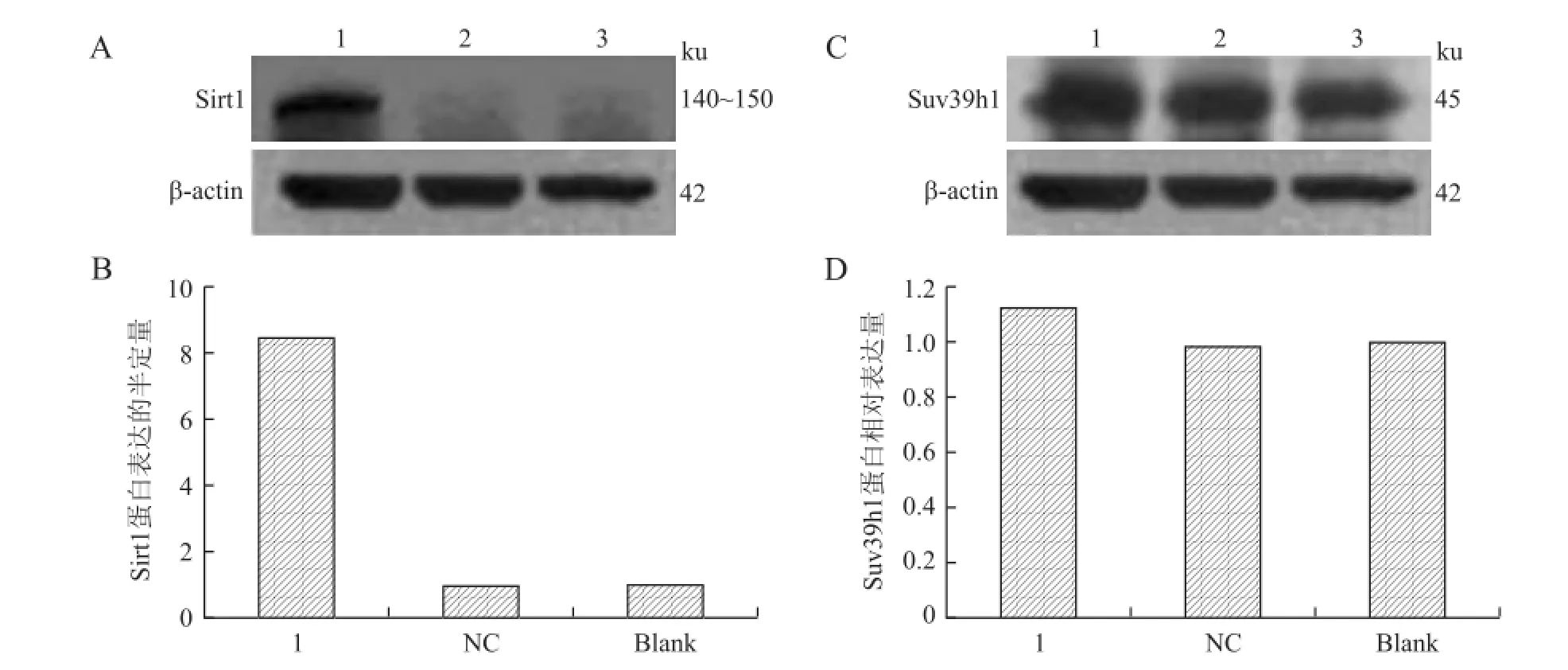
图4 Sirt1与Suv39h1蛋白表达水平变化Fig.4 Changes of Sirt1 and Suv39h1 protein expressions
3 讨 论
Suv39h1是第1个被发现的特异性H3K9甲基转移酶,组蛋白H3K9的甲基化在异染色质形成及基因转录调控中具有重要作用。近年的一些研究表明,H3K9甲基化失衡与白血病、胃癌等一些癌症发生发展相关。Set7/9介导Suv39h1甲基化,导致了异染色质松散和基因组不稳定。有研究人员确定了蛋白质甲基转移酶Set7/9是Suv39h1活性的一个独特调控因子,证实Set7/9介导了Suv39h1赖氨酸105和123位点特异性甲基化[7]。
此外,朱卫国等[8]鉴别出Set7/9也是Sirt1的独特调控因子,细胞内Set7/9与Sirt1相互作用显著增强,从而抑制了Sirt1与p53的相互作用,导致p53乙酰化水平相应增高,p53介导的转录激活增强。研究结果表明除甲基化作用依赖的机制,Set7/9还可通过Sirt1间接调控p53的功能。p53作为抑癌基因,与一些肿瘤的发生相关,去乙酰化,甲基化等表观遗传学修饰对其功能有影响[9-10]。
因此,有必要对Set7/9在肝癌细胞中的功能进行研究。shRNA是多种小分子干扰RNA制备方法中较好的可以进行长期基因沉默研究的方法[11-13]。脂质体介导的sh RNA进入细胞的方法往往转染效率低,而通过慢病毒包装进行转染在基础研究中得到广泛应用[14]。慢病毒是在人类免疫缺陷病毒-1(HIV-1)的基础上改造形成的逆转录病毒[15],能够将目的基因整合到染色体上,得到稳定的细胞株。
sh RNA为环状结构,在细胞内经酶切产生小分子干扰RNA,后者的反义链在细胞内与特定蛋白质结合形成RISCs(RNA-induced silencing complexes),通过反义链与特异mRNA序列互补结合,剪切mRNA导致mRNA降解,从而在细胞内产生特定基因的沉默效果[16-17]。其具有抑制效果确切、严格序列特异性及针对性强等优势。有效干扰序列设计是sh RNA构建的关键,本实验设计的靶序列参考已有文献报道,并进行sh RNA构建,实验证明为有效干扰靶点。
总之,本研究立足近年来发现Set7/9催化的非组蛋白甲基化作用能引起蛋白稳定性改变和基因表达变化等多种分子生物学效应,这些分子效应涉及到染色体结构、细胞周期及凋亡等方面,构建了Set7/9-shRNA质粒,研究在肝癌细胞系Hep G2中该基因的功能,并初步探索与其相关的蛋白的表达变化情况。结果显示,Sirt1蛋白表达水平提高8.4倍,Suv39h1蛋白表达水平升高1.1倍。这两种蛋白分别具有去乙酰化和甲基化的功能,因此其表达水平的变化暗示在HepG2细胞系中Set7/9基因可能会对其活性和细胞周期等产生影响,这有待更深入的研究。
[1]HSU SH,YEH ML,WANG SN,et al.New insights in recurrent HCV infection after liver transplantation[J].Clin Dev Immunol,2013,2013:890517.
[2]HU Z,ZHOU J,WANG H,et al.Survival in liver transplant recipients with hepatitis B-or hepatitis C-associated hepatocellular carcinoma:the Chinese experience from 1999 to 2010[J]. PLoS One,2013,8(4):e61620
[3]KURASH JK,LEI H,SHEN Q,et al.Methylation of p53 by Set7/9 mediates p53 acetylation and activity in vivo[J].Molecular Cell,2008,29:392-400.
[4]ESTEVE PO,CHIN HG,BENNER J,et al.Regulation of DNMT1 stability through SET7-mediated lysine methylation in mammalian cells[J].Proc Natl Acad Sci USA,2009,106:5076-5081.
[5]GUO HB,GUO H.Mechanism of histone methylation catalyzed by protein lysine methytransferase SET7/9 and origin of product specificity[J].Proc Natl Acad Sci USA,2007,104(21):8797-8802.
[6]QIAN C,ZHOU MM.SET domain protein lysine methyltransferases:structure,specificity and catalysis[J].Cell Mol Life Sci,2006,63(23):2755-2763.
[7]WANG DL,ZHOU JY,LIU XY,et al.Methylation of SUV39H1 by SET7/9 results in heterochromatin relaxation and genome instability[J].Proc Natl Acad Sci USA,2013,110(14):5516-5521.
[8]LIU X,WANG D,ZHAO Y,et al.Methyltransferase Set7/9 regulates p53 activity by interacting with Sirtuin 1(SIRT1)[J]. Proc Natl Acad Sci USA,2011,108(5):1925-1930.
[9]HUANG J,PEREZ-BURGOS L,PLACEK BJ,et al.Repression of p53 activity by Smyd2 mediated methylation[J].Nature,2006,444(7119):629-632.
[10]SCOUMANNE A,CHEN X.Protein methylation:a new mechanism of p53 tumor suppressor regulation[J].Histol Histopathol,2008,23(9):1143-1149.
[11]姜海艳,王玉梅,关伟军.乳腺癌多药耐药机制及RNAi技术的应用[J].中国实验诊断学,2009,13(4):561.
[12]YU JY,DERUITER SL,TURNER DL,et al.RNA interference by expression of short-interfering RNAs and hairpin RNAs in mammalian cells[J].Proc Nat Acad Sci USA,2002,99(9):6047-6052.
[13]LIU F,HE CW,ZHANG YF,et al.RNA interference by expression of short hairpin RNAs suppresses bcl-x L gene expression in nasopharyngeal carcinoma cells[J].Acta Pharmacol Sin,2005,26(2):228-234.
[14]GHOSH SS,GOPINATH P,RAMESH A,et al.Adenoviral vectors:a promising tool for gene therapy[J].Appl Biochem Biotechnol,2006,133(1):9-29.
[15]LOIS C,HONG E J,PEASE S,et al.Germline transmission and tissue specific expression of transgenes delivered by lentiviral vectors[J].Science,2002,295(5556):868-872.
[16]FANG CH,LIU J,FAN YF,et al.Outcomes of hepatectomy for hepatolithiasis based on 3-dimensional reconstruction technique[J].J Am Coll Surg,2013,217(2):280-288.
[17]易敏,黄文娟,宋彩虹,等.丙型肝炎病毒核心蛋白抑制sh RNA介导的RNA干扰作用[J].第三军医大学学报,2012,34(12):1162-1166.
(编辑 卓选鹏)
Construction of shSet7/9 vector and its function in HepG2
MA Ke-jun1,SHI Xing-chen2,LI Ping1,LI Xiao-qiang3,REN Wen1,QIN Long3,SHI Xin-he1
(1.Central Laboratory,Lanzhou University Second Hospital,Lanzhou 730030;2.Department of Orthopaedics,Lanzhou Second Hospital,Lanzhou 730030;3.Pharmacy,Lanzhou University Second Hospital,Lanzhou 730030,China)
ObjectiveTo silence human gene Set7/9 and screen out stable transfection cell line in hepatocellular carcinoma cell line Hep G2 so as to investigate the impact of down-regulation of Set7/9 in cell line HepG2 and provide experimental foundation for studies on the effect of set7/9 in HepG2.MethodsThe target oligo was designed and synthesized;sh RNA interference vector and the control vector were constructed and transfected into Hep G2 cells;the stable transfection cells were screened out.Then Real-time PCR and Western blot were performed to detect the silence of Set7/9 according to both gene expression and protein expression level.ResultsThe sh RNA interference vector was constructed and transfected into Hep G2 cells successfully.Compared with that in the negative control group,the expression of Set7/9 was dramatically downregulated(P<0.05). Meanwhile,the expression of related protein Sirt1 and Suv39h1 was upregulated 8.4 folds and 1.1 fold,respectively.ConclusionDownregulation of Set7/9 expression can upregulate Sirt1 and Suv39h1,suggesting that Set7/9 may af fect the activity of Hep G2 cell lines.
Set7/9;Hep G2 cell;Western blot;methyltransferase;vector construction;hepatocellular carcinoma
R735.7
A
10.7652/jdyxb201506008
2015-01-06
2015-05-06
2013年兰州大学中央高校基本科研业务费(No.lzujbky-2013-144)
Supported by the Fundamental Research Funds for the Central Universities(No.lzujbky-2013-144)
施鑫鹤.E-mail:shixh@lzu.edu.cn
优先出版:http://www.cnki.net/kcms/detail/61.1399.R.20150929.0859.010.html(2015-09-29)

