猪源粪肠球菌的基因型及耐药性分析
董栋,商军,钱晓璐,田恺
猪源粪肠球菌的基因型及耐药性分析
董栋1,2,商军1∗,钱晓璐1,田恺1
(1.上海市兽药饲料检测所,上海201103;2.上海海洋大学,上海201306)
为了解上海地区规模化猪场中粪肠球菌的耐药性和基因型,2013年3月-2014年5月从上海地区10个规模化养猪场收集到分离鉴定的粪肠球菌133株,采用微量肉汤稀释法对12种抗菌药物进行药敏试验,采用PCR技术分别检测分离株中β-内酰胺类抗生素耐药相关基因(TEM)、氨基糖苷类抗生素耐药相关基因[aac(6’)/aph(2”)和aph(3’)-Ⅲ和ant(6)-Ⅰ]、四环素耐药相关基因(tetM)、红霉素耐药相关基因(ermB和mefA)和万古霉素耐药相关基因(vanA和vanB)。结果显示,在133株粪肠球菌中,耐药基因ermB、tetM、TEM、ant(6)-Ⅰ、aac(6’)/aph2”、aph(3’)-Ⅲ、vanA的检出率分别为:95.5%(127/133)、91.0%(121/133)、63.2%(84/133)、45.9%(61/133)、42.1%(56/133)、24.1%(32/133)、3.0%(4/133);未检出mefA和vanB基因的菌株。分离菌株对12种抗菌药物的耐药性较为严重,且以6~9耐为主要耐药株,占81.2%。试验表明,上海地区猪源粪肠球菌多重耐药现象严重,携带抗菌药物相关耐药基因是导致分离株耐药的主要原因。
粪肠球菌;耐药性;基因型;抗菌药物
粪肠球菌球是一种重要的机会致病菌,在自然界中广泛分布,常栖居于人和动物的肠道中。粪肠球菌可导致人和动物多个脏器的感染,包括尿路感染、皮肤软组织感染、盆腔感染、腹腔感染、伤口感染、菌血症、心内膜炎和脑膜炎[1-2]。由于其质粒、转座子和突变株的发生,粪肠球菌易被诱导产生新的耐药性,对许多抗菌药物可产生获得性耐药,如对高水平的β-内酰胺、氨基糖苷、糖肽类(万古霉素)、四环素、红霉素、利福平和氯霉素容易产生获得性耐药[3]。随着兽医临床上抗菌药物被广泛应用,导致粪肠球菌耐药菌株不断出现,甚至对多种抗菌药物同时耐药的多重耐药菌株也屡见不鲜,细菌耐药性的出现和耐药细菌的感染常使经验性治疗难以奏效,近年来,猪感染粪肠球菌发病和死亡的报道也越来越多[4-6],给兽医临床抗感染治疗带来极大的挑战,对动物和人类的健康造成严重威胁。而粪肠球菌是表示抗菌药物耐药性水平的一种革兰氏阳性指示菌,因此有必要对猪源分离粪肠球菌的耐药性和基因型进行分析和研究,可为科学制定抗菌感染的防治方案及细菌耐药性的监测提供必要的理论依据。为此对从上海地区10个规模化养猪场分离得到的133株粪肠球菌进行了研究分析。
1 材料与方法
1.1 菌株来源 2013年3月-2014年5月从上海地区6个区县10个规模化养猪场肛门样本中分离得到的粪肠球菌133株;质控菌株:粪肠球菌ATCC 29212,美国标准菌种保藏中心。所有菌株置于20%甘油肉汤中,-20℃保存。
1.2 试剂与试药 冻干型细菌定量药敏(MIC)测试盒及MH肉汤,天津市金章科技发展有限公司;9700 PCR扩增仪,美国AB公司;PCR引物合成、相关试剂、试剂盒及蛋白酶K,上海英骏公司;GelDoc 2000凝胶图像分析系统,美国BIO-RAD公司;低温离心机,德国Beckman公司;VITEK 2 Compact全自动微生物分析系统及配套细菌鉴定卡(GN卡),法国生物梅里埃公司;普通营养肉汤及营养琼脂,北京陆桥技术有限责任公司;肠球菌显色琼脂,美国BD公司。
1.3 方法
1.3.1 采样 以灭菌棉签拭子采猪肛门样品,置入运送培养基中,保存时间不超过48 h。每个养殖场采30~50份拭子样本,共400份样本。
1.3.2 粪肠球菌分离,纯化与鉴定 取拭子样品用接种棒直接接种于肠球菌显色琼脂平板,(36±1)℃培养18~24 h,挑取黑色带光泽的可疑菌落、显色琼脂上纯化一代。纯化后可疑菌落接种营养琼脂平板纯化,(36±1)℃培养16~18 h。对于已纯化的菌落采用生物梅里埃VITEK 2 Compact全自动微生物分析系统鉴定出133株。
1.3.3 药敏试验 按照美国临床检验标准委员会(CLSI)推荐的微量肉汤稀释法进行实验操作和药敏结果结果判断[7-9]。直接从过夜不超过24 h培养的平板上挑取3~5个纯新鲜菌落,均匀悬浮于1.5 mL细菌稀释管中,再将该菌液调整于0.5麦氏浓度,使菌液的细菌含量1×108CFU/mL,然后稀释为1×105~2×105CFU/mL左右,接种于含不同药物浓度的96孔冻干型细菌定量药敏(MIC)测试盒中,(36±1)℃温箱中培养16~20 h,按同法做粪肠球菌ATCC 29212的质量控制,观察结果。对所测得的MIC结果经统计学处理,得出MIC50、MIC90及MIC范围,测定结果按MIC判定标准判定为敏感(S)、中介(I)和耐药(R)。药敏试验结果的MIC判定标准和质控范围见表1。
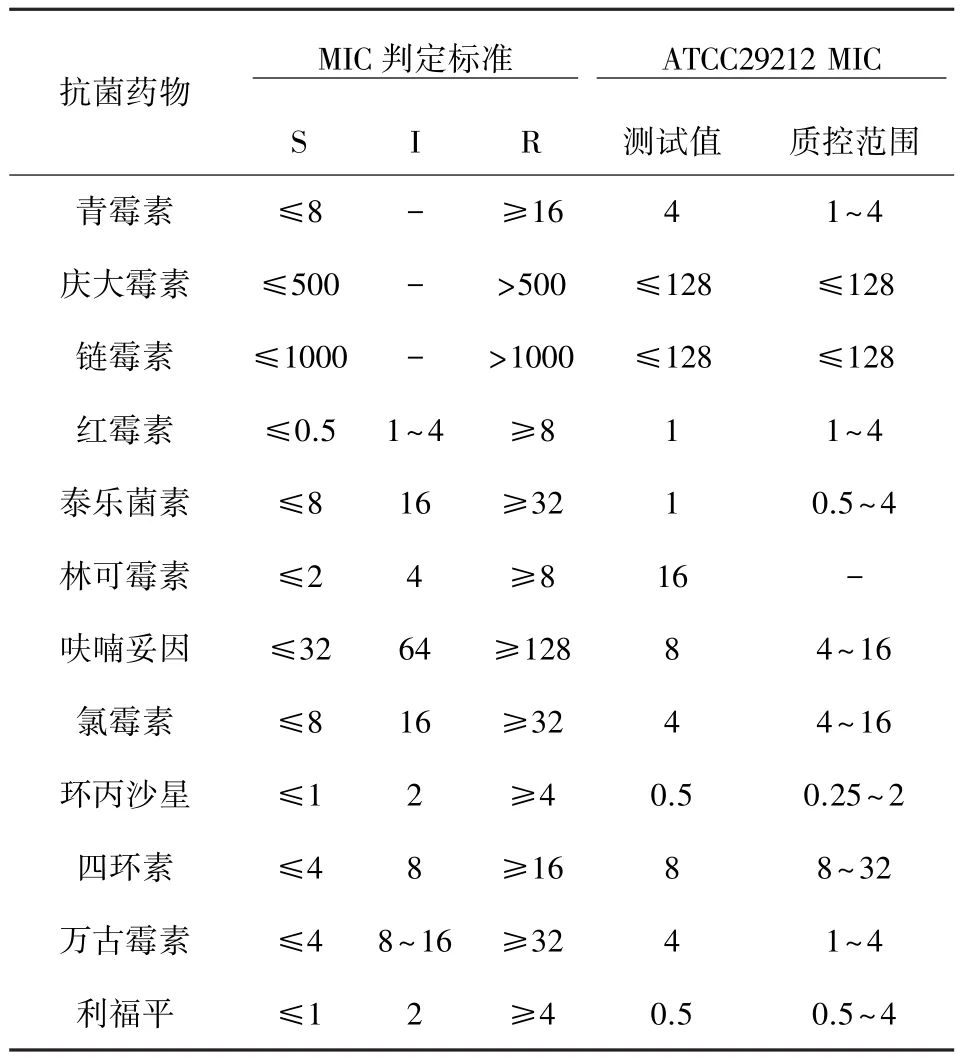
表1 粪肠球菌对12种抗菌药物药敏试验的M IC判定标准和质控范围mg/L
1.3.4 基因检测 (1)引物设计:在GenBank中查询肠球菌属的9种耐药基因的基因序列(表2),运用Primer5.0系列分析软件设计引物。(2)DNA模板制备:采用传统的煮沸法提取质粒DNA[10],吸取上清作为PCR扩增模板。(3)PCR 25μL反应体系:其中模板5μL,上、下游引物各0.5μL,dNTP 100 mol/L,其他成分按常规配比加入后,双蒸水补至25μL。各种耐药基因扩增的循环参数为:95℃预变性2 min;94℃变性30 s;50℃退火40 s;72℃延伸40 s;循环35次;72℃再延伸5 m in。其中ermB基因循环参数按93℃预变性2 min,93℃变性30 s;37℃退火90 s;72℃延伸100 s;循环35次;72℃再延伸10 min。取各个耐药基因PCR产物5μL,以2%的琼脂糖凝胶上100 V电泳20 min,在凝胶成像系统检验结果。
2 结果
2.1 药敏试验 结果见表3和图1。

表2 耐药基因PCR引物系列的设计

表3 12种抗菌药物对133株粪肠球菌的抗菌活性及粪肠球菌对抗菌药物的耐药率
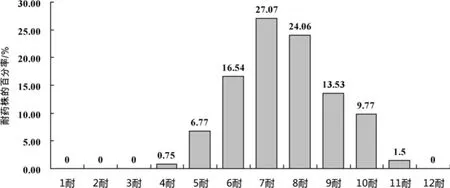
图1 133株粪肠球菌对12种抗菌药物的多重耐药性
133株粪肠球菌对12种抗菌药物的耐药率情况为:耐药率超过90%的有林可霉素、红霉素、泰乐菌素和四环素,超过70%的有氯霉素,超过60%的有〛青霉素,超过50%的有链霉素、利福平和环丙沙星,超过40%的有庆大霉素,低于10%的有呋喃妥因和万古霉素。133粪肠球菌耐药谱复杂,所耐的抗菌药物种类变化大,共得到72个耐药谱,多重耐药性和交叉耐药性耐药株以6耐~9耐为主,占81.2%,表现出较为严重的耐药性。值得注意的是,在4株万古霉素耐药粪肠球菌中,有3株表现为对万古霉素高度耐药(MIC>64 mg/L),且对链霉素和庆大霉素表现为高度耐药(MIC>2048 mg/L),高水平庆大霉素(MIC>500mg/L)和(或)链霉素(MIC>1000mg/L)耐药检出比例较高,为大于60%,且两对者同时耐药的有32株。
2.2 耐药基因检测 结果见表4。5133株粪肠球菌中,除未检出mefA和vanB基因外,vanA、aac(6’)/aph(2”)、ant(6)-Ⅰ、aph(3’)-Ⅲ、tetM、ermB和TEM基因的检出率范围为3.0%~95.5%,基因阳性相对表型阳性的符合率范围为78.2%~100%。
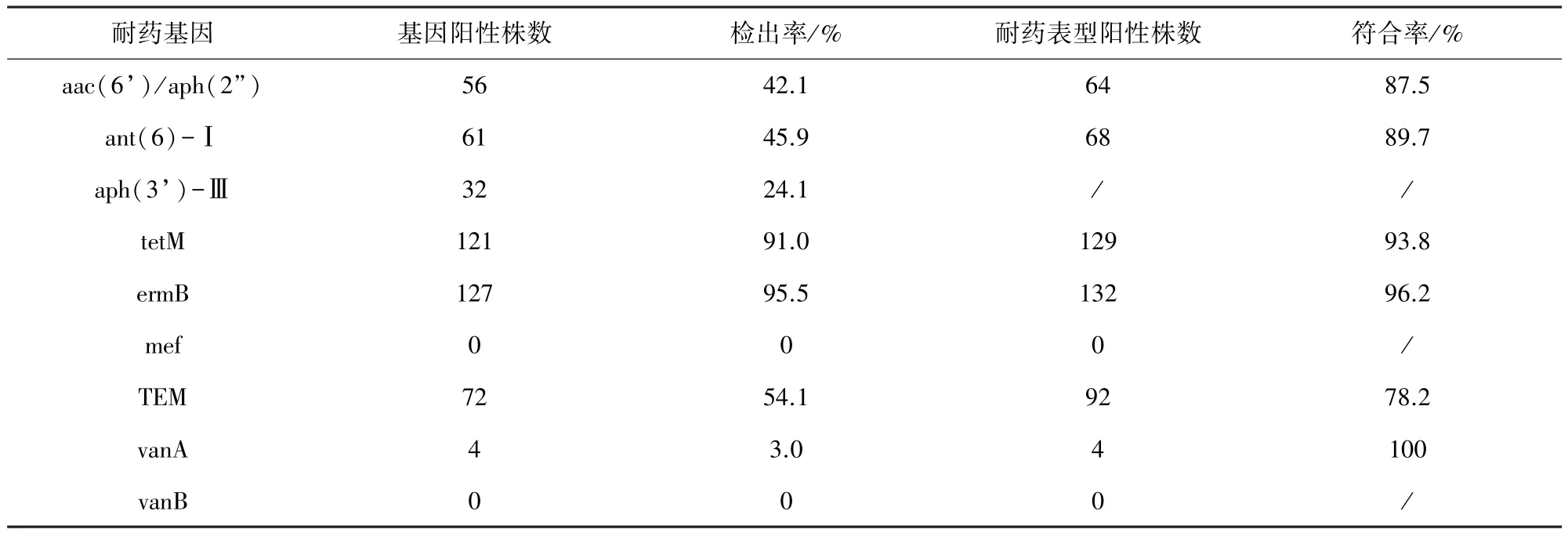
表4 133株粪肠球菌耐药基因的检测率和符合率
3 讨论
从药敏结果分析,除对呋喃妥因和万古霉素敏感外,对其余抗菌药物都呈现不同程度的耐药性,且多重耐药性和交叉耐药性严重,与相关报道一致[1,11-12]。这与近年来随着肠球菌属对抗菌药物耐药性的逐渐上升,又出现了耐万古霉素肠球菌(VRE)和高水平氨基糖苷类耐药(HLAR)[13-15],给兽医临床治疗带来了困难情况相一致。
从耐药基因检测结果分析,本试验133株粪肠球菌中,tetM基因的检出率为91.0%,基因阳性相对表型阳性的符合率为93.8%,说明tetM基因的存在是粪肠球菌分离株对四环素耐药的主要因素。ermB基因的检出率为95.5%,高于相关文献报道[1,15],未检测出mefA基因,基因阳性相对表型阳性的符合率为96.2%,可见粪肠球菌对红霉素耐药仍以ermB基因的作用机制为主。有5株红霉素耐药粪肠球菌未检出ermB基因,是否携带ermB基因家族其他类型基因导致红霉素耐药,还需要进一步研究。对庆大霉素耐药的64株分离株中aac(6’)/aph(2”)基因的符合率为87.5%,链霉素耐药的68株粪肠球菌中ant(6)-I基因的符合率为89.7%,aph(3’)-Ⅲ基因32株,检出率为24.1%,双功能酶基因aac(6’)/aph(2”)和ant(6)-Ⅰ基因的符合率均大于80%,与文献报道相一致[16],说明aac(6’)/aph(2”)和ant(6)-Ⅰ介导了粪肠球菌分离株的高水平氨基糖苷类耐药(HLAR)。TEM基因的检出率为63.2%,基因型阳性的菌株表型均为阳性符合率为78.2%,表明携带TEM基因是导致粪肠球菌分离株对青霉素类耐药的主要原因。有4株耐万古霉素的粪肠球菌中检出vanA基因,未检测出vanB基因。
目前,肠球菌属已是医学临床感染分离率较高的细菌,呈多重耐药,给临床治疗带来困难。本研究采集猪源粪肠球菌,从耐药表型和基因水平检测分析发现,携带耐药基因是导致粪肠球菌分离株对相应抗生素耐药的主要因素,因此加强细菌耐药基因的检测,可为科学制定抗菌感染的防治方案及细菌耐药性的监测提供必要的理论依据。
[1] 齐亚银,张莉,王静梅,等.动物源粪肠球菌对7种抗生素耐药表型及耐药基因检测[J].中国兽医学报,2010,30(7):962-963.
[2] 刘燕霏,杨建德,赵瑞利.粪肠球菌动物致病的研究进展[J].家畜生态学报,2013,34(11):75-76.
[3] 瞿婷婷,陈亚岗,俞云松.肠球菌耐药性研究[J].浙江预防医学,2004,16(7):3-5.
[4] 林荣高.湖南省猪源粪肠球菌基因多样性分析[D].湖南长沙:湖南农业大学,2011:7-22.
[5] 黎满香.感染猪的粪肠球菌的分离鉴定及部分特性和诊断方法研究[D].湖南长沙:湖南农业大学,2010:4-12.
[6] Donabedian SM,PerriM B,Abdujamilova N,et al.Characterization of vancomycin-resistant Enterococcus faecium isolated from swine in three Michigan counties[J].JClin Microbiol,2010,48(7):4156-4160.
[7] Clinical and Laboratory Standards Institute(CLSI).Performance standards for antimicrobial susceptibility testing:twentieth informational supplement[S].2013:M100-S23.
[8] Clinical and Laboratory Standards Institute(CLSI).Development of in vitro susceptibility testing criteria and quality control parameters for veterinary antimicrobial agents[S].2008:M37-A3.
[9] Clinical and Laboratory Standards Institute(CLSI).Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically[S].2012:M07-A9.
[10]李水法,王华钧,孙小军等.肠球菌属耐药基因检测[J].中华医院感染学杂志,2011,21(1):14.
[11]Oprea SF,Zaidi N,Donabedian SM,et al.Molecular and clinical epidemiology of vancomycin resistant Enterococcus faecalis[J].JAnti-microb Chemother,2004,53(4):626-630.
[12]Qu T T,Chen Y G,Yu Y S,et al.Genotypic diversity and epidemiology of high-level gentamicin resistant Enterococcus in a Chinese hospital[J].J Infect,2006,52(2):124-130.
[13]刘长连,徐韫健,梁权辉.氨基糖苷类高水平耐药肠球菌耐药基因的检测与研究[J].中国微生态学杂,2013,25(3):298.
[14]罗润齐,叶晓光,王若伦.粪肠球菌耐药谱5年动态观察与分析[J].广州医药杂,2008,39(2):47-48.
[15]刘晶,朱文君,赵娟.肠球菌多重耐药性及耐药基因的研究[J].中国卫生检验杂志,2011,21(8):2059.
[16]孟峻,张军力,郭素芳.耐高浓度庆大霉素肠球菌检测及药敏结果分析[J].中华医院感染学杂志,2009,19(19):2607-2609.
(编辑:李文平)
Analysis of Genotype and Antim icrobial Resistance in Enterococcus faecalis Isolated from Pigs
DONG Dong1,2,SHANG Jun1∗,QIAN Xiao-lu1,TIAN Kai1
(1.Shanghai Institute for Veterinary Drug&Feeds Control,Shanghai 201103,China;2.Shanghai Ocean University,Shanghai 201306,China)
This study was conducted to investigate the antibiotic susceptibility and resistance genotypes in Enterococcus faecalis strains isolated from ten Large-scale pig farms in Shanghai.133 strains of E.faecalis were collected between March 2013 and May 2014.The susceptibility testingwas performed by brothmicrodilution,and the genotypes ofβ-lactam antibiotics resistance-associated gene(TEM),aminoglycoside genes[aac(6’)/aph(2”),aph(3’)-Ⅲand ant(6)-Ⅰ],tetracycline gene(tetM),erythromycin genes(ermB and mefA)and vancomycin genes(vanA and vanB)were analyzed by PCR.The positive rate of the resistance genes ermB,tetM,TEM,ant(6)-Ⅰ,aac(6’)/aph2”,aph(3’)-Ⅲ,vanA in the 133 strains of E.faecalis tested were 95.5%(127/133),91.0%(121/133),63.2%(84/133),45.9%(61/133),42.1%(56/133),24.1%(32/133),and 3.0%(4/133),respectively.No mefA and vanB gene strains were detected.12 kinds of antibacterial drug resistance were relatively serious and 6~9 resistance as themain drug resistant strains were detected,accounting for 81.2%.This study shows thatmultidrug resistance of E.faecalis was a serious issue,and resistance genes harbored were the very important reasons for resistance to antibiotics in E.faecalis.
Enterococcus faecalis;antimicrobial resistance;genotype;antimicrobial agents
2014-10-27
A
1002-1280(2015)02-0013-05
S852.61+1
董栋,硕士研究生,从事动物源细菌耐药性的研究。
商军。E-mail:sjshvdc@163.com

