圭亚那柱花草新EST—SSR标记验证及其在柱花草属上的转移
丁西朋 罗小燕 贾庆麟 白昌军
摘 要 柱花草(Stylosanthes spp.)是我国南方热带地区优良豆科牧草。为获得高效的柱花草分子标记,本课题组利用热研5号圭亚那柱花草(Stylosanthes guianensis)转录组测序数据开发了2008个EST-SSR标记。为验证新EST-SSR标记的有效性及在柱花草属中的通用性,本研究从中随机挑选117个EST-SSR标记并分析了其在圭亚那柱花草中的有效性及其在其他8种柱花草中的可转移性。结果表明,有98个EST-SSR标记可获得有效扩增,其中96个在不同柱花草种质间有多态性,每个标记检测出的等位基因2~11个,共检测到等位基因456个。98个EST-SSR标记在其他8种柱花草间的可转移率为82.2%~100%,其中69个标记在8种柱花草间都能有效扩增。
关键词 柱花草;EST-SSR;标记开发;遗传多样性分析
中图分类号 S541 文献标识码 A
Transferability of Novel EST-SSR Markers in Stylosanthes
guianensis Across the Species of Stylosanthes
DING Xipeng, LUO Xiaoyan, JIA Qinglin, BAI Changjun*
Institute of Tropical Crops Genetic Resources, CATAS, Danzhou, Hainan 571737, China
Abstract To obtain effective molecular markers for Stylosanthes which is one of the most excellent legume forages, 2008 EST-SSR markers were developed using the transcriptomic sequences from S. guianensis cv. Reyan No.5. To assess the usefulness and universality of these novel EST-SSR markers within Stylosanthes genus, 117 EST-SSR markers were selected to determine the usefulness in S. guianensis and the transferability among other close eight species. The results showed that 98 of 117 EST-SSR markers were able to produce reliable bands with expected sizes and 96 markers were polymorphic, with 2~11 alleles among different Stylosanthes accessions. The transferability of 98 validated EST-SSR markers across other close eight species was very high, ranging from 82.2% to 100%, and 69 markers of them were common in all eight species of Stylosanthes. The results would provide a basis for the application of EST-SSR in genetic diversity, construction of genetic maps, and molecular marker-assisted selection breeding in stylo species.
Key words Stylosanthes spp.; EST-SSR; Marker-development; Genetic diversity
doi 10.3969/j.issn.1000-2561.2015.10.006
分子标记是作物遗传学研究和分子标记辅助育种的重要工具,SSR标记具有数量丰富、多态性高、共显性、操作简单等多个优点,在水稻、拟南芥、玉米等多种植物中被开发并应用于遗传多样性分析、指纹图谱构建、遗传连锁图谱构建和分子标记辅助育种等方面[1-2]。根据来源不同,SSR标记可分为基因组SSR(genomic-SSR)和 EST-SSR两种[3]。EST-SSR标记是基于EST 序列开发的SSR标记,与传统的genomic-SSR标记开发相比较,不仅开发速度快,且成本低。由于EST-SSR来源于表达的基因组区域,其多态性可能与基因功能直接相关,从而强化了EST-SSR标记在遗传学领域研究中的应用[4]。此外,由于EST序列比基因组DNA序列相对保守,所以EST-SSR标记相对于genomic-SSR标记在不同种间的通用性更高,但多态性较低[5]。近年来,随着EST序列的迅速发展,基于EST的SSR标记开发也在日趋增加。目前,EST-SSR标记已在苜蓿[6-7]、羊草[8]、木豆[9]、黑麦草[10]等牧草中得到开发和应用。
柱花草(Stylosanthes spp.)又名笔花豆,为多年生豆科植物,原产于拉丁美洲。因其适应性强、耐贫瘠、抗旱、饲草产量高、营养价值高,兼具改良土壤、水土保持的功效,是全球热带及亚热带地区广泛种植的优良豆科牧草之一[11]。柱花草属包含约50个种,常用的栽培品种主要来自圭亚那柱花草(Styosanthes guianensis)、有钩柱花草(S. hamata)、糙柱花草(S. scabra)和头状柱花草(S. macrocephala)等。1957年,我国开始柱花草的引种选育与栽培研究,先后选育出维拉诺有钩柱花草(S. hamata ‘Verano)、热研5号圭亚那柱花草(S. guianensis ‘Reyan No. 5)、西卡糙柱花草(S. scabra‘Seca)等12个柱花草优良品种[12]。目前,国内外柱花草遗传学研究的报道以RAPD、AFLP和ISSR等分子标记为主,柱花草SSR标记开发及验证的相关报道非常有限,极大地限制了该标记在柱花草遗传学研究领域中的应用[13]。本课题组已利用热研5号圭亚那柱花草转录组测序数据开发了EST-SSR标记2008个,本研究从中随机挑选117个EST-SSR标记,验证了新开发的EST-SSR标记的有效性和实用性,并分析了其在不同柱花草种间的可转移性,为利用EST-SSR 标记开展柱花草遗传学研究提供参考。
1 材料与方法
1.1 材料
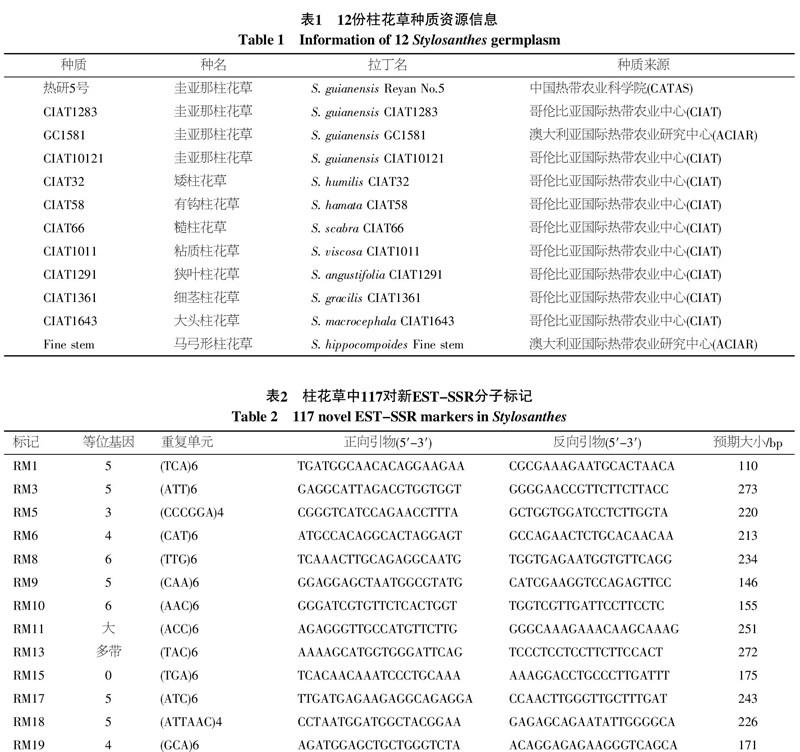
1.1.1 供试材料 来自9个不同柱花草种的12份柱花草种质(表1),包括4份圭亚那柱花草种质和8份其他不同种的柱花草种质,均由中国热带农业科学院热带作物品种资源研究所热带牧草研究中心收集。
1.1.2 试剂 挑选的117对EST-SSR引物由上海生工生物工程技术服务有限公司合成(表2),试验中Taq DNA聚合酶、dNTPs、10×PCR Buffer(含Mg2+)及DL 2 000均购自TaKaRa公司。
1.2 方法
1.2.1 材料种植及取样 将去皮的柱花草种子置于80 ℃水中浸泡5 min,然后在25 ℃恒温条件下的人工智能气候箱中进行发芽,发芽后移植至14 L塑料盆中进行营养液培养。培养1个月后,每份种质随机挑选5株,对叶片进行混合取样,置于液氮保存,用于DNA抽提。
1.2.2 基因组总DNA抽提 利用改良的CTAB法从柱花草叶片中提取基因组总DNA[14],用1.0%琼脂糖凝胶电泳检测基因组DNA,然后用NanoVue超微量分光光度计(GE Healthcare)测定其浓度,将DNA浓度稀释至100 ng/μL后置于-20 ℃冰箱中备用。
1.2.3 PCR扩增及产物检测 PCR扩增反应体系为20 μL,包括2.0 μL 10×PCR Buffer(含Mg2+),1.0 μL模板DNA(100 ng/μL),0.4 μL正向引物 (10 μmol/L),0.4 μL反向引物(10 μmol/L),1.2 μL dNTPs(2.5 mmol/L),0.3 μL Taq DNA 聚合酶(5 U/μL),剩余体积用ddH2O补足。PCR反应扩增程序为:94 ℃预变性5 min;94 ℃变性30 s,55 ℃退火45 s,72 ℃延伸45 s,循环35次;最后72 ℃延伸10 min,4 ℃保存。扩增产物用8% PAGE(Polyacrylamide Gel Electrophoresis,聚丙烯酰胺凝胶电泳)电泳后,采用硝酸银法染色,最后照相记录[14]。
1.3 数据分析
扩增产物片段大小以DL 2 000 DNA marker作为分子量参考来读取,对大小合适、清晰且易于辨认的谱带采用“0,1”系统记录其位置,无条带记为“0”,有条带记为“1”。所记录的原始数据汇总到Excel表格中并形成数据矩阵,用于扩增条带的多态性比较和聚类分析。利用NTSYS-pc 2.10e软件分析柱花草种质间的遗传相似系数(Genetic Similarity, GS),然后利用GS值采用非加权组平均法(Unweighted Pair-group Method with Arithmetic Means, UPGMA)对供试材料进行聚类分析[15]。利用POPGENE 1.32软件计算每个位点的遗传多样性参数,包括等位基因数、观察杂合度(Ho)、期望杂合度(He)和Shannon指数[16]。每个位点的多态性信息含量(Polymorphic Information Content, PIC)按公式PIC=1-∑k Pi2进行计算,k表示每个位点检测到的等位基因数目,Pi表示第i个等位基因在供试材料中出现的频率[17]。
2 结果与分析
2.1 柱花草新EST-SSR标记的有效性
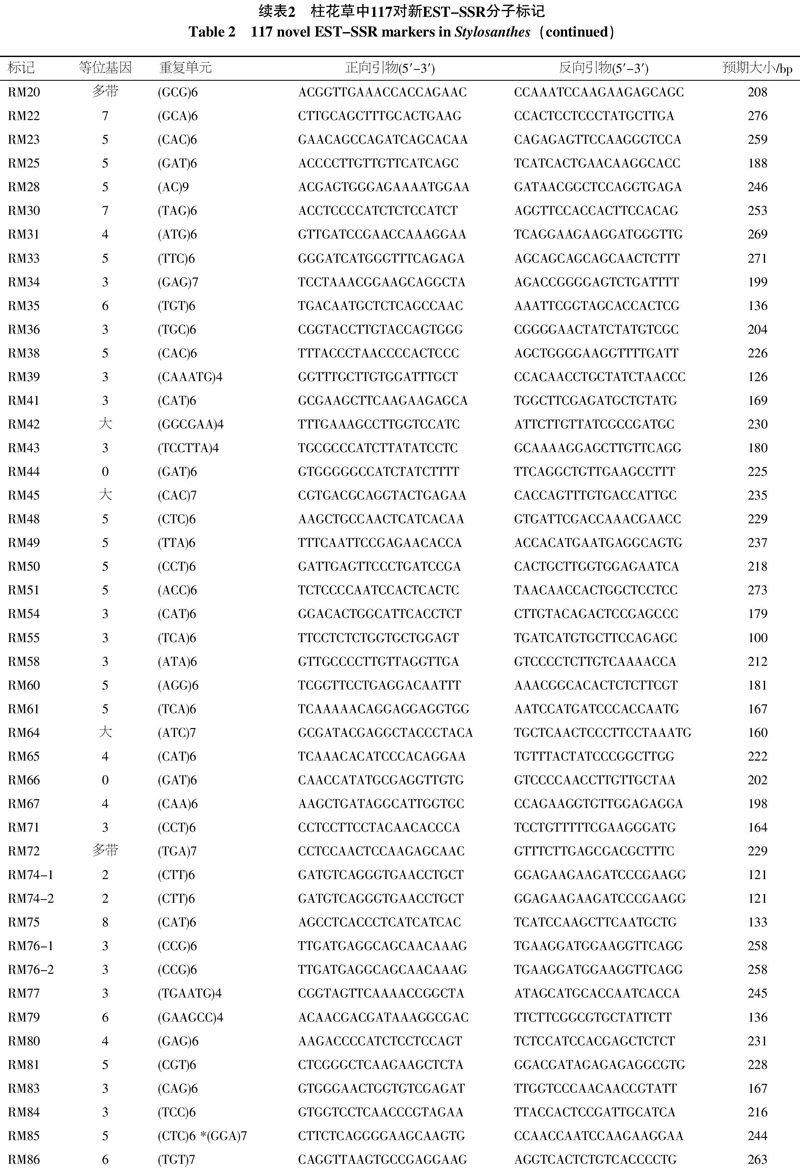
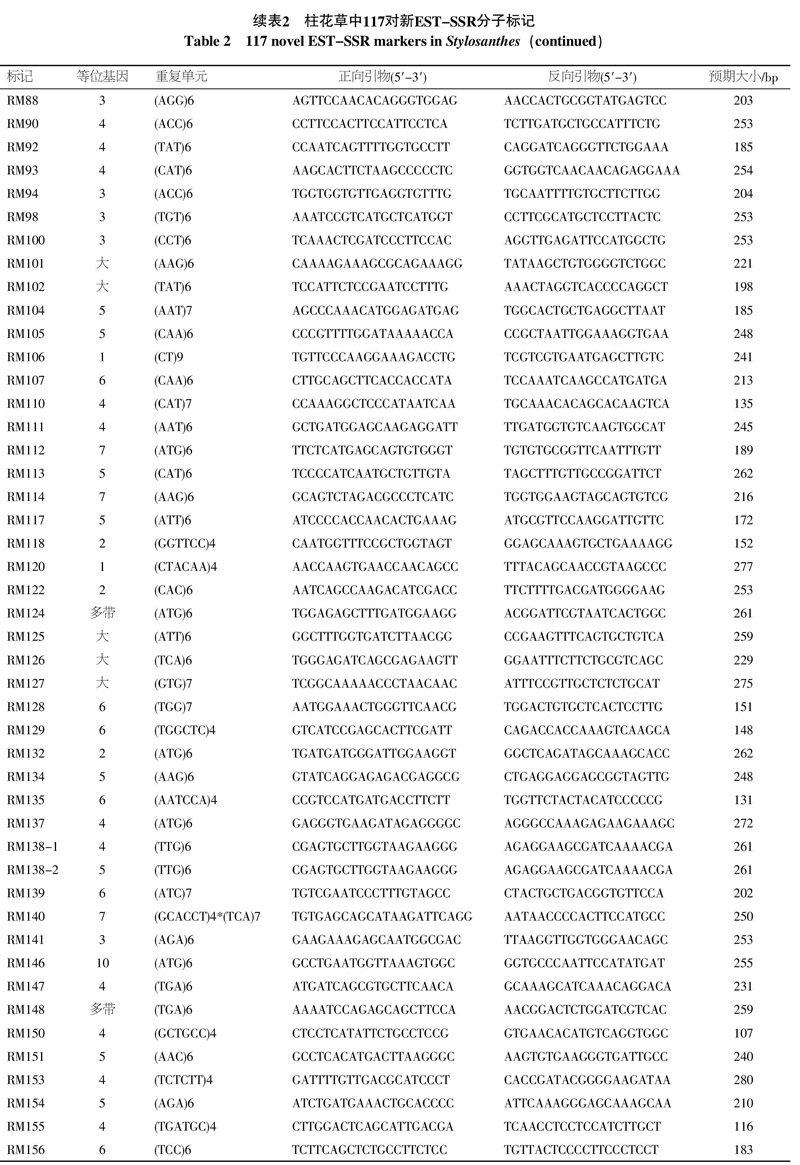
在转录测序数据拼接过程中存在的误差、EST对应DNA 序列存在的内含子或引物存在的非特异性结合位点等原因,都会引起其对应EST-SSR标记无效。本研究结果表明:在117对EST-SSR引物中,98对在4份圭亚那柱花草种质中均可获得大小合适、条带清晰的谱带,确定为有效引物,占总引物的83.76%。剩下的19对中有9对扩增产物偏大、5对(RM15、RM44、RM66、RM164和RM170)没有产物、5对(RM13、RM20、RM72、RM124和RM148)有多条带(表2)。
2.2 圭亚那柱花草新EST-SSR标记在8种柱花草间的可转移性
不同的EST-SSR标记在柱花草种间的转移率不同。在98个有效引物检测到的101个SSR位点中,有31个位点至少在1个柱花草种中无扩增产物(表3),其中有10个SSR位点只在1种柱花草中检测不到,可转移率为87.5%;有10个SSR位点在2种柱花草中不能被检测到,可转移率为75.0%;有4个SSR位点在3种柱花草中检测不到,可转移率为62.5%;有2个SSR位点(RM132和RM153)在4种柱花草中检测不到,可转移率为50.0%;有4个SSR位点在6种柱花草中不能被检测到,可转移率为25.0%;只有1个SSR点(RM74-1)在7种柱花草中不能被检测到,可转移率为12.5%。剩下的70个位点(占总位点数的69.31%)在供试的其他8个柱花草种中都可以检测到(表3),说明大部分新EST-SSR标记在不同柱花草种中具有较高的通用性。
98个有效EST-SSR引物在8种不同柱花草种间的可转移性存在较大的差异,可转移率为82.2%~100%(表3)。其中,在大头柱花草(S. macrocephala)中可检测到有效位点83个,可转移率为82.2%,在粘质柱花草(S. viscosa)和狭叶柱花草(S. angustifolia)中均可检测到有效位点84个,可转移率为83.2%。相比而言,在有钩柱花草(S. hamata)和糙柱花草(S. scabra)中有92个有效位点可被检测到,可转移率达到91.1%,在矮柱花草(S. humilis)和细茎柱花草(S. gracilis)中有94个有效位点可被检测到,可转移率达到93.1%,而在马弓形柱花草(S. hippocampoides)有效引物的可转移率达到了100%。由此可见,不同柱花草种的遗传背景存在明显差异,因而检测得到EST-SSR位点的数量和位置也表现出差别,说明筛选获得的可转移性EST-SSR引物可以对不同柱花草种进行有效区分。
2.3 EST-SSR标记特征分析
在98对有效引物中共检测到SSR位点101个,其中RM74、RM76和RM138均扩增出2个SSR位点(表2)。在101个检测到的SSR位点中,只有2个位点(RM106和RM120)在12份柱花草种质中没有多态性,其他99个位点均有多态性,共检测到456个等位基因,平均每个位点4.61个等位基因(表2)。其中6个位点有2个等位基因,21个位点有3个等位基因,20个位点有4个等位基因,29个位点有5个等位基因,13个位点有6个等位基因,7个位点有7个等位基因,RM75、RM146和RM163分别有8、10和11个等位基因(图1)。
利用POPGENE 1.32软件分析每个位点的遗传多样性参数,结果显示,99个多态性位点的观察杂合度(Ho)的变化范围为0.000~0.400,平均值为0.083,而期望杂合度(He)的变化范围为0.159~0.928,平均值为0.658(表4)。各多态性位点的PIC值变化范围为0.153~0.889,平均为0.629,其中PIC<0.25的只有2个分子标记(RM74-2和RM172),而PIC>0.5的有78个位点,占总多态性位点的78.79%。位点RM74-2的PIC值最低为0.153,RM146的PIC值最高为0.889(表4)。各多态性位点的Shannon指数变异范围为0.287~2.268,其平均值为1.228,同样表明了新开发EST-SSR标记较好的多态性。
2.4 遗传多样性分析
利用NTSYS-pc 2.10e分析软件,根据98个有效引物的数据分析12份柱花草种质的遗传多样性,结果表明,所有供试材料间的遗传相似系数GS变化范围为0.57~0.87,平均为0.69,说明这12份柱花草种质间的遗传多样性比较丰富。其中,圭亚那柱花草热研5号与圭亚那柱花草GC1581的遗传相似性最大,GS值为0.87,这可能是因为它们来自圭亚那柱花草种中相同的亚种(圭亚那亚种);其次,糙柱花草CIAT66和粘质柱花草CIAT1011的遗传相似性也较大,GS值为0.80,可能是由于粘质柱花草是糙柱花草祖先之一的原因[18];而圭亚那柱花草GC1581和糙柱花草CIAT66的遗传相似性最小,GS值为0.57。
基于多态性SSR引物的UPGMA聚类分析结果表明,12份来自不同种的柱花草种质可以明显地被分为2大类(图2)。热研5号柱花草、GC1581柱花草、CIAT1283柱花草、CIAT10121柱花草、Fine stem柱花草和CIAT1361柱花草被归为一类,其中前4份种质都属于圭亚那柱花草,Fine stem柱花草属于马弓形柱花草,CIAT1361柱花草属于细茎柱花草,虽然它们属于不同的种,但是它们具有相同类型的基因组(G类)[17]。剩下的矮柱花草CIAT58、狭叶柱花草CIAT1291、有钩柱花草CIAT58、糙柱花草CIAT66、粘质柱花草CIAT1011和大头柱花草CIAT1643被归为一类。其中有钩柱花草CIAT58、糙柱花草CIAT66和粘质柱花草CIAT1011虽然属于不同的种,倍性也不相同,但拥有相同类型的基因组(有钩柱花草A类,糙柱花草AB类,粘质柱花草A类)[18],所以它们的亲缘关系比较近。
3 讨论与结论
本研究检测的117对EST-SSR引物中,98对可获得大小合适、条带清晰的谱带,有效引物比例(83.76%)和甘薯中EST-SSR引物的有效引物比例(84.6%)相近[19],高于木豆(71.24%)[9]、苜蓿(62.96%)[7]和茶叶(59.9%)[20],但低于苎麻(98.0%)[21]。引物没有扩增产物可能是因为EST-SSR位点对应的DNA序列中存在较大内含子或不同剪切方式造成的,扩增产物偏大可能是由于EST-SSR位点对应的DNA序列中存在内含子造成的,而扩增条带偏多可能是因为引物非特异性结合引起的。
不同SSR引物对应序列的保守性不一样,所以不同SSR引物在柱花草种间的可转移性不同。本研究98对有效引物共检测到101个SSR位点,在8种柱花草间的可转移率为12.5%~100%,平均可转移率为89.36%。Chandra等[12]开发的21个genomic-SSR标记在柱花草种间的可转移率为0~100%,平均仅为45.24%,而20个EST-SSR标记在柱花草种间的可转移率为40%~100%,平均为82.0%。由于不同柱花草种遗传背景的差异,SSR引物在不同柱花草种间的可转移性存在较大差异。本研究有效EST-SSR标记在8种不同柱花草中的可转移率为82.2%~100%,平均为89.63%。Santos等[22]开发的genomic-SSR标记在3种柱花草间的可转移率为35%~50%,平均为40.0%。Chandra等[12]开发的genomic-SSR标记在4种柱花草间的可转移率为38.1%~61.9%,平均为45.25%,而EST-SSR标记在5种柱花草间的可转移率为78.9%~94.7%,平均为85.72%。无论是单个标记还是在不同种间,EST-SSR标记的可转移率都明显大于genomic-SSR。
据Bostein等[23]提出的衡量分子标记多态性标准,当PIC>0.5时,该分子标记为高多态性标记,当0.5>PIC>0.25时,该分子标记为中多态性标记,当PIC<0.25时,该分子标记为低多态性标记。本研究结果显示99个多态性位点中,PIC>0.5的有78个位点,占总多态性位点的78.79%,表明新开发的EST-SSR标记具有很好的多态性和应用价值。
聚类分析可将12份柱花草种质分为2大类(图2),这与利用STS标记[24-25],RFLP标记[25-26]和ITS标记[27]对柱花草种间亲缘关系的研究结果相一致。在Ⅰ类中,3种柱花草的基因组都属于G类。马弓形柱花草和细茎柱花草原本都被视为圭亚那柱花草的一个亚种[18],Calles和Schultze-Kraft[28]将细茎柱花草重新定义为一个种,而马弓形柱花草至今无定论。在Ⅱ类中,虽然有钩柱花草CIAT58、糙柱花草CIAT66和粘质柱花草CIAT1011属于不同的种,但它们拥有相同类型的基因组(A类),所以亲缘关系比较近[18]。
参考文献
[1] Zane L, Bargelloni L, Patarnello T. Strategies for microsatellite isolation: a review[J]. Molecular Ecology, 2002, 11(1): 1-16.
[2] Kalia R K, Mai M K, Kalia S, et al. Microsatellite markers: an overview of the recent progress in plants[J]. Euphytica, 2011, 177(3): 309-334.
[3] Tan C, Wu Y, Taliaferro C M, et al. Development of simple sequence repeat markers for bermudagrass from its expressed sequence tag sequences and preexisting sorghum SSR markers[J]. Molecular Breeding, 2012, 29(1): 23-30.
[4] Varshney R K, Graner A, Sorrells M E. Genic microsatellite markers in plants: features and applications[J]. Trends in Biotechnology, 2005, 23(1): 48-55.
[5] Ellis J R, Burke J M. EST-SSRs as a resource for population genetic analyses[J]. Heredity, 2007, 99: 125-132.
[6] Liu Z, Chen T, Ma L, et al. Global transcriptome sequencing using the Illumina platform and the development of EST-SSR markers in autotetraploid alfalfa[J]. PLoS One, 2013, 8(12): e83549.
[7] Wang Z, Yu G, Shi B, et al. Development and characterization of simple sequence repeat(SSR)markers based on RNA-sequencing of Medicago sativa and in silico mapping onto the M. truncatula genome[J]. PLoS One, 2014, 9(3): e92 029.
[8] Chen S, Huang X, Yan X, et al. Transcriptome analysis in sheepgrass(Leymus chinensis): a dominant perennial grass of the Eurasian Steppe[J]. PLoS One, 2013, 8(7): e67974.
[9] Dutta S, Kumawat G, Singh B P, et al. Development of genic-SSR markers by deep transcriptome sequencing in pigeonpea[Cajanus cajan(L.)Millspaugh][J]. BMC Plant Biology, 2011, 11: 17.
[10] Studer B, Asp T, Frei U, et al. Expressed sequence tag-derived microsatellite markers of perennial ryegrass(Lolium perenne L.)[J]. Molecular Breeding, 2008, 21(4): 533-548.
[11] 蒋昌顺.柱花草的研究进展[J]. 热带作物学报, 2005, 26(4): 104-108.
[12] 白昌军, 刘国道, 陈志权, 等. 热研20号太空柱花草选育研究报告[J]. 热带作物学报, 2011, 32(1): 33-41.
[13] Chandra A, Tiwari K K, Nagaich D, et al. Development and characterization of microsatellite markers from tropical forage Stylosanthes species and analysis of genetic variability and cross-species transferability[J]. Genome, 2011, 54(12): 1 016-1 028.
[14] 张 龙, 丁西朋, 严琳玲, 等. 8种柱花草属牧草SSR-PCR反应体系优化及引物筛选[J]. 草业科学, 2014, 31(2): 233-242.
[15] Rohlf F J. NTSYS-pc Numerical taxonomy and multivariate analysis system, version 2.10[M]. New York: Exeter Software, 2000.
[16] Yeh F C, Yang R C, Boyle T. POPGENE, version 1.32: the user friendly software for population genetic analysis[M]. Molecular Biology and Biotechnology Centre, University of Alberta, Edmonton, AB, Canada, 1999.
[17] Varshney R K, Grosse I, Hahnel U, et al. Genetic mapping and BAC assignment of EST-derived SSR markers shows non-uniform distribution of genes in the barley genome[J]. Theoretical and Applied Genetics, 2006, 113(2): 239-250.
[18] Maass B L, Sawkins M. History, relationships and diversity among Stylosanthes species of commercial significance[M] // Chakraborty S ed. High-yielding anthracnose-resistant Stylosanthes for agricultural systems. Australia: Australian Centre for International Agricultural Research(ACIAR), 2004: 9-26.
[19] Wang Z, Li J, Luo Z, et al. Characterization and development of EST-derived SSR markers in cultivated sweetpotato(Ipomoea batatas)[J]. BMC Plant Biology, 2011, 11: 139.
[20] Tan L Q, Wang L Y, Wei K, et al. Floral transcriptome sequencing for SSR marker development and linkage map construction in the tea plant(Camellia sinensis)[J]. PLoS One, 2013, 8(11): e81 611.
[21] Liu T, Zhu S, Fu L, et al. Development and characterization of 1, 827 expressed sequence tag-derived simple sequence repeat markers for ramie(Boehmeria nivea L. Gaud)[J]. PLoS One, 2013, 8(4): e60 346.
[22] Santos M O, Karia C T, Resende R M, et al. Isolation and characterization of microsatellite loci in the tropical forage legume Stylosanthes guianensis(Aubl.)Sw[J]. Conservation Genetics Resources, 2009, 1(1): 43-46.
[23] Botstein D, White R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. The American Journal of Human Genetics, 1980, 32(3): 314-331.
[24] Vander Stappen J, Weltjens I, Van Campenhout S, et al. Genetic relationships among Stylosanthes species as revealed by sequence-tagged site markers[J]. Theoretical and Applied Genetics, 1999, 98(6-7): 1 054-1 062.
[25] Liu C J, Musial J M, Thomas B D. Genetic relationships among Stylosanthes species revealed by RFLP and STS analyses[J]. Theoretical and Applied Genetics, 1999, 99(7-8): 1 179-1 186.
[26] Gillies A C M, Abbott R J. Phylogenetic relationships in the genus Stylosanthes(Leguminosae)based upon chloroplast DNA variation[J]. Plant Systematics and Evolution, 1996, 200(3-4): 193-211.
[27] Vander Stappen J, Marant S, Volckaert G. Molecular characterization and phylogenetic utility of the rDNA external transcribed spacer region in Stylosanthes(Fabaceae)[J]. Theoretical and Applied Genetics, 2003, 107(2): 291-298.
[28] Calles T, Schultze-Kraft R. Re-establishment of Stylosanthes gracilis(Leguminosae)at species level[J]. Kew Bulletin, 2010, 65(2): 233-240.

