单核苷酸多态性芯片与染色体核型分析在唐氏筛查高风险孕妇产前诊断中的比较研究*
白小艺, 章 钧, 田 琪, 林俊伟, 侯红瑛△
(中山大学附属第三医院 1产科,2产科实验室,广东 广州 510630)
单核苷酸多态性芯片与染色体核型分析在唐氏筛查高风险孕妇产前诊断中的比较研究*
白小艺1, 章 钧2, 田 琪2, 林俊伟2, 侯红瑛1△
(中山大学附属第三医院1产科,2产科实验室,广东 广州 510630)
目的: 探讨单核苷酸多态性芯片(SNP array)在唐氏筛查高风险孕妇胎儿染色体分析中的应用价值。方法: 选取312例因唐氏筛查高风险的孕妇,行羊膜腔穿刺术后获得羊水,对羊水进行G显带核型分析和SNP array检测,比较核型分析与SNP array检测结果,并按年龄分组比较拷贝数变异(CNVs)的发生率差别。结果: 核型分析和SNP array均准确发现2例21三体(0.64%),6例核型分析提示染色体平衡重组(1.92%)的样本经SNP array分析证实不存在重排片段重复或缺失。在303例核型正常的胎儿羊水细胞中,SNP array检测发现176例CNVs,其中良性CNVs 106例,临床意义不明确的CNVs(VOUS)61例,新发CNVs(denovoCNVs)9例,未发现已知的致病性CNVs。唐氏筛查高风险组与唐氏筛查高风险合并高龄组CNVs的分布差别无统计学意义(P>0.05)。此外,本研究中首次报道14种CNVs。结论: SNP array 可进一步确定核型分析的平衡易位是否存在染色体微缺失/重复。在核型正常的胎儿中,SNP array可检测出大量拷贝数异常,发现14种新的CNVs但现有数据库无法判断其临床意义,需进一步研究确认。此外,孕妇年龄对胎儿基因组中新发CNVs的发生率无明显影响。
单核苷酸多态性芯片; 染色体核型分析; 拷贝数变异; 产前诊断
20世纪70年代,以G显带技术为主的染色体核型分析在产前诊断中开始应用,可减少染色体数目和结构异常的胎儿出生的数量, 提高人口出生素质。但每年仍有0.65%新出生人口存在染色体异常导致先天性疾病的发生,0.2%新出生人口存在染色体重排并影响其生殖功能[1-2]。核型分析能检测出染色体数目异常,不平衡结构畸形,明显的平衡结构异常等染色体畸变,但核型分析存在检测深度低、耗时长、培养失败率较高、判读结果主观及易发生人为错误等缺点,使它在产前诊断的应用存在一定的局限性[3]。
近年来,以微阵列-比较基因组杂交(array comparative genomic hybridisation,aCGH)和单核苷酸多态性芯片(single nucleotide polymorphism array,SNP array)为平台的染色体微阵列技术(chromosome microarray analysis,CMA)用于临床细胞遗传学领域及染色体病的研究,可对染色体亚微结构异常进行诊断定位。CMA在产后发育迟缓/智力障碍、自闭症谱系障碍和多发性先天畸形疾病中检测出比核型分析高15%~20%检出率,可取代G显带分析技术,成为一线检测手段[4-6]。但其在产前诊断中,特别是对于唐氏筛查高风险的孕妇进行产前诊断的应用价值尚不明确。为了研究SNP array是否比传统核型分析对遗传物质的异常具有更高检出率,我们对312例因唐氏筛查高风险而行产前诊断的孕妇胎儿样本进行了核型分析和SNP array检测,探讨SNP array在唐氏筛查高风险孕妇人群中的应用价值。
材 料 和 方 法
1 研究对象
2013年3月~2014年8月在中山大学附属第三医院因唐氏筛查高风险(早孕或中孕唐氏筛查21三体风险值≥1∶270)行羊膜腔穿刺术进行产前诊断的孕妇,排除染色体培养失败和(或)SNP array分析失败以及拒绝行SNP array检测的病例,共收集312例孕妇的羊水标本成功进行核型分析及SNP array检测。312例孕妇的一般情况如表1所示。
表1 行SNP array检测312例孕妇的一般情况
Table 1.Basic information of 312 pregnant women for SNP array analysis

VariableData(Mean±SD)Data[n(%)]Age(year)31.85±5.13—Gestationalweeks20.01±2.08—Parity Nulliparity—170(54.49)Multiparity—142(45.51)Indication DSabnormality—210(67.31)DSabnormality+advancedage—102(32.69)
DS: Down’s syndrome.
2 核型分析实验步骤
行羊膜腔穿刺术抽取羊水20 mL,分离后加入Gibco羊水培养基,置于37 ℃、5% CO2中培养10~14 d,收获羊水细胞经过消化、固定、制片、烤片、显带与染色制得玻片。最后在油镜下计数分析细胞核型。每1标本计数20个核型,至少分析3个核型,记录核型分析结果。
3 SNP array检测过程
2013年3月1日~2013年6月5日共有50例孕妇羊水细胞SNP array采用Illumina Human Cyto SNP12芯片(含30万个遗传标记探针)进行分析,2013年6月6日~2014年8月31日共有262例孕妇羊水细胞SNP array采用OmniZhongHua-8(含90万个遗传标记探针)进行分析。基本步骤包括提取羊水细胞DNA,DNA扩增、片段化、沉淀、重悬、与芯片杂交、芯片的延伸、染色及包被、芯片扫描及数据分析等。SNP array发现的拷贝数变异(copy number variants,CNVs)>100 kb则通过实时荧光PCR、多重连接探针扩增(multiplex ligation-dependent probe amplification,MLPA)或高通量测序进行确定。对CNVs的性质和判读参考实验室内部数据库、在线公共数据库DECIPHER、ISCA、DGV和Pubmed。根据检索的结果及父母的比对,将CNVs分为良性(benign)CNVs、致病性(pathogenicity)CNVs、不明意义的CNVs(variants of unknown significance,VOUS)、未包含基因片段的无义(no significance)CNVs及新发现(denovo)CNVs[5]。
4 统计学处理
采用统计软件SPSS 19.0处理,定量资料的描述性分析以均数±标准差(mean±SD)表示,两组间分类资料的比较使用2检验,以P<0.05为差异有统计学意义。
结 果
1 核型分析结果
312例孕妇羊水细胞经过培养后均成功进行核型分析,303例胎儿核型呈正常核型,占所有孕妇97.12%; 21三体综合征的胎儿2例,占0.64%;染色体臂间倒位5例,占1.60%,其中发生在5号染色体1例,9号染色体4例;嵌合型染色体易位1例,是发生在8号和11号染色体之间的易位;嵌合型marker染色体1例,见表2。
2 SNP array结果分类
在312例孕妇的羊水细胞进行SNP array检测,排除<100 kb的检测结果,归纳相同种类的CNVs后,共检测出325种≥100 kb的CNVs,其中87种CNVs内未包含或覆盖基因,即为无义CNVs,占26.77%;在238种 包含基因的CNVs中,其中良性CNVs有179种,占55.08%; VOUS有45种,占13.84%;未发现明确致病性CNVs。在检测出的CNVs中,良性和无义的CNVs占多数,为81.85%(266/325)。上述CNVs中未在数据库中有报道我们新发现的CNVs有14种,占4.31%,见表3。
表2 312例唐氏筛查高风险孕妇羊水细胞核型分析结果
Table 2.Chromosome karyotyping results of 312 pregnancy with Down’s syndrome screening abnormality

KaryotypenProportion(%)46,XX15449.3646,XY14947.7647,XX,+2120.6446,XX,inv(5)(p15.3q11)10.3246,XX,inv(9)(p11q13)10.3246,XY,inv(9)(p11q13)30.9647,XX,+mar/46,XX10.3246,XX,t(8;11)(q12.2;p15)/46,XX10.32
Inv: inversion; t:translocation; mar:marker chromosome.
表3 312例孕妇羊水细胞SNP array 分析结果
Table 3.SNP array analysis results of 312 pregnancy with Down’s syndrome screening abnormality

CNVsnProportion(%)Nosignificance8726.77Benign17955.08Pathogenicity00VOUS4513.84Denovo144.31Total325100.00
3 对染色体畸变检测结果比较
染色体畸变,即染色体数目和结构异常,核型分析检出率为2.88%(9/312),包括数目异常3例(2例为21三体和1例marker染色体)和结构异常(染色体臂间倒位5例和易位1例),见表2。SNP array检测出2例21三体的羊水细胞,与核型分析结果一致;对于平衡染色体重排,包括核型能检测出的倒位和易位,在我们的研究中,检出率为0,能够进一步确定所检测样本的染色体重排为平衡的,不存在染色体片段的缺失和重复。本次研究中,有一例嵌合型含marker染色体的病例,含marker的细胞比例为26.67%,该病例SNP array未能检测出具有临床意义的CNVs,这也更加确定该marker染色体中不包含染色体微重复/缺失,证实该marker染色体不具有致病性。总体看来,SNP array可检测出非整倍体数目异常,对于平衡的染色体重排不能检出,见表4。
表4 核型分析与SNP array检测染色体畸变结果比较
Table 4.Comparison of the results between karyotyping and SNP array in detecting chromosome abnormality

TypeKaryotyping(n)SNParray(n)21trisomy22Inversion50Translocation10Mosaicmarkerchromosome10Total92
4 303例染色体核型正常的胎儿SNP array的结果
在303例核型正常(46,XX或46,XY)的胎儿中,对其SNP array的结果进行进一步的分析。在统计过程中,若胎儿既有良性CNVs又有VOUS时,将其归类为VOUS组;若胎儿既有VOUS又有新发现的CNVs,将其归类为VOUS组;若胎儿既有良性CNVs又有新发现的CNVs,将其归类为新发现CNVs。在303例核型正常的胎儿中,106例胎儿CNVs为良性,更加确定其遗传物质不具有致病性;61例胎儿CNVs为意义不明的CNVs。此外,在9例胎儿中发现现有数据库尚未报道的CNVs,为我们首次报道的CNVs。同时,我们将年龄进行分组,探讨高龄与CNVs性质的关系发现,高龄不会导致VOUS发生风险增高(P>0.05),见表5。
5 新发现的CNVs
在本次研究中,我们发现14种未在现有数据库中有报道的CNVs,如表6所示。这些CNVs的意义不明,需要我们对其进行追踪,以明确这些CNVs的意义。同时,这些CNVs也可丰富我们当地及国际数据库,为以后的研究者提供数据参考。
讨 论
近年来,随着 aCGH 和 SNP array技术出现和不断成熟, CMA迅速发展成为一个了解基因和患者遗传病因的有力工具[7]。传统核型分析检测深度平均只有7~10 Mb,染色体微阵列分析可以检测出5~100 kb的亚微结构 CNVs,比核型分析的检测阈值提高了至少100倍。染色体微阵列分析在产后发育迟缓/智力低下、多发先天性畸形、自闭症谱系疾病中已取代核型分析成为主要的检测方法,可以检出比核型分析多10%~15%的染色体亚微结构重排[5]。但染色体微阵列分析在临床产前诊断中的应用仍缺乏大样本前瞻性的队列研究,在产前诊断中的适用人群、应用指征、结果解释等方面尚未得到一致的意见。2013年美国妇产科医师协会和母胎医学会发布了染色体微阵列分析在产前诊断中应用的实践指南,建议在产前超声发现胎儿有一个或多个结构异常的孕妇行介入性产前诊断时,推荐使用染色体微阵列分析,可取代核型分析;在超声提示胎儿结构正常的孕妇行介入性产前诊断时,可使用核型分析或染色体微阵列分析[3]。

表5 303例核型正常的胎儿SNP array的结果比较
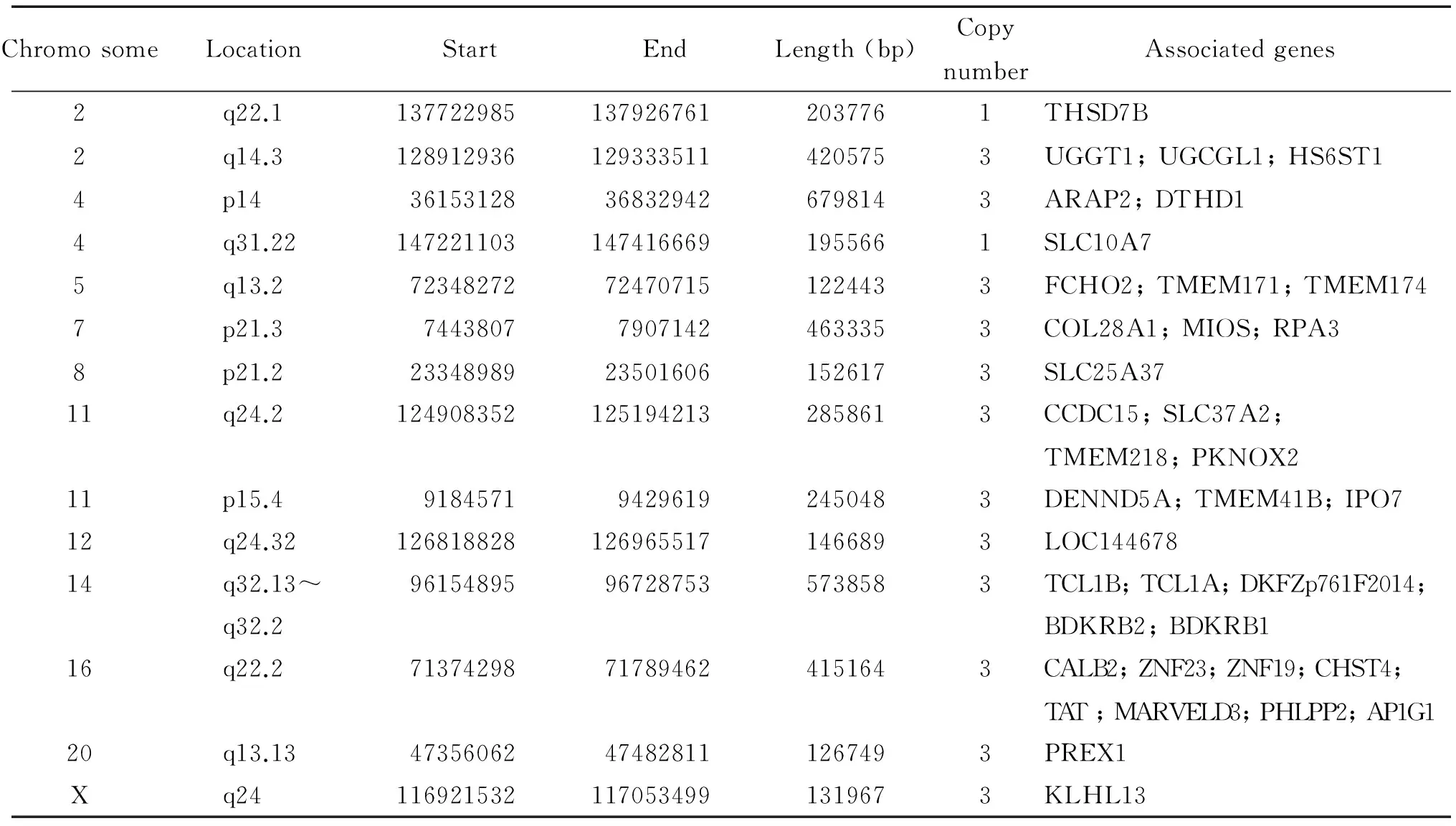
表6 14种新发现的CNVs
在本次研究中,我们成功地对312例因唐氏筛查高风险行介入性产前诊断的孕妇进行了核型分析和SNP array检测,对比SNP array和核型分析对染色体畸变的检测结果,核型分析和SNP array均一致地检测出2例(0.64%)21三体(47,XX,+21)胎儿,这与Wapner等[8]研究结果一致,SNP array能准确检测非整倍体数目异常。染色体平衡易位和倒位的发生率为0.7%,由于染色体的平衡重组,未涉及遗传物质的获得或丢失,现有染色体微阵列分析技术不能检测出染色体的平衡重组[9]。Wapner等[8]在4 282例患者中,核型分析发现了40例染色体平衡重组,CMA均未成功检出。在我们的研究中,SNP array也未能检测出6例(1.92%)核型发现的染色体平衡(包括5例染色体臂间倒位和1例易位),这也就更加确定这6例胎儿的染色体重组是平衡的,未涉及遗传物质的获得和丢失。此外,遗传性平衡易位导致胎儿产生明显的表型的风险较低,一般在胎儿本身不会有致病性,但是可能会对胎儿的成年后的生育产生影响[8, 10],这需要在产前咨询中与父母准确解释。
在染色体微阵列分析中, VOUS增加了临床咨询和处理的复杂性,同时,这也可能导致患者及家属心理上的焦虑以及造成正常妊娠的终止[11]。关于VOUS的检测率,各个研究报道不一致,平均为2.3%[9, 12-13]。造成VOUS检出差异的原因之一在于使用的染色体微阵列分析的平台的检测深度不一致,CMA检测率越高,VOUS的数量越多。Hillman等[14]对比了12个队列研究发现,VOUS的检出率与CMA整体检出率是呈正相关关系(Spearman系数=0.81,P<0.01)。减少VOUS的结果,可通过检测双亲的染色体、规范CNVs的判断标准以及及时更新数据库的信息等,可将VOUS控制在1%范围内[10]。我们的研究中,在61例(20.13%)胎儿中发现VOUS,明显高于国外的其它研究,其中原因之一为未对胎儿双亲进行SNP array的检测。由于SNP array的费用比较昂贵,是传统核型分析的2~3倍,以及对SNP array的价值的认识不足,胎儿双亲绝大多数拒绝继续行SNP array,这是我们此次研究的不足之处。再者,我们研究中86.47%(262/303例)的SNP array使用的是OmniZhongHua-8芯片,含有90万个遗传标记探针,该芯片首次在我们研究中使用,可参考的数据信息不足。因此,可能造成本次研究中VOUS的检出率远远高于文献报道。在后续的研究中,我们继续追踪胎儿的超声检查结果,以及出生后的情况,以明确这部分VOUS的意义。
目前,对于母体血清学异常的孕妇行胎儿染色体微阵列分析的研究较少,且研究中的样本量少。对于母体血清学异常,文献报道,胎儿致病性CNVs检出率为1.54%(0~5.2%),共 28/1 810例。对于高龄孕妇行CMA检测,致病性CNVs检出率为1.16%(0.3~1.7%),共74/6 359例[8-10, 12-13, 15-17]。在我们研究中唐氏筛查高风险的孕妇行SNP array检测,未检出致病性CNVs,检出率为0,这与Fiorentino等[9]及Lee等[15]的报道一致。因此,在唐氏筛查异常的孕妇行介入性产前诊断时,进行SNP array检测是否能提高检出率,SNP array对这部分孕妇是否有价值仍需要进一步更大样本的研究。
CNVs在人类基因组中是很常见的。不同的人种、民族等,可能会有不同的CNVs富集。在本次研究中,我们首次报道14种新发现的CNVs,在现有的数据库中未找到类似的CNVs。对于这部分CNVs,我们应进行双亲的检测,以确定这部分CNVs是家族遗传的还是新突变的;对于新突变的CNVs以及这部分CNVs的意义,需对胎儿进行超声检查以及出生后情况的追踪,以明确该部分CNVs的性质,丰富CMA数据库,以便CMA在产前诊断中得到更好的应用。
总之,本次研究是首次在中国大陆地区唐氏筛查高风险的孕妇中进行大样本SNP array检测。SNP array能准确检测非整倍体数目异常,不能检测染色体平衡重组,SNP array 可进一步确定核型分析的平衡易位是否存在微缺失/重复。在核型正常的胎儿中,我们发现20.13% 的胎儿中出现VOUS,未检测出致病性CNVs。同时,我们首次报道检测出14种新发现CNVs,而VOUS和denovoCNVs的临床意义需要进一步跟踪认识。
[1] Alberman ED, Creasy MR. Frequency of chromosomal abnormalities in miscarriages and perinatal deaths[J]. J Med Genet, 1977, 14(5): 313-315.
[2] Obstet Gynecol. ACOG Practice Bulletin No. 88, December 2007. Invasive prenatal testing for aneuploidy[J]. Obstet Gynecol, 2007, 110(6):1459-1467.
[3] Obstet Gynecol. Committee Opinion No. 581: the use of chromosomal microarray analysis in prenatal diagnosis[J]. Obstet Gynecol, 2013, 122(6):1374-1377.
[4] Valduga M, Philippe C, Bach SP, et al. A retrospective study by oligonucleotide array-CGH analysis in 50 fetuses with multiple malformations[J]. Prenat Diagn, 2010, 30(4): 333-341.
[5] Miller DT, Adam MP, Aradhya S, et al. Consensus statement: chromosomal microarray is a first-tier clinical diagnostic test for individuals with developmental disabilities or congenital anomalies[J]. Am J Hum Genet, 2010, 86(5): 749-764.
[6] de Vries BB, Pfundt R, Leisink M, et al. Diagnostic genome profiling in mental retardation[J]. Am J Hum Genet, 2005, 77(4):606-616.
[7] 陈 瑛,蔡光伟. 采用微阵列-比较基因组杂交进行产前诊断的局限性和困难[J]. 中华医学遗传学杂志, 2011, 28(1): 47-51.
[8] Wapner RJ, Martin CL, Levy B, et al. Chromosomal microarray versus karyotyping for prenatal diagnosis[J]. N Engl J Med, 2012, 367(23): 2175-2184.
[9] Fiorentino F, Caiazzo F, Napolitano S, et al. Introducing array comparative genomic hybridization into routine prenatal diagnosis practice: a prospective study on over 1 000 consecutive clinical cases[J]. Prenat Diagn, 2011, 31(13): 1270-1282.
[10]Breman A, Pursley AN, Hixson P, et al. Prenatal chromosomal microarray analysis in a diagnostic laboratory; experience with >1 000 cases and review of the literature[J]. Prenat Diagn, 2012, 32(4): 351-361.
[11]Friedman JM. High-resolution array genomic hybridization in prenatal diagnosis[J]. Prenat Diagn, 2009, 29(1): 20-28.
[12]Shaffer LG, Dabell MP, Fisher AJ, et al. Experience with microarray-based comparative genomic hybridization for prenatal diagnosis in over 5 000 pregnancies[J]. Prenat Diagn, 2012, 32(10):976-985.
[13]Scott F, Murphy K, Carey L, et al. Prenatal diagnosis using combined quantitative fluorescent polymerase chain reaction and array comparative genomic hybridization analysis as a first-line test: results from over 1 000 consecutive cases[J]. Ultrasound Obstet Gynecol, 2013, 41(5): 500-507.
[14]Hillman SC, Mcmullan DJ, Hall G, et al. Use of prenatal chromosomal microarray: prospective cohort study and systematic review and meta-analysis[J]. Ultrasound Obstet Gynecol, 2013, 41(6): 610-620.
[15]Lee CN, Lin SY, Lin CH, et al. Clinical utility of array comparative genomic hybridisation for prenatal diagnosis: a cohort study of 3 171 pregnancies[J]. BJOG, 2012, 119(5): 614-625.
[16]Oneda B, Baldinger R, Reissmann R, et al. High-resolution chromosomal microarrays in prenatal diagnosis significantly increase diagnostic power[J]. Prenat Diagn, 2014, 34(6): 525-533.
[17]Kan AS, Lau ET, Tang WF, et al. Whole-genome array CGH evaluation for replacing prenatal karyotyping in Hong Kong[J]. PLoS One, 2014, 9(2): e87988.
抑制糖原合成酶激酶3可诱导人胰腺癌细胞的促存活自噬信号
糖原合成酶激酶3(glycogen synthase kinase-3, GSK3)是一种在细胞中普遍表达的丝氨酸-苏氨酸激酶,具有多种功能,其中包括对糖原代谢和转录过程的调节。最近Marchand等证明抑制GSK3可通过JNK-cJUN信号通路引起人胰腺癌细胞的凋亡。在此基础之上,他们发现抑制GSK3可通过增强自噬/溶酶体网状系统的活性来诱导促生存信号,制衡死亡信号。在胰腺癌细胞内,自噬溶酶体生物合成的主要转录调节因子、转录因子EB(transcription factor EB, TFEB)均受GSK3的调控。类似于mTOR信号通路的抑制,GSK3抑制剂促进TFEB的核定位,通过内生丝氨酸/苏氨酸磷酸化酶的活动导致TFEB的去磷酸化。然而,GSK3和mTOR信号通路的抑制对TFEB磷酸化的影响其机制和途径是不同的,这表明TFEB受多种激酶和/或磷酸酶的调控。尽管对TFEB磷酸化的影响各异,GSK3和mTOR的抑制剂都可以促进14-3-3分离和TFEB的核定位。定量质谱分析进一步揭示了TFEB与依赖于GSK3和mTOR抑制剂的核蛋白的密切关联,同时也表明它们对TFEB的转录调节功能具有积极的影响。最终,在成熟的胰腺癌细胞可见TFEB充分的核定位,而下调TFEB的表达量明显破坏胰腺癌细胞在非锚泊性方式下的生长能力。此外,TFEB限制性细胞对GSK3抑制引起的凋亡更加敏感。以上研究结果除了提供GSK3对TFEB调控的关键证据外,还揭示了胰腺癌细胞中GSK3所调控的新功能。
J Biol Chem, 2015, 290(9):5592-5605(周 晗)
Comparison between single nucleotide polymorphism array and karyotyping in prenatal diagnosis in Down’s screening abnormal pregnancy
BAI Xiao-yi1, ZHANG Jun2, TIAN Qi2, LIN Jun-wei2, HOU Hong-ying1
(1DepartmentofObstetrics,2LaboratoryofObstetrics,TheThirdAffiliatedHospitalofSunYat-senUniversity,Guangzhou510630,China.E-mail:gdgzhhyy@163.com)
AIM: To evaluate the clinical application of single nucleotide polymorphism array (SNP array) in prenatal diagnosis for screening the abnormality of women with Down’s syndrome (DS). METHODS: The amniotic fluid samples (n=312) collected by amniocentesis for the DS screening abnormality women were tested by karyotyping and SNP array analysis, respectively. The findings of karyotyping and SNP array analysis were compared. RESULTS: Two cases of trisomy 21 were identified by karyotyping and SNP array analysis, but SNP array analysis failed to identify 6 cases of chromosome balanced structural rearrangement. SNP detec ted 176 cases copy number variants (CNVs) in 303 cases normal karyotype were detected by SNP, including 106 benign CNVs, 61 variants of unknown significance (VOUS), 9denovoCNVs, and none of them was pathogenic. The distribution difference of CNVs in DS screening positive group and DS screening positive plus advanced maternal age group was not statistically significant (P>0.05). Furthermore, we reported 14 kinds of CNVs for the first time in population. CONCLUSION: SNP array can further assure chromosome microduplication/microdeletion. In normal karyotype fetus of prenatal diagnosis, SNP can detect some clinical significant CNVs.
Single nucleotide polymorphism array; Karyotyping; Copy number variants; Prenatal diagnosis
1000- 4718(2015)04- 0707- 06
2014- 12- 16
2015- 03- 12
广东省科技计划(No. 2009B060700107); 中山大学达安基因股份有限公司广州市医学诊断技术和产品创新及应用协同创新重大专项合作费(No. 201400000004-4)
R714
A
10.3969/j.issn.1000- 4718.2015.04.024
△通讯作者 Tel: 020-85252172; E-mail: gdgzhhyy@163.com

