结肠癌组织IL-10和IFN-γ mRNA的表达及其临床意义
邓菲丹 罗建业
结肠癌患者机体内免疫微环境的变化已经受到人们的重视,其中辅助性T细胞(Th)亚群Th1/Th2的失衡在恶性肿瘤免疫逃逸与免疫耐受中非常重要[1]。在哺乳动物细胞基因组中,约有3%~5%的胞嘧啶被甲基化,基因甲基化异常与结肠癌变化密切相关[2],因此制定结肠癌Th1、Th2相关细胞因子DNA甲基化谱对结肠癌的早期诊断有重要意义。本文通过检测结肠癌患者结肠组织中Th1/Th2型细胞因子白细胞介素-10(IL-10)和干扰素-γ(IFN-γ)基因启动子区甲基化状态及转录活性,旨在探讨DNA甲基化与基因表达的关系及其在结肠癌中的作用,为结肠癌的诊断和治疗提供新的靶点。
1 资料与方法
1.1 一般资料 随机选取2011年1月-2014年6月本院住院部就诊的80例结肠癌患者组织为结肠癌组、80例相应癌旁组织为癌旁组和100例正常结肠组织为对照组。结肠癌患者年龄34~75岁,中位年龄49岁,男55例,女25例,病理类型均为鳞癌,根据WHO标准分级[3],高分化鳞癌14例,中分化鳞癌42例,低分化鳞癌24例。对照组32~75岁,中位年龄49岁。收集三组患者的结肠组织标本,其中对照组标本来源于结肠息肉组织,结肠癌组标本来源于结肠活检和手术治疗,癌旁组标本来源于结肠癌旁组织手术,结肠癌患者所有手术标本均经过本院病理学专家明确诊断,在患者手术中取病理标本,标本放入液氮中速冻后-80 ℃条件下保存待检,样本获得了伦理委员会批准并签署知情同意书。
1.2 方法
1.2.1 目标标本相关的DNA和RNA提取 均按试剂盒说明书提供的方法提取三组结肠组织DNA与RNA,并采用琼脂糖凝胶电泳并鉴定DNA和RNA完整性。
1.2.2 IL-10和IFN-γ基因启动子区甲基化检测 按照EZ DNA Methylation-GoldTM Kit试剂盒(购自美国Zymoresearch公司)操作说明对DNA进行甲基化修饰及纯化。在NCBI线上检索IFN-γ(J00219)和IL-10(AF418271)基因启动子区序列,Methyl Primer Express V2.0软件进行分析核对参考物[4],亚硫酸氢盐测序法(BSP)检测IL-10基因启动子。基化特异性PCR(MSP)检测IFN-γ基因启动子区非甲基化及甲基化基因,IL-10和IFN-γ基因启动子区序列一甲基化特异性PCR和RT-PCR 引物序列如表1所示。基化结果判断:出现甲基化特异性引物扩增产物判断定为甲基化,仅非甲基化特异性引物扩增产物判断定为非甲基化。

表1 IL-10和IFN-γ基因启动子区序列一甲基化特异性PCR和RT-PCR 引物序列
1.3 统计学处理 数据采用SPSS 17.0软件进行统计学分析,计量资料以 (±s)表示,采用t检验,计数资料采用 字2检验,P<0.05为差异有统计学意义。
2 结果
2.1 三组结肠组织IL-10和IFN-γ mRNA的表达比较 三组IL-10和IFN-γ mRNA 水平比较,差异均有统计学意义(P<0.05),其中结肠癌组和癌旁组IL-10 mRNA均高于对照组(P<0.05),IFN-γ mRNA均低于对照组(P<0.05),结果见表2。

表2 三组结肠组织IL-10和IFN-γ mRNA的表达比较(x-±s) copy/mL
2.2 三组IL-10和IFN-γ基因启动子区甲基化率比较 三组结肠组织IL-10基因启动子区-320、-350、-352、-373位点甲基化率进行相互比较,差异均无统计学意义(P>0.05);三组结肠组织基因启动子区-110、-185位点甲基化率进行相互比较,差异均有统计学意义(P<0.05),其中结肠癌组和癌旁组IL-10基因启动子区-110均高于健康对照组(P<0.05),结肠癌组IL-10基因启动子区-185低于健康对照组(P<0.05),结果见表3;IFN-γ基因启动子区甲基化率结肠癌组>癌旁组>对照组,差异均有统计学意义(P<0.05),结果见表3;三组结肠组织提取DNA的IFN-γ基因启动子区甲基化状态见电泳图1。
2.3 IL-10基因启动子区 通过在线的TFSEARCH软件分析发现,IL-10基因启动子区包含有STAT3或Ets-1、HSF、CCAAT-box、HSF与AP-4、ER共 5个转录因子结合域,见图2。
3 讨论
机体Th1介导的细胞免疫在体内抑制肿瘤生发展中扮演的重要角色,而Th1向Th1漂移,将造成机体免疫抑制状态,使肿瘤能够逃避免疫监视[5-6]。IFN-γ是Th1类分泌的主要较早抗肿瘤细胞因子之一,且协助肿瘤坏死因子-α(TNF-α)诱导Th0细胞向Th1极化;IL-10是Th2类分泌免疫抑制细胞因子,是一种抑制或者下调的Th1型免疫反应的细胞因子,具有多种抑制抗肿瘤免疫应答的物生作用,使肿瘤细胞免疫耐受。IL-10高表达则促进机体肿瘤细胞生长发育。笔者研究结果表明,结肠癌患者IL-10表达水平明显比健康者强,抗肿瘤相关的IFN-γ则低表达,这说明,机体正处于明显的Th1/Th2失衡状态,Th0细胞向Th2漂移极化现象,T细胞对肿瘤抗原肽的无能状态,这是结肠癌患者免疫监视功能严重低下的最主要原因。

表3 三组结肠组织IL-10和 IFN-γ基因启动子区甲基化率比较 例(%)
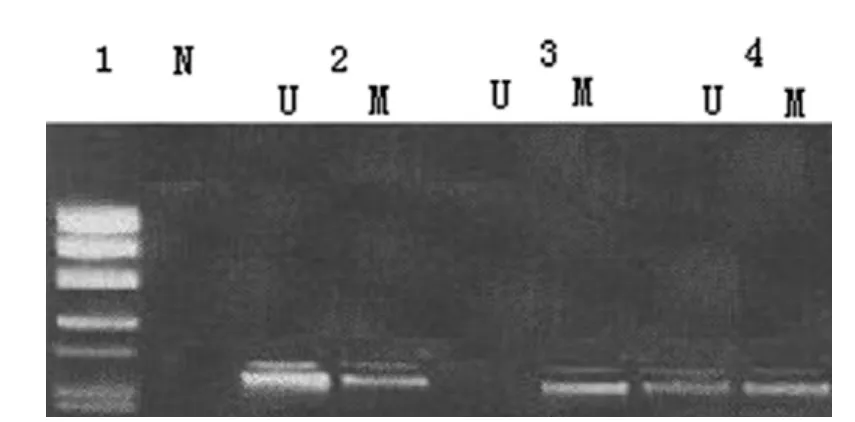
图1 MSP检测三组结肠组织IFN-γ基因启动子区甲基化状态注:1为电泳标记,N为空白对照,U为非甲基化,M为甲基化,2为对照组,3为结肠癌组,4为癌旁组

图2 IL-10基因启动子区与转录因子结合位点预测
DNA甲基化主要在mRNA转录基因水平抑制细胞因子基因的表达,CpG岛甲基化与其所在基因的表达呈反比,即CpG岛甲基化程度越高,mRNA基因表达水平越低,反之则越高[7-8]。本研究对三组不同结肠组织的IL-10和IFN-γ基因的甲基化状态进行在线的TFSEARCH软件分析,结肠癌患者结肠癌组织IL-10基因启动子区-185位点甲基化率明显较低,使IFN-γ甲基化率异常性增高,这与张磊昌等[9]相关研究报道一致,说明DNA甲基化导致基因表达异常,丧失对细胞生长的抑制作用及对异常细胞的诱导凋亡作用,造成免疫功能低下,促使癌细胞的过度增殖,从而可能导致了肿瘤的发生。
DNA甲基化在生物界是一种正常的自然修饰方式,在DNA甲基转移酶的催化下,将甲基转移到特定碱基上的过程DNA甲基化引起抑癌基因转录抑制,其中约70%~80%的甲基化发生在CpG序列上,结肠癌组织IL-10基因CpG岛的甲基化率较低,这与结肠癌细胞中发现IL-10基因的转录因子SP1、CCAAT-box、STAT3和Ets-1有关[9-10]。因此,本研究采用BSP进一步论证了甲基化对IL-10基因的转录活性的影响,在结肠癌组织中IL-10基因启动子高甲基化的发生率明显高于正常健康组织,IL-10的表达调控受多种因素的影响,IL-10的DNA甲基化修饰在这个过程中起重要作用[11-12]。笔者通过DNA序列的转录因子结合位点预测,发现结肠癌组的IL-10的DNA甲基-110、-185位点的甲基化率明显高于对照组,他们影响5个转录因子位点相结合活性,从而造成结肠癌的IL-10基因启动因子活性增高致使IL-10基因高的表达,并且负调控IFN-γ mRNA的表达,对结肠癌细胞的增生、凋亡、分化起负性调控作用,使结肠癌细胞免疫耐受,逃避机体免疫监视。
综上所述,IL-10和IFN-γ基因启动子区甲基化状态使IL-10基因高的表达和IFN-γ mRNA低的表达,使结肠癌患者处于明显的Th1/Th2失衡状态,Th0细胞向Th2漂移极化现象,与结肠癌发生发展密切相关。
[1] 陈远崇,陈竹雨.结直肠癌患者血清Th1/Th2细胞因子水平变化及其临床意义研究[J].中国全科医学杂志社,2011,14(12):4136-4138.
[2]陈绍勤,陈志华,林素勇,等.5-杂氮-2’-脱氧胞苷对人结直肠癌HCT216细胞KISS2基因表达及侵袭迁移的影响[J].肿瘤,2014,34(1):26-32.
[3]王广华.腹腔镜结肠癌手术应用于结肠癌不同分期的合理性探究[J].中国实用医药,2012,7(28):89.
[4] Subramaniam M M,Chan J Y,Soong R,et a1.RUNX3 inactivation by frequent promoter hypermethylation and protein misloealization constitute an early event in breast cancer progression[J].Breast Cancer Res Treat,2009,113(1):l13-121.
[5] 木尔扯尔,文彬,黄一凡.β-catenin 与 wisp-1在结肠癌中的表达及意义[J].现代肿瘤医学,2014,22(2):366-370.
[6]何文.外周血T细胞亚群检测在恶性肿瘤中的应用[J].临床医学,2012,32(4):98-99.
[7]林昀,张云丽,胡昕,等.PEG10基因在肝癌组织中高表达的分子机制及其功能的研究[J].肿瘤,2013,33(1):28-35.
[8]杨涛,邵江河,李启英.树突状细胞联合CIK 细胞应用于恶性实体瘤治疗的临床疗效观察[J].肿瘤,2010,30(8):700-705.
[9]张磊昌,何纯钢,曹云飞,等.散发性结直肠癌组织p33ING1b启动子甲基化情况及其意义[J].中国全科医学,2014,17(14):1617-1620.
[10]高燕,张厚德,林菊生,等.人肝癌父系表达基因10的遗传印记状态[J].中华肝脏病杂志, 2O10,18(12):894-899.
[11]金奎,陈雄飞,周宁新.DC联合CIK免疫治疗术后晚期胆管癌72例疗效观察[J].中国全科医学,2014,17(14):1617-1620.
[12] Zhang Y,Lyver E R,Nakamaru-Ogiso E,et al.Dre2,aconserved eukaryotic Fe/S cluster protein,functions in cytosotic Fe/S protein biogenesis[J].Mol Cell Biol,2008,28(12):5569-5582.

