牦牛AQP9基因CDS全长序列克隆及其生物信息学特征分析
刘健锋,丁艳平,伍志伟,王建林,邵宝平*
(1.兰州大学生命科学学院,动物学与发育生物学研究所,兰州 730000;2.西北师范大学生命科学学院,兰州 730070;3.甘肃中医学院 系统生物学与中医药转化研究所,兰州 730020)
牦牛AQP9基因CDS全长序列克隆及其生物信息学特征分析
刘健锋1,丁艳平2,伍志伟3,王建林1,邵宝平1*
(1.兰州大学生命科学学院,动物学与发育生物学研究所,兰州 730000;2.西北师范大学生命科学学院,兰州 730070;3.甘肃中医学院 系统生物学与中医药转化研究所,兰州 730020)
为了研究高原动物对青藏高原极端生境的适应机理,并为探讨高原动物适应机制提供基本数据。本研究运用基因克隆技术对牦牛脑AQP9基因CDS全长序列进行克隆,采用生物信息学方法进行分析,AQP9基因和编码序列特征进行了以下几方面的预测和分析:其物化性质、疏水性、跨膜结构和蛋白二级结构。结果表明,牦牛AQP9的CDS含有一个885 bp的开放阅读框,编码295个氨基酸;牦牛AQP9基因编码蛋白分子量和理论等电点分别为31.89 ku和6.21,其编码蛋白含有6次跨膜结构,属于疏水性蛋白;二级结构由α-螺旋、延伸链、β-折叠及无规则卷曲构成;牦牛AQP9基因编码氨基酸序列与黄牛、藏羚羊、绵羊等物种间同源性较高,系统进化情况与其亲缘关系远近一致。本研究将为牦牛低氧适应性研究提供一定的基础资料。
牦牛;AQP9基因;CDS区;分子克隆;生物信息学
AQP9(水通道蛋白9,AQP9)是水通道蛋白家族第二亚类水甘油通道蛋白(包括AQP3、AQP7、AQP9和AQP10)的成员之一[1]。1997年,H.Kuriyama等[2]在进行人类脂肪组织基因序列系统分析过程中首次发现AQP9。近年的研究表明,AQP9可以透过嘌呤(腺嘌呤)、嘧啶(尿嘧啶和化学疗法中使用的氟尿嘧啶)及一元羧酸(乳酸和β-羟基丁酸)[3-4];AQP9对一元羧酸,比如乳酸和β-羟基丁酸的通透效率与体内pH相关,尤其在pH为5.5时通透性急剧增加[3];AQP9还可以促进砷类金属的运输,这表明AQP9可能是哺乳动物细胞摄取亚砷酸盐的主要途径[5]。AQP9主要分布于星形胶质细胞和儿茶酚胺能神经元细胞膜上[6],而儿茶酚胺神经元的电位活动在甘油和乳酸浓度上升时会被改变,包括摄食行为的最终改变。综上所述,除水分子外,AQP9对尿素、甘油、多元醇、乳酸等多种小分子溶质均有通透性,可参与组织中水的转运、水电解质的平衡调节、渗透压的感知及能量代谢等[6-11]。
缺氧是引发众多类型脑疾病,尤其是脑水肿发生、发展的重要因子。脑是机体适应其栖息地进行生存、生产及繁衍的“指令中枢”,又是对环境氧最敏感的器官。脑神经细胞作为进化最古老的细胞,“水和能量的代谢”在维持其正常生理功能中发挥了非常重要的生理作用。大量研究表明低氧可上调AQP9蛋白的表达且AQP9的表达与脑水肿程度呈正相关[1,7,10,12-13]。牦牛作为体型最大、运动剧烈且好动的半野生动物,其对高寒、低氧极端生境的适应早已引起国内外广大学者的关注[14-15]。但是,到目前为止,在国内外还尚未见到有关“牦牛脑AQP9基因CDS全长序列及其相关生物信息学特征”的研究报道。本研究运用基因克隆、表达质粒构建及生物信息学分析等技术和方法,对牦牛AQP9基因进行了CDS全长序列克隆及生物信息学特征分析。本研究结果将为明晰高原动物对青藏高原高寒、低氧等极端环境的适应机理,进一步探讨高原动物脑神经细胞在其极端生境中水和能量代谢的分子机理,以及为人类高原疾病的预防和治疗研究提供基础资料。
1 材料与方法
1.1 试验材料
试验动物为健康成年甘南牦牛(3~4岁,体重约225 kg,海拔>3 400 m)。试验材料采于甘肃省甘南州玛曲县屠宰场,在动物屠宰后,立即取其头沿矢状面开颅取脑,用无菌生理盐水将脑快速冲洗干净后剪成小块迅速放入液氮中带回实验室,-80 ℃超低温冰箱长期保存。
1.2 分子生物学试剂
Trizol试剂、TaKaRa反转录试剂盒、TaKaRa PrimeSTAR HS DNA 聚合酶、DL5000 DNA marker、DL2000 DNA marker、E.coliDH5ɑ、DNA凝胶回收试剂盒、限制性内切酶EcoR I和KpnI及质粒提取试剂盒购自宝生物工程(大连)有限公司;Kan抗生素、DNase I和DEPC购自Sigma公司(美国),pEGFP-C1载体和感受态细胞购自北京全式金生物技术有限公司。
1.3 方法
1.3.1 牦牛大脑皮质总RNA的提取 采用Trizol法提取大脑皮质总RNA,DNase I去除基因组中残留的DNA,1.5%琼脂糖凝胶电泳及紫外分光光度计检测总RNA的质量,-70℃保存备用。
1.3.2 引物设计与合成 参考GenBank中黄牛AQP9基因cDNA序列设计特异性引物P1和P2,即P1:5′-GGAATTCATGCAGCCTGAGATGGA-ACAAA-3′;P2:5′-GGGGTACCTTACATGATT-GCATTCAGTTCATATTT-3′,横线部分分别为EcoR I和KpnI内切酶酶切位点序列。扩增引物由宝生物工程(大连)有限公司合成。
1.3.3 RT-PCR扩增 以牦牛大脑皮质总RNA为模板进行逆转录反应,总反应体系10 μL:5×PrimeScript Buffer 2 μL,RNase free dH2O 7 μL,500 ng·μL-1总RNA 1 μL,混匀后,37 ℃ 15 min,85 ℃ 5 s条件下进行逆转录反应,将产物4 ℃保存。以逆转录产物为模版,P1、P2为引物,扩增AQP9基因。PCR反应体系25 μL:5×Prime Star Buffer 5 μL,dNTP Mixture 2 μL,上下游引物各0.5 μL,TaKaRa PrimeSTAR HS 0.25 μL,cDNA模板1 μL,RNase-Free dH2O 15.75 μL。PCR反应条件:98 ℃预变性2 min;98 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1 min,共30个循环;72 ℃延伸5 min,4 ℃保存。用l%的琼脂糖凝胶电泳检测PCR产物后,用DNA片段回收试剂盒对PCR产物回收。
1.3.4AQP9基因的克隆 回收的PCR产物在T4 连接酶的作用下与pEGFP-C1载体进行过夜连接12~16 h。将连接产物转化至感受态E.coliDH5ɑ中,37 ℃,100 r·min-1培养1 h,涂于含有Kan的LB固体培养基上,37 ℃培养12 h。挑取阳性单克隆接种于含有Kan的LB液体培养基中,37 ℃,180 r·min-1过夜培养。用“质粒提取试剂盒”提取质粒后用EcoR I和KpnI内切酶进行双酶切鉴定。鉴定成功后将质粒送往上海生工公司进行测序。1.3.5 生物信息学分析AQP9基因开放阅读框(ORF)采用NCBI的ORF Finder程序分析,序列比对及同源性分析使用DNAMAN软件及在线NCBI网站(http://www.ncbi.nlm.nih.gov);蛋白质特性预测使用Bioedit及Lasergene7.1软件包中的Protean软件;亲水性疏水性预测采用Epasy服务器上的protscale程序(http://web.expasy.org/protscale/);蛋白质二级结构预测采用法国里昂CNRS的SOPMA软件(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl? page=npsa_sopma.html),并利用SWISS-MODEL软件(http://swiss-model.expasy.org/)进行三级结构预测;使用MEGA 4.0软件构建系统进化树,置信度用Boot-strap检验(重复次数为1 000)。
2 结 果
2.1 牦牛AQP9基因的PCR扩增
牦牛大脑皮层总RNA用1.5%琼脂糖凝胶电泳检测到28S、18S和5S 3条带(图略),OD260 nm/OD280 nm平均为1.95,表明RNA完整性较好,没有蛋白质或DNA污染,且纯度和浓度适合RT-PCR。经特异性引物PCR扩增的产物通过1%琼脂糖凝胶电泳检测,如图1所示,DNA片段在1 000 bp左右,可能还含有编码区两侧序列,与预期DNA片段(885 bp)大小并不矛盾,条带清晰且特异性良好,可以进行切胶回收进一步做克隆。
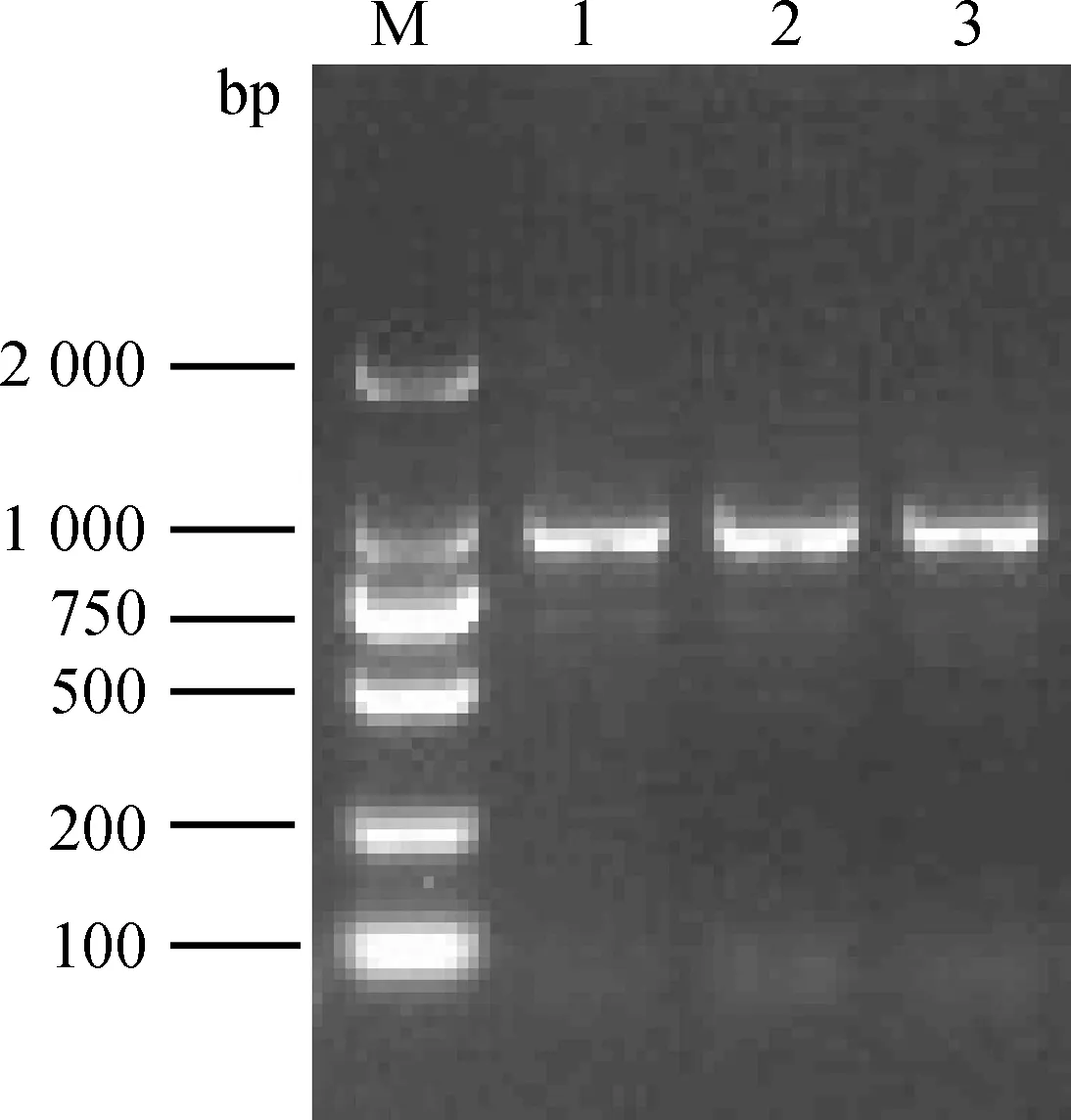
M.DNA相对分子质量标准;1~3. PCR扩增产物M.DNA marker;1-3. PCR products图1 AQP9基因PCR扩增产物的电泳图Fig.1 The PCR amplification of AQP9 gene
2.2 牦牛AQP9基因克隆及重组子的鉴定
提取菌液的质粒并用EcoR I和KpnI内切酶双酶切鉴定重组质粒,得到一条约5 000 bp的pEGFP-C1线性片段和约900 bp的插入片段(图2),与预期相符。
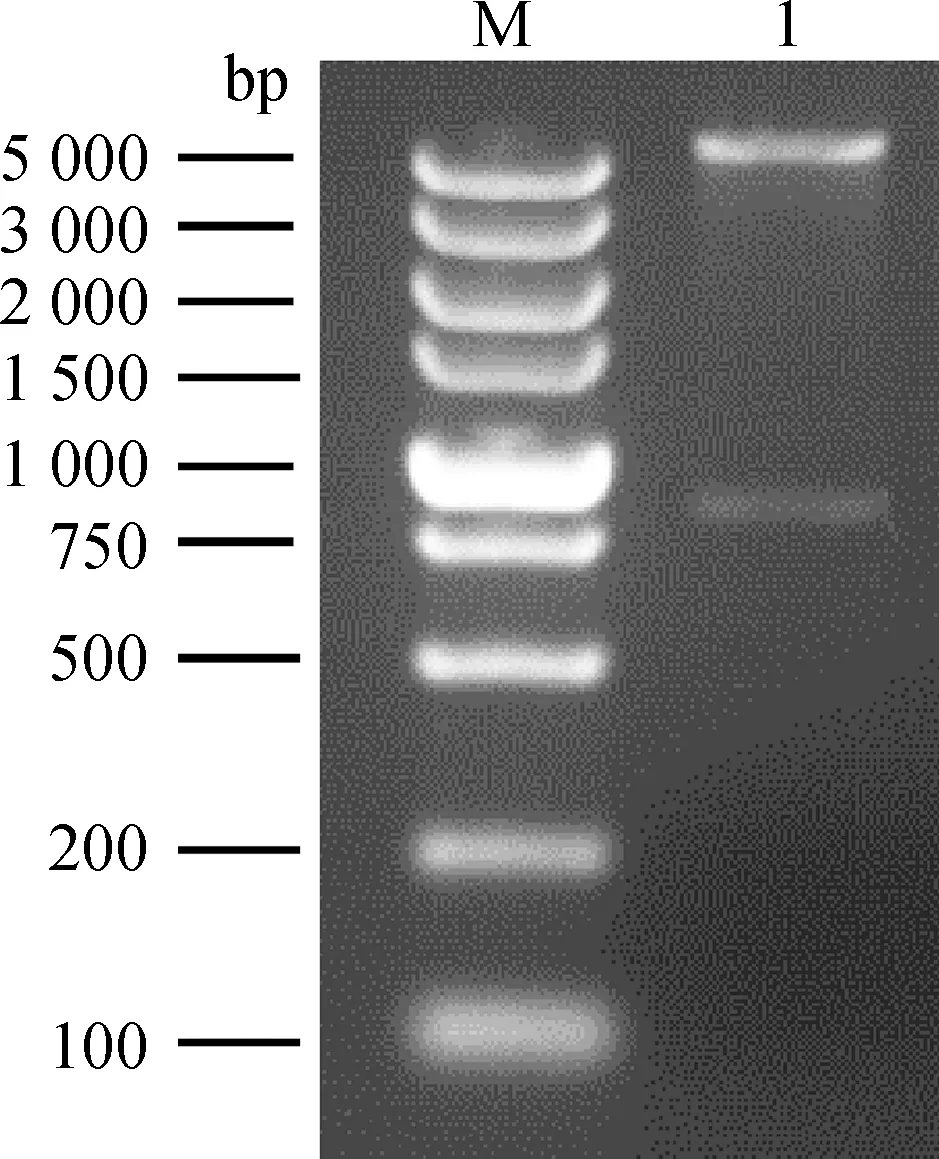
M.DNA相对分子质量标准;1.重组质粒的双酶切片段M.DNA marker;1.Recombinant plasmid digested by double digestion图2 重组质粒的双酶切鉴定Fig.2 Restriction endonuclease digestion of recombinant plasmid
2.3 牦牛AQP9基因的序列测定及分析
将双酶切鉴定正确的阳性重组质粒送往上海生工公司测序。测序结果表明,克隆得到的序列在NCBI上进行同源性搜索,所得到的目的基因为牦牛AQP9编码区全长(CDS),为885 bp,编码295个氨基酸,其中3个半胱氨酸(Cys)。应用NCBI的ORF Finder程序对牦牛AQP9基因序列开放阅读框分析,分析表明AQP9基因含有一个长度为885 bp的开放阅读框,编码295个氨基酸,起始密码子为ATG,终止密码子为TAA(图3)。
2.4 牦牛AQP9蛋白的生物信息学分析
2.4.1 牦牛AQP9蛋白的理化性质 运用Protparam软件分析了牦牛AQP9蛋白的基本理化性质,其分析表明:蛋白分子量为31 892.4 u,理论等电点为6.21;含有20种基本氨基酸,其中含量最高的是Ala(11.5%),含量最低的是Cys(1.0%);有带负电荷的残基23个,带正电荷的残基20个;280 nm的摩尔消光系数为34 045 mol·cm-1;蛋白的平均亲水系数为0.569。2.4.2 牦牛AQP9蛋白的亲水性/疏水性 运用Expasy服务器中ProtScale程序对牦牛AQP9的亲水性/疏水性进行了分析,如图4所示,第160位异亮氨酸(Ile)疏水性最强(最高分值3.800);第271位组氨酸(His)亲水性最强(最低分值为-3.2)。

图3 牦牛AQP9基因的碱基和氨基酸序列Fig.3 The nucleotide and amino acid sequences of yak AQP9 gene
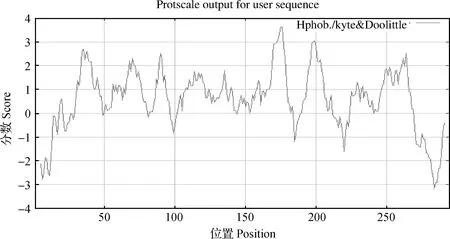
图4 牦牛AQP9蛋白疏水性分析Fig.4 Analysis of hydrophobicity of yak AQP9 protein
2.4.3 牦牛AQP9蛋白的跨膜区分析 运用TMHMM server 2.0分析表明,如图5所示,该蛋白为6次跨膜蛋白,即1~26位氨基酸为胞内部分;27~49氨基酸为第1个跨膜螺旋;50~58氨基酸在胞外;59~81氨基酸为第2个跨膜螺旋;82~101为胞内部分;102~124为第3个跨膜螺旋;125~157为胞外部分;158~180为第4个跨膜螺旋;181~192为胞内部分;193~215为第5个跨膜螺旋;216~242为胞外部分;243~265为第6个跨膜螺旋;266~295为胞内部分。
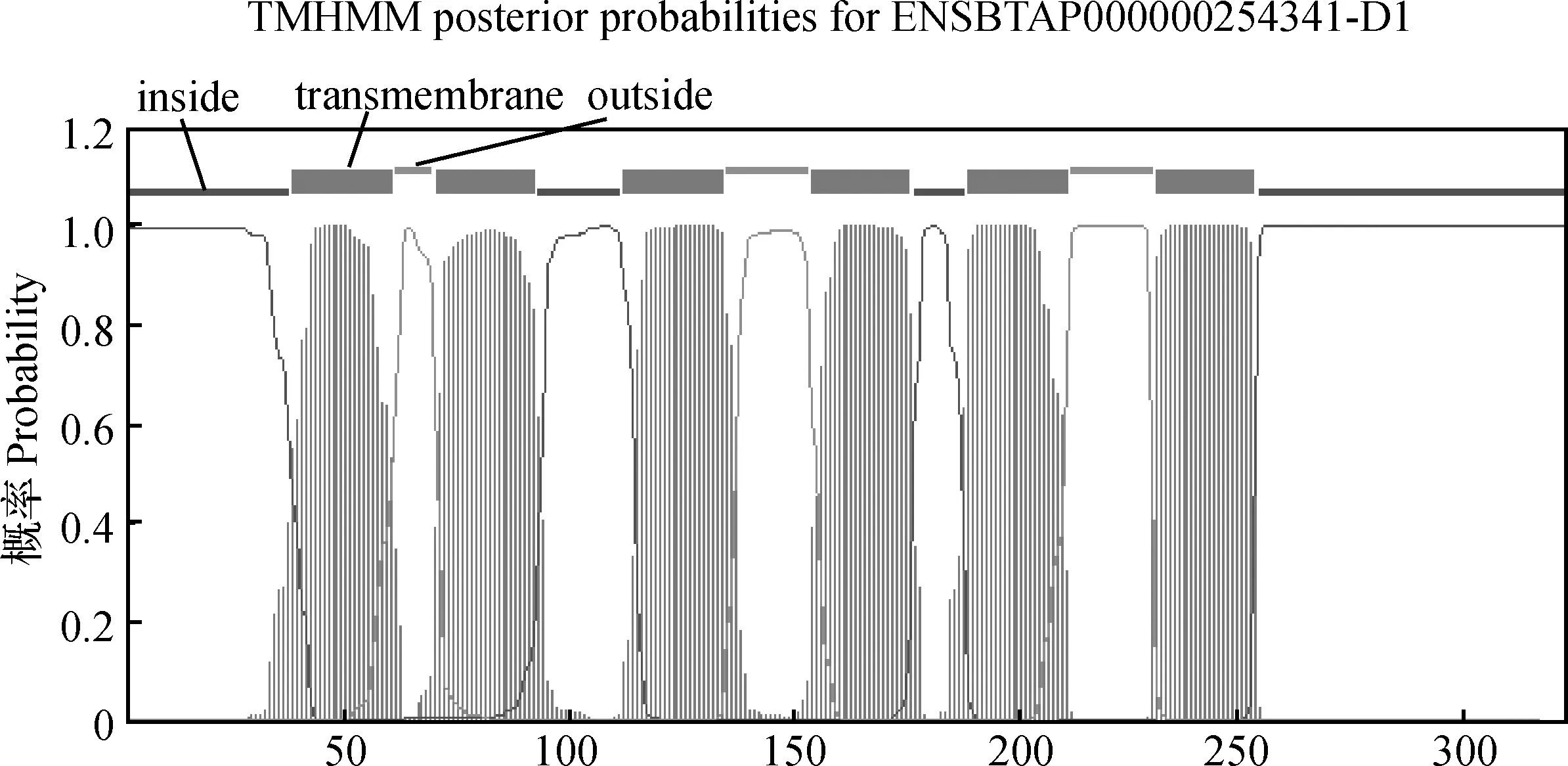
图5 牦牛AQP9蛋白跨膜区域Fig.5 Transmembrane region of yak AQP9 protein
2.4.4 牦牛AQP9蛋白的高级结构预测 如图6所示,运用SOPMA服务器分析表明,牦牛AQP9的二级结构由α-螺旋、延伸、β-折叠及无规则卷曲4种结构组成,其各种结构所占比例分别是38.18%、24.32%、4.73%及32.77%,其中α-螺旋主要分布在位于细胞内的氨基酸两端。运用SWISS-MODEL进行同源建模,如图7所示,牦牛AQP9蛋白的三级结构也是由α-螺旋、延伸、β-折叠和无规则卷曲构成,这与二级结构预测结果一致。
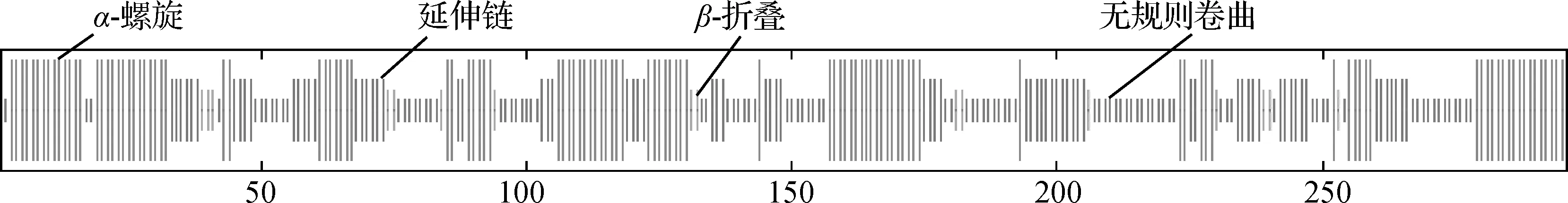
图6 牦牛AQP9蛋白二级结构Fig.6 Secondary structure of yak AQP9 protein
2.4.5 牦牛AQP9蛋白同源性及系统发育分析 通过NCBI数据库中收集下载7个物种的AQP9基因编码蛋白序列,采用MegAlign软件进行同源性分析,发现牦牛AQP9基因编码蛋白与其他物种AQP9基因编码蛋白具有较高的同源性,牦牛AQP9氨基酸序列与黄牛、藏羚羊、绵羊、野猪、野骆驼、人、及狼的同源性分别为99.8%、97.1%、96.7%、89.6%、88.2%、87.6%及87.5%。用NJ法构建了上述8个物种AQP9的基因系统发育树,如图8所示,牦牛与黄牛、藏羚羊及绵羊在系统发育树中距离最近。
3 讨 论
随着人类基因组测序工作的基本完成,基因组学的研究从结构基因组学过渡到了功能基因组学[16]。cDNA的测序已成为人们了解编码基因结构与功能的关键所在。生物信息技术为试验研究提供了重要的线索,对随后的研究起到了“事半功倍”的作用。随着基因组序列信息的日益丰富,计算方法和数据库的不断完善,生物信息学将在基因全长cDNA克隆和分析中扮演更加重要的角色。1988年,B.M.Denker首先从哺乳动物的红细胞膜上发现了一个28 ku的疏水性跨膜蛋白[17]。1991年G.M.Preston等[18]完成了第一个水通道蛋白cDNA的分子克隆和功能鉴定,证明了哺乳动物的细胞膜上有可以特异性转运水的结构,从而促进了一个新的研究领域的发展。迄今为止,在哺乳动物组织中已发现13种AQP (AQP0-AQP12)。AQPs的发现对生物有机体内物质跨膜转运和细胞内外环境平衡调节机制的研究及临床代谢类障碍疾病病理的发现、认识和治疗,具有划时代的意义。
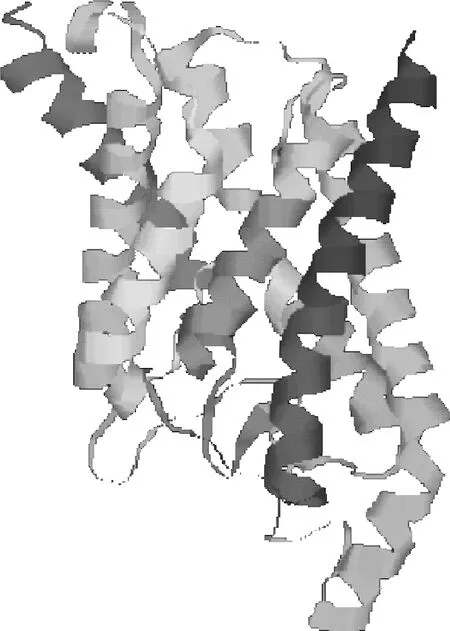
图7 牦牛AQP9蛋白三级结构Fig.7 Putative tertiary structure of yak AQP9 protein
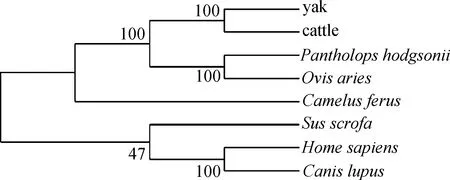
图8 NJ法构建的AQP9基因系统发生树Fig.8 Phylogenetic tree of AQP9 gene by NJ method
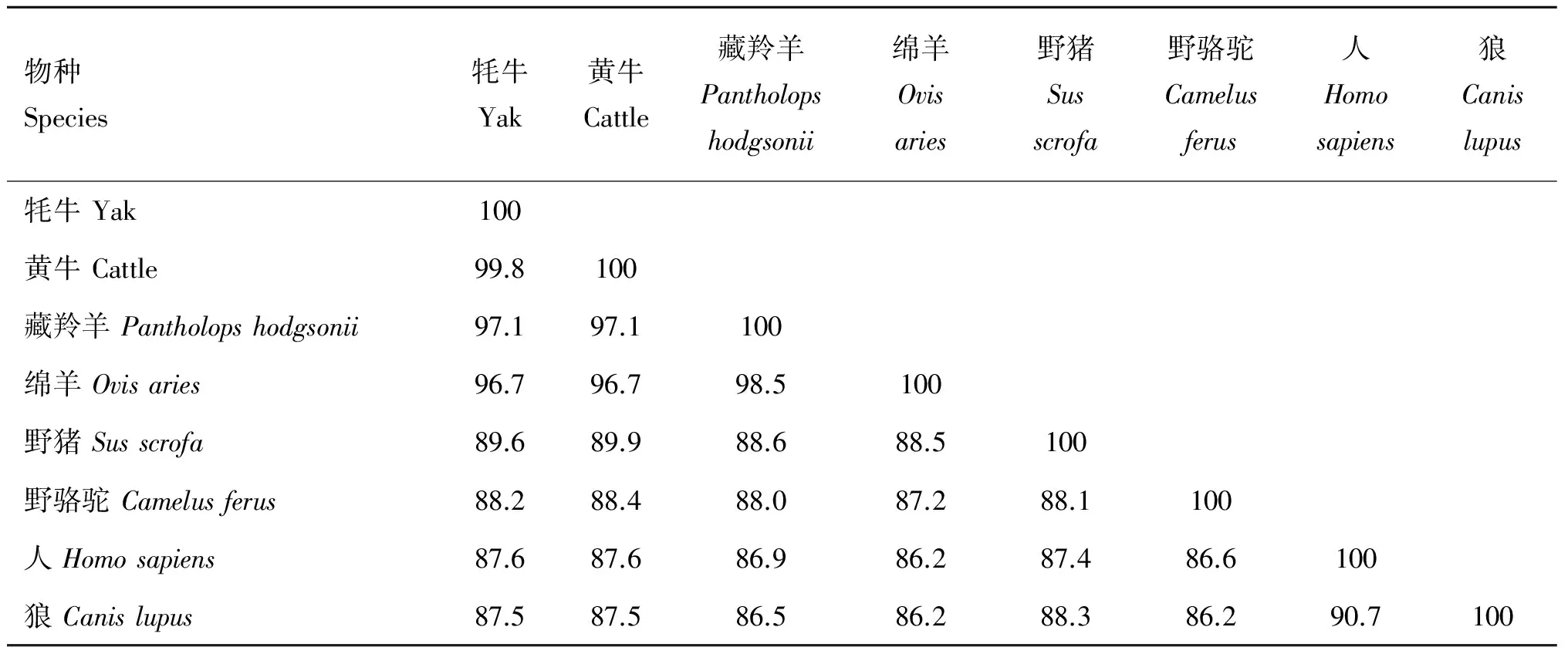
表1 牦牛与其他7个物种AQP9的氨基酸序列同源性比较
本研究克隆得到的牦牛AQP9基因编码区序列具有特定的起始密码子(ATG)和终止密码子(TAA),是牦牛AQP9基因编码区全长序列,编码AQP9蛋白。牦牛AQP9基因含有一个长度为885 bp的开放阅读框,编码295个氨基酸。通过对牦牛AQP9基因编码氨基酸序列跨膜结构进行分析表明,肽链中含有6个串联的疏水跨膜区,且这些跨膜区富含α-螺旋,缺少β-折叠结构,这与水通道蛋白家族其他成员结构相似[19]。该蛋白具有水通道蛋白家族保守的序列特征,其二级结构由α-螺旋、延伸、β-折叠以及无规则卷曲4种结构模式组成,且α-螺旋和无规则卷曲分别占33.18%和24.32%,这与其他AQPs结构相似[19]。牦牛AQP9基因编码氨基酸序列中含有两个天冬酰胺-脯氨酸-丙氨酸(Asn-Pro-Ala,NPA)结构,NPA为AQP家族共有的特征结构,这与G.M.Preston等[18]发现的结果相一致,且这两个NPA序列在空间上围绕通道中轴呈点对称分布。依据氨基酸分值越低亲水性越强和分值越高疏水性越强的规律可知,AQP9蛋白整体疏水性较强,仅两端膜内的部分氨基酸具有较好的亲水性。同源建模结果表明,牦牛AQP9蛋白的高级结构是由相同的4个亚基组成的同源四聚体,这与鼠的高级结构类似[20]。
序列同源性在一定程度上反映物种间亲缘关系的远近。通过序列比对和系统进化分析发现,牦牛AQP9蛋白与其他物种AQP9蛋白具有较高的同源性,这与物种进化的结果相一致,表明AQP9基因可能是由同一个祖先基因进化而来,同时也反映了AQP9蛋白在不同物种结构上的稳定性对生物体功能的重要性。尽管牦牛和黄牛AQP9蛋白氨基酸序列间同源性很高,但还是存在两个氨基酸的差异。另外,大量研究表明,在脑神经细胞中AQP9蛋白同时参与水和能量代谢最重要的膜通道蛋白[1,7,10,12-13];而且,AQP9蛋白还在脑水肿的发生、发展及其清除过程中也发挥着重要作用[13,21-22]。由此推测,在青藏高原极端低氧环境中,AQP9蛋白在牦牛脑神经细胞的水和能量代谢及其高原脑水肿抗性的调控中发挥中非常重要的作用,但其具体调控机理有待进一步研究。
本研究利用分子克隆手段获得牦牛AQP9基因的编码区序列并首次通过生物信息学相关技术和方法发现该蛋白含有六次跨膜结构,属于疏水性蛋白;二级结构由α-螺旋、延伸、β-折叠及无规则卷曲构成;牦牛AQP9蛋白与黄牛、藏羚羊、绵羊等物种间同源性较高,本研究将为牦牛低氧适应性研究提供一定的基础资料。
[1] BADAUT J.Aquaglyceroporin 9 in brain pathologies[J].Neuroscience,2010,168:1047-1057.
[2] KURIYAMA H,KAWAMOTO S,ISHIDA N,et al.Molecular cloning and expression of a novel human aquaporin from adipose tissue with glycerol permeability[J].BiochemBiophysicResCommun,1997,241:53-58.
[3] TSUKAGUCHI H,SHAYAKUL C,BERGER U V,et al.Molecular characterization of a broad selectivity neutral solute channel[J].JBiolChem,1998,273:24737-24743.
[4] TSUKAGUCHI H,WEREMOWICZ S,MORTON C C,et al.Functional and molecular characterization of the human neutral solute channel aquaporin-9[J].AmJPhysiol,1999,277:F685-696.
[5] LIU Z,SHEN J,CARBREY J M,et al.Arsenite transport by mammalian aquaglyceroporins AQP7 and AQP9[J].ProcNatlAcadSciUSA,2002,99:6053-6058.
[6] BADAUT J,PETIT J M,BRUNET J F,et al.Distribution of Aquaporin 9 in the adult rat brain:preferential expression in catecholaminergic neurons and in glial cells[J].Neuroscience,2004,128:27-38.
[7] BADAUT J,REGLI L.Distribution and possible roles of aquaporin 9 in the brain[J].Neuroscience,2004,129:969-979.
[8] WARTH A,MITTELBRONN M,HULPER P,et al.Expression of the water channel protein aquaporin-9 in malignant brain tumors[J].ApplImmunohistochemMolMorphol,2007,15:193-198.
[9] DIBAS A,YANG M H,BOBICH J,et al.Stress-induced changes in neuronal Aquaporin-9 (AQP9) in a retinal ganglion cell-line[J].PharmacolRes,2007,55:378-384.
[10] BADAUT J,HIRT L,GRANZIERA C,et al.Astrocyte-specific expression of aquaporin-9 in mouse brain is increased after transient focal cerebral ischemia[J].JCerebBloodFlowMetab,2001,21:477-482.
[11] ZELENINA M.Regulation of brain aquaporins[J].NeurochemInt,2010,57:468-488.
[12] BADAUT J,BRUNET J F,REGLI L.Aquaporins in the brain:from aqueduct to “multi-duct”[J].MetabBrainDis,2007,22:251-263.
[13] 邵宝平,余华晟,王建林.AQP9在脑疾病中的研究进展[J].动物医学进展,2012,33(1):87-91. SHAO B P,YU H S,WANG J L.Progress on Aquaglyceroporin 9 in Brain Pathologies[J].ProgressinVeterinaryMedicine,2012,33(1):87-91.(in Chinese)
[14] HAN X T,HAN X T,XIE A Y,et al.Effects of high altitude and season on fasting heat production in the yakBosgrunniensorPoephagusgrunniens[J].BrJNutr,2002,88:189-197.
[15] SHAO B,LONG R,DING Y,et al.Morphological adaptations of yak (Bosgrunniens) tongue to the foraging environment of the Qinghai-Tibetan plateau[J].JAnimSci, 2010,88:2594-2603.
[16] KIM S K.Functional genomics:the worm scores a knockout[J].CurrBiol,2001,11:R85-87.
[17] DENKER B M,SMITH B L,KUHAJDA F P,et al.Identification,purification,and partial characterization of a novel Mr 28 000 integral membrane protein from erythrocytes and renal tubules[J].JBiolChem,1988,263:15634-15642.
[18] PRESTON G M,AGRE P.Isolation of the cDNA for erythrocyte integral membrane protein of 28 kilodaltons:member of an ancient channel family[J].ProcNatlAcadSciUSA,1991,88:11110-11114.
[19] MOON C,PRESTON G M,GRIFFIN C A,et al.The human aquaporin-CHIP gene.Structure,organization,and chromosomal localization[J].JBiolChem,1993,268:15772-15778.
[20] SUZUKI M,TANAKA S.Structure and diverse functions of vertebrate aquaporins[J].Seikagaku,2014,86:41-53.
[21] ROJEK A M,SKOWRONSKI M T,FUCHTBAUER E M,et al.Defective glycerol metabolism in aquaporin 9 (AQP9) knockout mice[J].ProcNatlAcadSciUSA,2007,104:3609-3614.
[22] HIGASHIDA T,PENG C,LI J,et al.Hypoxia-inducible factor-1alpha contributes to brain edema after stroke by regulating aquaporins and glycerol distribution in brain[J].CurrNeurovascRes,2011,8:44-51.
(编辑 郭云雁)
Cloning and Bioinformatics Analysis of the CDS of YakAQP9 Gene
LIU Jian-feng1,DING Yan-ping2,WU Zhi-wei3,WANG Jian-lin1,SHAO Bao-ping1*
(1.InstituteofZoologyandDevelopmentalBiology,SchoolofLifeSciences,LanzhouUniversity,Lanzhou730000,China;2.SchoolofLifeScience,NorthwestNormalUniversity,Lanzhou730070,China;3.InstituteofSystemsBiologyandTCMTransformation,GansuTraditionalChineseMedicalCollege,Lanzhou730020,China)
The aim of this study was to investigate the adaption mechanism of plateau animals to extreme environment of the Qinghai-Tibet plateau,and to provide some basic data for exploring the adaption mechanism of plateau animals.The coding region sequence of yakAQP9 was cloned by molecular cloning technique and analyzed by bioinformatics in this study.Some characteristics of theAQP9 gene and encoded protein sequences were predicted and analyzed in the following aspects as the general physical and chemical properties,hydrophobicity,transmembrane structure and secondary structure.The results showed that the CDS of yakAQP9 contains a complete ORF (885 bp) encoding 295 amino acids.The putative molecular weight and theory isoelectric point ofAQP9 gene coding protein in yak were 31.89 ku and 6.21,respectively.The protein encoded by yakAQP9 contains 6 transmembrane domains,and it was a hydrophobic protein.The secondary structures are mainly composed of α-helix,extended strand,β-turn and random coil.Deduced amino acid sequences ofAQP9 gene between yak and cattle,sheep and other species were high homology,and the phylogenetic distance consistent with their genetic relationship.This study will provide certain basic data for studying hypoxia adaptation of yak.
yak;Aquaporin9(AQP9);gene;CDS region;molecular cloning;bioinformatics
10.11843/j.issn.0366-6964.2015.07.008
2014-08-27
国家自然科学基金项目(青年基金:31000190;地区基金:31060141);兰州大学中央高校基本科研业务费专项
刘健锋(1988-),男,重庆忠县人,硕士生,主要从事高原动物适应性机理研究,E-mail:andyandhope@gmail.com
*通信作者:邵宝平,博士,副教授,硕导,主要从事高原动物适应性机理研究,E-mail:shaobp@lzu.edu.cn
S823.8+5.2
A
0366-6964(2015)07-1134-07

