靶向猪ATGL 基因的miRNA预测及鉴定
戴丽荷,褚晓红,路伏增,黄孙平,徐如海
(浙江省农业科学院畜牧兽医研究所,浙江省畜禽遗传育种工程技术研究中心,杭州 310021)
靶向猪ATGL基因的miRNA预测及鉴定
戴丽荷,褚晓红,路伏增,黄孙平,徐如海*
(浙江省农业科学院畜牧兽医研究所,浙江省畜禽遗传育种工程技术研究中心,杭州 310021)
本试验旨在初步筛选出调控猪脂肪甘油三酯脂肪酶基因(ATGL)的miRNA。利用生物信息学分析预测10条靶向ATGL基因的猪miRNAs,然后通过装载含猪ATGL基因3′UTR序列来构建pMIR-Luc报告基因载体,以及通过装载含反向miRNA种子区对应的3′UTR序列来构建3个突变载体作为阴性对照,以pRL-TK载体作为内参,与候选miRNA模拟物共转染HEK 293T细胞,最终利用双荧光素酶报告基因法来检测荧光素酶活性。试验成功构建了含猪ATGL基因3′UTR序列的重组载体pMIR-ATGL-3′UTR,双荧光素酶报告基因试验显示ssc-miR-769-3p、ssc-miR-1343、ssc-miR-7137-3p、ssc-miR-671-5p、ssc-miR-149能下调293T细胞中pMIR-ATGL-3′UTR的荧光素酶活性,而点突变试验证实了ssc-miR-1343与ssc-miR-7137-3p能通过与猪ATGL基因3′UTR上预测的种子区结合下调荧光素酶活性。结果表明,sc-miR-1343与ssc-miR-7137-3p通过靶向结合3′UTR对猪ATGL基因有下调作用。
猪;ATGL基因;miRNA;双荧光素酶报告基因系统;3′UTR
脂肪甘油三酯脂肪酶(Adipose triglyceride lipase,ATGL)是2004年由3个不同的研究小组在人和小鼠中几乎同时发现的一种新的脂肪分解酶,官方命名为PNPLA2,又名为TTS-2.2(iPLA2ζ),desnutrin和PEDF-R等[1-3]。当机体需要能量时,在脂肪分解酶的作用下,甘油三酯被逐级分解,最终以甘油和游离脂肪酸的形式进入血液,完成脂动员过程[4]。ATGL的作用发生在脂肪动用时水解甘油三酯的第一步,为激素敏感脂肪酶(Hormone sensitive lipase,HSL)提供甘油二酯底物,是脂肪组织和其他组织中甘油三酯水解的起始步骤的限速酶[5-6]。ATGL与HSL协调作用,才能使哺乳动物脂肪组织中贮存的甘油三酯发生脂解。
microRNA(miRNA)是一类约22个核苷酸长度的单链非编码RNA,它通过靶向结合靶基因mRNA的3′非编码区(3′untranslated region,3′UTR)的特定位点从而抑制该转录本的翻译,最终调节特定的生理功能[7-8]。miRNA是基因表达的转录后调节者,是揭示性状形成分子机制的一个新靶点。
ATGL是启动脂肪动员的一个关键脂肪分解酶,其表达或活性上的轻微变异均有可能影响猪背膘厚和瘦肉率,研究ATGL基因的转录后调控对深入研究该基因的表达调控尤为重要。而有关猪ATGL基因相关的miRNA研究,目前还未见报道。本研究利用生物信息学分析,反向预测ATGL基因靶向的猪miRNA,并通过构建含ATGL基因3′UTR的双荧光素酶报告载体,利用双荧光素酶报告基因系统和点突变试验筛选出负性调控ATGL基因表达的miRNA,为ATGL在脂肪代谢中的功能研究奠定基础。
1 材料与方法
1.1 主要试剂与载体
人胚肾上皮细胞系HEK 293T细胞为本实验室保存;pMIR-REPORTTMLuciferase载体购自Ambion;pRL-TK质粒、双荧光素酶报告基因检测试剂盒(Dual-luciferase Reporter Assay System)和pGEM-T载体购自Promega;凝胶回收及质粒抽提试剂盒均购自Axygen;Platinum Taq DNA Polymerase 和LipofectamineTM2000转染试剂购自Invitrogen;T4 DNA连接酶、DNA marker、限制性内切酶SpeI和Hind III购自Fermentas;各种引物、ssc-miRNA mimics及阴性对照由Invitrogen公司合成。
1.2 生物信息学预测与猪ATGL基因3′UTR作用的miRNA
根据miRBase(http://www.mirbase.org/)数据库公布的猪miRNA(Release 21,2014),结合MiRanda(http://www.microrna.org/)、RNAhybrid(http://bibiserv.techfak.uni-bielefeld.de/rnahybrid/submission.html)、RNA22(http://cbcsrv.watson.ibm.com/rna22.html)和MicroInspector(http://bioinfo.uni-plovdiv.bg/microinspector/)等软件,预测在ATGL基因3′UTR存在潜在标靶关系的猪源miRNA,选出可能性最大的10条miRNAs作为候选miRNA进行后续试验。
1.3 报告基因载体构建
根据NCBI猪ATGL基因序列(EF648448)进行扩增测序验证后,合成猪ATGL基因3′UTR序列,加上SpeI/Hind III酶切位点,克隆至pMIR-LUC载体中,命名为pMIR-ATGL-3′UTR(野生型,简写为WT)。
靶基因双荧光素酶突变载体的构建主要是通过PCR的方法替换掉靶基因上的各miRNA结合位点。以pMIR-ATGL-3′UTR载体为模板,根据种子区设计突变引物(表1),分别与ATGL基因3′UTR引物扩增得到靶基因3′UTR序列结合位点上下游片段,以上下游片段混合物作为模板,PCR扩增无结合位点的靶基因3′UTR突变序列,然后通过连接pGEM-T载体测序验证,最后克隆至pMIR-LUC载体上,构建ATGL靶位点突变报告载体pMIR-ATGL-MT1(简写为MT1)、pMIR-ATGL-MT2(MT2)和pMIR-ATGL-MT3(MT3)。
1.4 细胞转染与双荧光素酶检测
293T 细胞用含10%胎牛血清的DMEM 高糖培养液,于37℃、5% CO2条件下培养和传代。细胞瞬时转染按照lipofectamine 2000 转染操作说明书进行。转染前1 d接种6 孔板,每孔细胞量约5×105个,培养过夜后对每孔细胞分别转染9组质粒,并设空白对照,具体试验分组:(1)无miRNA无转染试剂的空白对照;(2)mimic Negative Control、pMIR-ATGL-3′UTR和pRL-TK;(3)ssc-miR-769-3p mimic、pMIR-ATGL-3′UTR和pRL-TK;(4)ssc-miR-1343 mimic、pMIR-ATGL-3′UTR和pRL-TK;(5)ssc-miR-7137-3p mimic、pMIR-ATGL-3′UTR和pRL-TK;(6)ssc-miR-671-5p mimic、pMIR-ATGL-3′UTR和pRL-TK;(7)ssc-miR-339-5p mimic、pMIR-ATGL-3′UTR和pRL-TK;(8)ssc-miR-149 mimic、pMIR-ATGL-3′UTR和pRL-TK;(9)ssc-miR-1249 mimic、pMIR-ATGL-3′UTR和pRL-TK;(10)ssc-miR-1296-5p mimic、pMIR-ATGL-3′UTR和pRL-TK。将转染完成后细胞培养板中的培养液弃去,按照双荧光素酶检测试剂盒说明,检测萤火虫荧光素酶和海肾荧光素酶活性,计算每个转染3个平行样的相对发光比率 (Relative light unit,RLU),并计算标准差。根据得到的比值来比较不同样品间报告基因的激活程度。
表1 构建含猪ATGL基因3′UTR突变序列的荧光素酶载体所用的突变引物
Table 1 Primers for constructing luciferase reporter gene vectors containing porcineATGLmutational 3′UTR area

引物Primer突变引物序列(5′→3′)Primersequence对应miRNACorrespondingmiRNAMT1⁃RMT1⁃FACTGGGGGGCCCGGCCCTCAGCACACCCTGGGCAGGGCCCTGGAGAAGGCTGCTTCTCCAGGGCCCTGCCCAGGGTGTGCTGAGGGCCGGGCCCCCCAGTCssc⁃miR⁃769⁃3pMT2⁃RMT2⁃FGTCTCACCCCACCTCACCGGGCGGGGAGATCCCCGCCCGGTGAGGTGGGGTGAGACCCCCCCCCTCCCTssc⁃miR⁃7137⁃3p和ssc⁃miR⁃1343MT3⁃RMT3⁃FCAGGTGCCGCAGCCCCCGGGGCCGTCGGGGGGAAGGGGAGATGAAGCATCTCCCCTTCCCCCCGACGGCCCCGGGGGCTGCGGCACCTGCssc⁃miR⁃671⁃5p
下划线和方框部分表示针对预测的不同miRNA的种子区进行碱基互补突变后的碱基序列
Underlines and boxes indicate mutant nucleotides in primers by complementing seed sequences for different miRNA
同样的方法对点突变试验设计试验分组:(1)无miRNA无转染试剂的空白对照;(2)mimic Negative Control、pMIR-ATGL-3′UTR和pRL-TK;(3)ssc-miR-769-3p mimic、pMIR-ATGL-3′UTR和pRL-TK;(4)ssc-miR-769-3p mimic、pMIR-ATGL-MT1和pRL-TK;(5)ssc-miR-1343 mimic、pMIR-ATGL-3′UTR和pRL-TK;(6)ssc-miR-1343 mimic、pMIR-ATGL-MT2和pRL-TK;(7)ssc-miR-7137-3p mimic、pMIR-ATGL-3′UTR和pRL-TK;(8)ssc-miR-7137-3p mimic、pMIR-ATGL-MT2和pRL-TK;(9)ssc-miR-671-5p mimic、pMIR-ATGL-3′UTR和pRL-TK;(10)ssc-miR-671-5p mimic、pMIR-ATGL-MT3和pRL-TK。
1.5 统计学处理
所有数据均以“平均数(Mean)±标准差(SD)”表示。统计学分析方法采用SAS8.0的t检验和GLM方差分析,P<0.05有显著性差异,P<0.01有极显著差异。为消除试验误差,每个表达载体分别做3次独立转染,每次转染做2个复孔,取平均值。
2 结 果
2.1 生物信息学预测猪ATGL基因靶向miRNAs
本研究利用生物信息学反向预测了猪ATGL基因靶向的miRNAs。根据预测结果按照自由能最小排序,排在前10的有ssc-miR-769-3p、ssc-miR-1343、ssc-miR-7137-3p、ssc-miR-671-5p、ssc-miR-339-5p、ssc-miR-4334-3p、ssc-miR-339、ssc-miR-149、ssc-miR-1249、ssc-miR-1296-5p,均与ATGL基因3′UTR存在互补结合位点(表2),但由于ssc-miR-339-5p、ssc-miR-4334-3p和ssc-miR-339在猪ATGL基因3′UTR上的互补结合位点相同,所以这3条miRNAs中挑选ssc-miR-339-5p作为代表。因此,选取ssc-miR-769-3p、ssc-miR-1343、ssc-miR-7137-3p、ssc-miR-671-5p、ssc-miR-339-5p、ssc-miR-149、ssc-miR-1249和ssc-miR-1296-5p这8个miRNAs作为调控猪ATGL基因的候选miRNA进行下一步筛选。
表2 与猪ATGL基因3′UTR有互补结合位点的可能miRNAs
Table 2 Potential miRNAs targeting 3′UTR of porcineATGLgene
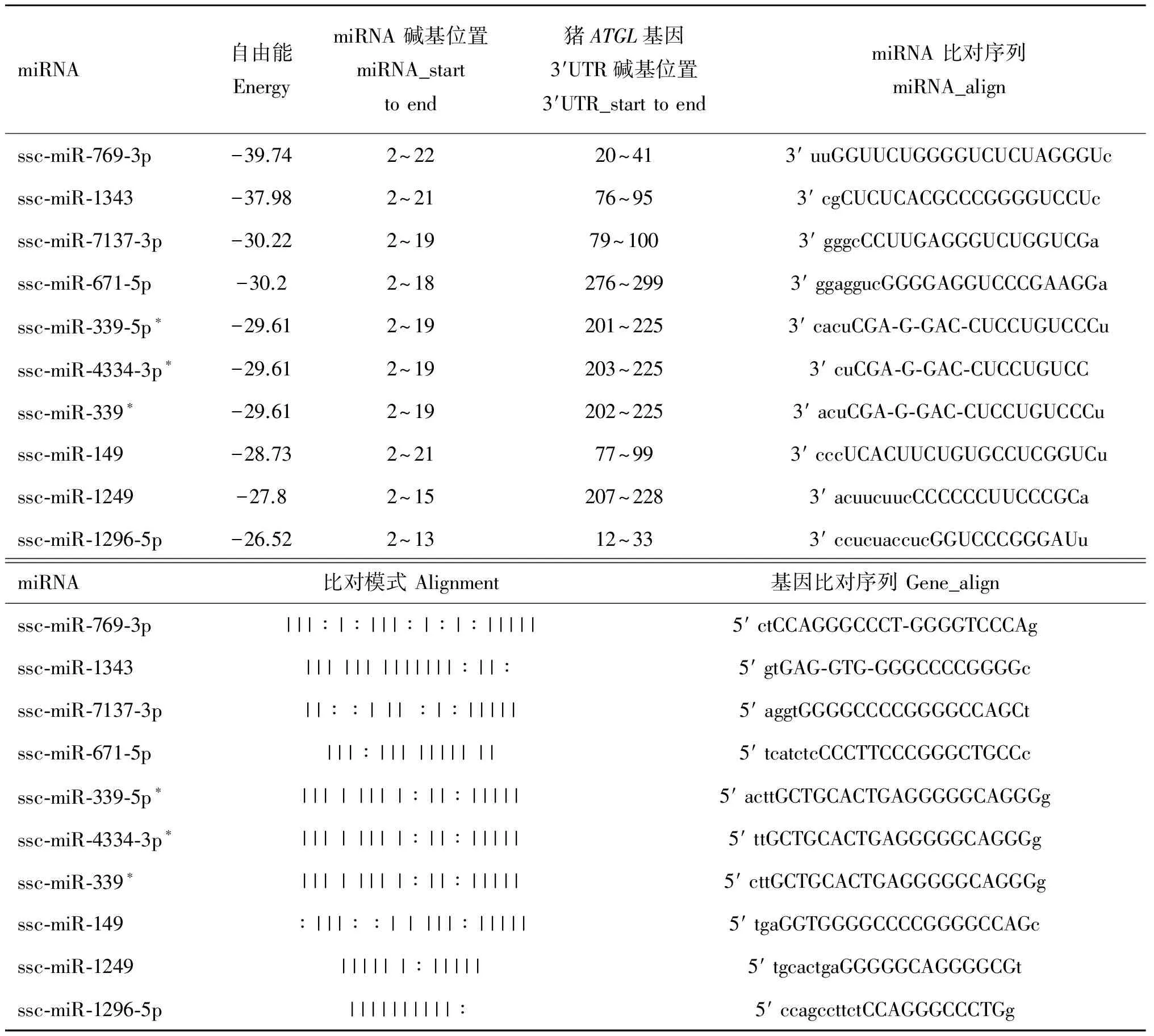
miRNA自由能EnergymiRNA碱基位置miRNA_starttoend猪ATGL基因3′UTR碱基位置3′UTR_starttoendmiRNA比对序列miRNA_alignssc⁃miR⁃769⁃3p-39.742~2220~413′uuGGUUCUGGGGUCUCUAGGGUcssc⁃miR⁃1343-37.982~2176~953′cgCUCUCACGCCCGGGGUCCUcssc⁃miR⁃7137⁃3p-30.222~1979~1003′gggcCCUUGAGGGUCUGGUCGassc⁃miR⁃671⁃5p-30.22~18276~2993′ggaggucGGGGAGGUCCCGAAGGassc⁃miR⁃339⁃5p∗-29.612~19201~2253′cacuCGA⁃G⁃GAC⁃CUCCUGUCCCussc⁃miR⁃4334⁃3p∗-29.612~19203~2253′cuCGA⁃G⁃GAC⁃CUCCUGUCCssc⁃miR⁃339∗-29.612~19202~2253′acuCGA⁃G⁃GAC⁃CUCCUGUCCCussc⁃miR⁃149-28.732~2177~993′cccUCACUUCUGUGCCUCGGUCussc⁃miR⁃1249-27.82~15207~2283′acuucuucCCCCCCUUCCCGCassc⁃miR⁃1296⁃5p-26.522~1312~333′ccucuaccucGGUCCCGGGAUumiRNA比对模式Alignment基因比对序列Gene_alignssc⁃miR⁃769⁃3p|||∶|∶|||∶|∶|∶|||||5′ctCCAGGGCCCT⁃GGGGTCCCAgssc⁃miR⁃1343|||||||||||||∶||∶5′gtGAG⁃GTG⁃GGGCCCCGGGGcssc⁃miR⁃7137⁃3p||∶∶|||∶|∶|||||5′aggtGGGGCCCCGGGGCCAGCtssc⁃miR⁃671⁃5p|||∶||||||||||5′tcatctcCCCTTCCCGGGCTGCCcssc⁃miR⁃339⁃5p∗||||||||∶||∶|||||5′acttGCTGCACTGAGGGGGCAGGGgssc⁃miR⁃4334⁃3p∗||||||||∶||∶|||||5′ttGCTGCACTGAGGGGGCAGGGgssc⁃miR⁃339∗||||||||∶||∶|||||5′cttGCTGCACTGAGGGGGCAGGGgssc⁃miR⁃149∶|||∶∶|||||∶|||||5′tgaGGTGGGGCCCCGGGGCCAGcssc⁃miR⁃1249||||||∶|||||5′tgcactgaGGGGGCAGGGGCGtssc⁃miR⁃1296⁃5p||||||||||∶5′ccagccttctCCAGGGCCCTGg
*.ssc-miR-339-5p、ssc-miR-4334-3p、ssc-miR-339预测与猪ATGL基因3′UTR有相同的互补结合模式,因此选取ssc-miR-339-5p为代表
*.ssc-miR-339-5p,ssc-miR-4334-3p and ssc-miR-339 had the same seed sequences binding to 3′UTR of porcineATGLgene,so ssc-miR-339-5p was chosen randomly for next research
2.2 猪ATGL基因3′UTR双荧光素酶靶标载体与突变载体的构建
经测序验证后,构建含有猪ATGL基因3′UTR序列的pMIR-ATGL-3′UTR载体,利用SpeI和Hind III进行双酶切鉴定得到451 bp的3′UTR片段及6.4 kb的pMIR-report骨架载体。同样的方法验证构建好的ATGL基因3′UTR双荧光素酶靶标突变载体(图1)。
2.3 miRNA对猪ATGL基因3′UTR的调控
重组载体pMIR-ATGL-3′UTR分别与8种ssc-miRNA mimics转染293T细胞系,同时设置无任何同源关系的miRNA作为阴性对照(NC),36 h后用双荧光素酶检测试剂盒检测,结果如图2所示。经配对资料t检验,ssc-miR-769-3p、ssc-miR-1343、ssc-miR-7137-3p、ssc-miR-671-5p、ssc-miR-149对猪ATGL基因3′UTR抑制效果差异显著,其中ssc-miR-1343+pMIR-ATGL-3′UTR共转染组的相对荧光素酶活性较NC+pMIR-ATGL-3′UTR共转染组降低了48.8%(P<0.01),ssc-miR-7137-3p共转染组降低了42.3%(P<0.01),ssc-miR-671-5p组降低了32.8%(P<0.01),ssc-miR-769-3p降低了19.4%(P<0.01),ssc-miR-149组降低了9.4%(P<0.05);而pMIR-ATGL-3′UTR 分别与ssc-miR-339-5p、ssc-miR-1249和ssc-miR-1296-5p共转染时,其荧光比率较对照组没有显著差异(P>0.05),说明这3种miRNAs对ATGL基因3′UTR无抑制作用。从最开始预测的10种miRNAs中,初步筛选出5种miRNAs(ssc-miR-769-3p、ssc-miR-1343、ssc-miR-7137-3p、ssc-miR-671-5p、ssc-miR-149)可通过ATGL3′UTR靶位点降低pMIR-ATGL-3′UTR的表达。

M.DNA相对分子质量标准;1.pMIR-ATGL-3′UTR载体;2.pMIR-ATGL-MT1载体;3.pMIR-ATGL-MT2载体;4.pMIR-ATGL-MT3载体 M.DNA marker;1.pMIR-ATGL-3′UTR vector;2.pMIR-ATGL-MT1 vector;3.pMIR-ATGL-MT2 vector;4.pMIR-ATGL-MT3 vector图1 双酶切鉴定pMIR-ATGL-3′UTR验证载体和突变载体Fig.1 Dual digestion of pMIR-ATGL-3′UTR vector and mutation vectors

*表示与mimic NC共转染组比较差异显著(P<0.05);**表示差异极显著(P<0.01);所有转染组以海肾荧光素酶报告基因载体转染内参照*.P<0.05;**.P<0.01;Renilla luciferase reporter vector was used as an internal control in all transfections图2 不同miRNA mimics分别与pMIR-ATGL-3′UTR共转染293T细胞的相对荧光素酶活性Fig.2 Relative luciferase activity of pMIR-ATGL-3′UTR reporter cotransfected with various miRNA mimics in 293T celles
为了进一步验证各miRNAs与ATGL3′UTR结合的靶位点,本试验选取了抑制作用较明显的ssc-miR-769-3p、ssc-miR-1343、ssc-miR-7137-3p和ssc-miR-671-5p,并针对不同miRNAs对预测种子区分别进行了反义突变(表2)。Ssc-miR-1343与ssc-miR-7137-3p的靶位点结合序列邻近,设计构建同一个3′UTR突变体命名为pMIR-ATGL-mut2(MT2),而ssc-miR-769-3p和ssc-miR-671-5p对应的突变体分别命名为pMIR-ATGL-mut1(MT1)和pMIR-ATGL-mut3(MT3)。
不同ssc-miRNA与pMIR-ATGL-3′UTR(WT)或pMIR-ATGL-mut(MT)共转染的双荧光素酶活性差异比较见图3和表3。与转染NC的对照组相比,4种ssc-miRNAs都能显著或极显著地下调pMIR-ATGL-3′UTR的相对荧光素酶活性,验证了4种miRNA都能靶向结合于猪ATGL基因3′UTR,并对其表达进行调控,但对预测的种子区分别进行突变后的回复效果却不一样。当ssc-miR-769-3p与WT共转染时,其相对荧光素酶活性比转染NC+WT的对照组下降了41.6%,差异极显著(P<0.01),与MT1共转染时,又回复了49.5%(P<0.05),但与NC组仍存有显著差异(P<0.01),说明预测的种子区影响到了ssc-769-3p与3′UTR的结合,并对ATGL3′UTR产生了一定的下调效果,但效果并不是很明显;ssc-miR-1343与WT共转染时,其相对荧光素酶活性下降了36.3%(P<0.01),与MT2共转染时,又回复了87.8%(P<0.01),与NC组差异不显著(P>0.05);ssc-miR-7137-3p与WT共转染时其相对荧光素酶活性下降了23.3%(P<0.01),与MT2共转染时,又回复了116.4%(P<0.01),与NC组相当(P>0.05);ssc-miR-671-5p与WT共转染时,其相对荧光素酶活性下降36.9%(P<0.01),而共转染MT3时,相对荧光素酶活性却没有回复,与WT共转染组没有差异(P>0.05),说明突变并没有影响ssc-miR-671-5p与ATGL3′UTR其他位置的结合,且结合位点并非预测的种子区域。结果说明,ssc-miR-1343和ssc-miR-7137-3p能通过预测的“种子区”靶向结合于猪ATGL基因3′UTR,并对其表达进行调控。

A、B、C.不同大写字母表示差异极显著,P<0.01。mimic NC.miRNA模拟物阴性对照;WT.无突变的pMIR-ATGL-3′UTR载体;MT.针对不同miRNA进行种子区突变后的pMIR-ATGL-mut载体,其中与ssc-miR-769-3p对应的是MT1载体,与ssc-miR-1343和ssc-miR-7137-3p对应的是MT2载体,与ssc-miR-671-5p对应的是MT3载体。表3同Different letters denoting significant difference between groups:A,B,C.P<0.01.Mimic NC.miRNA mimic negtive control;WT.pMIR-ATGL-3′UTR vector as a wild type;MT.pMIR-ATGL-mut vectors,in which MT1 has the mutated seed sequence for ssc-miR-769-3p,MT2 has the mutated seed sequence for ssc-miR-1343and ssc-miR-7137-3p,MT3 for ssc-miR-671-5p.The same as table 3图3 种子区突变前后4种ssc-miRNAs对荧光素酶表达的影响Fig.3 Relative luciferase activity of various reporters in the presence or absence of miRNA seed sequence in 293T cells
表3 不同ssc-miRNA与pMIR-ATGL-3′UTR(WT)或pMIR-ATGL-mut(MT)共转染的荧光素酶活性差异比较
Table 3 Difference comparison of relative luciferase activity in 293T cells cotransfected between different ssc-miRNAs and corresponding reporter vectors

相对荧光素酶比值RLU均值±标准差Mean±SDP(NC⁃WT)1P(WT⁃MT)2P(NC⁃MT)3ssc⁃miR⁃769⁃3pmimic+WTssc⁃miR⁃769⁃3pmimic+MT116.79±0.4922.71±0.49<0.00010.00020.0001ssc⁃miR⁃1343mimic+WTssc⁃miR⁃1343mimic+MT218.32±0.5627.47±0.56<0.0001<0.00010.1673ssc⁃miR⁃7137⁃3pmimic+WTssc⁃miR⁃7137⁃3pmimic+MT222.06±0.6329.84±0.630.00030.00010.2606ssc⁃miR⁃671⁃5pmimic+WTssc⁃miR⁃671⁃5pmimic+MT318.13±0.5218.34±0.52<0.00010.7845<0.0001mimicNC28.73±0.49
1、2、3.各组间的差异比较P值1,2,3.Pvalue among various groups
3 讨 论
miRNA是基因表达的转录后调节者,是揭示性状形成分子机制的一个新靶点。到目前为止,在最新的miRBase版本(Release 21)中已发现的猪miRNAs有382种,明显低于已鉴定的人(1 881种)和小鼠(1 193种)的miRNAs数量,而其中绝大多数的生物学功能尚不完全清楚。不断积累的证据显示,miRNA在脂肪代谢中发挥重要功能,不但调控动物脂肪的分化,还参与脂类代谢[9-11]及与脂类代谢相关的激素内分泌调节[12]。在动物体内敲除miR-14基因,导致甘油三酯和甘油二酯的含量增加,过量表达则减少[9]。抑制肥胖小鼠模型中miR-122的表达除了降低血浆胆固醇水平外,会明显促进脂肪变性,且脂肪生成基因表达减少[11]。miR-1792通过靶位点Rb2/pl30的3′UTR作用,能加快脂肪前体细胞3T3-L1细胞分化速度,增加甘油三酯的沉积[13]。对猪miRNA的研究中,李国喜[14]对7和240日龄荣昌猪背部皮下脂肪组织miRNA转录组进行测序分析,共筛选到227种猪保守miRNAs,同时确定miR-103参与脂类代谢和脂肪细胞分化,并提出miR-103可能通过靶作用于维甲酸诱导14基因(RAI14)促进猪前体脂肪细胞分化。在猪脂肪细胞分化的过程中,miR143通过作用靶点细胞外调节激酶5(ERK5)促进脂肪细胞分化[15],也能促进甘油三酯的分解,抑制甘油和游离脂肪酸的合成,进而促进脂滴沉积[16];miR-27a能抑制脂质的合成代谢,促进脂质的分解代谢,进而阻碍猪脂肪细胞脂滴的沉积[16];猪miR-181a能通过靶位点TNFα基因的3′UTR作用,能加速脂滴聚集,增加甘油三酯的沉积,调控脂肪发生[17]。这些说明miRNA也作为脂肪代谢调控网络中的一份子发挥着重要的生物学作用。脂肪的发育程度与猪瘦肉率关系密切,所以研究miRNA对脂肪代谢的调控机制意义重大。
ATGL能催化细胞内甘油三酯降解的起始步骤,是启动脂肪动员的一个关键脂肪分解酶。如果能筛选到参与调控ATGL基因表达的miRNA,并分析其调控机制,人们就可以明确这些miRNA在脂肪代谢中所扮演的角色,从而无论是明确ATGL脂解机理还是在开发潜在的饲料添加剂方面都有重要价值。S.K.Das博士利用双荧光素酶活性分析发现,miR-124a能通过与小鼠ATGL基因的3′UTR结合,从而下调小鼠ATGL基因 mRNA表达[18]。但由于猪和人的ATGL基因在3′UTR存在较大的差异,对猪ATGL基因miRNA的研究显得很有必要。
通过靶标预测软件,获得可能与猪ATGL基因3′UTR互作的10个候选miRNAs。但在软件预测的基础上,仍需要通过试验的方法来验证生物信息学预测的是否准确。目前最为常用的miRNA靶位点鉴定方法是荧光素酶报告基因法。利用miRNA mimics及对照来验证预测的10种miRNAs是否与猪ATGL基因的3′UTR段结合,发现5种miRNAs(ssc-miR-769-3p、ssc-miR-1343、ssc-miR-7137-3p、ssc-miR-671-5p、ssc-miR-149)的共转染组相对荧光活性比对照组显著降低(P<0.05)。其中miR1343和miR7137-3p对ATGL3′UTR报告基因载体的相对荧光活性降低显著,分别达到了48.8%和42.3%,而miR769-3p则只降低9.4%。有些报道中,miRNA对一些靶基因的表达量能抑制50%甚至更多[19],这可能和miRNA与靶基因的结合程度有关,有些结合程度较高,有些结合程度较低[20]。继续进行靶位点突变试验,进一步缩小范围检测这些miRNA对其结合位点突变的ATGL基因3′UTR荧光素酶报告载体活性的影响,发现miR-1343和miR-7137-3p对ATGL的抑制作用因为种子区的突变得以回复,进一步说明了这一作用是由于miRNA与ATGL基因3′UTR的作用而产生,且相应的种子区是miR-1343和miR7137-3p与ATGL结合起关键作用的碱基,因此确定了ssc-miR1343和 ssc-miR-7137-3p与ATGL结合的靶位点。miR-671-5p对ATGL的抑制作用没有因为突变得到回复,说明了miRNA-671-5p与ATGL3′UTR存在另外的结合位点而非预测的种子区。整个筛选过程也说明了生物信息学预测的结果存在一定的假阳性率,需要进一步的试验验证。
关于ssc-miR-1343和ssc-7137-3p的研究都还较少。G.Li等[21]采用Solexa测序技术发现ssc-miR-1343在7和240日龄荣昌猪背部皮下脂肪组织中表达;C.Chen等[22]采用相同的Solexa测序方法发现150日龄的大约克和梅山猪背部皮下脂肪组织中表达ssc-miR-1343,且通过茎环qPCR验证了梅山猪中ssc-miR-1343的表达量比大约克猪高出一倍;最新发现其在乳汁中也有表达[23]。这也都说明ssc-miR-1343与猪脂肪组织的发育和脂肪代谢有着某些联系,而本研究发现的ssc-miR-1343与猪ATGL3′UTR的互作模式也许能为这个联系提供一个答案,ssc-miR-1343可能通过调控ATGL的表达进而影响猪脂肪代谢。而对于ssc-miR-7137-3p的报道更少,其最早发现于猪感染胸膜肺炎放线杆菌后的肝组织中[24],后来发现在猪乳汁中也有表达[23],但还未见有关ssc-miR-7137-3p与脂肪代谢关系的报道。
由于每个miRNA可以有多个靶基因,而几个miRNAs也可以调节同一个基因[25]。这种复杂的调节网络既可以通过一个miRNA来调控多个基因的表达,也可以通过几个miRNAs的组合来精细调控某个基因的表达。本研究初步筛选出2种miRNAs对猪ATGL具有较显著的调控作用,但其具体的调控网络仍需进行更深入的研究,而且还有待于进一步检测ssc-miRNA-1343和ssc-miR-7137-3p对猪ATGL蛋白水平的调控。
4 结 论
本研究成功构建了猪ATGL基因3′UTR的荧光素酶验证载体和突变载体,初步筛选出与猪ATGL基因3′UTR 互作的5种miRNAs (ssc-miR-769-3p、ssc-miR-1343、ssc-miR-7137-3p、ssc-miR-671-5p、ssc-miR-149),并通过突变试验发现ssc-miR-1343和ssc-miR-7137-3p能通过预测的“种子区”靶向结合于猪ATGL基因3′UTR,并下调其表达,为进一步发掘调控猪ATGL基因表达的miRNA提供了试验基础。
[1] ZIMMERMANN R,STRAUSS J G,HAEMMERLE G,et al.Fat mobilization in adipose tissue is promoted by adipose triglyceride lipase[J].Science,2004,306:1383-1386.
[2] JENKINS C M,MANCUSO D J,YAN W,et al.Identification,cloning,expression,and purification of three novel human calcium-independent phospholipase A2 family members possessing triacylglycerol lipase and acylglyceroltransacylase activities[J].JBiolChem, 2004,279(47):48968-48975.
[3] VILLENA J A,ROY S,SARKADI-NAGY E,et al.Desnutrin,an adipocyte gene encoding a novel patatin domain-containing protein,is induced by fasting and glucocorticoids:ectopic expression of desnutrin increases triglyceride hydrolysis[J].JBiolChem,2004,279(45):47066-47075.
[4] LANGIN D,DICKER A,TAVERNIER G,et al.Adipocyte lipases and defect of lipolysis in human obesity[J].Diabetes,2005,54(11):3190-3197.
[5] LAKE A C,SUN Y,LI J L,et al.Expression,regulation,and triglyceride hydrolase activity of Adiponutrin family members[J].JLipidRes,2005,46:2477-2487.[6] HAEMMERLE G A,LASS R,ZIMMERMANN G,et al.Defective lipolysis and altered energy metabolism in mice lacking adipose triglyceride lipase[J].Science,2006,312:734-737.
[7] BARTEL D P.MicroRNAs:genomics,biogenesis,mechanism,and function[J].Cell,2004,116(2):281-297.[8] 郑永霞,焦炳华.MiRNA 的生物形成及调控基因表达机制[J].生命的化学,2010,30(6):821-826. ZHENG Y X,JIAO B H.Biogenesis of microRNA and mechanism of the regulation of gene expression[J].ChemistryofLife,2010,30(6):821-826.(in Chinese)
[9] XU P,VERNOOY S Y,GUO M,et al.The Drosophila microRNA Mir-14 suppresses cell death and is required for normal fat metabolism[J].CurrBiol,2003,13(9):790-795.
[10] WILFRED B R,WANG W X,NELSON P T.Energizing miRNA research:a review of the role of miRNAs in lipid metabolism,with a prediction that miR-103/107 regulates human metabolic pathways[J].MolGenetMetab,2007,91(3):209-217.
[11] ESAU C,DAVIS,MURRAY S F,et al.MiR-122 regulation of lipid metabolism revealed byinvivoantisense targeting[J].CellMetab,2006,3(2):87-98.
[12] POY M N,ELIASSON L,KRUTZFLDT J,et al.A pancreatic islet-specific microRNA regulates insulin secretion[J].Nature,2004,432(7014):226-230.
[13] WANG Q Y,LI C,WANG J H,et al. MiR-1792 cluster accelerates adipocyte differentiation by negatively regulating tumor suppressor Rb2/ p130[J].ProcNatlAcadSciUSA, 2008,105(8):2889-2894.
[14] 李国喜.猪脂肪组织发育相关miRNA的鉴定及miR-103生物学功能的初步研究[D].杨凌:西北农林科技大学,2011. LI G X.Identification of microRNAs related to the swine adipose tissue development and primary research on the biological roles of miR-103[D].Yangling:Northwest Agriculture and Forestry University,2011.(in Chinese)
[15] ESAU C,KANG X,PERALTA E, et al.MicroRNA-143 regulates adipocyte differentiation[J].JBiolChem, 2004,279(50):52361-52365.
[16] 李鹏昊.MiR-27a、miR-143对猪脂肪沉积和miR-1、miR-206对肌肉肥大的调控研究[D].雅安:四川农业大学,2011. LI P H.Roles of miR-1、miR-206、miR-27a、miR-143 in the fat deposition and muscular hypertrophy of swine[D].Yaan:Sichuan Agricultural University,2011.(in Chinese)
[17] LI H,CHEN X,GUAN L,et al.MiRNA-181a regulates adipogenesis by targeting tumor necrosis factor-α (TNF-α) in the porcine model[J].PLoSONE,2013,8(10):e71568.
[18] DAS S K.Role of lipolysis in cancer associated cachexia and regulation of lipolysis by microRNA 124a[D].Graz:Medical University of Graz,2011.
[19] ZHAO Y,RANSOM J F,LI A,et al.Dysregulation of cardiogenesis,cardiac conduction,and cell cycle in mice lacking miRNA-1-2[J].Cell,2007,129(2):303-317.[20] BARTEL D P.MicroRNAs:genomics,biogenesis,mechanism,and function[J].Cell,2004,116(2):281-297.[21] LI G,LI Y,LI X,et al.MicroRNA identity and abundance in developing swine adipose tissue as determined by Solexa sequencing[J].JCellBiochem,2011,112(5):1318-1328.
[22] CHEN C,DENG B,QIAO M,et al.Solexa sequencing identification of conserved and novel microRNAs in backfat of large white and chinese Meishan pigs[J].PLoSONE,2012,7(2):e31426.
[23] CHEN T,XI Q Y,YE R S,et al.Exploration of microRNAs in porcine milk exosomes[J].BMCGenomics,2014,15:100.
[24] PODOLSKA A,ANTHON C,BAK M,et al.Profiling microRNAs in lung tissue from pigs infected withActinobacilluspleuropneumoniae[J].BMCGenomics,2012,13:459.
[25] CHO W C S.OncomiRs:the discovery and progress of microRNAs in cancers[J].MolCancer,2007,6(2):60-67.
(编辑 郭云雁)
Prediction and Validation of miRNA Targeting PorcineATGLGene
DAI Li-he,CHU Xiao-hong,LU Fu-zeng,HUANG Sun-ping,XU Ru-hai*
(ZhejiangEngineeringResearchCenterforLivestockandPoultryGeneticBreeding,InstituteofAnimalHusbandryandVeterinaryScience,ZhejiangAcademyofAgriculturalSciences,Hangzhou310021,China)
To preliminary screen out ssc-miRNAs targeting porcine adipose triglyceride lipase gene (ATGL),10 ssc-miRNAs targetingATGLwere predicted by bioinformatics analysis.3′UTR of porcineATGLgene was inserted into the pMIR-Luc target reporter vector and transiently co-transfected with candidate miRNA mimics into HEK 293T cells,using pRL-TK vector as an internal control reporter.Three other reporter vectors containing the mutated ssc-miRNAs targeting sites by reversing the seed region of targeting sites were created as negative controls.The dual luciferase reporter assay system was used to evaluate the activity of luciferase.The results showed that pMIR-ATGL-3′UTR recombinant vector was successfully constructed which contained 3′UTR of porcineATGLgene.Dual luciferase reporter assay showed that ssc-miR-769-3p,ssc-miR-1343,ssc-miR-7137-3p,ssc-miR-671-5p and ssc-miR-149 could down-regulate the luciferase activity of pMIR-ATGL-3′UTR vector in 293T cell,while mutant assay verified ssc-miR-1343 and ssc-miR-7137-3p could down-regulate the luciferase activity by targeting the predicted seed region of 3′UTR of porcineATGLgene.These results indicated that ssc-miR-1343 and ssc-miR-7137-3p could down-regulate the expression of porcineATGLgene by targeting its 3′UTR.
pig;ATGLgene;miRNA;dual luciferase reporter system;3′UTR
10.11843/j.issn.0366-6964.2015.08.002
2014-11-21
国家自然科学基金(31201783);“十二五”农村领域国家科技计划课题(2011AA100304-1);浙江省自然基金(Y3100569);浙江省育种专项(2012C12906-5)
戴丽荷(1982-),女,浙江天台人,助理研究员,博士,主要从事猪遗传育种研究,E-mail:dailihe505@126.com
*通信作者:徐如海,E-mail:xuruhai@163.com
S828.2
A
0366-6964(2015)08-1281-09

