菲律宾蛤仔EST_SSR标记与生长性状的相关分析
牛泓博,聂鸿涛,朱德鹏,杨 凤,闫喜武
大连海洋大学,辽宁省贝类良种繁育工程技术研究中心, 大连 116023
菲律宾蛤仔EST_SSR标记与生长性状的相关分析
牛泓博,聂鸿涛,朱德鹏,杨 凤,闫喜武*
大连海洋大学,辽宁省贝类良种繁育工程技术研究中心, 大连 116023
研究利用20 个微卫星标记对菲律宾蛤仔斑马蛤F2代家系107 个个体进行遗传多样性分析,并对标记位点与生长相关性状进行分析。在20 个微卫星位点共检测到41 个等位基因,各位点等位基因数为2—3 个,等位基因片段大小为109—430 bp,平均等位基因数为2.05 个。平均有效等位基因数为1.71个,观测杂合度平均值为0.504,期望杂合度的平均值为0.431,平均多态信息含量为0.324。经卡方检验,3 个位点SSR11,SSR164和SSR213的基因型分布显著偏离了孟德尔定律(P<0.01)。运用SPSS 20.0对20 个微卫星位点与菲律宾蛤仔斑马蛤家系生长性状的相关性(壳长、壳宽、壳高和体重)进行连锁显著性检验。结果表明,SSR9位点与壳高存在显著的相关关系(P<0.05),SSR135和SSR164位点与壳宽呈显著相关(P<0.05),SSR142位点与体重呈显著性相关(P<0.05)。研究结果可为菲律宾蛤仔的分子标记辅助选育提供参考。
菲律宾蛤仔; 微卫星标记; 生长性状; 相关分析
菲律宾蛤仔(Ruditapesphilippinarum)隶属于软体动物门(Mollusca),双壳纲(Bivalvia),异齿亚纲(Heterodonta),帘蛤目(Veneroida),帘蛤科(Veneridae),蛤仔属(Ruditapes),是我国传统四大养殖贝类之一,广泛分布在我国南北沿海。据统计,2010年菲律宾蛤仔世界产量达到360多万t[1],我国年产量在300 万t左右,占世界菲律宾蛤仔产量的90%以上[2]。由于其营养丰富、味道鲜美,近年来市场需求量也日益增大。然而由于人工苗种的累代养殖,菲律宾蛤仔养殖群体的种质逐渐下降,养殖产业迫切需要菲律宾蛤仔的优良品种。近年来,菲律宾蛤仔的遗传改良技术取得了一定的成绩,如选择育种和杂交育种等[2-6]。传统育种方法对目标性状缺乏准确可靠的早期预选手段,造成育种盲目性大、效率低。而紧密连锁的DNA标记为实现水产动物目标性状的早期选择提供了有效途径。因此,研究与水产动物重要经济性状紧密连锁的分子标记,特别是对缺乏遗传连锁图谱的水产动物DNA标记,就显得十分重要。
微卫星(microsatellites)又称简单序列重复(Simple Sequence Repeat, SSR),一般由1—6 bp的简单序列重复排列组成。由于微卫星具有多态性丰富、共显性遗传、遵循孟德尔分离定律、易于(Polymerase Chain Reaction) PCR扩增等优点,是分子遗传学研究的理想分子标记,已广泛应用于水产动物的重要经济性状相关分子标记筛选、(Quantitative Trait Locus) QTL定位等方面[7-15]。在海洋贝类,虾夷扇贝[16]和马氏珠母贝[17]等已报道了生长性状与微卫星标记的相关性分析。目前,对菲律宾蛤仔的研究,主要集中在生物学、生态学和养殖技术等方面[2]。然而,有关菲律宾蛤仔重要经济性状的分子基础研究还刚刚起步,标记与生长性状的相关分析迄今还未见报道。
本实验利用20 对微卫星标记对菲律宾蛤仔斑马蛤家系进行遗传多样性分析,并对标记位点与生长性状相关性进行分析,筛选出对斑马蛤生长有利的基因型,实现分子标记和生长性状的连锁分析。为菲律宾蛤仔优良品系的进一步选育,以及遗传图谱的构建和QTL定位等提供参考。
1 材料与方法
1.1 实验材料
实验所用斑马蛤材料为2011年大连石河菲律宾蛤仔野生群体中挑选的斑马蛤为亲本,交配产生F1代群体,上选F1代群体中大的个体,采用巢式设计建立了30 个F1家系,本实验所用家系为第8 组家系,随机选取其中107 个个体,对所有个体的壳长、壳宽、壳高和体重进行测量。取部分足尖组织,固定于95%乙醇中用于DNA的提取。
1.2 DNA提取及微卫星分析
DNA的提取采用苯酚-氯仿-异戊醇法抽提DNA,并用1%琼脂糖凝胶电泳进行检测。DNA保存于-20 ℃备用。PCR反应体系:反应总体积为10 μL,其中DNA模板1 μL、上下游引物(10 μmol/L) 各0.4 μL、easytaq MIX 5 μL、ddH2O 3.2 μL。实验中所用微卫星引物的核心序列及退火温度见表1。PCR反应程序为:94 ℃ 5 min,94 ℃ 40 s,退火温度40 s,72 ℃ 40 s,30 个循环;72 ℃ 延伸10 min,4 ℃保存。PCR扩增产物经12%聚丙烯酰胺凝胶电泳检测,银染显色。凝胶在扫描仪上成像,拍照保存后对电泳谱带进行分析。等位基因大小用100 bp marker为参照标准对电泳条带进行判读。
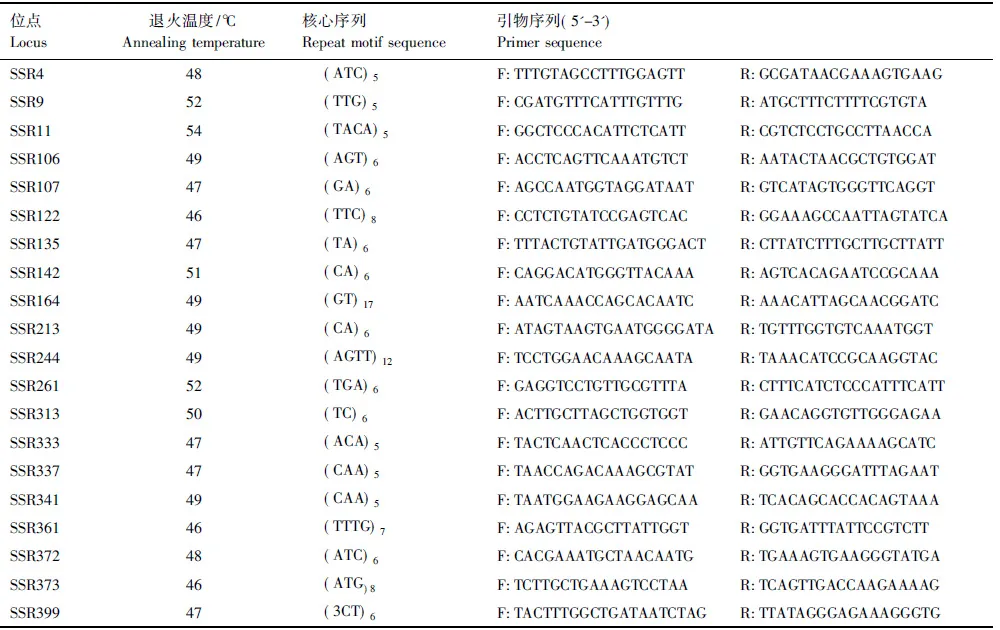
表1 菲律宾蛤仔20个微卫星引物序列和退火温度Table 1 Characteristics of the 20 polymorphic microsatellite loci from Ruditapes philippinarum
1.3 统计分析
用PopGene3.2软件进行数据处理、遗传多样性分析, 计算等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)及期望杂合度(He)。根据Botstein等[18]的方法可计算多态信息含量(PIC)。利用PIC-CALC计算软件,根据等位基因频率来计算每个位点的多态信息含量,其计算公式如下:
式中,n为某一位点上的等位基因数;pi、pj分别为群体中第i和第j个位点的等位基因频率,j=i+1。
用SPSS20.0软件中的一般线性模型(GLM) 对菲律宾蛤仔斑马蛤家系的生长相关性状与20个微卫星标记相关性进行分析。其线性模型如下:
yij=μ+gi+εij
式中,yij为斑马蛤某性状第i个标记第j个个体表型值,μ为群体平均值,gi为第i个标记的效应值,εij为随机误差。
2 结果
2.1 性状的表型值分布
经测量得到的壳长、壳宽、壳高和体重4 个生长性状,均符合正态分布,可直接进行相关性分析。正态分布信息见表2。
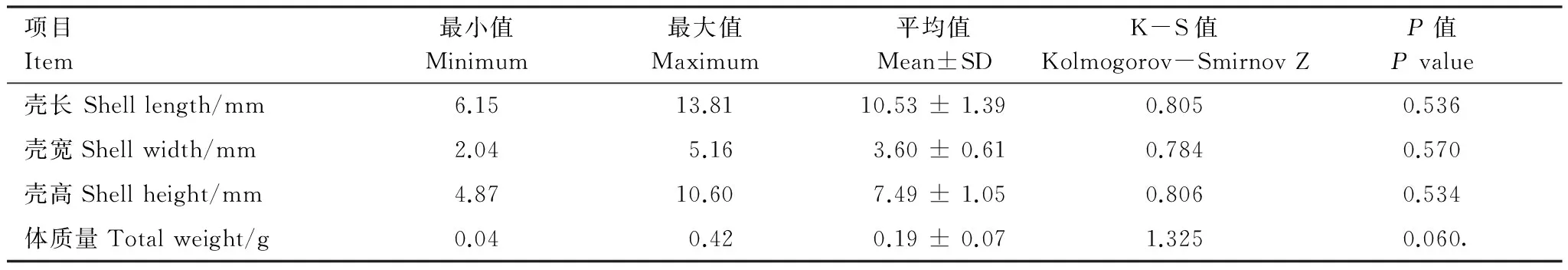
表2 壳长、壳宽、壳高和体重的表型数据及正态分布统计检验Table 2 The phenotypic data of shell lenth, width, height, body weight and normal distribution test
2.2 微卫星位点基因型
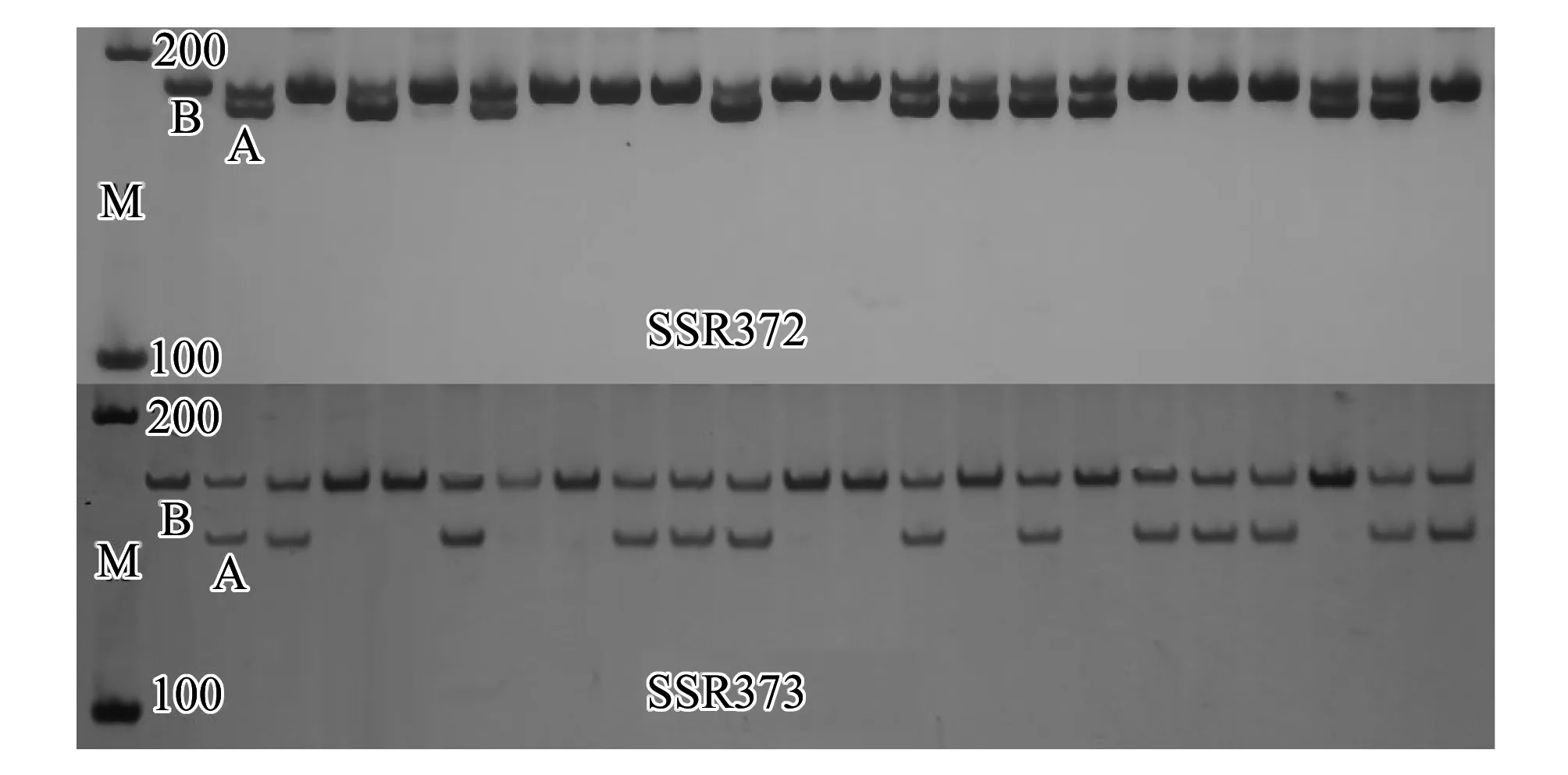
图1 引物SSR372和SSR373在斑马蛤F2 家系部分个体的聚丙烯电泳图谱Fig.1 Electrophoresis pattern of PCR product of partial individuals in the F2 family of Ruditapes philippinarum amplified with microsatellite marker SSR372 and SSR373
利用父母本和8个子代进行引物筛选,经PCR 扩增、聚丙烯酰胺凝胶电泳,从32 对微卫星引物中筛选得到20 对扩增条带清晰、稳定,具有多态性的位点。检测了父母本和107 个F2子代个体在20 个微卫星位点的基因型(表3)。SSR372和SSR373位点在菲律宾蛤仔斑马蛤家系部分个体中的扩增结果见图1。
20 个微卫星位点共检测到41 个等位基因,片段大小为109—430 bp,各位点的等位基因数2—3 个,平均等位基因数2.05 个,平均有效等位基因数为1.71 个。各位点的期望杂合度、观测杂合度、基因型分布和多态信息含量等值见表3。20 个位点的期望杂合度平均值为0.431,观测杂合度平均值为0.504,多态信息含量平均值为0.324。在20 个位点中,有2 个位点SSR213和SSR261的观测杂合度低于期望杂合度,表现出纯合子过剩、杂合子不足现象。另外18 个位点的观测杂合度则高于期望杂合度,其中SSR11和SSR164的观测杂合度与期望杂合度的差异较大。经卡方检验,有17 个位点的基因型比例符合孟德尔分离定律,另外3 个位点SSR11,SSR164和SSR213的基因型分布显著偏离了孟德尔定律(P<0.01)(表3)。
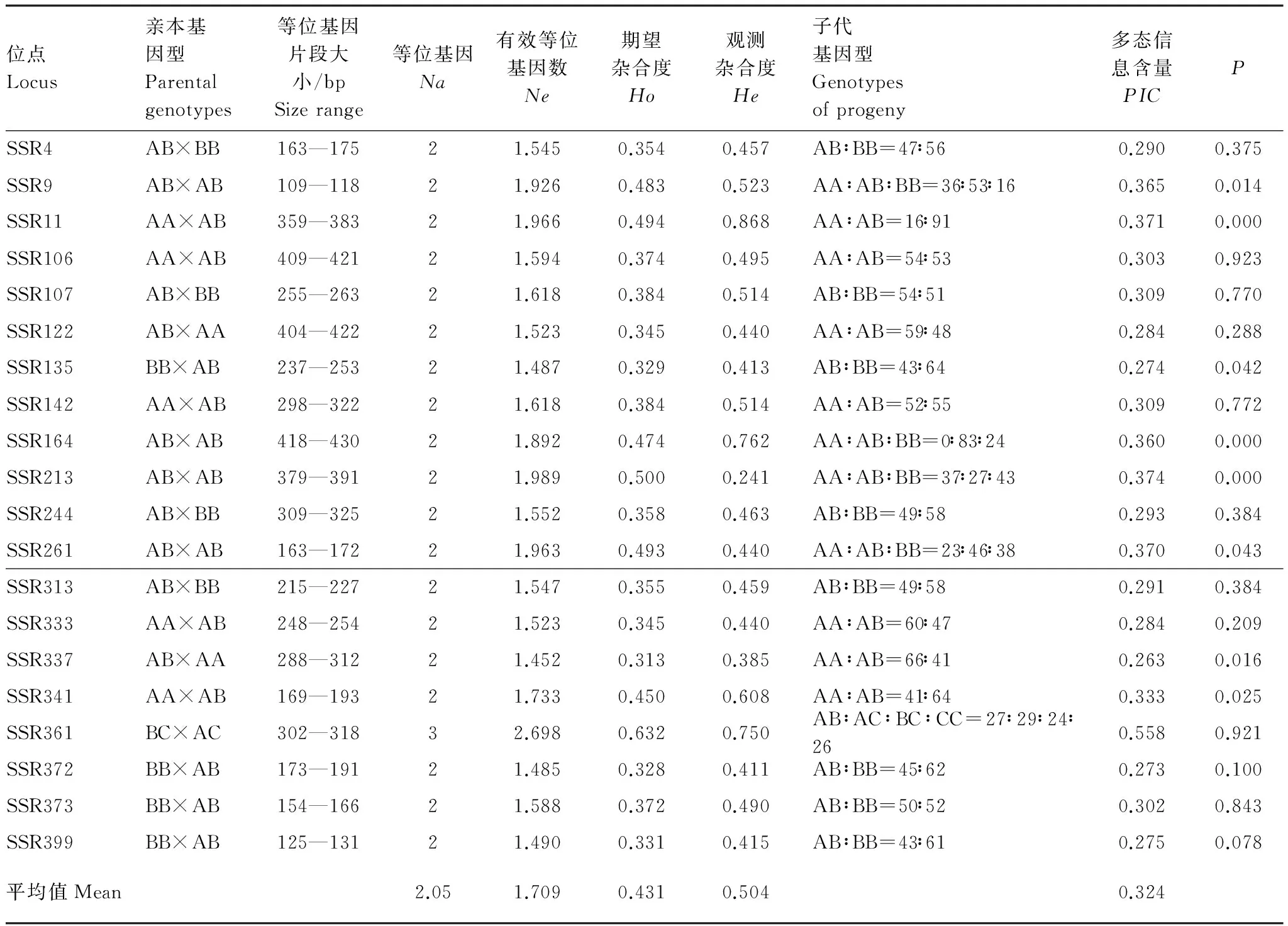
表3 菲律宾蛤仔20个微卫星位点在斑马蛤F2家系中的统计信息Table 3 Statistic information for 20 microsatellite loci in an F2 family of Ruditapes philippinarum
Na: The number of alleles;Ne: Number of effective alleles;Ho: Observed heterozygosity;He: Expected heterozygosity; PIC: Polymorphism information content;P为基因型分离比的卡方检验;加粗P值代表偏分离的基因型分离比 (P<0.01)
2.3 微卫星标记与生长性状的相关性分析
用SPSS 20.0软件中的一般线性模型(General Linear Model, GLM),对微卫星位点基因型与斑马蛤主要生长性状(壳长、壳宽、壳高和体重)之间的相关性进行分析,经方差分析检验呈显著性差异的位点。在20 对微卫星位点中,共找出4 个与斑马蛤生长相关的位点(表4)。SSR9位点与壳高呈显著关联,SSR135和SSR164与壳宽显著相关,SSR142位点与体重呈显著性相关。由于相关分析得到的微卫星位点除SSR9号引物外其他3 个位点基因型个数都小于3,故使用SPSS中compare means过程进行不同位点表型值的比较。在SSR9位点上,AA基因型的壳长,壳宽,壳高和体重4 项生长指标平均值均高于其他基因型个体,说明AA基因型对斑马蛤生长起正效应。另外,SSR135位点上的BB基因型、SSR142位点上的AA基因型和SSR164位点上的AB基因型,在4 项生长指标中的平均值均高于各自位点中的其他基因型,说明4个基因型在各自位点上对斑马蛤的生长具有正效应。
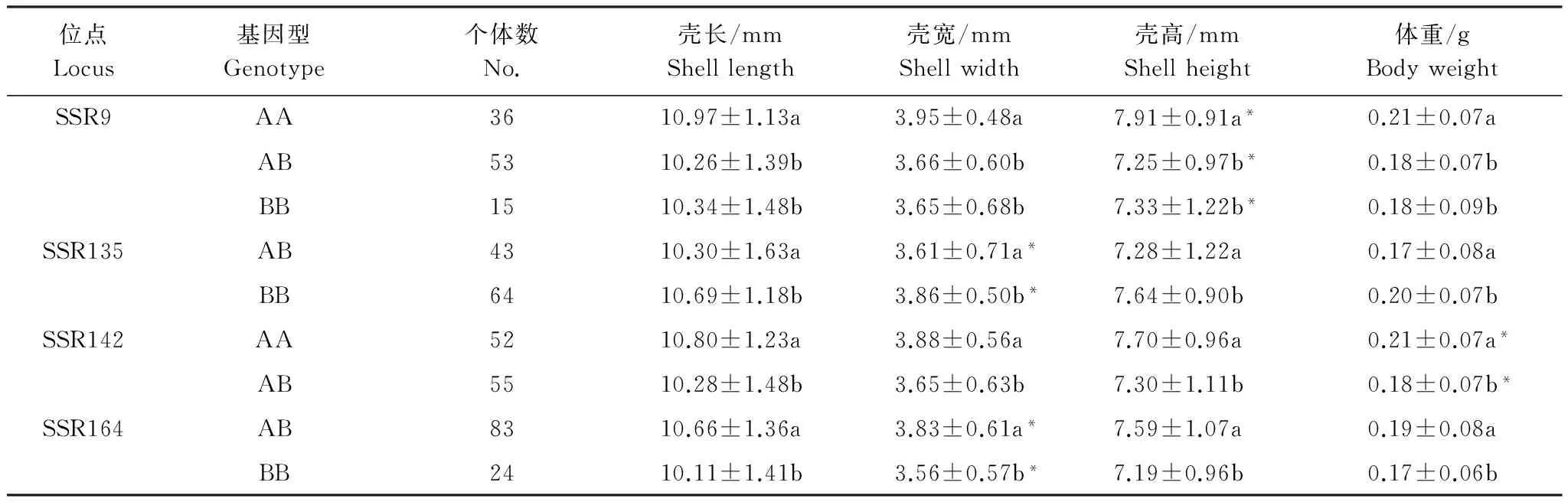
表4 20个微卫星位点不同基因型主要生长性状的平均值及多重比较Table 4 Means and multiple comparisons of major growth traits with different genotypes at 20 microsatellites
上标字母表示在同一位点中不同基因型之间差异显著(P<0.05),无字母表示基因型间差异不显著,*表示与生长相关的位点
3 讨论
群体的遗传多样性主要表现在杂合度、等位基因数和多态信息含量三个方面。杂合度大小可以反映出遗传结构变异程度的高低,杂合度期望越高,群体的遗传结构越复杂[19]。本实验中,观测杂合度和期望杂合度的平均值分别为0.504和0.431,说明实验家系的遗传变异和遗传多样性较丰富。从等位基因来看,有效等位基因越接近所检测到的等位基因的绝对值,说明等位基因在群体中的分布越均匀[20]。本研究在20 个微卫星位点共检测到41 个等位基因,其中有效等位基因数为34 个,接近于实际等位基因数,表明所检测的微卫星位点的等位基因在群体中分布较均匀。多态信息含量可作为一个衡量遗传标记所包含的或能提供的遗传信息容量的指标。一般认为,在某一群体中,当PIC> 0.5时该位点表现为高度多态,0.25 在许多海洋贝类中,经常会出现偏分离的等位基因,这似乎是海洋贝类中普遍存在的现象。在本实验中,发现3 个微卫星位点的基因型分离比显著偏离孟德尔遗传定律(P<0.01),部分位点偏分离的原因可能与生活环境和人工选择有关,再有可能是由隐性致死基因、取样或基因型判读错误等因素引起[22]。大部分微卫星位点的期望杂合度与观测杂合度基本接近,说明群体内的基因型频率未发生大的改变。但有2 个位点SSR11和SSR164的观测杂合度明显大于期望杂合度,在SSR164发现了一种纯合子完全缺失的现象(基因型AA为0),说明该位点附近可能存在着与之紧密连锁的隐性致死基因[23]。 菲律宾蛤仔的壳长、壳宽、壳高、体重等性状属于数量性状,数量性状受多个基因的控制,其遗传基础比较复杂且容易受环境的影响。标记与性状之间的连锁分析,是根据标记位点的基因型以及数量性状的表型对个体进行显著性检验,差异显著则说明标记与数量性状存在关联[24]。因此,如果一个群体的性状差异显著,或两个群体的差异很大,就可以通过标记与性状的相关分析,找出性状与一个或多个标记的遗传相关,一旦发现显著相关,即可认为存在一个数量性状位点,从而实现从表型到基因型选择育种的转变[25]。借助微卫星标记进行性状连锁分析或 QTL 定位,在水产动物育种中已也有较多报道[10-17]。 目前,菲律宾蛤仔遗传标记的开发较少, 不足以满足遗传作图和QTL 定位的需要。所以,有必要应用有限的微卫星标记对菲律宾蛤仔生长相关性状进行连锁分析。在本研究中,共找到了4 个与生长性状相关的位点,SSR9位点与壳高存在显著的相关关系(P<0.05),SSR135和SSR164与壳宽显著相关(P<0.05),SSR142与体重显著相关(P<0.05)。在这些关联中,出现了几个标记同一个性状相关,说明这些位点存在多因一效的现象,并符合数量性状的相关理论。对上述位点不同基因型的多重比较表明,SSR9位点的AA基因型、SSR135位点的BB基因型、SSR142位点的AA基因型和SSR164位点的AB基因型都与壳长、壳宽、壳高和体重性状呈正相关。目前,分子标记在菲律宾蛤仔中的应用主要集中于遗传多样性研究[26-27],有关菲律宾蛤仔性状相关分子标记的筛选迄今未见报道。本文筛选出菲律宾蛤仔生长性状相关的微卫星标记,为菲律宾蛤仔分子标记辅助选育提供参考。 [1] FAO. Yearbook of fishery statistics summary tables. 2011, Available from ftp://ftp.fao.org/fi/stat/summary/default.htm#aqua. [2] 张国范, 闫喜武. 蛤仔养殖学. 北京: 科学出版社, 2010. [3] 闫喜武, 张跃环, 霍忠明, 孙焕强, 杨凤, 张国范. 不同壳色菲律宾蛤仔品系F2的表型性状. 水产学报, 2010, 34(6): 701-709. [4] 闫喜武, 张跃环, 孙焕强, 霍忠明, 孙欣, 杨凤, 张国范. 菲律宾蛤仔两道红与白斑马品系的三元杂交. 水产学报, 2010, 34(8): 1190-1197. [5] Yan X W, Huo Z M, Yang F, Zhang G F. Heritability of larval and juvenile growth for two stocks of Manila clamRuditapesphilippinarum. Aquaculture Research, 2013, doi: 10.1111/j.1365-2109.2012.03250.x. [6] Zhao L Q, Yan X W, Huo Z M, Yang F, Zhang G F. Divergent selection for shell length in the Manila clam,Ruditapesphilippinarum. Journal of the World Aquaculture Society, 2012, 43(6): 878-884. [7] Cnaani A, Hallerman E M, Ron M, Weller J I, Indelman M, Kashi Y, Graham A E G, Hulata G. Detection of a chromosomal region with two quantitative trait loci, affecting cold tolerance and fish size, in an F2tilapia hybrid. Aquaculture, 2003, 223(1/4): 117-128. [8] Rodriguez M F, LaPatra S, Williams S, Famula T, May B. Genetic markers associated with resistance to infectious hematopoietic necrosis in rainbow and steelhead trout (Oncorhynchusmykiss) backcrosses. Aquaculture, 2004, 241(1/4): 93-115. [9] 高国强, 常玉梅, 韩启霞, 池炳杰, 李明云, 薛良义, 梁利群. 大黄鱼耐低温性状相关微卫星标记的筛选. 遗传, 2010, 32(3): 248-253. [10] 张天时, 刘萍, 李健, 孔杰, 王清印. 中国对虾与生长性状相关微卫星DNA分子标记的初步研究. 海洋水产研究, 2006, 27(5): 34-38. [11] 樊佳佳, 白俊杰, 李小慧, 何小平, 何小燕, 李胜杰, 叶星, 吴立新. 大口黑鲈生长性状的微卫星DNA标记筛选. 遗传, 2009, 31(5): 515-522. [12] 张研, 梁利群, 常玉梅, 侯宁, 鲁翠云, 孙效文. 鲤鱼体长性状的QTL定位及其遗传效应分析. 遗传, 2007, 29(10): 1243-1248. [13] 王桂兴, 刘永新, 孙效文, 刘海金. 牙鲆微卫星分子标记与生长性状的相关性分析. 东北农业大学学报, 2009, 40(7): 77-84. [14] 刘贤德, 韦信键, 蔡明夷, 刘洋, 王志勇. 大黄鱼22个微卫星标记在F1家系中的分离方式及与生长性状的相关分析. 水产学报, 2012, 36(9): 1322-1330. [15] 刘磊, 李健, 刘萍, 赵法箴, 高保全, 杜盈, 马春艳. 三疣梭子蟹微卫星标记与生长相关性状的相关性分析. 水产学报,2012, 36(7): 1034-1041. [16] 张曼, 陈蒙, 姬南京. 虾夷扇贝家系群体遗传结构及其微卫星标记与经济性状相关性的分析研究. 中国农学通报, 2012, 28(20): 125-130. [17] 邓岳文, 高远镇, 王学颖, 杜晓东. 马氏珠母贝生长性状与EST-SSR标记的关联分析. 农业生物技术学报, 2013, 21(1): 77-88. [18] Botstein D, White R L, Skolnick M, Davis R W. Construction of genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32(3): 314-331. [19] Yang M S, Hertel H, Schneck V. Genetic diversity and population structure ofRobiniapseudoacaciaprovenances from middle Europe. Acta Ecologica Sinica, 2004, 24(12): 2700-2706. [20] Hines H C, ZiKakis J P, Haenlein G F, Kiddy C G, Trowbridge C L. Linkage relationships among loci of polymorphism in blood and milk of cattle. Journal of Dairy Science, l 981, 64(1): 71-76. [21] Wright S. Evolution and the Genetics of Populations: Variability Within and Among Natural Population. Chicago: University of Chicago Press, 1978. [22] Launey S, Hedgecock D. High genetic load in the Pacific oysterCrassostreagigas. Genetics, 2001, 159(1): 255-265. [23] Nie H T, Li Q, Kong L. Centromere mapping in the Pacific abalone (Haliotisdiscushannai) through half-tetrad analysis in gynogenetic diploid families. Animal Genetics, 2012, 43(3): 290-297. [25] 钟金城, 陈智华. 分子遗传学与动物育种. 成都: 四川大学出版社, 2001. [26] 虞志飞, 闫喜武, 杨霏, 王金海, 张跃环, 杨凤, 张国范. 菲律宾蛤仔大连群体不同世代的遗传多样性. 生态学报, 2011, 31(15): 4199-4206. [27] 闫喜武, 虞志飞, 秦艳杰, 杨霏, 王金海, 张跃环, 杨凤, 张国范. 菲律宾蛤仔EST-SSR标记开发及不同地理群体遗传多样性. 生态学报, 2011, 31(15): 4190-4198. Identification of EST_SSR markers associated with growth-related traits in the Manila clamRuditapesphilippinarum NIU Hongbo, NIE Hongtao, ZHU Depeng, YANG Feng, YAN Xiwu* EngineeringandTechnologyResearchCenterofShellfishBreedingofLiaoningProvince,DalianOceanUniversity,Dalian116023,China The Manila clam,Ruditapesphilippinarum, which is widely distributed along the coasts of China, is an economically important marine bivalve species in China′s aquaculture industry. The world production of this species was 3.6 million metric tons in 2010. China is the first largest country in the world in terms of production of the Manila clam, producing about 3.0 million metric tons annually, which accounts for about 90% of global production. This species has several pedigrees including White, Zebra, Liangdao Red and Marine Red distributing in the coastal areas in North China. Microsatellites or simple sequence repeats (SSRs) are tandemly repeated motifs of 1—6 genetic base pairs. Microsatellite markers are powerful molecular markers due to their high polymorphism, stability, and co-dominance, and they are used widely in studies of genetic diversity, parentage assignment, genetic linkage map construction, and trait-related marker screening. In this study, 20 microsatellite DNA markers were used to analyze the genetic diversity of 107 individuals of a Zebra F2 pedigree ofR.philippinarum. Forty-one alleles were detected, and the number of alleles (Na) was 2—3 at each locus (average, 2.05). The effective number of alleles (Ne) was 1.71, and the DNA fragment length was 109—430 base pairs. The mean values of observed heterozygosity (Ho), expected heterozygosity (He), and polymorphism information content (PIC) were 0.504, 0.431, and 0.324, respectively. The probability value of the chi-square test showed that three loci significantly deviated from Mendelian segregation (P<0.01), which suggested that these loci might link with the adaptive gene, and two loci (SSR11 and SSR164) may link with recessive homozygous lethal genes. The general linear model procedure in SPSS20.0 was used to analyze the correlation between the 20 microsatellites and growth-related traits ofR.philippinarum(i.e., shell length, shell width, shell height, and body weight). Four loci were significantly related to the growth traits (P<0.05): SSR135 and SSR164 were significantly related to shell width (P<0.05), SSR9 was significantly related to shell height (P<0.05), and SSR142 was significantly related to total weight (P<0.05). Favorable genotypes for each growth trait were identified by a multiple comparison among the loci. Allele A for SSR9 had a significant impact on shell height (P<0.05) and had the highest phenotype value; thus, it can be used as a molecular marker for selective breeding. SSR135 and SSR164 had a significant impact on shell width and total weight (P<0.05), respectively, and allele B for SSR135 and allele A for SSR142 had a positive effect on growth-related traits. The following four genotypes of these loci had a favorable effect on growth-related traits: AA for SSR9, BB for SSR135, AA for SSR142, and AB for SSR164. The trait-related microsatellite loci identified in this study will be valuable for marker-assisted breeding ofR.philippinarum. Ruditapesphilippinarum; microsatellites; growth-related traits; correlation analysis 国家自然科学基金(31302183); 国家高技术研究发展计划“863”项目(2012AA10A400); 现代农业产业技术体系建设专项(CARS-48) 2013-05-16; 日期:2014-04-25 10.5846/stxb201305161083 *通讯作者Corresponding author.E-mail: yanxiwu@dlou.edu 牛泓博,聂鸿涛,朱德鹏,杨凤,闫喜武.菲律宾蛤仔EST_SSR标记与生长性状的相关分析.生态学报,2015,35(6):1910-1916. Niu H B, Nie H T, Zhu D P, Yang F, Yan X W.Identification of EST_SSR markers associated with growth-related traits in the Manila clamRuditapesphilippinarum.Acta Ecologica Sinica,2015,35(6):1910-1916.

