东乡野生稻耐冷渐渗系DNA甲基化水平和模式变化研究
温秀芳,戴亮芳,赵 俊,罗向东,邓晓娟,张帆涛,谢建坤
(江西师范大学 生命科学学院,南昌 330022)
东乡野生稻耐冷渐渗系DNA甲基化水平和模式变化研究
温秀芳,戴亮芳,赵 俊,罗向东*,邓晓娟,张帆涛,谢建坤
(江西师范大学 生命科学学院,南昌 330022)
根据水稻全基因组和特定位点的CpG岛序列设计引物,采用McrBC酶酶切DNA,以东乡野生稻耐冷渐渗系(IL5335和IL5423)及其双亲为试材,研究其基因组和特定位点的DNA甲基化水平和模式变化特征,探讨甲基化变异对野生优异基因渐渗的影响。结果显示:(1)在覆盖全基因组的83个CpG岛中,IL5335和IL5423的基因组甲基化频率分别为46.6%和53.8%,低于受体亲本协青早B的62.6%;大部分(75.9%~80.7%)受体亲本的甲基化模式在两耐冷渐渗中能稳定遗传,另外一些位点发生了甲基化模式的改变,主要表现为脱甲基化(13.3%~18.1%)和过甲基化(4.4%~6.0%)。(2)在耐冷QTLs区间,两耐冷渐渗系的甲基化水平为13.3%~26.7%,远低于受体亲本的61.4%;它们在该区域的甲基化模式变异主要为脱甲基化(33.3%~40.0%),高于全基因组的平均变异率。(3)分析逆转座子Houba和Osr14区域的51个CpG岛发现,耐冷渐渗系在该区域具有较高频率的甲基化修饰和较低的甲基化模式变异。研究表明,种间杂交渐渗诱发了受体亲本广泛的甲基化水平和模式变异,为野生优异基因的有效利用提供了新的机遇和挑战。
东乡野生稻;耐冷渐渗系;DNA甲基化;McrBC酶
DNA甲基化(DNA methylation)是真核生物DNA中一种可逆且能遗传的表观遗传修饰,广泛存在于细菌和动植物基因组中[1]。DNA甲基化通常被认为是基因组DNA的一种防御型机制[2]。但事实上,在植物的生长发育过程中DNA甲基化修饰起着非常重要的作用,它能关闭或启动某些基因的转录表达程序,参与基因的表达调控、组蛋白修饰、染色体印记、着丝粒和端粒结构的维持等生命活动[3]。因此,DNA甲基化的遗传与可塑性对生物体维持正常的生命活动至关重要。
研究表明,外界因素可诱发植物基因组甲基化变异,如远缘杂交、转基因、离体培养、环境胁迫等[4-5]。前人对拟南芥、棉花、大米草、玉米等种间杂交后代的研究发现,外源物种DNA的导入可诱发受体物种基因组广泛的DNA甲基化水平和模式的变化,引发相关基因的表达或沉默,进而引起种间杂交后代的基因表达的表型与其双亲具有显著差异[6-7]。因此,DNA甲基化水平和模式的改变一方面为种间杂交创造新的遗传变异提供了新的育种途径和思路,但同时对野生优异基因的有效利用提出了新的挑战。为此,阐明种间杂交后代中甲基化模式的变异的遗传与分子机制具有重要的理论和实践意义。
东乡野生稻是一种全球分布最北的普通野生稻(OryzarufipogonGriff.),蕴藏大量栽培稻品种改良所没有的优异基因,如高产、强耐冷、抗旱等[8]。利用东乡野生稻创制水稻(O.sativaL.)新种质已成为改良栽培稻重要农艺性状和拓宽水稻遗传基础的重要课题。此前本课题组通过种间杂交和多次回交、自交获得了一些携带野生稻优异基因的抗旱、耐冷、耐低磷渐渗系[8-9]。随后的研究发现,这些高世代渐渗系中伴随一些非孟德尔式的表观遗传变异,如高世代(BC1F9)强耐冷渐渗系IL5243和IL5335在株型、株高以及谷粒颜色等形态学性状还存在一定变异或分离,其逆转录酶的序列结构和荧光定量的相对表达量与供体亲本有很大的差异[10],减数分裂也存在落后染色体及双核仁等不稳定现象[8]。为了更全面地了解野生稻优异基因在向栽培稻渐渗过程中发生的遗传与表观遗传变异特征,本研究基于水稻全基因组序列信息,对水稻全基因组的CpG岛进行分析,利用CpG两侧的序列设计甲基化特异性PCR扩增引物,分析东乡野生稻耐冷渐渗系的在种间杂交渐渗过程中所发生甲基化水平和模式变化特征。在此基础上,基于我们此前鉴定的东乡野生稻耐冷基因粗定位QTLs结果[11]和耐冷渐渗系中已发生逆转录酶活性变化的转座子结果[10],对渐渗系基因组的特定区域的CpG岛甲基化水平和模式变化特征作进一步分析,探讨DNA甲基化水平和模式变化对外源优异基因渐渗的影响,为东乡野生稻耐冷基因在实际育种中的充分利用提供有益信息。
1 材料和方法
1.1 材 料
本课题组此前以东乡野生稻(O.rufipogonGriff.)为供体亲本,以栽培稻‘协青早B’(O.sativaL.cv.XieqingzaoB)为受体亲本杂交获得种间杂种F1。种间杂种F1随后与受体亲本进行回交得BC1F1,经单粒传连续自交得BC1F9代群体,该渐渗群体经耐冷鉴定筛选,获得强耐冷渐渗系IL5335和IL5243[8]。本文以东乡野生稻耐冷渐渗系(IL5335和IL5423)及其双亲为试材,研究其基因组和特定位点DNA甲基化水平和模式变化特征。
1.2 CpG岛甲基化特异扩增引物设计
利用Rice Genome Annotation Project(http://rice.plantbiology.msu.edu/)中水稻基因组信息,均匀地选择分布在12条染色体上不同BAC中的DNA序列,结合我们对耐冷基因粗定位结果[11],利用程序Methyl Primer Express v1.0在这些序列中寻找CpG岛,针对这些富含CG序列的DNA利用Primers 3 Input(version 4.0)设计了98对甲基化特异性PCR扩增引物,其中15对为耐冷基因QTL定位区间的引物,其余83条为覆盖全基因组的甲基化特异性扩增引物。耐冷粗定位区间为第1号染色体上的RM246-RM1095,4号染色体上的S47M-S410M,12号染色体上的RM519-RM270[11]。另外,我们此前发现耐冷渐渗系中逆转座子Houba和Osr15已发生逆转录酶活性变化[10],为此根据各染色体上Houba和Osr15转座区域的DNA序列,设计特异引物51对。引物序列交于上海英俊公司合成。
1.3 甲基化引物扩增分析
采用CTAB法提取水稻基因组DNA。DNA检测合格后用McrBC酶于37 ℃酶切(约16 h以上),随后65 ℃温育1 h使酶灭活终止酶切,McrBC酶酶切体系50 μL,包含10×NBE buffer 5 μL,100×BSA 0.5 μL,GTP(100 mmol/L)0.5 μL,McrBC酶(10 U/μL)1 μL,DNA 1 μL,灭菌ddH2O 42 μL。酶切产物用ddH2O稀释10倍备用,并用阳性和阴性引物检测酶切是否完全。阳性引物是针对Tos17基因设计的(正向引物:GCTACCCGTTCTTGGACTAT;反向引物:CTGAAATCGGAGCACTGACA);阴性引物是针对不易发生甲基化的激酶位点设计的(正向引物:CGACTAAACCACTCCAA-TCATC;反向引物:CCAATCAAAACTTCTCCTGTAA)。酶切时设空白对照,即酶切体系中McrBC酶用ddH2O代替,其他体系相同[12]。
McrBC酶切完全DNA样品用于甲基化引物扩增,扩增反应总体积为15 μL,包含10×buffer 1.5 μL,MgCl2(25 mmol/L)0.9 μL,dNTP(2.5 mmol/L)1.2 μL,甲基化引物(5 μmol/L)2.0 μL,Taqpolymerase(5 U/μL)0.2 μL,McrBC酶切后的模板DNA(25 ng/μL)2 μL,灭菌水7.2 μL。PCR扩增条件为96 ℃预变性5 min,96 ℃变性30 s,58 ℃退火30 s,72 ℃延伸45 s,35个循环后,72 ℃延伸10 min,最后4 ℃保存5 min。PCR产物用1.0%的琼脂糖凝胶检测,并利用凝胶成像分析系统进行拍照,每对引物对所有材料的扩增重复3次。对照组实验中未扩增出来的材料同样也进行3次重复扩增实验。
1.4 数据统计分析
没有扩增条带的记为“0”,表示该DNA片段具有甲基化位点;扩增出目的条带的记为“1”,表示该DNA片段无甲基化位点。CpG甲基化模式变化分为A、B、C三类,其中A类表示受体亲本‘协青早B’的甲基化模式在渐渗后代中被稳定遗传,包括A1、A2两个亚类;受体亲本‘协青早B’中无甲基化的位点在渐渗后代变为甲基化位点的情况定为B类,即发生了过甲基化变异;C类表示‘协青早B’为甲基化位点,而渐渗后代中变无甲基化位点,即发生了脱甲基化变异。
2 结果与分析
2.1 基因组DNA酶切效果检测
为了确定McrBC酶对参试材料的基因组DNA酶切是否完全,本研究分别采用阳性和阴性引物对酶切产物进行扩增检测。阳性引物可扩增基因组最易发生甲基化且被切开的Tos17基因,如果酶切不完全,材料可扩增出1条700 bp左右的条带;阴性引物是针对不易发生甲基化的激酶位点设计的,可扩增出约200 bp左右的片段。本研究的McrBC酶切产物检测结果表明(图1,A),渐渗系及其双亲的酶切产物的阳性引物扩增均无700 bp的扩增产物,用阴性引物扩增时出现1条200 bp的条带,表明渐渗系及其双亲的McrBC酶切产物可用于进一步甲基化检测。
2.2 耐冷渐渗系基因组甲基化水平和模式变化特点
本研究利用覆盖全基因组12条染色体上的83对特异性甲基化引物,对McrBC酶酶切后的双亲和东乡野生稻耐冷渐渗系(IL5335和IL5423)进行特异扩增,分析其甲基化水平和模式变化特点。结果表明,受体亲本‘协青早B’、供体亲本东乡野生稻和两耐冷渐渗系都具有较高频率的DNA甲基化(45.4%~62.6%)。其中,受体亲本‘协青早B’的甲基化修饰程度要远高出供体亲本东乡野生稻,分别为62.6%和45.4%(表1和图1,B);耐冷渐渗系(IL5335和IL5243)的甲基化水平介于双亲之间,且与受体亲本存在明显差异,分别为46.6%和53.8%。IL5335的甲基化水平与东乡野生稻比较接近,两者无显著性差异。表明种间杂交渐渗诱发了受体亲本的甲基化水平发生了显著变化,而且在不同的渐渗系中甲基化水平变异也存在明显差异。
进一步分析这83个CpG岛甲基化模式的遗传和变异特征发现,渐渗系的CpG岛甲基化模式变异主要分为A、B、C三类(表2),其中A类表示受体亲本‘协青早B’的甲基化模式在渐渗中能稳定遗传,分为A1、A2两个亚类。A1和A2分别表示未甲基化位点的遗传和甲基化位点的遗传模式。B类和C类分别表示耐冷渐渗系中发生了过甲基化变异和脱甲基化变异。统计分析表明,大部分受体亲本‘协青早B’甲基化模式在两耐冷渐渗系IL5335和IL5243中能稳定遗传,遗传率分别为75.9%和80.7%。两耐冷渐渗系与受体亲本相比,分别有20和16个CpG岛位点发生了甲基化模式变异,并主要表现为脱甲基化变异(C类)和过甲基化变异(B类)。其中IL5335和IL5243脱甲基化频率分别为18.1%和13.3%;过甲基化变化(B类)分别为4.4%和6%。表明种间杂交渐渗诱发了受体亲本的甲基化模式发生了显著变异。
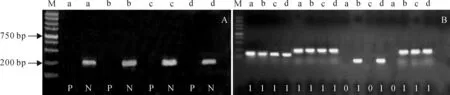
图1 参试材料McrBC酶切效果检测(A)与DNA甲基化分析部分电泳图(B)M.Marker;P、N对应的泳道分别表示阳性和阴性引物对参试材料的扩增;a~d对应泳道的材料分别为栽培稻‘协青早B’、东乡野生稻、IL5335和IL5243;0对应的泳道表示无扩增条带,表示该位点DNA含有甲基化,1对应的泳道表示扩增出目的条带,表示该位点无DNA甲基化
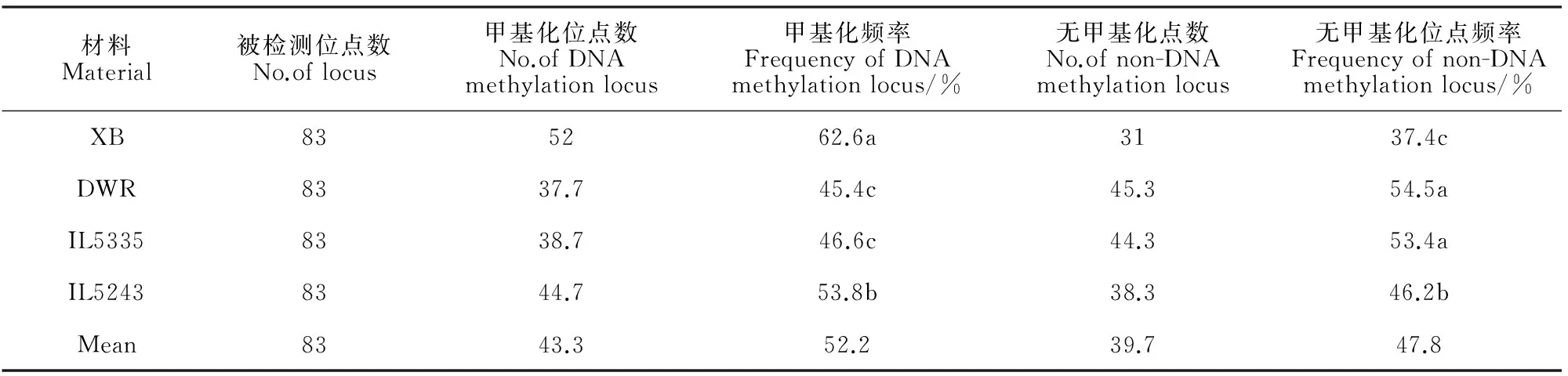
表1 耐冷渐渗系及其双亲基因组DNA甲基化水平特征
注:XB.协青早B;DWR.东乡野生稻;Mean为平均值;同列不同字母表示0.05水平显著性差异;下同。
Note:XB.OryzasativaL.cv.Xieqingzao B;DWR.Dongxiang wild rice.Mean:average values of the different material.The different normal letter within the same column represents significant difference at 0.05 level.The same as below.

表2 耐冷渐渗系基因组DNA甲基化模式的遗传与变异特点
注:XB(-)和ILs(-)分别表示在XB和ILs中未扩增出目的条带,表示在相应的位点中发生了DNA甲基化;XB(+)和ILs(+)分别表示在XB和ILs中能扩增出目的条带,表示在相应的位点未发生DNA甲基化;每列括号前的数据表示位点数,括号内表示其频率;下同。
Note:XB(-) and ILs(-) represent no band had been amplified in XB and ILs,respectively,which means that it’s DNA methylation site;XB(+) and ILs(+) represent bands could be amplified in XB and ILs,respectively,which means that it’s non-methylation site.The data before brackets was the number of locus,and in the brackets was its frequency;The same as below.
2.3 耐冷粗定位区间DNA甲基化变化特点
基于我们前期东乡野生稻耐冷QTLs的粗定位结果,对渐渗系的耐冷粗定位区间的CpG进行甲基化水平和模式特征分析。结果发现,受体亲本‘协青早B’在耐冷粗定位区间的甲基化水平达53.3%,与全基因组CpG岛甲基化水平(61.4%)无显著性差异。东乡野生稻和两耐冷渐渗系(IL5335和IL5243)在该区间的甲基化水平分别为20.0%、13.3%和26.7%,低于其全基因组CpG岛甲基化的平均水平(表1和表3)。受体亲本‘协青早B’的甲基化模式(A1和A2)大部分在两耐冷渐渗系中能得到较稳定的遗传,分别为70.0%和60.0%。与受体亲本相比,IL5335和IL5243耐冷渐渗系中也发现一定比例的甲基化遗传模式变异,并主要表现为脱甲基化(C类),分别为40%和33.3%,高于全基因甲基化模式的变异的平均水平(表1和表3)。
2.4 逆转座子区域DNA甲基化变化特点
基于我们此前的研究结果,针对耐冷渐渗系(IL5335和IL5243)中已发生逆转录酶活性变化的逆转座子Houba和Osr15区域设计了51对甲基化引物,进一步研究双亲及耐冷渐渗系在特定相关区域的甲基化水平和模式变化特征。研究发现,在Houba和Osr15逆转座子区域,‘协青早B’、东乡野生稻、IL5335和IL5243的甲基化水平分别为62.7%、56.9%、66.7%和49.0%,均高于全基因组DNA甲基化的平均水平(表1和表4)。在Houba和Osr15区域,‘协青早B’、IL5335和IL5243之间的甲基化水平无显著差异,但均与东乡野生稻之间存在显著差异。IL5335和IL5243甲基化模式大部分继承(86.6%)大部分与受体亲本’协青早B’一致,并主要表现为A1类型的遗传模式。在转座子区域,IL5335和IL5243的甲基化模式总变异率(包括过甲基化和脱甲基化)分别为15.7%和9.8%,均低于其他区域甲基化模式的变异率。表明耐冷渐渗系在Houba和Osr14转座子区域具有较高频率的甲基化修饰和较低的甲基化模式变异率,这利于转座子和基因组结构的稳定,为渐渗系优异性状的转移和性状稳定奠定基础。
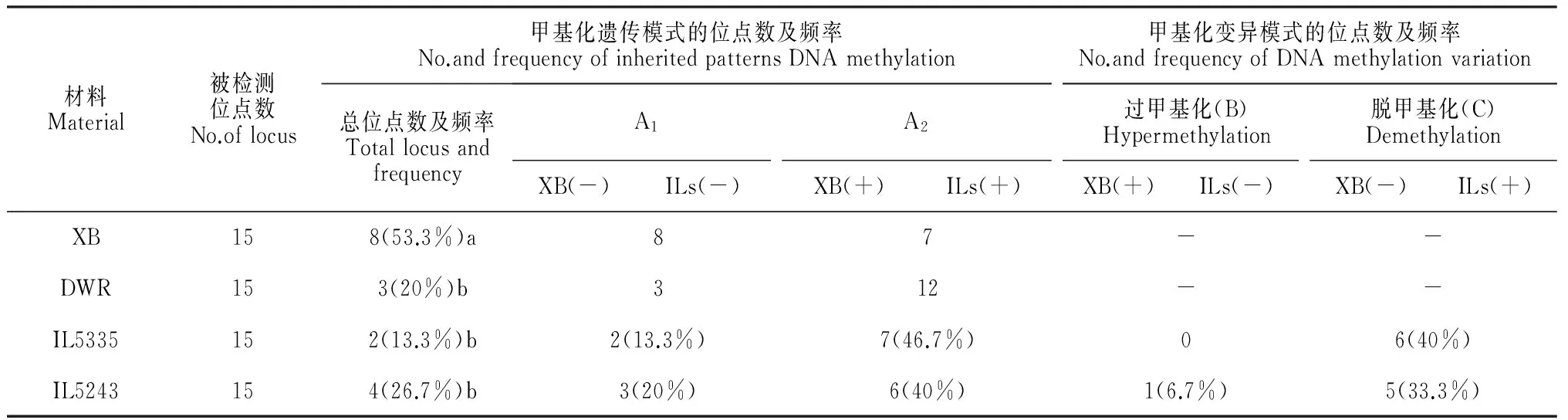
表3 耐冷渐渗系在耐冷区间DNA甲基化水平和模式的遗传与变异特点
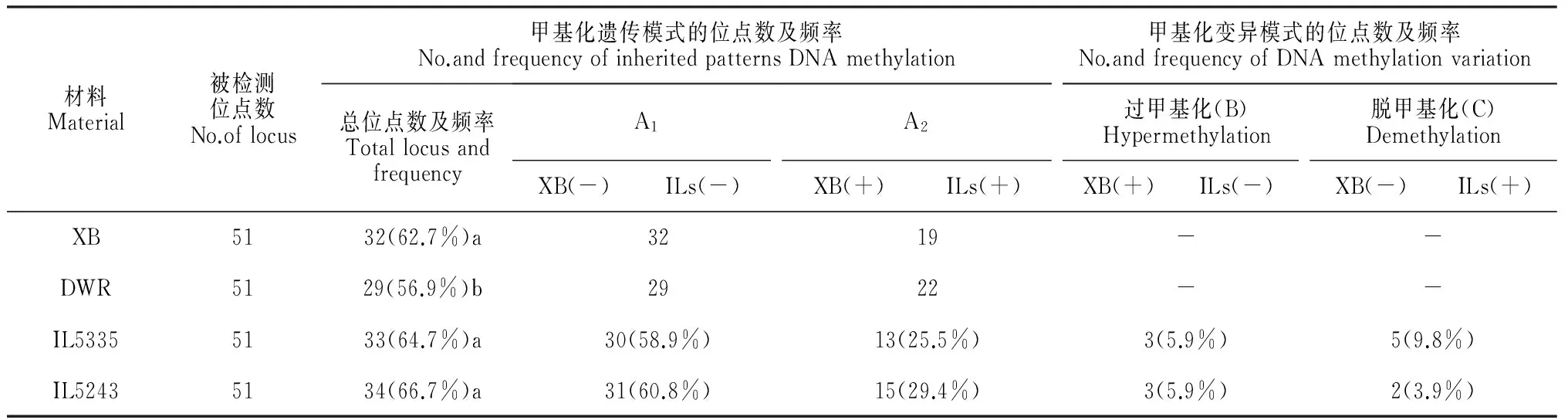
表4 耐冷渐渗系在逆转座子区域DNA甲基化水平和模式的遗传与变异特点
3 讨 论
外源DNA渐渗会给受体基因组带来一种强烈的“冲击”(Genome shock),这种冲击不仅表现在渐渗系后代中会产生各种基于基因组序列变化的遗传学变异,同时还可能发生许多表观遗传学变异[13]。远缘杂交育种实践告诉我们,表观遗传学变异在许多作物新品种改良创造了很多新的抗性和新表型,为作物遗传育种提供了新途径,因而对这些变异发生的分子机制深入研究将有助于作物品种改良[6,14]。作为表观遗传修饰的重要内容,DNA甲基化可在阻断遗传信息传递而不改变DNA序列的情况下引起生物体形态性状的变化。阐明远缘杂交渐渗过程中基因组的甲基化水平和模式变异特征及分子机制可为物种的多样性维持机制和分子进化机制提供理论基础,同时也对新品种的培育提供有益信息,相关研究已成为表观遗传学研究的热点[15-16]。
McrBC是一种甲基化内切酶,可特异切割含有甲基化胞嘧啶的DNA单链或双链,但不作用于非甲基化的DNA。结合PCR技术,此方法不仅可以检测绝大部分甲基化的胞嘧啶(如5-甲基胞嘧啶,5-羟甲基胞嘧啶或N4-甲基胞嘧啶),还可针对特异位点设计引物,研究特定染色体区域的甲基化特征,是检测胞嘧啶甲基化的有利工具[17]。本研究利用该技术分析耐冷渐渗系全基因组83个CpG岛和66个特定区域CpG岛(15耐冷区间和51转座子区间)发现,两耐冷渐渗后代稳定遗传了回交亲本的大部分甲基化水平和模式,但也有一部分位点发生了DNA甲基化模式的变异,包括过甲基化和脱甲基化,但不同区域CpG位点甲基化水平和变异模式有明显差异,这与水稻[14-15]、拟南芥[18]、大米草[19]以及玉米[20]等植物杂交后代的甲基化研究相似。与其他DNA甲基化检测方法(如甲基化敏感多态性,MSAP)相比,McrBC酶切法检测得到的DNA甲基化频率略低一些[4,21],但该技术技术稳定、重复性好,能精确研究特定位点、特定基因的甲基化水平和模式变异特征,是研究DNA甲基化水平和模式变化对外源优异基因渐渗影响的有效手段。
通常认为基因处于甲基化修饰状态时表达抑制,而处于非甲基化状态时表达活跃,但无论基因处于何种状态,都可能使渐渗后代产生相关有利性状的增强或者产生亲本没有的有利新表型,这取决于特异位点的甲基化增强或降低对这些性状的影响[6-7]。本研究结果表明,两耐冷渐渗系与受体亲本‘协青早B’相比,渐渗后代甲基化变异包括脱甲基化和过甲基化两种方式,并以脱甲基化为主,东乡野生稻和两耐冷渐渗后代IL5335和IL5243在耐冷QTLs粗定位区间的甲基化水平明显低于普通区间的CpG岛甲基化水平(分别为20.0%、13.3%和26.7%),并且发生甲基化模式变化的位点中有53.8%是在该区域,推测优异渐渗系在其形成过程中发生了更为广泛的DNA甲基化模式变化,但耐冷QTLs区间的优异基因在杂交渐渗过程中的DNA甲基化变异特征和分子机制还需进一步深入的研究。另外,我们此前发现渐渗系中Houba和Osr14转座子的活性较高,而本研究的结果表明,各参试材料在Houba和Osr14转座子区域的甲基化水平较其他区域要略高,这是否与DNA甲基化沉默转座子的活性、促进渐渗系性状稳定有关也还有待于进一步研究。
[1] DONG Z Y,WANG Y M,ZHANG Z J,etal.Extent and pattern of DNA methylation alteration in rice lines derived from introgressive hybridization of rice andZizanialatifoliaGriseb[J].TheoreticalandAppliedGenetics,2006,113:196-205.
[2] GOLL M G,BESTOR T H.Eukaryotic cytosine methyltransferases[J].AnnualReviewofBiochemistry,2005,74:481-51.
[3] NYBOM H,WEISING K,ROTTER B.DNA fingerprinting in botany:past,present,future[J].InvestigativeGenetics,2014,5:1-35.
[4] CHEN L Z,CHEN J F.Changes of cytosine methylation induced by wide hybridization and allopolyploid in cucumis[J].Genome,2008,51(10):789-799.
[5] LI L Q(李丽琴),FU CH H(付春华),ZHAO CH F(赵春芳),etal.Genetic and epigenetic variations ofTaxusmediaL.cv.hicksii during process of dedifferentiation[J].PlantPhysiologyCommunications(植物生理学通讯),2009,45(6):544-548(in Chinese).
[6] SANETOMO R,HOSAKA K.Reciprocal differences in DNA sequence and methylation status of the pollen DNA between F1hybrids ofSolanumtuberosum×S.demissum[J].Euphytica,2011,182:219-229.
[7] ABID G,MUHOVISKI Y,JACQUEMIN J M,etal.Changes in DNA-methylation during zygotic embryogenesis in interspecific hybrids of beans(Phaseolusssp.)[J].PlantCellTissOrganCulture,2011,105:383-393.
[8] LUO X D,JIAN S R,CAO J F,etal.Identification and molecular cytology analysis of cold tolerance introgression lines derived fromOryzasativaL.mating withO.rufipogonGriff[J].Euphytica,2012,187:461-469.
[9] JIAN SH R(简水溶),WAN Y(万 勇),LUO X D(罗向东),etal.Genetic analysis of cold tolerance at the seedling stage in Dongxiang wild rice(Oryzarufipogon)[J].ChineseBulletinofBotany(植物学报),2011,46(1):21-27(in Chinese).
[10] CHEN Y L(陈雅玲),LUO X D(罗向东),ZHANG F T(张帆涛),etal.Cloning and expression analysis of retrotransposon reverse transcriptase in introgression lines from Dongxiang wild rice[J].ChineseBulletinofBotany(植物学报),2013,48(2):138-144(in Chinese).
[11] 简水溶.东乡野生稻耐冷性状的QTL定位[D].南昌:江西师范大学,2011.
[12] SCHUMACHER A,KAPRANOV P,KAMINSKY Z,etal.Microarray-based DNA methylation profiling:technology and applications[J].NucleicAcidsResearch,2006,34:528-542.
[13] DENG X J,LUO X D,CHEN Y L,etal.Genetic diversity and genetic changes in the introgression lines derived fromOryzasativaL.mating withO.rufipogonGriff[J].JournalofIntegrativeAgriculture,2012,11(7):1 059-1 066.
[14] YASUDA K,TSUKIYAMA T,KARKI S,etal.Mobilization of the active transposon mPing in interspecific hybrid rice betweenOryzasativaandO.glaberrima[J].Euphytica,2013,192:17-24.
[15] KANTAMA L,JUNBUATHONG S,SAKULKOO J,etal.Epigenetic changes and transposon reactivation in Thai rice hybrids[J].MolecularBreeding,2013,31:815-827.
[16] WANG W S,PAN Y J,ZHAO X Q,etal.Drought-induced site-specific DNA methylation and its association with drought tolerance in rice(OryzasativaL.)[J].JournalofExperimentalBotany,2011,62:1 951-1 960.
[17] ZILBERMAN D,HENIKOFF S.Genome-wide analysis of DNA methylation patterns[J].Development,2007,134:3 959-3 965.
[18] MADLUNG A,MASUELLI R W,WATSON B,etal.Remodeling of DNA methylation and phenotypic and transcriptional changes in syntheticArabidopsisallotetraploids[J].PlantPhysiology,2002,129:733-746.
[19] SALMON A,AINOUCHE M L,WENDEL J F.Genetic and epigenetic consequences of recent hybridization and polyploidy inSpartina(Poaceae)[J].MolecularEcology,2005,14:1 163-1 175.
[20] SEKHON R S,CHOPRA S.Progressive loss of DNA methylation releases epigenetic gene silencing from a tandemly repeated maizeMybgene[J].Genetics,2009,181:81-91.
[21] DONG Z Y,WANG Y M,ZHANG Z J,etal.Extent and pattern of DNA methylation alteration in rice lines derived from introgressive hybridization of rice andZizanialatifoliaGriseb[J].TheoreticalandAppliedGenetics,2006,113:196-205.
(编辑:宋亚珍)
DNA Methylation Level and Pattern Changes in Cold ToleranceIntrogression Lines Derived fromOryzarufipogonGriff.
WEN Xiufang,DAI Liangfang,ZHAO Jun,LUO Xiangdong*,DENG Xiaojuan,ZHANG Fantao,XIE Jiankun
(College of Life Science,Jiangxi Normal University,Nanchang 330022,China)
In the present study,DNA methylation specific PCR primers were designed according to the sequences of CpG island region throughout 12 chromosomes and the specific genomic position.In order to investigate the changes of DNA methylation level and pattern in the cold tolerance introgression lines (ILs,IL5335 and IL5423) and their parents,we used these special primers to amplify the previously digested genomic DNA by McrBC enzyme.(1)Analysis of 83 pairs of primers distributed across the 12 chromosomes of rice showed that the frequency of DNA methylation in IL5335 and IL5423 was to 46.6% and 53.8%,respectively,which was lower than that of recurrent parent (OryzasativaL.cv.Xieqingzao B).Most of DNA methylation patterns of recurrent parent could be inherited in the ILs.At the same time,some of methylation patterns have changed including demethylation (13.3-18.1%) and hypermethylation (4.4%-6.0%).(2)In the QTLs region related to cold tolerance,the frequency of DNA methylation in IL5335 and IL5423 was 13.3%-26.7%,which was much lower than that of recurrent parent.The frequency of DNA methylation patterns changes of the two ILs in the QTLs region was mainly for demethylation (33.3%-40.0%),which was higher than that of the average variation frequency of whole-genome.(3)Further analysis on 51 pairs of primers based on two retrotransposons (HoubaandOsr14) demonstrated that there was higher level of frequency of DNA methylation and lower frequency of methylation pattern changes.These results suggested that the extensive DNA methylation level and pattern changes have been happened during the interspecific hybridization and alien gene introgression,which would provide useful information for efficiently exploiting and using the interesting wild genes for rice enhancement.
Dongxiang wild rice;cold tolerance introgression lines;DNA methylation;McrBC enzyme
1000-4025(2015)04-0694-07
10.7606/j.issn.1000-4025.2015.04.0694
2014-12-27;修改稿收到日期:2015-01-25
国家自然科学基金(31260255);江西省教育厅科技计划项目(GJJ12184);江西省科技支撑计划(20122BBF60064);江西省青年科学家培养计划(20112BCB23007)
温秀芳(1989-),女,在读硕士研究生,主要从事植物遗传与分子生物学研究。E-mail:1025108610@qq.com
*通信作者:罗向东,博士,教授,主要从事植物遗传与分子生物学研究。E-mail:xdluolf@163.com
Q789
A
——东乡三中发展掠记

