水稻超短根突变体ssr1的遗传分析和基因定位
吴 晶,项显波,陆开形,朱世华,丁沃娜*
(1 宁波大学 海洋学院,浙江宁波 315211;2 宁波大学 科学技术学院,浙江宁波 315211)
水稻超短根突变体ssr1的遗传分析和基因定位
吴 晶1,项显波1,陆开形2,朱世华2,丁沃娜2*
(1 宁波大学 海洋学院,浙江宁波 315211;2 宁波大学 科学技术学院,浙江宁波 315211)
该研究从甲基磺酸乙酯(EMS)诱变的籼稻‘Kasalath’突变体库中筛选到1个根系超短的突变体,命名为ssr1(supershortroot1),8 d苗龄突变体的主根和不定根长度分别只有野生型的8.89%和2.29%,其不定根发生正常,但侧根的发生和伸长都受到严重抑制,且根毛也非常短。此外,ssr1植株整体矮小,株高不到野生型的一半。遗传分析结果表明,该突变性状由1对隐性核基因控制。利用图位克隆技术将SSR1基因定位在第9染色体的STS(sequence tagged site)分子标记9g7047K和9g7290K之间,物理距离约为243 kb,在定位区间共发现39个预测基因,经分析其中没有已克隆的根系发育基因。对SSR1的定位为进一步克隆该基因和阐明水稻根构型的分子机理奠定了基础。
水稻;超短根;遗传分析;基因定位
根是植物长期适应陆地条件而形成的一个重要器官,由主根、侧根、不定根和根毛组成,主根、侧根和不定根又分为根基、根体和根尖三大区域。根尖是每条根生长最旺盛的部分,由成熟区、伸长区、分生区和根冠四部分组成,根构型是指同一根系中所有的根在生长介质中的空间造型和分布[1]。种子植物的根系一般分为直根系和须根系,大部分双子叶植物的根系为直根系,大部分单子叶植物的根系为须根系。根的功能主要有锚定植株,吸收和运输水分、无机盐和有机物,合成及分泌一些次生代谢产物[2]。
水稻是中国最重要的经济粮食作物,其种植面积约占经济粮食总种植面积的40%,总产量约占经济粮食总产量的一半。随着对水稻根系研究的深入,根系对整个植株尤其对地上部的调节作用越来越受到关注,这些调节作用可能是由根系合成的一些次生代谢产物来完成。研究表明,根系对植株的调节作用非常复杂,主要包括:(1)调控气孔开关;(2)调节叶片光合作用;(3)控制叶片衰老进程等[3-5]。水稻根系形态对产量的影响已得到广泛的认同[6-7],对水稻根系构型的研究为作物改良和提高水稻产量提供了新思路。
近年来,国内外通过T-DNA插入、EMS诱变等方法筛选到了一系列与水稻根系发育相关的突变体,部分基因已被克隆,这些基因绝大多数属于核单基因隐性遗传,极少数属于核单基因半显性或核单基因显性遗传。它们在水稻12条染色体上都有分布,第1~4染色体上分布较多,每条染色体上有7~9个;第5~8、11、12染色体次之,有3~6个;第9、10染色体上则分布较少,只有1~2个,其中定位在第9染色体上的基因只有编码葡糖胺-6-磷酸乙酰转移酶的OsGNA1[8]。已克隆的这些基因功能各不相同,它们参与了不同的信号通路,表明根系发育的调控机制很复杂,目前人们对其认知还很有限。更多水稻根系构型突变体的发掘和基因功能的研究将有利于根系生长发育调控网络的构建,为水稻根系育种提供宝贵的遗传材料和基因资源。
超短根突变体ssr1是从EMS诱变的籼稻‘Kasalath’突变体库中筛选而来,该突变体苗期的地上部明显矮化,主根、不定根和根毛的伸长受到严重抑制,侧根数量也极少,只有2~3条。本研究对ssr1进行了初步的表型鉴定、遗传分析和基因定位,为进一步克隆该基因和研究其功能奠定了基础。
1 材料和方法
1.1 水稻材料及培养条件
水稻超短根突变体ssr1是由籼稻‘Kasalath’经EMS诱变后筛选得到。培养条件:光照12 h,湿度70%,白天30 ℃,夜晚22 ℃。水稻培养液的配方参照Yoshida等[9],培养8 d后观察表型,用Nikon D70s相机进行整个植株及根部的拍摄,主根的根茎结合部用体视镜(Leica MZ95,Germany)进行观察。样本统计数为20株。
1.2 遗传分析和定位群体的构建
将突变体ssr1与野生型‘Kasalath’杂交获得F1,观察F1表型并判断显隐性。F1自交获得F2,培养8 d后观察并统计F2植株长短根的分离比,并进行卡方检测。突变体ssr1与粳稻‘日本晴’杂交,F1自交获得的F2群体里分离出来的超短根植株用于基因定位。
1.3 水稻突变基因的分子定位
采用简易TPS法[10]从水稻叶片中提取亲本、F1以及F2分离群体突变株的DNA。
在已公布的简单重复序列(SSR)标记网站(http://www.gramene.org/)选取了138对均匀覆盖水稻12条染色体且具有‘Kasalath’与‘日本晴’多态性的SSR引物进行粗定位。根据浙江大学提供的‘Kasalath’与‘日本晴’全基因组SNP分析结果,设计STS引物。
PCR扩增体系:1 μL模板DNA、1 μL 10×PCR buffer、1.2 μL 25 mmol/L MgCl2、0.3 μL 2.5 mmol/L dNTP、0.3 μL 10 μmol/L上下游引物、0.1 μL 5 U/mLTaqDNA聚合酶、5.8 μL超纯水。PCR反应条件:94 ℃预变性4 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,34个循环;72 ℃延伸5min。PCR产物用于6%非变性聚丙烯酰胺凝胶190 V电泳2.5 h,银染后显色分析并拍照。
2 结果与分析
2.1 突变体的表型鉴定
8 d苗龄ssr1突变体的主根和不定根都很短,长度分别只有野生型的8.89%和2.29%,其不定根发生能力正常,但侧根的发生受到严重抑制,只在靠近根基部位有2~3根,且长度也是显著变短。在体视镜下观察发现ssr1的侧根比野生型粗,且根毛也非常短。此外,ssr1植株整体矮小,株高不到野生型的一半(图1和表1)。
2.2 遗传分析
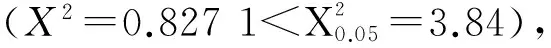
2.3 SSR1基因定位
选用ssr1ב日本晴’的F2群体作为定位群体,取30株F2群体中分离的超短根表型植株分别提取DNA,然后各取等量DNA混成一个突变体库。以两亲本、F1和突变体库的DNA做模板,对125对均匀覆盖水稻12条染色体的多态性SSR引物进行PCR扩增。发现SSR1与第9染色体上的RM105引物连锁。
根据浙江大学提供的‘Kasalath’与‘日本晴’全基因组SNP分析结果,在RM105引物附件设计了5对STS引物(表2),以ssr1ב日本晴’的F2群体中挑选的264个超短根植株的单株DNA做模板,PCR扩增RM105和新设计的STS引物,最终把SSR1基因定位在9g7047K和9g7290K引物之间,物理距离约为243 kb,该区间覆盖了3个BAC克隆:OSJNBa0044K01、P0592C05和OJ138_H04(图2)。根据网站(http://rice.plantbiology.msu.edu/cgi-bin/gbrowse/rice/)提供的水稻基因注释信息,在定位的区域中共发现39个预测基因:16个功能基因、7个反转录转座子、3个转座子、3个假定蛋白和10个未知表达蛋白,经分析这39个预测基因中没有已克隆的根系发育基因(表3),因此SSR1是一个调控水稻根系发生的新基因。
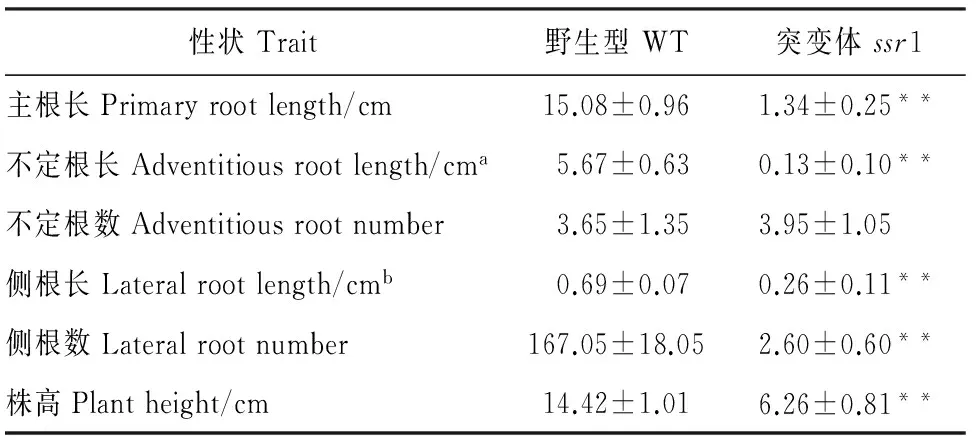
表1 8 d苗龄的野生型和突变体ssr1的表型参数
注:a.最长3根不定根长度的平均值;b.主根上最长10根侧根长度的平均值;**.表示突变体和野生型在0.01水平上差异显著。
Note:a.Average of three longest adventitious roots on each plant;b.Average of ten longest lateral roots on each primary root;**.Significant difference between WT andssr1 at the 0.01 probability level.
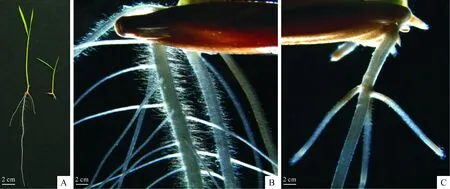
图1 8 d苗龄的野生型和突变体ssr1的表型鉴定A.野生型(左)和ssr1(右)的全株照;B、C.野生型(B)和ssr1(C)根茎结合部的体视镜观察
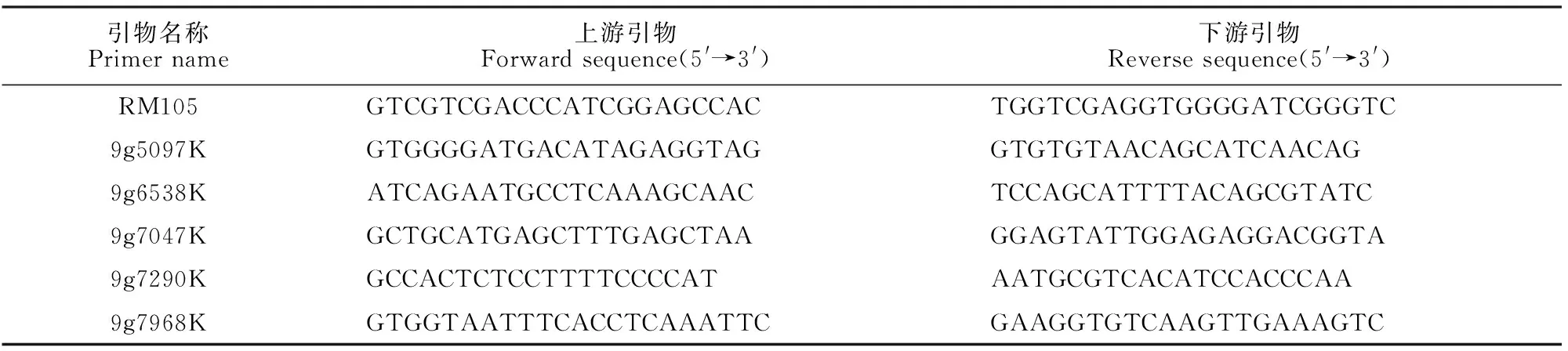
表2 具有多态性的SSR及STS标记序列
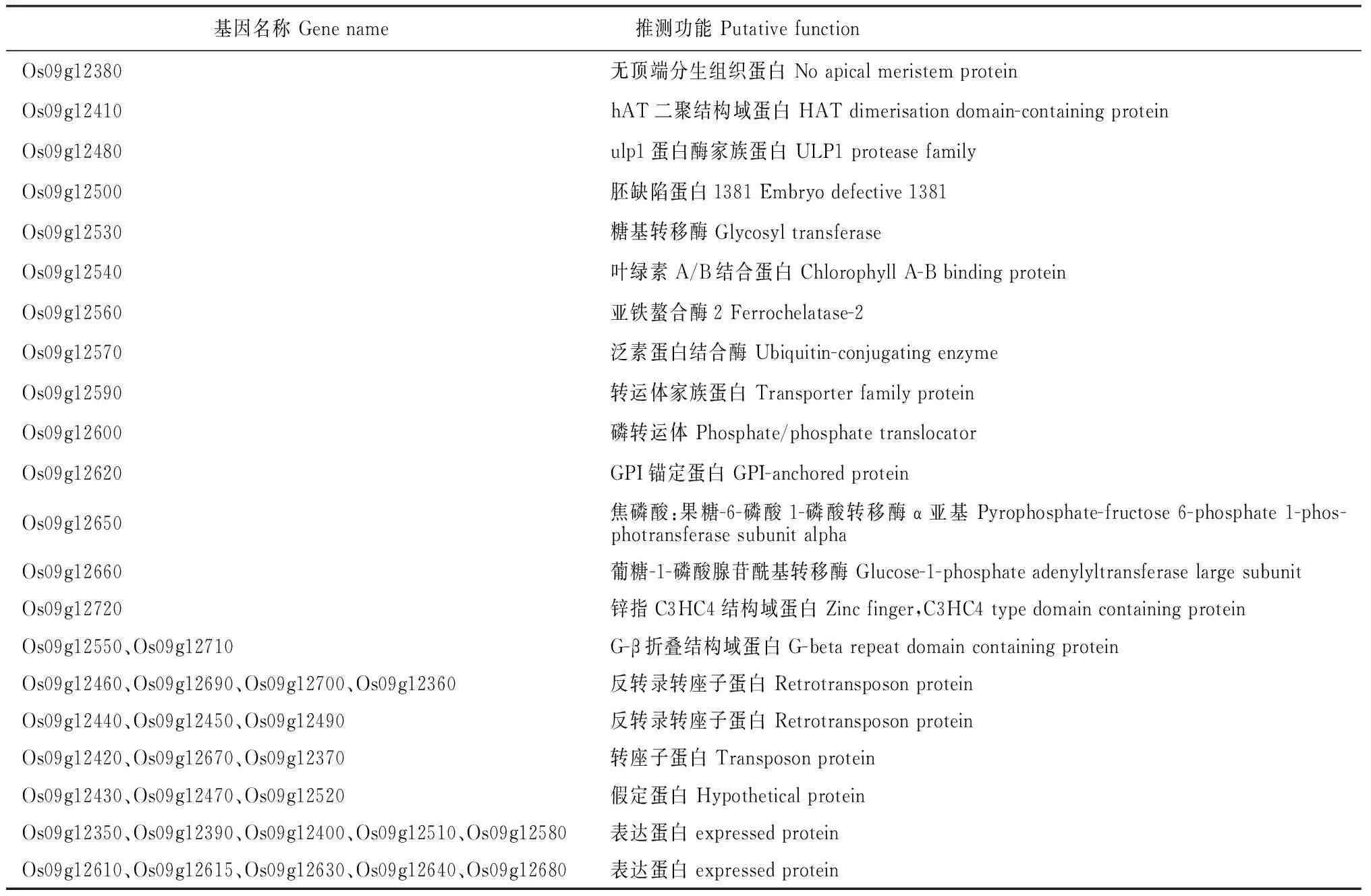
表3 定位区间内基因及其推测功能
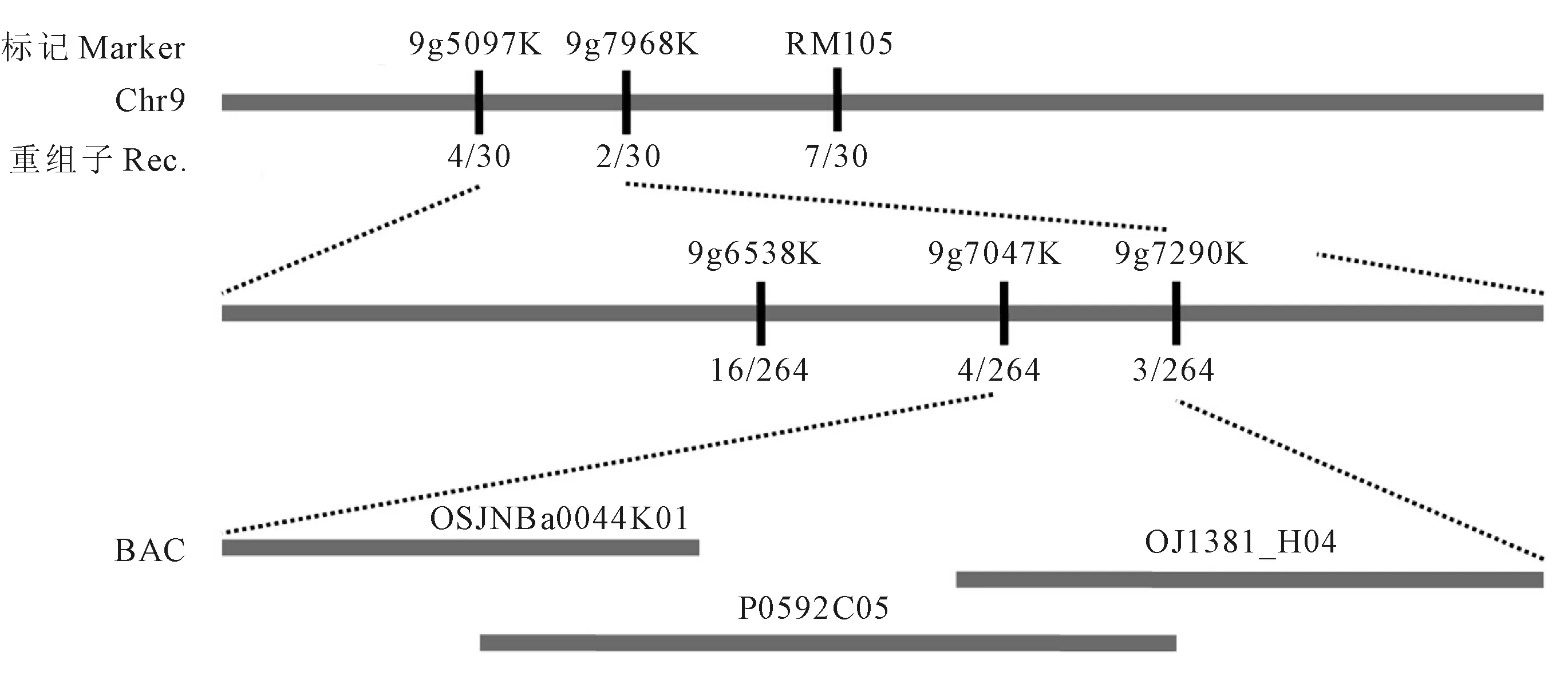
图2 SSR1基因定位
3 讨 论
对水稻根系发育相关基因的功能研究是解析其根系构型的重要途径之一。Scarpella等[11]发现RAL1基因突变导致水稻主根缺失,不定根与侧根数减少,且长度变短,分蘖数也显著减少。Jia等[12]克隆的OsCyt-inv1基因突变造成超短根表型,侧根变得粗壮且数目降低,地上部变短,育性下降。TDD1基因功能缺失后主根长度只有野生型的1/5,不定根长度变短且数量减少,叶片变窄,地上部矮化严重[13]。GLR3.1、OsGNA1和LRL6-1基因突变后突变体苗期的主根长度只有野生型的1/4左右,且地上部的生长受到不同程度的影响[8,14-15]。OsDGL1、OsGLU3、OsCKI1和OsARF12基因突变后的植株主根长度减短一半以上,且不定根与侧根的生长受到影响,地上部也相应变得矮小[16-19]。OsCAND1、OsWOX11和CLR5基因突变导致主根长度为野生型的2/3左右,不定根数也不同程度的减少,其中OsCAND1功能缺失的突变体存活不到2周[20-22]。OsMT2b、OsSPR1、OsTUA2、OsGatB和OsCOW功能缺失后突变体主根长度略微减短,不定根与侧根的长度或数量及育性都受影响,植株也表现矮化[23-27]。OsJMJ714编码的组蛋白去甲基化酶,OsAGAP编码的ARF-GTP 酶激活蛋白过表达的转基因植株均表现出主根长度略微减短,不定根、侧根长度减短且数量减少,地上部矮化[28-29]。
本研究从EMS诱变的籼稻‘Kasalath’突变体库中筛选到一个主根和不定根都超短的突变体ssr1,主根长度还不到野生型的1/10,侧根数量少且变短变粗,此外,根毛也很短。遗传分析结果表明该突变性状由1对隐性核基因控制。比较发现ssr1表型与OsCyt-inv1功能缺失所形成的水稻超短根表型很相似,不同的是Oscyt-inv1突变体的侧根数目比ssr1多一些,但是长度更短[12]。OsCyt-inv1也属于单基因核隐性遗传,定位在水稻第2染色体上,编码了一个碱性/中性蔗糖转化酶,细胞学实验发现短根原因主要是由于细胞伸长受阻所造成的,糖分析结果显示突变体中蔗糖不能降解成己糖,给突变体加入外源葡萄糖培养时,突变体的超短根表型最终恢复正常[12]。
本实验经图位克隆技术最终将SSR1定位在第9染色体的分子标记9g7047K和9g7290K之间,物理距离约为243 kb,目前在第9染色体上已克隆的根系基因只有OsGNA1[14],它位于第9染色体长臂的后段,而SSR1基因位于第9染色体长臂的前段,因此SSR1是一个新的根系发育相关基因。经分析定位区间包含39个预测基因,对SSR1的定位为进一步克隆该基因和阐明水稻根构型分子机理奠定了基础。
[1] LYNCH J P.Root architecture and plant productivity[J].PlantPhysiology,1995,109(1):7-13.
[2] 严小龙.根系生物学原理与应用[M].北京:科学出版社,2007:13-22.
[3] HUANG R F(黄荣峰),WANG X CH(王学臣).Advances in mechanism of stomatal movements[J].ChineseJournalofApplied&EnvironmentalBiology(应用与环境生物学报),1996,2(3):320-326(in Chinese).
[4] DAVIES W J,ZHANG J.Root signals and the regulation of growth and development of plants in drying soil[J].AnnualReviewPlantPhysiologyandPlantMolecularBiology,1991,42:55-76.
[5] DUAN L SH(段留生),HAN B W(韩碧文),HE ZH P(何钟佩).The effects of correlation between leaf and other organs on leaf senescence[J].ChineseBulletinofBotany(植物学通报),1998,15(1):43-49(in Chinese).
[6] COUDERT Y,PÉRIN C,COURTOIS B,etal.Genetic control of root development in rice,the model cereal[J].TrendsinPlantScience,2010,15(4):219-226.
[7] MORITA S,IWABUCHI A,YAMAZAKI K.Relationships between the growth direction of primary roots and yield in rice plants[J].JapaneseJournalofCropScience,1986,55(4):520-525.
[8] JIANG H W,WANG S M,DANG L,etal.A novel short-root gene encodes a glucosamine-6-phosphate acetyltransferase required for maintaining normal root cell shape in rice[J].PlantPhysiology,2005,138(1):232-242.
[9] YOSHIDA S,FORNO D A,COCK J H,etal.Laboratory manual for physiological studies of rice[M].Manila,Philippines:The International Rice Research Institute,1976:62.
[10] ZHANG X Q,ZOU J S,ZHU H T,etal.Genetic analysis and gene mapping of an early flowering and multi-ovary mutant in rice(OryzasativaL.)[J].Hereditas,2008,30(10):1 349-1 355.
[11] SCARPELLA E,RUEB S,ANNEMARIE H,etal.TheRADICLELESS1 gene is required for vascular pattern formation in rice[J].Development,2003,130(4):645-658.
[12] JIA L Q,ZHANG B T,MAO C Z,etal.OsCYT-INV1 for alkaline/neutral invertase is involved in root cell development and reproductivity in rice(OryzasativaL.)[J].Planta,2008,228(1):51-59.
[13] SAZUKA T,KAMIYA N,NISHIMURA T,etal.A rice tryptophan deficient dwarf mutant,tdd1,contains a reduced level of indole acetic acid and develops abnormal flowers and organless embryos[J].ThePlantJournal,2009,60(2):227-241.
[14] LI J,ZHU S H,SONG X W,etal.A rice glutamate receptor-like gene is critical for the division and survival of individual cells in the root apical meristem[J].PlantCell,2006,18(2):340-349.
[15] 钟威麒.水稻少侧根突变体lrl6-1的遗传分析与基因定位研究[D].杭州:浙江大学,2008.
[16] QIN C,LI Y Y,GAN J,etal.OsDGL1,a homolog of an oligosaccharyltransferase complex subunit,is involved in N-glycosylation and root development in rice[J].PlantCellPhysiology,2013,54(1):129-137.
[17] ZHANG J W,XU L,WU Y R,etal.OsGLU3,a putative membrane-bound endo-1,4-beta-glucanase,is required for root cell elongation and division in rice(OryzasativaL.)[J].MolecularPlant,2012,5(1):176-186.
[18] LIU W,XU Z H,LUO D,etal.Roles ofOsCKI1,a rice casein kinase I,in root development and plant hormone sensitivity[J].ThePlantJournal,2003,36(2):189-202.
[19] QI Y,WANG S,SHEN C,etal.OsARF12,a transcription activator on auxin response gene,regulates root elongation and affects iron accumulation in rice(Oryzasativa)[J].NewPhytologist,2012,193(1):109-120.
[20] WANG X F,HE F F,MA X X,etal.OsCANDl is required for crown root emergence in rice[J].MolecularPlant,2011,4(2):289-299.
[21] ZHAO Y,HU Y F,DAI M Q,etal.The WUSCHEL-related homeobox geneWOX11 is required to activate shoot-borne crown root dvelopment in rice[J].PlantCell,2009,21(3):736-748.
[22] KITOMI Y,ITO H,HOBO T,etal.The auxin responsive AP2/ERF transcription factorCROWNROOTLESS5 is involved in crown root initiation in rice through the induction ofOsRRl,a type-A response regulator of cytokinin signaling[J].ThePlantJournal,2011,67(3):472-484.
[23] YUAN J,CHEN D,REN Y J,etal.Characteristic and expression analysis of a metallothionein gene,OsMT2b,down-regulated by cytokinin suggests functions in root development and seed embryo germination of rice[J].PlantPhysiology,2008,146(4):1 637-1 650.
[24] JIA L Q,WU Z C,HAO X,etal.Identification of a novel mitochondrial protein,short postembryonic roots 1(SPR1),involved in root development and iron homeostasis inOryzasativa[J].NewPhytologist,2011,189(3):843-855.
[25] 何芬芳.水稻OsTUA2基因克隆与功能研究[D].杭州:浙江大学,2008.
[26] 张欢欢.水稻根系发育调控基因OsGatB的克隆与初步功能分析[D].杭州:浙江大学,2011.
[27] WOO Y M,PARK H J,SUUDI M,etal.Constitutively wilted 1,a member of the rice YUCCA gene family,is required for maintaining water homeostasis and an appropriate root to shoot ratio[J].PlantMolecularBiology,2007,65(1-2):125-136.
[28] 秦 巧.OsJMJ714调控水稻根系发育与生长素含量的功能研究[D].北京:中国农业科学院,2013.
[29] ZHUANG X L,JIANG J F,LI J H,etal.Over-expression of OsAGAP,an ARF-GAP,interferes with auxin influx,vesicle trafficking and root development[J].ThePlantJournal,2006,48(4):581-591.
(编辑:宋亚珍)
Genetic Analysis and Gene Mapping of a SuperShort Root Mutantssr1 in Rice (OryzasativaL.)
WU Jing1,XIANG Xianbo1,LU Kaixing2,ZHU Shihua2,DING Wona2*
(1 School of Marine Science,Ningbo University,Ningbo,Zhejiang 315211,China;2 College of Science and Technology,Ningbo University,Ningbo,Zhejiang 315211,China)
In this study,a mutant with super short primary and adventitious roots,less lateral roots and short root hairs was isolated from an EMS (ethyl methane sulfonate)-generated rice mutant library in the Kasalath background,designated asssr1 (supershortroot1).At the 8 d old stage,the length of primary and adventitious roots ofssr1 was only 8.89% and 2.29% of the wild type (WT),respectively.The initiation of adventitious roots ofssr1 was similar as WT,while the initiation and elongation of lateral roots were severely impaired and root hairs were also much shorter than that of WT.Moreover,ssr1 showed dwarf phenotype with plant height less than half of WT.Genetic analysis indicated that the mutant phenotype was controlled by a single recessive nuclear gene.Map-based cloning analysis locatedSSR1 to a 243 kb region between STS(sequence tagged site)markers 9g7047K and 9g7290K on chromosome 9.The region contains 39 putative genes with none reported to be related to root development of rice.This result will be helpful for the cloning ofSSR1 and further characterization of molecular genetic mechanisms underlying root architecture in rice.
rice (OryzasativaL.);super short root;genetic analysis;gene mapping
1000-4025(2015)04-0701-06
10.7606/j.issn.1000-4025.2015.04.0701
2014-11-27;修改稿收到日期:2015-01-28
国家自然科学基金(31371595);宁波市自然科学基金(2014A610198);宁波大学胡岚优秀博士基金
吴 晶(1988-),男,在读硕士研究生,主要从事水稻根系基因功能研究。E-mail:wujing999@163.com
*通信作者:丁沃娜,博士,副教授,主要从事水稻分子生物学研究。E-mail:dwn@zju.edu.cn
Q343.1+7
A

