鄱阳湖湖泊细菌群落组成及结构
——以松门山为例
寇文伯,黄正云,张 杰,刘倩纯,刘芳鹏,刘以珍,吴 兰,*
1 江西省分子生物学与基因工程重点实验室,南昌大学,南昌 330031 2 南昌大学生命科学学院,南昌 330031
鄱阳湖湖泊细菌群落组成及结构
——以松门山为例
寇文伯1,2,黄正云2,张 杰1,2,刘倩纯1,2,刘芳鹏1,2,刘以珍1,2,吴 兰1,2,*
1 江西省分子生物学与基因工程重点实验室,南昌大学,南昌 330031 2 南昌大学生命科学学院,南昌 330031
于2011年5月在鄱阳湖——松门山湖区采集底泥与表层水样,分别提取了表层水体浮游和底泥微生物基因组DNA,利用454高通量测序技术对细菌的16S rRNA基因进行了序列测定,分析了湖泊底泥细菌、水体浮游细菌群落结构特征。结果显示:底泥细菌OTUs(Operational Taxonomic Units)为1454,表层水体浮游细菌OTUs为269;底泥细菌群落比表层水体更加多样化,底泥细菌的物种数大大多于表层水体。同时,底泥细菌群落与表层浮游细菌群落结构存在显著差异。物种分类显示鄱阳湖底泥细菌种类隶属于20门,228属,其中优势种群为δ-变形菌纲(Deltaproteobacteria)、β-变形菌纲(Betaproteobacteria)和疣微菌门(Verrucomicrobia);表层水体浮游细菌隶属于13门116属,优势种群为β-变形菌纲、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)。结果进一步揭示,无论是浮游细菌群落还是沉积物细菌群落,优势细菌种群的基因型多样性更高。
细菌多样性;细菌群落组成;高通量测序;鄱阳湖
湖泊生态系统在全球及区域地球化学循环中都发挥着关键的作用,而细菌作为有机化合物的主要分解者, 通过分解有机污染物为小分子产物, 来影响和调节水体环境, 实现水质的净化[1],并在一定程度上反映水体污染特征及其生态功能演变,因此,细菌在湖泊生态系统中作用的研究日益受到重视。研究细菌群落结构和多样性,对了解群落结构间的动态变化及其生态功能、阐明群落与生境、功能间的具体联系及维护湖泊生态环境具有重要意义[2]。
鄱阳湖是我国最大的淡水湖泊,在涵养水源、调蓄洪水、调节气候、降解污染保护土壤、营养循环等方面具有重要生态功能[2],是目前少数未受到严重污染的淡水湖泊之一,也是具有国际意义的水禽繁殖与迁徙栖息地,具有维护生物多样性等特殊功能。目前针对鄱阳湖的研究主要集中在重金属含量、水文特征、水质分析及植被的生物多样性等方面[3-5],对湖泊水体和微生物的报道相对较少,仅见本实验室前期相关工作[6]。
此次工作采用454焦磷酸测序技术,研究了鄱阳湖松门山湖区底泥沉积物与表层水体中细菌的物种组成、群落结构及多样性。为进一步探讨鄱阳湖底泥与表层水体水质演变特征提供理论依据。
1 材料与方法
1.1 样品采集及处理
文章所用数据来自于2011年5月对鄱阳湖的调查,采样点为鄱阳湖—松门山处(29°12′26.9″N,116°11′28.0″E)(图1)。样品采集分别利用抓斗式采泥器和2 L有机玻璃采水器进行,采集底泥沉积物样品和湖泊表层0.5 m深水样。底泥和湖水样品均采用重复三点法取样,即湖中央为一点,从中央向两侧延伸200m至500m各1点。底泥与水样分别保存在聚乙烯瓶(70%酒精消毒处理)与聚乙烯薄膜自封袋中,低温运回实验室。
1.2 湖泊细菌群落结构测定
水样用滤膜过滤(0.22 μm,Milipore)后采用溶菌酶-SDS-冻融相结合的方法[7]进行微生物基因组DNA的提取,底泥微生物基因组DNA提取则直接利用土壤DNA提取试剂盒(Power SoilTMDNA Isolation Kit, MOBIO, USA)进行,将样品的3个重复样分别提取基因组DNA后再混合,以降低提取过程中的操作误差给实验结果带来的影响。细菌16S rRNA片段采用454 GS Junior测序仪进行测序(美国肯特州立大学完成),测序引物选用16S rRNA通用引物530F(5′-CCA TCT CAT CCC TGC GTG TCT CCG ACT CAG-3′)和1110R(5′-CCT ATC CCC TGT GTG CCT TGG CAG TCT CAG GGG TTN CGN TCG TTG-3′)(引物含有454接头和标签序列),PCR扩增区域为V3-V6区,扩增片段电泳检测后进行454测序。
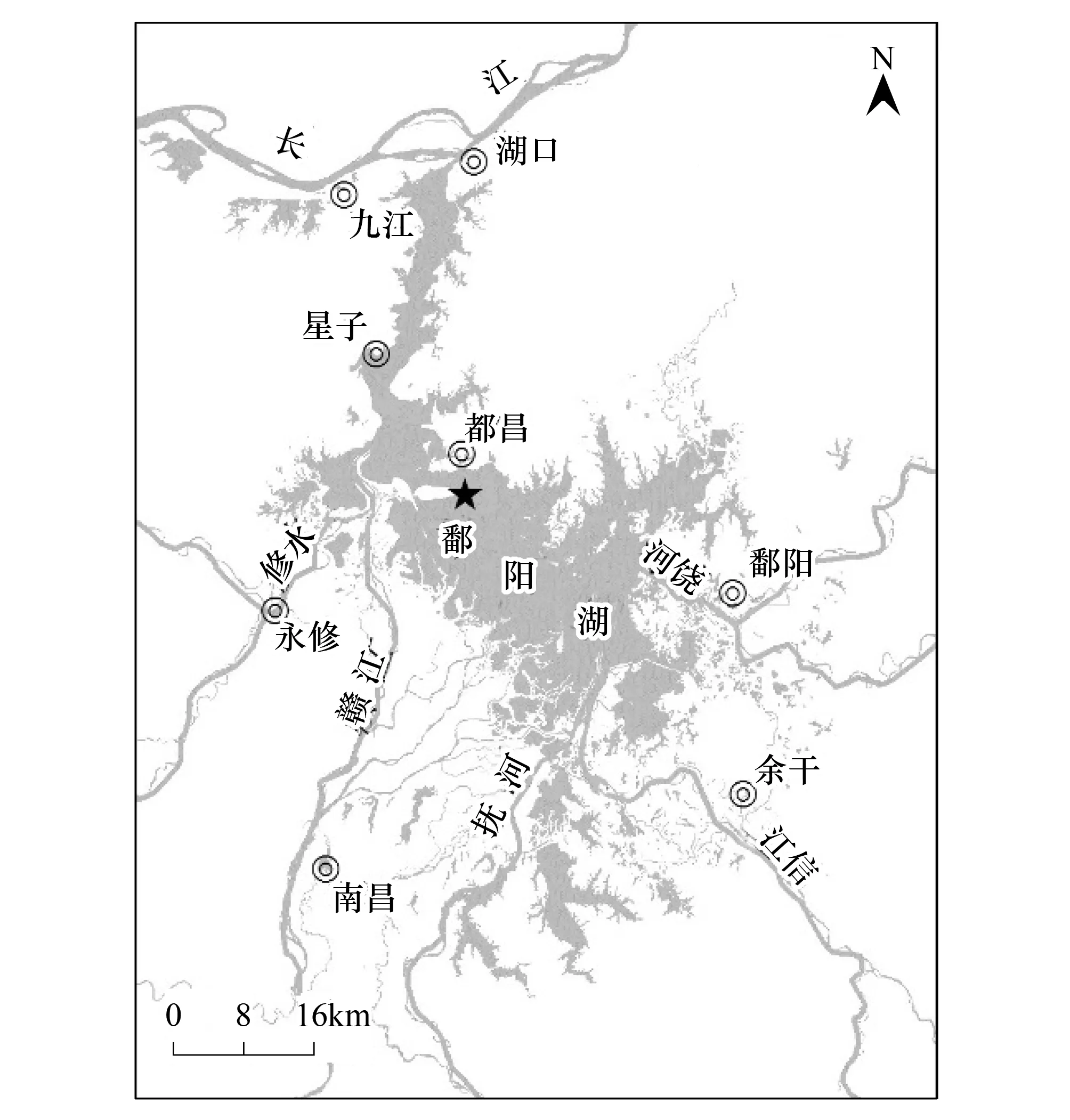
图1 鄱阳湖采样位置Fig.1 Sampling station in the Poyang Lake
1.3 序列分析
序列使用Mothur v1.33.1[8]软件包进行检查并去除嵌合体,筛选有效序列。将有效序列相似性≥97%的序列归为同一分类操作单元(OTU),绘制稀疏曲线,计算Chao1物种丰富度指数,覆盖度,在底泥与浮游细菌序列中随机抽取同等数目的序列后,计算shannon指数,invsimpson指数用以评价细菌群落的多样性及丰富度。对OTUs进行libshuff检验以检验水体与底泥细菌群落是否具有相同的结构。RDP v9[9]用于16S rRNA序列的分类,以高阶细菌分类法80%的置信度作为阈值。根据得到的菌种分类信息绘制水体与底泥菌种间的韦恩图。上述分析过程及图表绘制均由Mothur v1.33.1及Microsoft Excel 2013完成。对不同菌种的相对丰度与不同菌种OTUs的数目使用NCSS 2007[10]进行相关性分析。
1.4 序列索取号
将鄱阳湖代表性细菌16S rRNA序列提交至NCBI数据库,水体细菌序列号为KF008723-KF009052,底泥序列号为SRR1040439。
2 结果与分析
2.1 序列统计和细菌多样性
为了解鄱阳湖表层水体和底泥细菌群落结构的差异性,将样品微生物DNA分别进行了454焦磷酸测序,底泥细菌和表层浮游细菌得到的16S rRNA基因片段总数分别为3625/ 2126(表1),底泥细菌与表层浮游细菌中OTUs总数为1648。

表1 鄱阳湖底泥和表层水体细菌OTUs多样性分析Table 1 Analysis of OTUs diversity of bacterial communities from sediment and surface-water
SS:泥样品surface sediment;SW:表层水样surface water

图2 底泥与表层水体细菌稀疏曲线分析 Fig.2 Rarefaction curve analysis of OTUs of the bacterial from sediment and water column
覆盖度和稀疏曲线用来评估所测序列库容中环境微生物的种类和数量,结果见图2和表1。底泥细菌和表层浮游细菌的覆盖度分别为74.9%和93.8%(表1)。稀疏曲线结果显示:底泥细菌库容近似一条直线,没有随序列数量增加而达到一个平台;而表层浮游细菌稀疏曲线则趋于平缓(图2)。结果表明,底泥细菌与表层浮游细菌所测序列库容都可以较好的反映细菌群落的种类与数量,其中浮游细菌库容基本涵盖了湖泊水体中所有细菌种群,来自底泥的样品没有获得细菌群落的全部多样性。结果进一步提示,鄱阳湖底泥中蕴藏着丰富的微生物资源。
细菌群落结构的整体差异通过libshuff检验进行,结果表明底泥细菌和表层浮游细菌群落结构是显著不同的(P<0.025),底泥中OTU的数目明显高于表层水体中OTU数目。在底泥与浮游细菌数据中随机抽取同等数量的序列后,进一步通过多样性指数评估细菌群落多样性,在OTU水平,底泥与表层水体样品的Shannon指数和Invsimpson指数分别为6.51/4.18、484.41/20.66(表1),结果表明,底泥的细菌群落多样性显著高于浮游细菌,该结果进一步支持鄱阳湖底泥群落比浮游细菌群落更加多样化的结论。丰富度指数Chao1表明,底泥样品中的物种数要远多于表层水体,Chao1值预测底泥中OTU数目约为3177,为浮游细菌中的6倍。另外基于Chao1物种丰富度指数,底泥细菌中45%的OTUs被发现,浮游细菌中58%的OTUs被发现。
2.2 鄱阳湖细菌群落的物种组成
RDP v9对1648个OTUs的分类结果表明,鄱阳湖总数为5751的序列中至少有20个不同的细菌门,底泥细菌与浮游细菌门相对丰度如图3所示。其中鄱阳湖底泥细菌隶属于20个门,变形菌门(Proteobacteria, 47%)、疣微菌门(Verrucomicrobia, 13%)比例最大,其次为酸杆菌门(Acidobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)和厚壁菌门(Firmicutes),共占18%。浮游细菌隶属于13个门,变形菌门(51%)、拟杆菌门(22%)、放线菌门(16%)占主导地位,其次为疣微菌(5%),其他一些观察到的门类比例较低(6%),包括浮霉菌门(Planctomycetes, <0.6%)、芽单胞菌门(Gemmatimonadete, <0.4%)等。

图3 底泥与表层水体样品在门水平的细菌组成Fig.3 Bacterial composition at phylum level of sediment sample and water column sampleGammaproteobacteria γ-变形菌纲;Deltaproteobacteria δ-变形菌纲;Betaproteobacteria β-变形菌纲;Alphaproteobacteria α-变形菌纲;Acidobacteria 酸杆菌门;Actinobacteria 放线菌门;Armatimonadetes 装甲菌门;Bacteroidetes 拟杆菌门;Chlamydiae 衣原体门;Chlorobi 绿菌门;Chloroflexi 绿弯菌门;Deinococcus-Thermus 异常球菌-栖热菌门;Elusimicrobia 迷踪菌门;Fusobacteria 梭杆菌门;Gemmatimonadetes 芽单胞菌门;Nitrospira 硝化螺旋菌门;Planctomycetes 浮霉菌门;Spirochaetes 螺旋体门;Verrucomicrobia 疣微菌门;BRC1、TM7、WS3 未定菌;unclassified 未分类;other 其他细菌
与浮游细菌相比,底泥细菌群落的特点是物种更加多元。底泥细菌除包括了浮游细菌所具有的全部门类外,另有7个独有门,分别为绿菌门(Chlorobi)、绿弯菌门(Chloroflexi)、恐球菌-栖热菌门(Deinococcus-Thermus)、Elusimicrobia、BRC1、TM7、WS3。另外,底泥细菌群落涵盖的未知类群(18%)远大于浮游细菌群落的4%。值得一提的是,变形菌尽管都是底泥和浮游细菌中的优势种群,但它们在组成细菌群落中β-变形菌纲(14%)和δ-变形菌纲(20%)相对丰度比较大,细菌群落中则是β-变形菌纲(36%)占绝对优势。从细菌门的角度看,鄱阳湖松门山湖区底泥细菌与浮游细菌群落之间的种类组成差异显著。
为了解细菌在底泥与浮游细菌中的物种组成,在属水平进一步进行了细菌群落结构分析。在属水平,全部细菌序列至少分为267个不同的属,其中77个属为鄱阳湖的底泥与表层水体浮游细菌共有。151个属为底泥细菌独有,39个属为浮游细菌独有(图4)。

图4 底泥与表层水体样品在属水平的韦恩图 Fig.4 Venn diagram at genus level of sediment sample and water column sample
底泥细菌隶属于228个属,丰度≥2%的属为疣微菌门的Subdivision3generaincertaesedis(9.2%)、δ-变形菌纲的地杆菌属(Geobacter, 3.7%)、厌氧粘细菌属(Anaeromyxobacter,2.5%)(数据未列出),其中厌氧粘细菌属属于该地底泥独有属,浮游细菌未见。结果提示,该三大类属是鄱阳湖底泥细菌中的常见属。浮游细菌隶属于116个属,丰度≥2%的属为β-变形菌纲的食酸菌属(Acidovorax, 8.2%)、Polynucleobacter(3.8%)、氢噬胞菌属(Hydrogenophaga,3.7%);γ-变形菌纲的不动杆菌属(Acinetobacter,3.3%);鞘脂杆菌纲(Sphingobacteria)的Arcicella(2.5%)、Sediminibacterium(2.1%);放线菌纲的Ilumatobacter(2.3%)(数据未列出)。其中包含了食酸菌属和氢噬胞菌属的丛毛单胞科(Comamonadaceae)在鄱阳湖浮游细菌中占绝对优势(24.4%),且氢噬胞菌属、不动杆菌属、Polynucleobacter和Arcicella均为鄱阳湖浮游细菌独有,底泥细菌未见。
2.3 细菌门的相对丰富度与基因型多样性的关系
为了探讨为什么在一个特定环境中细菌组成的丰富度不同,本研究对细菌门丰度和基因型多样性之间的相关性进行了研究。结果见图5。结果表明:无论浮游还是沉积物细菌群落,优势细菌种群的基因型多样性更高,在底泥和浮游细菌中细菌门(phylum)的相对丰度与门内OTUs的数量均有很高的相关性(底泥P<0.01,r=0.887;表层水体P<0.01,r=0.981)。结果提示:不论是在鄱阳湖底泥还是水体中,相对丰度较大的细菌门比相对罕见的细菌门有更多的OTUs。并且底泥细菌与浮游细菌群落中相对丰富的细菌门的OTUs数目比较,底泥细菌多于浮游细菌。

图5 细菌门的相对丰度与OTUs数目的相关性分析Fig.5 Relative abundance of phyla as a function of the number of unique OTUs
3 讨论
鄱阳湖是浅水淡水湖泊,其底泥中最丰富的细菌为变形菌(47%),该结果与Miskin与Tamaki等人通过对两个浅水富营养化Priest Pot湖与Kasumigaura湖底泥细菌分析结果[11-12]相一致。变形菌为Priest Pot湖与Kasumigaura湖底泥细菌的最优势种群,且Kasumigaura湖变形菌中的主要微生物是δ-、β-、γ-,都与此次调查得到的结论相符合。本次对鄱阳湖松门山湖区的研究中,观察到浮游细菌中最丰富的细菌门类包括变形菌门(β-、α-、γ-)、拟杆菌门和放线菌门,该结果与此前本实验室在2006年10月对鄱阳湖老爷庙水域的调查工作[6]中得出的老爷庙水域细菌分属于六大主要簇群(变形菌、拟杆菌、放线菌等)的结果基本一致。结果提示,鄱阳湖不同区域水体细菌组成结构地域差异不明显,可能是鄱阳湖属于通江湖泊、水体流速较大所致。
就湖泊底泥和浮游细菌组成差异而言,目前研究较少。本文结果表明,鄱阳湖底泥细菌群落相比于浮游细菌群落具有更高的丰富度,并且在群落结构、种类组成上两者都有较大差异。本结论证实了此前Beier等人的研究成果[13]。Beier等人通过研究Fulda河流发现细菌群落在底泥与水体样品间显著不同。此外,Hu等人对九龙江流域河流生态系统的调查也发现了底栖细菌群落的丰富度与均匀度要比浮游细菌群落更高,可能的原因是湖泊底泥环境比水体更为复杂,且底泥和水体环境差异性显著所致[14]。另外,尽管变形菌门的细菌在鄱阳湖底泥细菌和浮游细菌中的比例都很高,但是从细菌属的角度来看,底泥细菌和浮游细菌中的优势种群并不相同。虽然细菌77个属在底泥细菌和浮游细菌中均被发现,但是大部分种类的细菌在底泥、浮游细菌间的分布并不均匀,如果在两个环境间比较这些共有的细菌属,会发现大部分细菌属在一个环境中的相对丰度要远大于另一个环境,即鄱阳湖底泥表层浮游细菌在细菌“属”层面没有明显的重叠。结果提示,基于“属”的水平,相对于浮游细菌,鄱阳湖松门山湖区底泥的细菌种类可能更加丰富多样,并且没有特别明显的优势种群。
地杆菌与厌氧粘细菌在鄱阳湖底泥细菌中是丰度相对较高的细菌属,地杆菌和厌氧粘细菌是两类隶属变形杆菌δ亚纲的微生物 Fe(Ⅲ)还原细菌,广泛分布于厌氧沉积环境,并且能够参于变价重金属的氧化还原、放射性金属元素稳定化及有机卤化物的生物代谢过程。Cummings等人对金属污染较严重(沉积了高浓度的Pb和Fe)的Coeur d′Alene淡水湖泊底泥研究中也发现地杆菌在该湖泊底泥中种类及数量都十分丰富[15],本研究结果与之类似,结果暗示Fe(Ⅲ)还原细菌可能会定殖在高金属浓度的环境中,提示鄱阳湖底泥可能受到一定程度的重金属污染。
细菌门的相对丰度与它们的多样性有关,按97%的相似性定义OTU是因为细菌群落并不只含有少数高丰度的基因型,而是含有大量独特的基因型。相对丰富度高的细菌门比相对丰富度低的细菌门有更多的OTU,说明环境中不同细菌群体的丰度随着该群体内不同基因型的数目而改变,即环境中优势细菌种群的基因型更加多样化。类似的结果也在Møller等人的研究[16]中被发现,进一步验证了Cohan等人提出的假说[17]:一个细菌谱系在生境中的生态成功不是因为其中少数竞争力非常强的物种,而是取决于它的多样性,在细菌“种”内存在着不同的生态型,造成生态型不同的原因则是环境的变化,不同生态型之间的基因交换使物种的竞争力得到增强。这个假说可以解释为什么有些细菌可以广泛的分布在各种环境中,比如淡水环境中的β-变形菌等。而高丰度的细菌种类是否也在地化循环及营养动态过程中扮演着更为重要的角色,则需要进一步的研究。
4 结论
(1)鄱阳湖松门山湖区细菌群落多样性在底泥细菌与浮游细菌之间有较大差异,底泥细菌群落更加多样化,物种更加丰富。且底泥与浮游细菌群落结构并不相同。
(2)松门山湖区底泥细菌群落主要由δ-变形菌、β-变形菌和疣微菌组成;浮游细菌群落则主要由β-变形菌、拟杆菌和放线菌组成。
(3)鄱阳湖松门山湖区底泥、浮游细菌群落分别与来自其他淡水湖泊底泥、水体环境中的细菌群落相似。其中会定殖在高金属浓度的环境中,具有变价重金属的氧化还原等功能的Fe(Ⅲ)还原菌地杆菌属细菌在底泥中相对丰度较大。
(4)底泥与浮游细菌群落在细菌门的相对丰度与其中OTUs的个数之间有显著相关性,即优势细菌种群的基因型多样性更高。
[1] 任丽娟, 何聃, 邢鹏, 王毓菁, 吴庆龙. 湖泊水体细菌多样性及其生态功能研究进展. 生物多样性, 2013, 21(4):421- 432.
[2] 崔丽娟. 鄱阳湖湿地生态系统服务功能价值评估研究. 生态学杂志, 2004, 23(4):47- 51.
[3] 张杰, 陈熙, 刘倩纯,吴兰. 鄱阳湖主要入湖口重金属的分布及潜在风险评价. 长江流域资源与环境, 2014, 23(1):95- 100.
[4] 陈晓玲, 张媛, 张琍, 陈莉琼, 陆建忠. 丰水期鄱阳湖水体中氮、 磷含量分布特征. 湖泊科学, 2013, 25(5):643- 648.
[5] 叶春, 赵晓松, 吴桂平, 王晓龙, 刘元波. 鄱阳湖自然保护区植被生物量时空变化及水位影响. 湖泊科学, 2013, 25(5):707- 714.
[6] 吴兰, 葛刚, 龚世杰, 万金保. 鄱阳湖老爷庙水域细菌群落组成分析. 长江流域资源与环境, 2011, 20(8):963- 969.
[7] Zhou J, Bruns M A, Tiedje J M. DNA recovery from soils of diverse composition. Applied and Environmental Microbiology, 1996, 62(2):316- 322.
[8] Schloss P D, Westcott S L, Ryabin T, Hall J R, Hartmann M, Hollister E B, Lesniewski R A, Oakley B B, Parks D H, Robinson C J, Sahl J W, Stres B, Thallinger G G, Van Horn D J, Weber C F. Introducing mothur:open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and Environmental Microbiology, 2009, 75(23):7537- 7541.
[9] Cole J R, Wang Q, Cardenas E, Fish J, Chai B, Farris R J, Kulam-Syed-Mohideen A S, McGarrell D M, Marsh T, Garrity G M, Tiedje J M. The Ribosomal Database Project:improved alignments and new tools for rRNA analysis. Nucleic Acids Research, 2009, 37(S1):D141- D145.
[10] Hintze J L. NCSS 2007 User′s Guide. Kaysville, Utah:NCSS, 2007.
[11] Miskin I P, Farrimond P, Head I M. Identification of novel bacterial lineages as active members of microbial populations in a freshwater sediment using a rapid RNA extraction procedure and RT-PCR. Microbiology, 1999, 145(8):1977- 1987.
[12] Tamaki H, Sekiguchi Y, Hanada S, Nakamura K, Nomura N, Matsumura M, Kamagata Y. Comparative analysis of bacterial diversity in freshwater sediment of a shallow eutrophic lake by molecular and improved cultivation-based techniques. Applied and Environmental Microbiology, 2005, 71(4):2162- 2169.
[13] Beier S, Witzel K P, Marxsen J. Bacterial community composition in Central European running waters examined by temperature gradient gel electrophoresis and sequence analysis of 16S rRNA genes. Applied and Environmental Microbiology, 2008, 74(1):188- 199.
[14] Hu A Y, Yang X Y, Chen N W, Hou L Y, Ma Y, Yu C P. Response of bacterial communities to environmental changes in a mesoscale subtropical watershed, Southeast China. Science of The Total Environment, 2014, 472:746- 756.
[15] Cummings D E, Snoeyenbos-West O L, Newby D T, Niggemyer A M, Lovley D R, Achenbach L A, Rosenzweig R F. Diversity of Geobacteraceae species inhabiting metal-polluted freshwater lake sediments ascertained by 16S rDNA analyses. Microbial Ecology, 2003, 46(2):257- 269.
[16] Møller A K, Søborg D A, Al-Soud W A, Sørensen S J, Kroer N. Bacterial community structure in High-Arctic snow and freshwater as revealed by pyrosequencing of 16S rRNA genes and cultivation. Polar Research, 2013, 32(1):1- 11.
[17] Cohan F M. What are bacterial species? Annual Review of Microbiology, 2002, 56(1):457- 487.
Bacterial community structure and composition in Lake Poyang:a case study in the Songmenshan Region, China
KOU Wenbo1,2, HUANG Zhengyun2, ZHANG Jie1,2, LIU Qianchun1,2, LIU Fangpeng1,2, LIU Yizhen1,2, WU Lan1,2,*
1KeyLaboratoryofMolecularBiologyandGeneticEngineeringinJiangxiProvince,NanchangUniversity,Nanchang330031,China2SchoolofLifeSciences,NanchangUniversity,Nanchang330031,China
Lacustrine ecosystems play a critical role in regional and global biochemical cycles. Bacteria are dominant contributors to biogeochemical cycles, participating in most of the decomposition of organic material in freshwater, and are critical for the maintenance of the biodiversity and stability of lacustrine ecosystems. In addition, bacterial biomass is an important component of natural lacustrine systems. Investigating the correlations between environmental variations and microbial communities is expected to reveal how microbes tolerate different types of environmental change and to increase our understanding about microbial ecology and evolution. Such information would also enhance our understanding about microbial adaptations to different environments and their function in these environments. Lake Poyang is the largest lake in China. As one of the few lakes that are still freely connected with the river and not heavily polluted, this lake is important for the maintenance of the unique biota of the Yangtze floodplain ecosystem. However, in contrast to the contamination and the water quality of Lake Poyang, little is known about the bacteria and their ecosystem functions in Lake Poyang. In this study, genomic DNA of the microbial community was extracted from sediment and water collected in May 2011 from Songmenshan Region, Lake Poyang, China. The benthic and planktonic bacterial community structures were examined by 454 pyrosequencing of the v4-v6 16S rRNA gene region. We used OTU-based analyses to identify changes in bacterial diversity and community structure. Chao1 estimator, inverse Simpson index, Shannon index, coverage, and the rarefaction curve were used to describe the richness and diversity of separate samples collected from different environments. The libshuff test was used to describe whether benthic and planktonic bacterial communities have the same structure. In addition, correlation analysis between the abundance of bacterial phyla and their diversity in each environment was performed to reveal whether they were related to each other. Overall, the dataset comprised 5751 sequences that were affiliated toBacteria; of these, 1454 and 269 OTUs were obtained from the sediment and water column, respectively, indicating that benthic bacterial communities have higher bacterial diversity and richness. The results also revealed different bacterial community structures between the sediment and water column. In benthic bacterial communities, 228 bacterial genera belonging to 20phyla, dominated byDeltaproteobacteria,Betaproteobacteria, andVerrucomicrobia, were found. A further 116 genera were obtained for bacterioplankton, which belonged to 13 phyla, withBetaproteobacteria,Bacteroidetes, andActinobacteriabeing dominant. Significant variations in the relative abundance of bacterial phyla and proteobacterial classes were observed between environments. At the genus level, significant differences were also found, the dominant genera in the benthic bacterial community wereSubdivision3generaincertaesedis,GeobacterandAnaeromyxobacteretc. In the bacterioplankton community, the dominant genera wereAcidovorax,Polynucleobacter,Hydrogenophaga,Acinetobacter, andArcicellaetc. Of note,Comamonadaceae, which includedAcidovoraxandHydrogenophagaetc, was consistently dominant in the bacterioplankton community. In addition,Anaeromyxobacterwas only detected in sediment, whilePolynucleobacter,Hydrogenophaga,Acinetobacter, andArcicellawere only detected in the bacterioplankton community. The results demonstrated that the relative abundances of bacterial communities at the phylum level are correlated with their diversity, and that the correlation between the relative abundance and number of unique OTUs was significant for benthic and planktonic bacterial communities.
bacterial diversity; bacterial community composition; pyrosequencing; Lake Poyang
国家自然科学基金(31360127,31060082); 南昌大学校级创新创业训练计划项目(2013011)
2014- 05- 03; < class="emphasis_bold">网络出版日期:
日期:2015- 05- 19
10.5846/stxb201405030873
*通讯作者Corresponding author.E-mail:wl690902@hotmail.com
寇文伯,黄正云,张杰,刘倩纯,刘芳鹏,刘以珍,吴兰.鄱阳湖湖泊细菌群落组成及结构——以松门山为例.生态学报,2015,35(23):7608- 7614.
Kou W B, Huang Z Y, Zhang J, Liu Q C, Liu F P, Liu Y Z, Wu L.Bacterial community structure and composition in Lake Poyang:a case study in the Songmenshan Region, China.Acta Ecologica Sinica,2015,35(23):7608- 7614.

