缺失PHO13基因与进化工程构建高效利用木糖酵母
汪晨宇,张国畅,陈 洵
(天津大学化工学院,天津 300072)
生物乙醇被认为是替代化石能源最洁净的液体燃料,近年来利用可再生资源生产生物乙醇受到越来越多的关注。相比传统的基于淀粉或者蔗糖的材料,木材废料和农业废弃物中的木质纤维素具有来源丰富,价格低廉并且不占用粮食资源等优点,木质纤维素生产乙醇已经成为了极具吸引力的生物乙醇生产途径[1-3]。然而作为广为人知的用来生产工业乙醇的微生物,酿酒酵母本身并不能利用木质纤维素中第二丰富的木糖,所以近20年来大量研究致力于利用酿酒酵母发酵木糖,其中主流方法是在酵母菌株内整合外源木糖还原酶(XR)和木糖醇脱氢酶(XDH)代谢通路,同时过表达内源木酮糖激酶(XK)编码基因。在XR/XDH代谢通路中木糖首先被依赖NADPH的XR转化成木糖醇,接着被依赖NAD+的XDH转化为木酮糖,这两步反应依赖的辅酶不同,造成的细胞内辅酶不平衡和木糖醇的积累是XR/XDH代谢通路的主要问题[4-7]。PHO13基因编码的蛋白同时具有蛋白磷酸化酶活性和对硝基苯磷酸盐的碱性磷酸化酶活性,研究表明缺失PHO13基因能提高酿酒酵母在木糖培养基里的生长速率和乙醇产量[8-9]。但是最初的研究是在实验室菌株上进行,并且对照菌的木糖代谢能力并不突出。同时,进化工程作为一个有力的提高工业微生物性状的方法,已经被应用于提高重组酿酒酵母利用木糖的能力[10-15]。因此,我们在重组工业菌株内缺失了PHO13基因,同时利用进化工程的方法,获得了能更加高效发酵木糖的工业菌株。酿酒酵母作为兼性厌氧微生物,能够在有氧条件利用木糖生长,在无氧条件下产出乙醇,所以我们选择有氧条件测试菌株生长情况,微好氧条件测试菌株木糖发酵性能,对所得菌株与出发菌株在不同木糖浓度培养基中进行了发酵实验,并测定了系列发酵数据。
1 实验材料与方法
1.1 菌株、培养基与培养条件
本研究中使用的酿酒酵母菌株和质粒列于表2。大肠杆菌Top10用于克隆和质粒的扩增。酿酒酵母KAM-2[16]作为出发菌株进行基因操作,诱变和进化工程筛选。大肠杆菌在包含浓度0.1 g/L氨苄青霉素的LB培养基(10 g/L胰化蛋白胨,5 g/L酵母提取物,10 g/L NaCl,pH值 7.0)37℃条件下培养。酵母菌在 YPD培养基(10 g/L酵母提取物,20 g/L蛋白胨,20 g/L葡萄糖)30℃条件下培养。YSCD培养基[6.7 g/L酵母基本氮源(YNB),根据营养缺陷型添加相应的氨基酸,20 g/L葡萄糖]用于酵母转化子的筛选,5-氟乳氢酸培养基用于URA3基因的反选择。YSCX培养基(6.7 g/L YNB,根据营养缺陷型添加相应的氨基酸,50 g/L木糖),YPX培养基(10 g/L酵母提取物,20 g/L蛋白胨,50或100 g/L木糖)用于进化工程和木糖发酵。
1.2 质粒和菌株构建
核酸操作使用标准的分子遗传技术,酵母的转化使用醋酸锂方法[17]。
从毕赤酵母NRRL7124和酿酒酵母W303菌株染色体中扩增出 XYL1,XYL2,XKS基因分别编码XR,XDH和XK。质粒 YIplac211-A8用启动子 pCCW12和pFBA1分别过表达 XYL1和 XKS,质粒 YI-plac211-Y8用启动子pCCW12和pTPI1分别过表达XYL2和 XKS,质粒 YIplac211-H8用启动子 pHXT7和pFBA1分别过表达XYL1和XYL2,将它们整合到酵母染色体上获得木糖利用重组酵母菌株KAM-6X(表 2)。
质粒pUC18-PHO13pt-URA用于缺失PHO13基因,用引物 pPHO13-U和 pPHO13-D(表1)通过PCR将PHO13启动子从W303染色体中扩增出来并插入到pUC18的 Hin d III与 Pst I酶切位点之间,同时用引物tPHO13-U和tPHO13-D(表1)扩增出PHO13终止子,插入到pUC18的BamHI与Eco RI酶切位点之间,得到质粒pUC18-PHO13pt。用引物URA-U和URA-D(表1)从质粒 YCplac33中扩增出筛选标记URA3基因,插入到 pUC18-PHO13pt的 Pst I和BamHI酶切位点之间,即得到质粒pUC18-PHO13pt-URA(表2)。然后用 Hin d III和 Eco RI双酶切质粒pUC18-PHO13pt-URA将其线性化后转化KAM-6X涂布CM-URA培养基,通过PHO13基因两端的同源序列与染色体发生同源重组用URA3替换PHO13基因,能在 CM-URA平板上生长的菌株为整合上URA3基因即PHO13缺失的转化子。接着将缺失PHO13的转化子涂在5-氟乳氢酸培养基中反选择URA3以排除URA3基因对生长的影响,由于 URA3基因会催化5-氟乳氢酸转化为对细胞有毒性的物质,因而能在5-氟乳氢酸平板上生长的为 URA3点突变失活的菌株,命名为KAM-6X(△PHO13)(表2)。
1.3 诱变和进化工程筛选
收集1 mL过夜培养的细胞(大约2×108个),无菌水冲洗后用0.1 mol/L的磷酸缓冲液(pH值7)重悬。将终体积分数为4%的EMS做为最适宜诱变剂量加入到细胞悬液中,震荡混匀。然后将混合体系在30℃条件下轻微摇动培养1 h,加入等体积新鲜的质量体积比(下同)为5%硫代硫酸钠终止诱变反应,离心收集诱变细胞,用5%硫代硫酸钠冲洗2次,重悬在无菌水中,进行下一步的进化筛选。进化包括3个阶段,每一轮都是以起始OD6000.1接入,生长至20后转接到新鲜的培养基中。第一阶段是在有氧条件下的含50 g/L木糖的YSCX培养基中进行了8轮,第二阶段是在微好氧条件下含50 g/L木糖的YPX培养基中进行了8轮,第三阶段在微好氧条件下含100 g/L木糖的YPX培养基中进行了12轮。将最后的细胞稀释涂布在含50 g/L木糖的YPX平板上,挑选较大的单菌落测试木糖发酵性能,获得最优发酵菌株PE(表2)。

表1 本研究中使用的引物Tab le 1 Primers used in this study
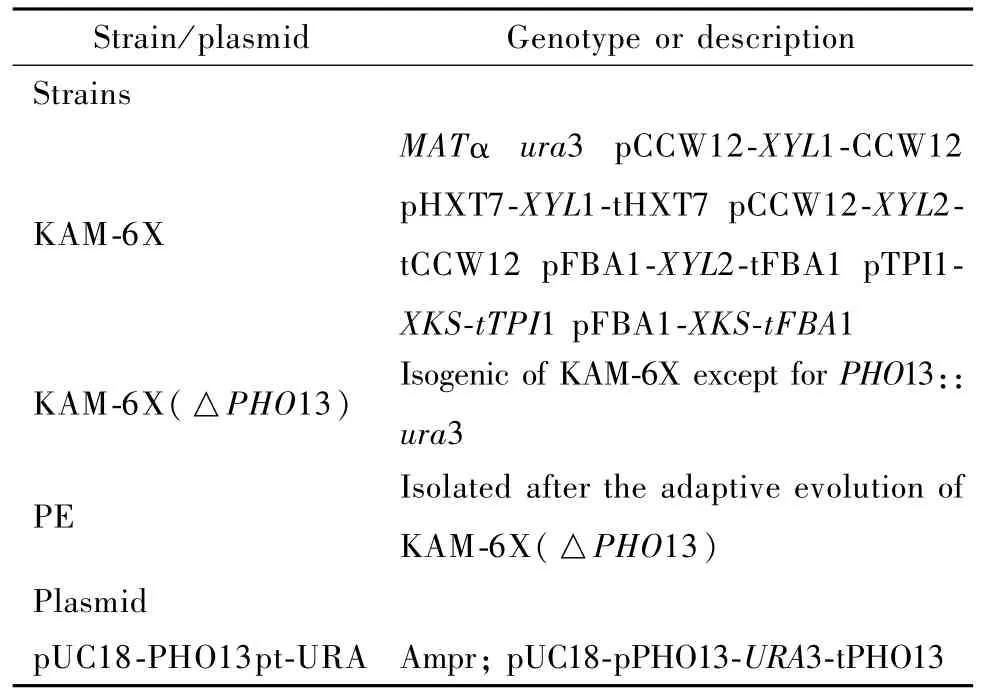
表2 本研究中使用的菌株和质粒Tab le 2 Strains and p lasmid used in this study
1.4 酶活测定
在含50 g/L木糖的YPX培养基中培养细胞至对数期,离心收集后用400μL预冷的裂解缓冲液重悬。将细胞转移至预先冷冻装有200μL玻璃珠的细胞破碎管,放入细胞破碎仪(Thermo Electron,USA)中以速度4档破碎30 s,重复3次。冷冻高速离心10 min,取上清液通过BioRad DC蛋白分析试剂盒(Bio-Rad,USA)测定蛋白浓度后用于酶活测定。酶活测定按照之前所报道的方法[7],使用CECIL 2000 series分光光度计(Bioquest,England)在 340 nm波长,30℃条件下测定反应曲线。所有酶活力的定义为每1 mg总蛋白每分钟转化NAD(P)H的微物质的量。NADH的摩尔吸光系数为 6.22 mol·mmol-1·cm-1。
1.5 木糖发酵及结果分析
菌株在YPD培养基中预培养12 h,离心收集细胞,用无菌水洗去葡萄糖后接入100 mL三角瓶中的40 mL发酵培养基。起始 OD为 1,干质量约为0.25 g/L。发酵温度30℃,好氧条件使用锡箔纸封口,微好氧条件使用封口膜封口,用0.5 mm的针头刺一个外排CO2的孔。每24 h取样用于代谢物组分分析。
代谢产物通过Waters Alliance 2695高效液相色谱(Waters, Milford, USA),HPX 87H交换色谱柱(BioRad,USA)和 Waters 2410示差检测器进行定量分析。以5 mmol/L的 H2SO4作为流动相,流速和柱温分别保持在0.4 mL/min和45℃。细胞干质量经过3次测量确定,用0.45μm预先称质量的玻璃纤维素膜过滤收集5 mL菌液,用无菌水过膜,放置于90℃烘箱14 h后称质量,细胞干质量即为两者质量之差。
2 结果与讨论
2.1 有氧条件下生长
我们首先测定了所得菌株在含50 g/L和100 g/L木糖的YPX培养基中有氧条件下的木糖消耗和生长情况,KAM-6X作对照。KAM-6X,KAM-6X(△PHO13)和PE的最大比生长速率,在50 g/L木糖浓度下分别为 0.153、0.261和 0.299 h-1,在100 g/L木糖浓度下分别为0.139、0.258和0.282 h-1,PE菌株比出发菌株在2种木糖浓度下比生长速率分别提高了95.43%和102.87%。在含50 g/L木糖的YPX中,前24 h内PE能耗掉36.12 g/L木糖,PHO13缺失菌能耗掉27.93 g/L木糖,而 KAM-6X只能耗掉8.16 g/L木糖。在100 g/L木糖的YPX中,前48 h内PE能耗掉70.25 g/L木糖,PHO13缺失菌能耗掉46.37 g/L木糖而KAM-6X只能耗掉41.5 g/L木糖。从图1能看出改造后的菌株在2种木糖浓度下都显示延迟期缩短,但指数期生长和耗糖情况差别不大,这与之前的报道一致[9]。
酶活测定所得如表3,PHO13缺失菌以及进化工程所得的菌株与出发菌株酶活没有大的差别,证明进化方法产生了一些新的偏好于代谢木糖的基因性状,也从另一角度证明了我们的进化方法可行。
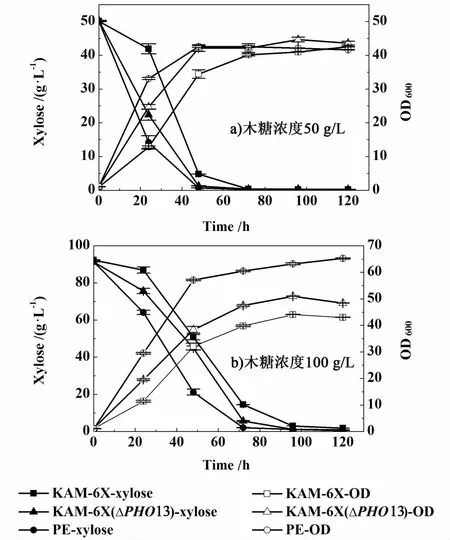
图1 菌株不同木糖浓度下有氧生长和木糖消耗情况Fig.1 G rowth and xylose consumption under aerobic condition in YPX
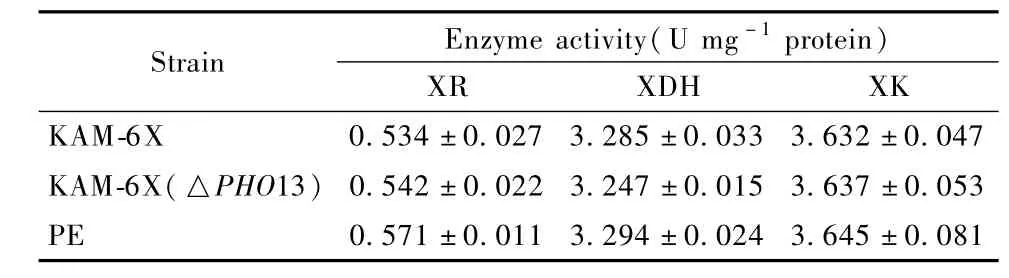
表3 酵母细胞培养粗提物中的体外酶活性Tab le 3 In vitro enzyme activities in crude extract fromcell cultu res of yeast strains
2.2 50 g/L木糖浓度下微好氧发酵
为了比较所得菌株的木糖发酵能力,我们在50 g/L的木糖浓度下进行了微好氧发酵实验。发酵数据如表4所示,PHO13缺失菌株能更快的消耗木糖[图2a)],产生更多的乙醇[图 2b)]和更少的木糖醇[图2c)],甘油产量的降低和乙酸产量的增加与之前的报道[8]一致。进化之后的菌株进一步提高耗糖速率[图2a)]和乙醇产量[图2b)],甘油产量继续减少,虽然木糖醇[图2c)]和乙酸产量稍微增加,但是糖醇转化率进一步提升。
2.3 100 g/L木糖浓度下KAM-6X(△PHO13)与PE微好氧发酵
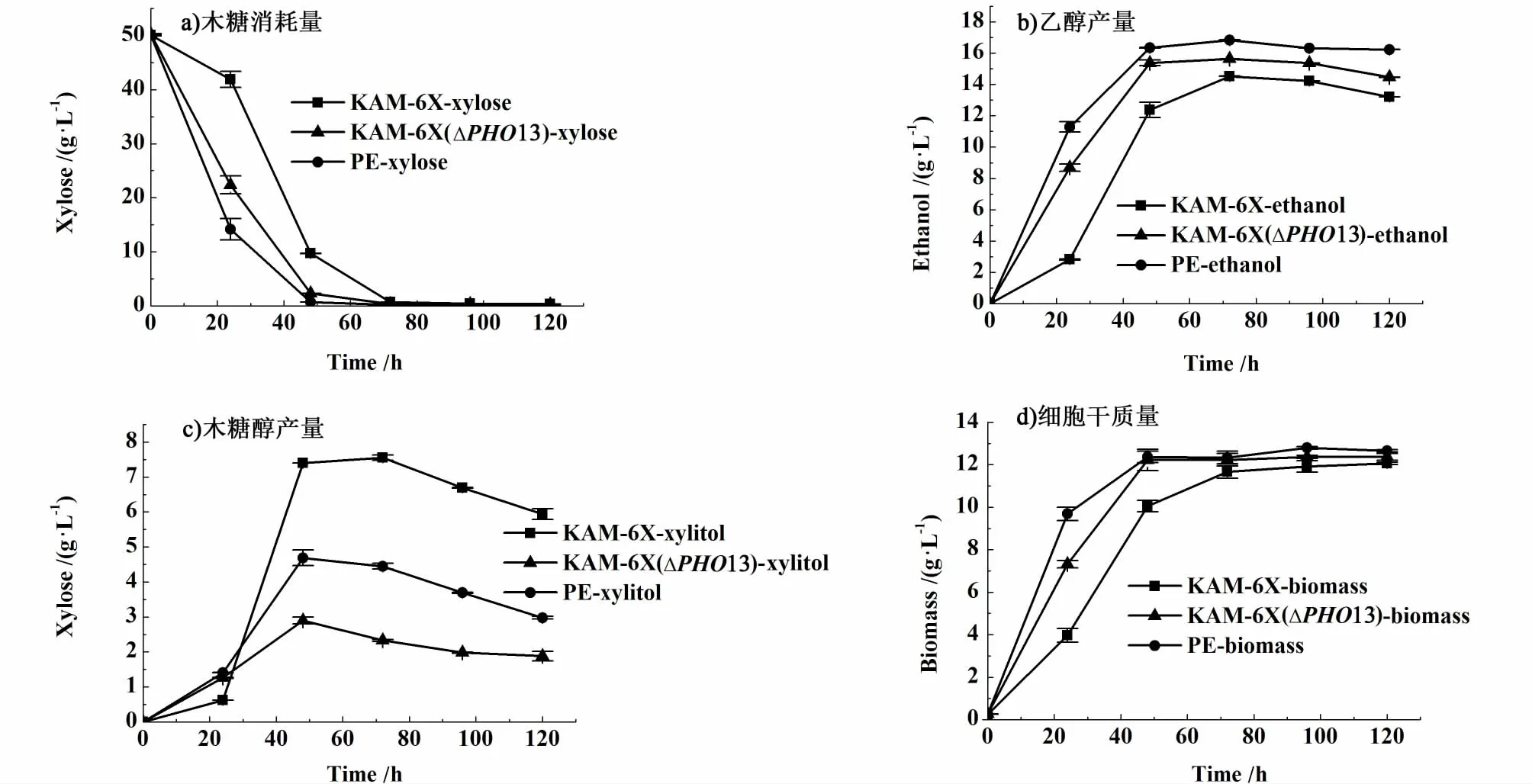
图2 菌株在50 g/L木糖浓度下微好氧发酵情况Fig.2 Xylose fermentation under oxygen-limited cond itions in YPX with 50 g/L xylose
由于 KAM-6X(△PHO13)和 PE能迅速耗掉50 g/L的木糖,我们接着测试了所得菌株在含100 g/L木糖的YPX中的发酵性能,结果如图3所示。与有氧条件下相比,微好氧条件下木糖消耗速率有所降低,KAM-6X(△PHO13)菌株能在 120 h消耗掉88.44 g/L的木糖,糖醇转化率为0.374 g/g,木糖醇转化率为0.059 g/g。PE菌株能在120 h能完全消耗掉95.33 g/L的木糖,但是糖醇转化率升高到0.382 g/g,木糖醇转化率也仅为0.066 g/g。结果证明,PE是一株高效利用木糖发酵的酿酒酵母工业菌株。
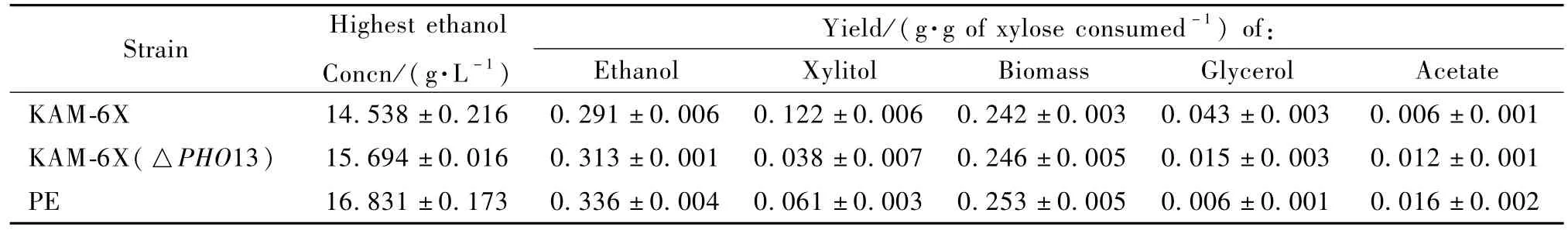
表4 50 g/L木糖浓度下微好氧木糖发酵结果Tab le 4 Resu lts of oxygen-limited batch fermentation of xylose(50 g/L)
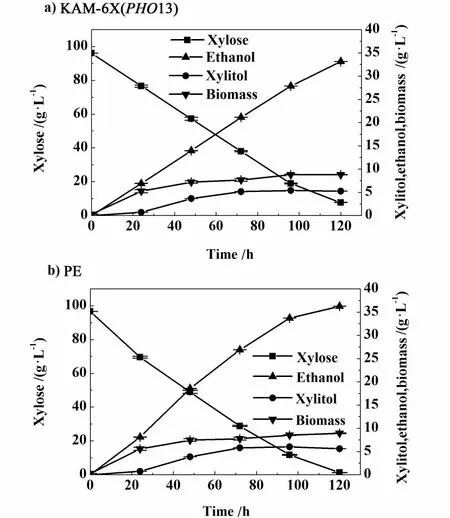
图3 100 g/L木糖浓度下微好氧发酵情况Fig.3 Xylose fermen tation under oxygen-limited cond itions in YPX with 100 g/L xylose
PHO13基因缺失被认为是调节了细胞的氧化还原水平,之前有报道PHO13缺失强烈抑制了呼吸缺陷菌株在木糖培养基中的生长[11],推测 PHO13缺失对木糖发酵的积极作用依靠细胞的呼吸作用,这和我们在微好氧条件下耗糖速率较慢的结果一致。在另一个报道中,PHO13缺失也提高了木糖利用速率和乙醇产出,但是甘油的产量却大大的提升了[9]。由此可以看出,不同菌株内氧化还原水平不同导致PHO13缺失对于不同基因背景的菌株效果有所差异。
我们在已经具有较强木糖利用能力的工业菌株内缺失了PHO13,有氧消耗木糖速率提高了将近1倍,微好氧发酵耗糖速度稍慢,糖醇转化率得到提升,木糖醇和甘油产量下降而乙酸的含量增加,这与最初的报道效果基本一致[8]。
通过进化工程,我们进一步提升了木糖消耗速率和糖醇转化率,这共分为三个阶段。第一阶段使用接近工业发酵营养成分的合成培养基,进行严格的筛选。第二阶段使用天然培养基避免了由于营养物质的限制造成的选择压力,使在微好氧条件下快速利用木糖成为唯一的筛选条件。第三阶段提高培养基中的木糖浓度到100 g/L,这样是为了拓宽木糖利用提升空间,获得能更加高效利用木糖的菌株。由于PHO13缺失菌株本身的木糖代谢能力已经很强,所以进化筛选得到的菌株提高的范围有限,但也是一种可行的提高木糖利用能力的改造方法。
3 结论
通过在具有较强木糖利用能力的工业酿酒酵母菌株中缺失 PHO13基因,结合进化工程方法,我们获得了一株高效利用木糖的工业菌株。有氧条件下最大比生长速率增加1倍,木糖消耗速度显著提高。微好氧发酵的副产物大量减少,糖醇转化率增加到0.382 g/g。结果证明PHO13基因缺失在不同于之前报道的基因背景的工业酵母菌株内也有提高木糖发酵能力的作用,结合合适的进化工程策略能够有效的构建高效利用木糖的工业菌株,所得菌株可以进一步分析改造以应用于工业生产。
[1]Ragauskas A J,Williams C K,Davison B H,et al.The path forward for biofuels and biomaterials[J].Science,2006, 311(5760): 484-489
[2]Stephanopoulos G.Challenges in engineering microbes for biofuels production [J].Science, 2007, 315(5 813): 801-804
[3]刘健,陈洪章,李佐虎.木糖发酵生产乙醇的研究[J].工业微生物,2001,31(2):36-41 Liu Jian, Chen Hongzhang, Li Zuohu.Ethanol production of xylose fermentation by Pichiastipitis[J].Industrial Microbiology, 2001, 31(2): 36-41(in Chinese)
[4]Hahn-Hägerdal B,Karhumaa K,Fonseca C,et al.Towards industrial pentose-fermenting yeast strains[J].Applied Microbiology and Biotechnology, 2007, 74(5):937-953
[5]Matsushika A,Inoue H,Kodaki T,et al.Ethanol production fromxylose in engineered Saccharomyces cerevisiae strains: Current state and perspectives[J].Applied Microbiology and Biotechnology, 2009, 84(1): 37-53
[6]Van Vleet JH,Jeffries TW.Yeastmetabolic engineering for hemicellulosic ethanol production[J].Current Opinion in Biotechnology, 2009, 20(3): 300-306
[7]Eliasson A,Christensson C,WahlbomC F,et al.Anaerobic xylose fermentation by recombinant Saccharomyces cerevisiae Carrying XYL1, XYL2, and XKS1 in mineral mediumchemostat cultures[J].Applied and Environmental Microbiology, 2000, 66(8): 3 381-3 386
[8]Van Vleet J H,Jeffries T W,Olsson L.Deleting the para-nitrophenyl phosphatase(pNPPase), PHO13, in recombinant Saccharomyces cerevisiae improves growth and ethanol production on D-xylose[J].Metabolic Engineering, 2008, 10(6): 360-369
[9]Fujitomi K,Sanda T, Hasunuma T, et al.Deletion of the PHO13 gene in Saccharomyces cerevisiae improves ethanol production fromlignocellulosic hydrolysate in the presence of acetic and formic acids, and furfural[J].Bioresource Technology, 2012, 111: 161-166
[10]Liu E, Hu Y.Construction of a xylose-fermenting Saccharomyces cerevisiae strain by combined approaches of genetic engineering,chemical mutagenesis and evolutionary adaptation[J].Biochemical Engineering Journal, 2010, 48(2): 204-210
[11]Shen Y,Chen X,Peng B,et al.An efficient xylose-fermenting recombinant Saccharomyces cerevisiae strain obtained through adaptive evolution and its global transcription profile[J].App lied Microbiology and Biotechnology, 2012, 96(4): 1 079-1 091
[12]Sonderegger M, Sauer U.Evolutionary engineering of Saccharomyces cerevisiae for anaerobic growth on xylose[J].Applied and environmental Microbiology, 2003,69(4): 1 990-1 998
[13]Wisselink H W, Toirkens MJ,Wu Q,et al.Novel evolutionary engineering approach for accelerated utilization of glucose, xylose, and arabinose mixtures by engineered Saccharomyces cerevisiae strains[J].Applied and Environmental Microbiology, 2009, 75(4): 907-914
[14]Zhou H, Cheng J, Wang B, et al.Xylose isomerase overexpression along with engineering of the pentose phosphate pathway and evolutionary engineering enable rapid xylose utilization and ethanol production by Saccharomyces cerevisiae[J].Metabolic Engineering,2012, 14(6): 611-622
[15]Scalcinati G,Otero JM,Vleet JR H,et al.Evolutionary engineering of Saccharomyces cerevisiae for efficient aerobic xylose consumption[J].FEMS Yeast Research,2012, 12(5): 582-597
[16]Zhang G, Liu J, Ding W.Decreased xylitol formation during xylose fermentation in Saccharomyces cerevisiae due to over expression of water-forming NADH oxidase[J].Applied and Environmental Microbiology, 2012,78(4): 1 081-1 086
[17]Sambrook JD.Molecular cloning: A laboratory manual[M].Third Edition.Cold Spring Harbor, NewYork:Cold Spring Harbor Laboratory Press,2001

