大豆查尔酮还原酶3基因(chr3)的克隆及体外催化活性分析
张 超,王丕武,张 卓
(吉林农业大学 a 农学院,b 生命科学学院, 吉林 长春130118)
大豆查尔酮还原酶3基因(chr3)的克隆及体外催化活性分析
张 超a,王丕武a,张 卓b
(吉林农业大学 a 农学院,b 生命科学学院, 吉林 长春130118)
【目的】 克隆大豆查尔酮还原酶3基因(chr3),并分析其编码蛋白体外催化活性,为研究大豆苷元的合成与调控研究提供参考。【方法】 以大豆品种“吉农17”为材料,采用RACE技术扩增大豆chr3基因,对其编码蛋白的结构和功能位点进行分析。将目的基因chr3连接到大肠杆菌载体pET28a上,然后转化到大肠杆菌BL21中,构建重组大肠杆菌原核表达载体BL21-pET28a-chr3,并通过高效液相色谱技术(HPLC)验证重组蛋白的催化性质及效率。【结果】 成功克隆chr3基因,其全长序列长1 416 bp (GenBank 登录号:KF927169),成功构建了大肠杆菌原核表达载体BL21-pET28a-chr3。HPLC检测结果表明,重组蛋白CHR3催化合成了异甘草素,使大豆异甘草素含量提高到了33.673 μmol/g。【结论】 分离鉴定了大豆chr3,其编码蛋白CHR3催化活性明显提高。
大豆;大豆苷元;查尔酮还原酶3基因;基因克隆;大肠杆菌
大豆苷元能够调节多项植物生理功能并增强植物对生态环境的适应能力,且在许多基础的植物生理过程中发挥重要作用。同时大豆苷元对于人体有着直接且重要的保健作用,如能够降低胆固醇含量和预防一些癌症[1-3]。
Welle等[4]首次在大豆中鉴定了查尔酮还原酶(Chalcone Reductase,CHR)及其催化功能。在紫花苜蓿等其他豆科植物中陆续分离鉴定了chr基因及其蛋白产物,并发现CHR特异性地存在于豆科植物中[5]。大豆苷元的生物合成过程是大豆异黄酮合成途径的一个分支。以香豆醛辅酶A和丙二酰基辅酶A为底物,经CHR与查尔酮合成酶(Chalcone Synthase,CHS)联合反应,得到产物4,2′,4′-三羟基查尔酮(异甘草素),异甘草素能够为查尔酮异构酶(Chalcone Isomerase,CHI)作用生成甘草素,构成异黄酮类化合物的15个碳原子的基本骨架,再在异黄酮合成酶(Isoflavone Synthase,IFS)作用下催化合成大豆苷元[6]。所以,CHR催化生成的化合物是大豆苷元合成所必需的前体物质[7]。同时,在缺乏CHR的情况下,前述几种酶蛋白的反应路径则会进入染料木素的合成途径中。Terrence等[8]研究证明,当通过RNAi技术干扰已知的大豆chr1基因的表达,仅导致大豆苷元含量明显减少,而染料木素等其他种类的异黄酮化合物含量则保持恒定或者升高。这一结果证明,大豆苷元与染料木素在生物体中的合成存在竞争关系。因此,探索大豆异黄酮合成代谢途径,克隆并鉴定chr基因功能对研究大豆苷元的合成与调控都具有重要的意义[9]。本研究采用RACE技术,克隆大豆查尔酮还原酶3基因(chr3),探讨新基因chr3在大豆苷元合成中的作用机理,以期为探索大豆苷元的合成机制和实际应用提供必要的基因资源。
1 材料与方法
1.1 植物材料与菌株
大豆品种“吉农17”、大肠杆菌DH5α、大肠杆菌BL21、克隆载体pMD18-T、原核表达载体pET28a均由吉林农业大学生物技术中心实验室保存。
1.2 主要试剂
3′ RACE试剂盒、5′ RACE试剂盒、PCR相关试剂、dNTP、T4 RNA连接酶、T4 DNA连接酶以及各种限制性内切酶,均购自TaKaRa公司;质粒提取试剂盒、胶回收试剂盒、异丙基-1-硫代-β-D-半乳糖苷(IPTG),均购自北京鼎国生物工程有限公司;Ni-NTA购自Pharmacia 公司;其他试剂和药品均为国产分析纯。
1.3 chr3基因cDNA全长片段的克隆[10-12]
1.3.1 核心片段 使用 RNAiso试剂盒(TaKaRa公司)提取大豆新鲜叶部组织的总RNA。按照RevertAid First Strand cDNA Synthesis Kit (FERMENTAS公司)操作说明合成cDNA,-20 ℃保存备用。PCR反应体系:Total RNA 3 μL,oligo(dT)181 μL,5×Reation Buffer 4 μL,RiboLock M-MuLV 逆转录酶1 μL,ddH2O 11 μL。42 ℃条件下孵育1 h,72 ℃条件下5 min终止反应。
扫描NCBI网站中大豆EST文库,获得chr基因的核心片段(GenBank登录号:TC365874),设计特异性引物Chr1-GSP1和Chr1-GSP2,Chr1-GSP1序列为5′-ATGTCGATCCCTATACCTT-3′,Chr1-GSP2序列为5′-TTAAGGAGACTTGTACCAC-3′。以逆转录的 cDNA 为模板,利用引物 Chr1-GSP1 和 Chr1-GSP2 通过 PCR 反应扩增目的基因核心片段。PCR 反应体系如下:10×TaqBuffer 2.5 μL,MgCl22.5 μL,dNTP Mix 0.5 μL,Chr1-GSP1 1 μL,Chr1-GSP2 1 μL,cDNA 1 μL,TaqDNA Polymerase 0.25 μL,ddH2O 28.75 μL。PCR扩增条件:94 ℃预变性 5 min;94 ℃变性40 s,55 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃延伸 8 min。
1.3.2 3′端cDNA片段 以大豆新鲜叶部组织的RNA为模板,按照3′-Full RACE Core Set Ver.2.0(TaKaRa公司)说明书进行3′ RACE。使用引物Chr1-GSP1和3′ RACE试剂盒中Outer Primer进行Outer PCR扩增反应,再以扩增产物为模板,使用引物Chr1-GSP2和3′RACE试剂盒中的Inner Primer进行Inner PCR反应,扩增得到目的基因3′端序列。Outer PCR反应体系:上述1.3.1的反转录反应液 1 μL,1×cDNA Dilution Buffer 8 μL,3′ RACE Control Outer Primer 2 μL,3′ RACE Outer Primer 2 μL,10×LA PCR Buffer 4 μL,MgCl23 μL,TaKaRa LATaq0.25 μL,ddH2O 28.75 μL。PCR扩增条件:94 ℃预变性5min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,20个循环;72 ℃延伸10 min。
Inner PCR反应体系:Outer PCR产物 1 μL,dNTP Mixture 8 μL,10×LA PCR Buffer 5 μL,3′ RACE Control Inner Primer 2 μL,3′ RACE Inner Primer 2 μL,MgCl25 μL,TaKaRa LATaq0.5 μL,ddH2O 26.5 μL。PCR扩增条件:94 ℃预变性3 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃延伸10 min。
1.3.3 5′端cDNA片段 以大豆新鲜叶部组织总RNA为模板,按照5′-Full RACE Kit(TaKaRa公司)说明书进行5′ RACE,通过 Tobacco Acid Pyrophosphatase(TAP)以去掉mRNA的5′帽子结构,使用T4 RNA 连接酶将5′ RACE Adaptor连接到mRNA的5′ 端后,使用5′ RACE Adaptor上的引物与核心序列的引物Chr1-GSP1进行RT-PCR反应,高特异性地扩增目的基因的5′ 端序列。Outer PCR反应体系:上述1.3.1的反转录反应液 2 μL,1×cDNA Dilution Buffer 8 μL,5′ RACE Control Outer Primer 2 μL,5′ RACE Outer Primer 2 μL,10×LA PCR Buffer 4 μL,MgCl23 μL,TaKaRa LATaq0.25 μL,ddH2O 28.75 μL。 PCR扩增条件:94 ℃预变性3 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,20个循环;72 ℃延伸10 min。
Inner PCR反应体系:Outer PCR产物1 μL,dNTP Mixture 8 μL,10×LA PCR Buffer 5 μL,5′ RACE Control Inner Primer 2 μL,5′ RACE Inner Primer 2 μL,MgCl25 μL,TaKaRa LATaq0.5 μL,ddH2O 26.5 μL。 PCR扩增条件:94 ℃预变性3 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,25个循环;72 ℃延伸10 min。
1.3.4 cDNA全长片段 将获得的3′和5′端cDNA片段进行拼接,设计扩增全长基因片段的特异性引物GSP3和GSP4。GSP3序列为5′-ATGTCGATCCCTATACCTT-3′,GSP4序列为5′-TTAAGGAGACTTGTACCAC-3′。以大豆新鲜叶部组织总RNA反转录的cDNA为模板,进行RT-PCR扩增。PCR 反应体系为:10×TaqBuffer 2.5 μL,MgCl22.5 μL,dNTP Mix 0.5 μL,GSP3 1 μL,GSP4 1 μL,cDNA 1 μL,TaqDNA Polymerase 0.25 μL,ddH2O 28.75 μL。PCR扩增条件除退火温度升高到56 ℃外,其余条件均与1.3.1节相同。用1%琼脂糖凝胶电泳检测扩增的产物。
1.4 chr3基因编码蛋白CHR3的结构与功能位点分析
应用在线程序ProMod Version 3.70 (http://beta.swissmodel.expasy.org/),分析chr3基因编码蛋白CHR3的氨基酸序列,预测chr3基因表达产物的基本性质及其催化功能。在NCBI网站中下载已知查尔酮还原酶基因序列,用DNAMAN软件对chr3基因序列与下载的已知查尔酮还原酶基因序列进行同源性比对,同时用NCBI网站在线分析程序ORF FINDER(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)对chr3基因序列进行开放阅读框分析,并寻找基因的保守域,通过DNAMAN软件将chr3基因开放阅读框翻译成氨基酸序列,用MEGA 4.0软件分析chr3基因编码蛋白CHR3与其他物种CHR蛋白质的同源性,构建系统进化树。
1.5 重组表达载体BL21-pET28a-chr3的构建
将目的基因Chr3连接到克隆载体pMD18-T上,构建载体pMD18T-chr3,以pMD18T-chr3载体为模板进行PCR反应,扩增带有设计酶切位点的目的基因。用SalⅠ和XhoⅠ双酶切扩增产物,经凝胶回收目的片段后,将其用T4 DNA连接酶连接至经同样双酶切处理的pET28a表达载体中,再转化大肠杆菌DH5α感受态细胞,挑取转化菌落,经PCR和双酶切鉴定,送至北京三博远志生物技术有限责任公司测序。将获得的重组表达载体命名为BL21-pET28-chr3。同时,以原核表达载体pET28a为阴性对照。
1.6 表达产物的分离纯化及SDS-PAGE电泳分析
将BL21-pET28a-chr3单菌落接种于5 mL的LB液体培养基(含100 mg/L卡那霉素)中,37 ℃条件下200 r/min振荡培养过夜。再将菌液接种于100 mL新鲜LB液体培养基(含100 mg/L卡那霉素)中,37 ℃、200 r/min振荡培养至OD600值约为0.6,加入IPTG至终浓度为5 mmol/L诱导表达,继续22 ℃、200 r/min振荡培养4 h。取表达产物,4 ℃、5 000 r/min离心10 min后收集菌体,用PBS重悬菌体,超声破碎,分别收集上清液和沉淀。将上清液通过Ni-NTA柱进行亲和层析,用10倍体积PBS溶液(20 mmol/L KH2PO4+0.2 mol/L NaCl,pH 7.6)平衡柱体,然后用含有30,200,400 mmol/L 咪唑的缓冲液(pH 7.6)进行梯度洗脱,收集各个洗脱峰,运用SDS-PAGE电泳分析收集成分。
1.7 chr3基因编码蛋白CHR3的功能鉴定与分析[13-16]
1.7.1 CHR3蛋白的制备及其催化反应 将1.5中构建的BL21-pET28a-chr3重组载体单菌落接种于5 mL的LB液体培养基(含100 mg/L卡那霉素)中,37 ℃条件下200 r/min振荡培养过夜。再将菌液接种于100 mL新鲜LB液体培养基(含100 mg/L卡那霉素)中,37 ℃振荡培养至OD600值约为0.6,加入IPTG至终浓度为5 mmol/L诱导表达后,继续于22 ℃、200 r/min振荡培养4 h。取表达产物,4 ℃离心后收集菌体,用PBS重悬菌体,超声破碎,收集BL21-pET28a-chr3菌液的处理液。称取0.5 g大豆粉,将其与2.5 mL BL21-pET28a-chr3重组质粒菌液混合,调整pH为6.5,18 ℃条件下200 r/min振荡处理45 min,将反应液立即置于80 ℃水浴中,终止催化反应。同时,以只含有pET28a质粒的BL21-pET28a为对照。
1.7.2 CHR3催化产物含量的测定 待1.7.1中大豆粉与重组蛋白CHR的催化反应终止后,将反应液转移到50 mL三角瓶中,先加入7.5 mL体积分数90%甲醇溶液,超声波处理之后浸泡过夜。过滤除去不溶的杂质,蒸发脱去萃取溶液中的溶剂,再加入乙醋酸盐缓冲液充分溶解提取物,同时加入适量的纤维素酶,37 ℃条件下过夜,确保除去所有修饰异甘草素糖苷的糖基。然后采用乙酸乙酯萃取酶水解液中的配基异甘草素,再将乙酸乙酯萃取相于N2中蒸干。将获得的样品溶解于甲醇溶液中,用0.22 μm有机滤膜抽滤,上样量为10 μL,用高效液相色谱技术(HPLC)进行定性与定量分析。
2 结果与分析
2.1 大豆chr3基因cDNA全长片段的克隆
2.1.1 核心片段 以大豆新鲜叶片总RNA转录的cDNA为模板,RT-PCR反应扩增目的基因的核心片段,得到长度为1 191 bp的片段(图1-A)。测序结果表明,扩增片段与NCBI中EST文库对应序列相似度为99%。
2.1.2 3′端cDNA片段 以大豆新鲜叶片总RNA转录的chr-1 cDNA为模板,使用特异性引物chr-1 GSP1分别与3′ RACE试剂盒中的引物扩增目的基因chr3 3′端片段,测序结果表明,获得的片段长度为1 294 bp,其中1 135 bp的区域与核心序列重叠,有159 bp的序列为新扩增的片段(图1-B)。
2.1.3 5′端cDNA片段 以大豆新鲜叶片总RNA转录的cDNA为模板,按照5′-Full RACE Kit说明书,扩增目的基因chr3的5′端片段,测序结果表明,得到的片段为533 bp,与核心片段有467 bp的重叠区域(图1-C)。
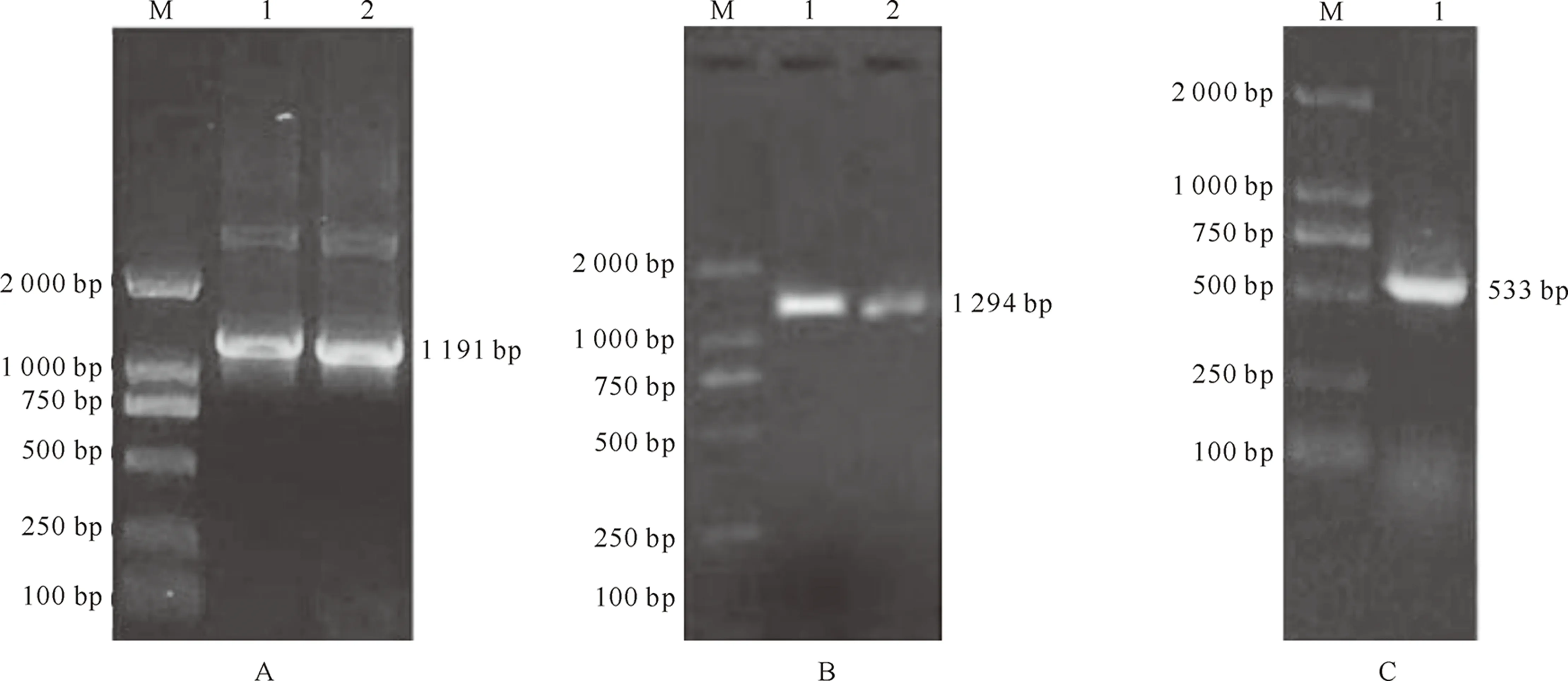
图1 大豆chr3基因的克隆A.核心片段;B.3′端cDNA片段;C.5′端cDNA片段;M.DNA标准分子量;1~2.PCR 产物
2.1.4 cDNA全长片段 将chr3 3′端cDNA片段和5′端cDNA片段的克隆测序结果进行拼接,去掉中间411 bp的重叠区域,得到chr3全长基因序列。设计扩增全长序列的特异性引物GSP3和GSP4,以大豆新鲜叶片的cDNA为模板,PCR反应扩增chr3基因的全长cDNA序列,得到长度为1 416 bp的片段,将其在GenBank上注册,登录号为KF927169(http://www.ncbi.nlm.nih.gov/nuccore/KF927169)。
2.2 大豆chr3基因编码蛋白CHR3的结构与功能位点预测
根据ProMod Version 3.70分析结果,理论预测chr3基因编码含有345个氨基酸残基的蛋白产物CHR3,其分子质量为32 189.42 u,理论等电点为5.90。对chr3基因编码蛋白CHR3的氨基酸序列分析发现,其中在48-65和72-155处共含有2个Aldo/Keto还原酶家族特征性催化位点 (Aldo/Keto reductase family signature),预示该蛋白可能属于Aldo/Keto型还原酶蛋白。同时构建chr3基因编码蛋白CHR3的3D立体结构,结果如图3所示。图3显示,chr3基因编码蛋白CHR3的3D立体结构中含有典型的Aldo/Keto型还原酶超家族(Aldo/Keto reductase superfamily,AKRs)的(α/β)8型桶式折叠结构。

图2 大豆chr3基因全长片段的PCR扩增结果 M.DNA标准分子量;1.PCR产物Fig.2 PCR product of soybean full chr3 geneM.DNA Marker;1.PCR product
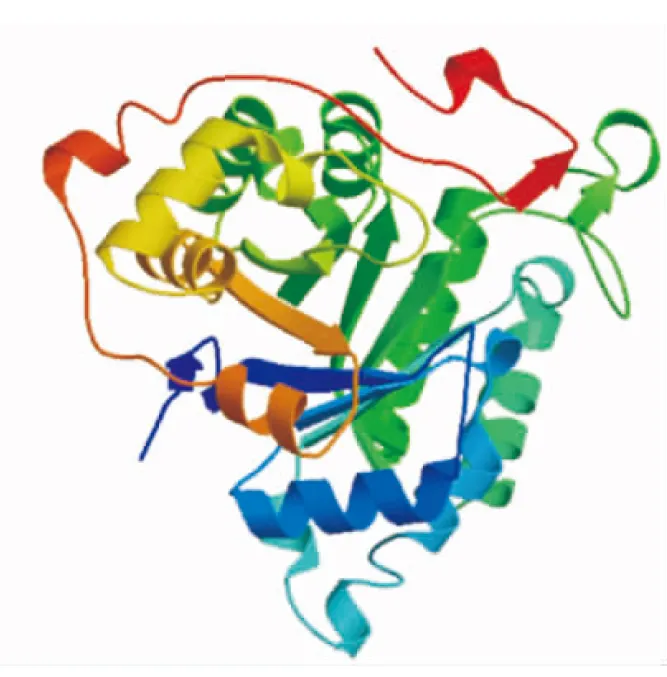
图3 预测的大豆chr3基因编码蛋白3D立体结构Fig.3 Three-dimensional structure of protein of soybean chr3 gene
氨基酸同源性分析表明,chr3基因编码蛋白CHR3与大豆已知的CHR1蛋白的同源性为82%,与CHR2蛋白的同源性为98%。系统进化树分析结果(图4)显示,CHR3的进化距离与CHR2更为相近,与CHR1稍远,相对于CHR1,CHR3与葛根、苜蓿等其他物种的进化距离较远,这也反映了大豆中不同CHR蛋白的进化趋势是有差异的。

图4 大豆chr3基因编码蛋白CHR3与其他物种CHR蛋白的系统进化树分支上的数字表示该树枝可信度的百分比
Fig.4 Phylogenetic tree of CHR protein Numbers on the cluster nodes are homology ratios among CHR verified by bootstrap
2.3 大豆重组蛋白CHR3在大肠杆菌中的表达与纯化
用在线分析程序ORF FINDER(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)对chr3基因序列进行开放阅读框分析,预测其长度为969 bp。经限制性内切酶SalⅠ、XhoⅠ酶切后,将chr3开放阅读框序列连接到原核表达载体pET28a,将重组载体转化到大肠杆菌BL21中,得到重组质粒BL21-pET28a-chr3,对其进行PCR和双酶切鉴定,结果(图5和图6)显示,重组质粒BL21-pET28a-chr3构建成功。

图5 大豆chr3基因原核表达载体 BL21-pET28a-chr3的PCR扩增结果 M.DNA标准分子; 1~4.BL21-pET28a-chr3的PCR产物;5.阴性对照 (pET28a)Fig.5 PCR identification of recombinant vector of soybean chr3M.DNA Marker;1-4.PCR products of BL21-pET28a-chr3 recombinant vector;5.Negative control (pET28a)
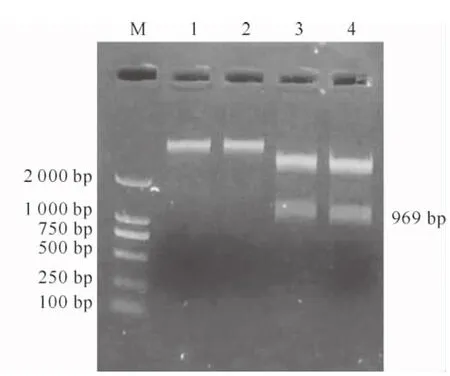
图6 大豆chr3基因原核表达载体 BL21-pET28a-chr3的双酶切鉴定结果 M.标准DNA分子;1,2.阴性对照 (pET28a);3,4.BL21-pET28a-chr3的SalⅠ、XhoⅠ双酶切产物Fig.6 Double digest identification of BL21-pET28a-chr3 recombinant vector of soybean chr3 M.DNA Marker;1,2.Negative control (pET28a);3,4.Products of BL21-pET28a-chr3 recombinant vector digested by SalⅠ and XhoⅠ
将构建的大肠杆菌工程菌BL21-pET28a-chr3用IPTG诱导表达4 h后,超声破碎样品,并用Ni-NTA柱进行亲和层析,将纯化的目的蛋白进行SDS-PAGE分析,结果(图7)显示,在诱导前没有目的条带出现,诱导但未纯化的样品中有分子质量约为32.5 ku的条带出现,与chr3基因表达产物的预期分子质量相符;此外,纯化后样品中只含有1条目的条带。经分析,表达产物约占菌体总蛋白的20%,说明CHR蛋白已能在大肠杆菌中正常表达。
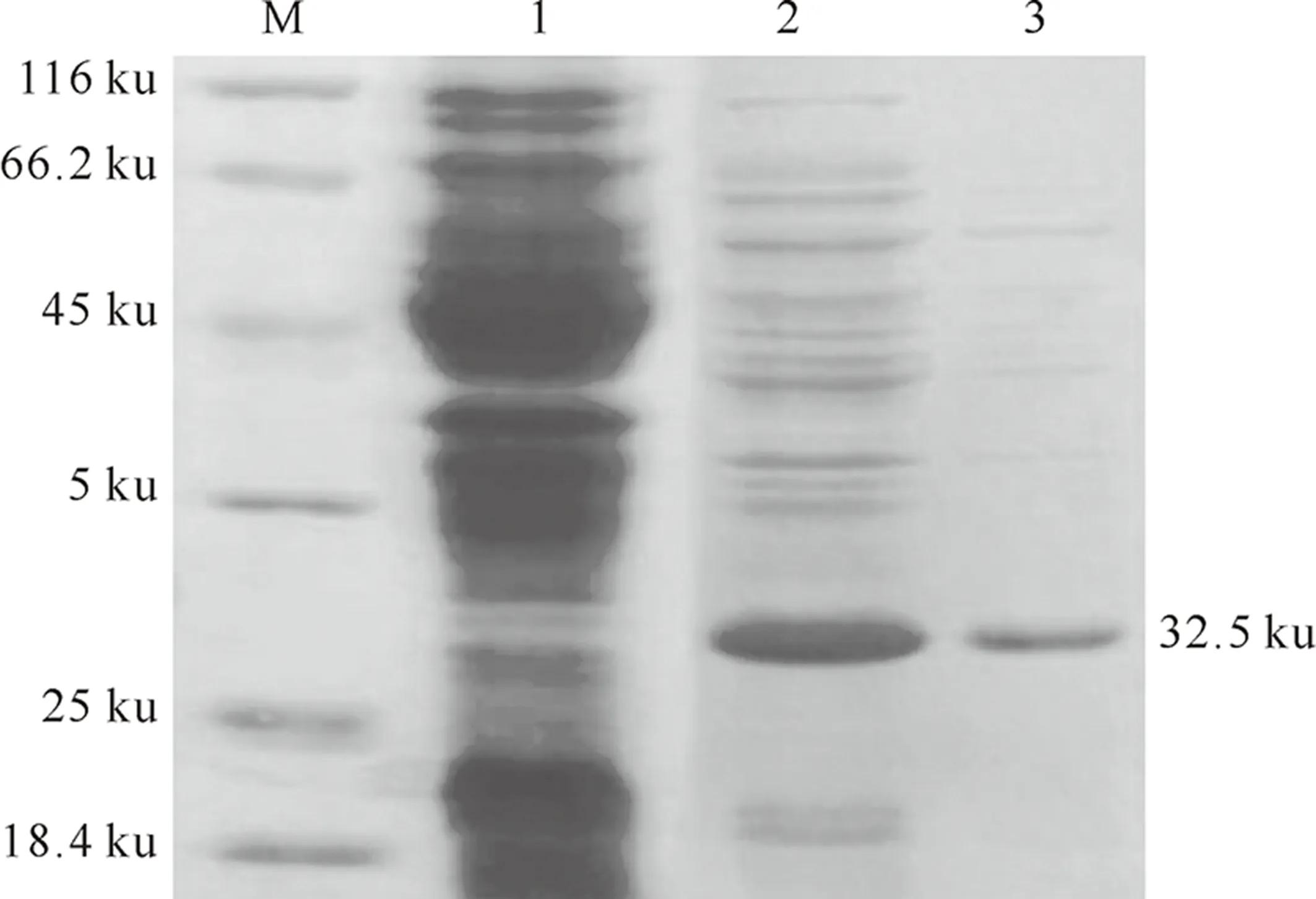
图7 大豆重组CHR3蛋白的SDS-PAGE鉴定M.标准低分子量蛋白;1.IPTG诱导前的表达蛋白;2.IPTG诱导后的表达蛋白;3.经过Ni-NTA柱纯化后的蛋白
2.4 大豆重组蛋白CHR3的催化功能
通过HPLC技术检测催化反应产物,鉴定基因chr3表达产物的催化功能。观察反应前后大豆粉中异甘草素的含量变化,结果如图8所示。由图8可以看出,异甘草素的保留时间为(5.374±0.1) min。在对照组中异甘草素峰面积为542 256,换算成含量为1.597 μmol/g,在重组蛋白CHR3处理的样品中,异甘草素峰面积则明显上升,为1 241 766,换算成含量为33.673 μmol/g。说明大肠杆菌表达的CHR3蛋白催化合成了异甘草素。
3 讨 论
CHR3行使催化作用时需要在NADPH存在情况下与查尔酮合成酶(CHS)联合作用。而对于这种联合作用机制目前尚不清楚。Dixon等[15]认为,在豆科植物中CHR与CHS都是以多基因型的形式存在于基因组中,并且其基因数量呈现出一种复杂的对应关系。迄今为止,在紫花苜蓿中发现了5种chr3基因;蒺藜苜蓿中发现了6种chr3基因;鹰嘴豆和黄芪等豆科植物也都分离得到2种以上chr基因,而大豆中至今仅确认了chr1(GenBank登录号:X55730)和chr2 (GenBank登录号:KF758935)2种基因。同时在大豆中已经至少发现了9种chs3基因。大豆中已知的chr3基因与chs3基因数量的不匹配限制了人们对于CHR与CHS联合作用机制的研究。
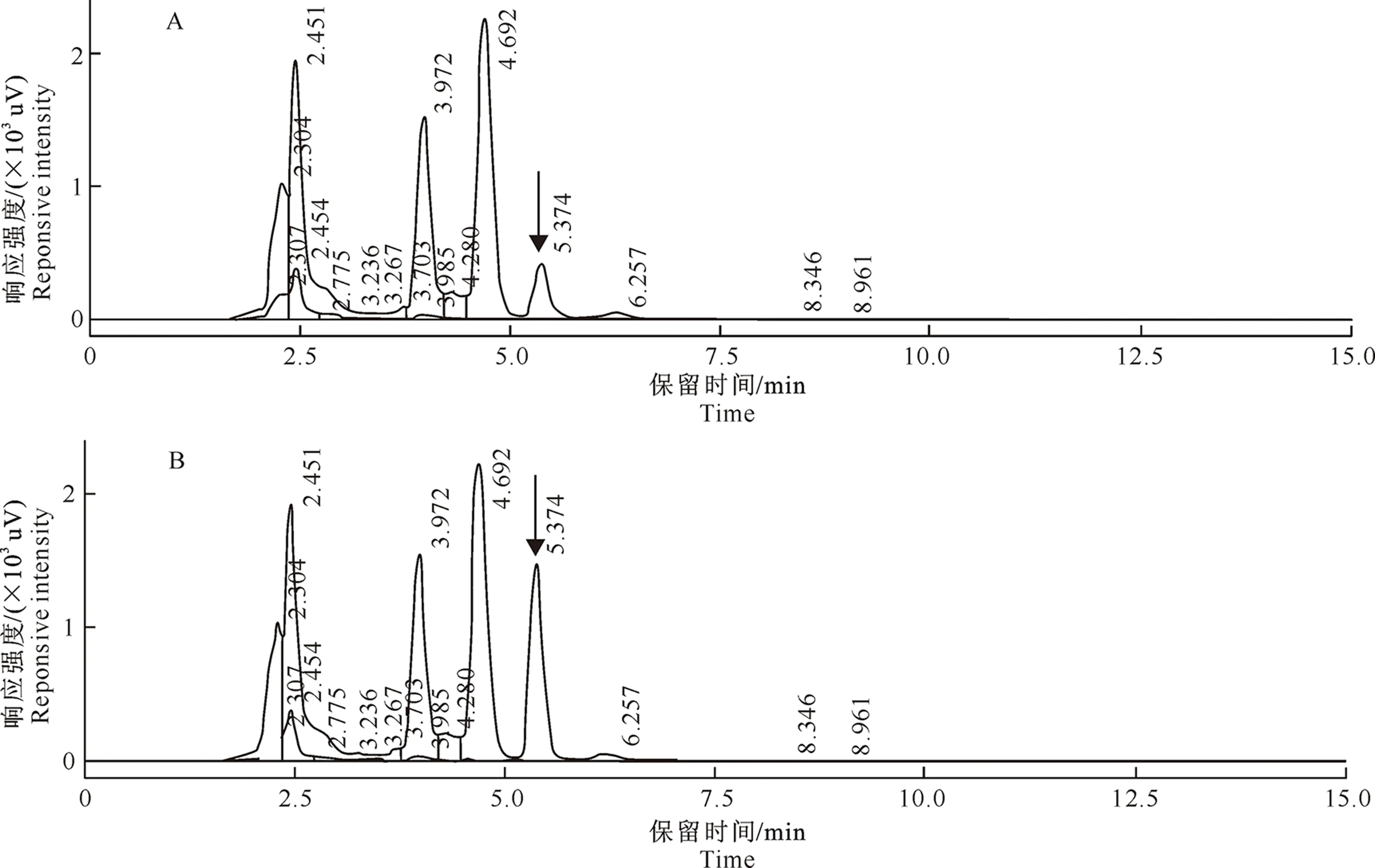
图8 重组蛋白CHR3催化后大豆粉中异甘草素含量的HPLC分析图谱
目前,已知的所有CHR都被验证属于AKRs超家族[17-18]。AKRs是自然界中三大氧化还原蛋白超家族中的一种。该蛋白超家族的结构特点是每个单体含有(α/β)8型桶式折叠结构,平均含有320个氨基酸残基,依靠NAD(P)(H)结合在催化位点上形成具有催化活性功能的酶蛋白[19-20]。另外分析显示所有CHR蛋白进化趋势相对保守,但在不同物种间还是有一定的差异。本研究分离得到能够编码345个氨基酸残基的新大豆chr3基因,预测其表达产物的肽链结构均符合上述特点。同时chr3基因编码蛋白CHR3在进化趋势上与chr1基因编码蛋白CHR1稍远,与chr2基因编码蛋白CHR2更为接近。本研究构建了含有chr3基因的原核表达载体pET28a-chr3,并将其重组到大肠杆菌BL21中并诱导表达,催化大豆粉中的底物合成了异甘草素,在对照组中异甘草素含量为 1.597 μmol/g,在重组菌处理的样品中异甘草素含量为33.673 μmol/g,重组菌的异甘草素含量是对照组的21.1倍,从而为确认chr3克隆成功,且其编码蛋白有明显的体外催化活性。
在大豆中,当染料木素不再进行代谢,直接贮存大豆苷元于细胞的液泡中,大豆苷元则作为大豆抗毒素类化合物合成必需前体物质被持续代谢。由于在大豆等豆科植物中大豆苷元含量少于染料木素,因此关于大豆苷元的相关研究和实际应用均落后于染料木素。关于大豆苷元的合成机理研究因为chr3基因资源不足而受到限制[21-23],本研究分离鉴定了一个新的大豆chr3基因,为研究大豆苷元的合成机理、基因调控和生产应用奠定了必要的基础。
[1] Harborne J B,Wiliams C A.Advances in flavonoid research since 1992 [J].Phytochemistry,2000,55(6):481-504.
[2] Gutierrez-Gonzalez J J,Wu X L,Gillman J D,et al.Intricate environment-modulated genetic networks control isoflavone accumulation in soybean seeds [J].BMC Plant Biology,2010,10(9):105-121.
[3] Ferrer J L,Austin M B,Stewart J C,et al.Structure and function of enzymes involved in the biosynthesis of phenylpropanoids [J].Plant Physiology and Biochemistry,2008,46(3):356-370.
[4] Welle R,Schroder G,Schillz E,et al.Induced plant responses to pathogen attack analysis and heterologous expression of the key enzyme in the biosynthesis of phytoalexins in soybean [J].European Journal of Biochemistry,1991,196(6):423-430.
[5] Liu G Y.Isolation,sequence identification and tissue expression profile of tow novel soybean(Glycinemax)genes-vestitone reductase and chalcone reductase [J].Molecular Biology Reports,2009,36(11):1991-1994.
[6] Wang X Q.Structure,function,and engineering of enzymes in isoflavonoid biosynthesis [J].Functional & Integrative Genomics,2011,11(4):13-22.
[7] Huang J X,Qu L J,Gu H Y,et al.A preliminary study on the origin and evolution of chalcone synthase (CHS) gene in angiospems [J].Acta Botanic Sinica,2004,46(1):10-19.
[8] Terrence L,Graham Madge Y,Senthil S,et al.RNAi silencing of genes for elicitation or biosynthesis of 5-deoxyisoflavonoids suppresses race-specific resistance and hypersensitive cell death in phytophthora sojae infected tissues [J].Plant Physiology,2007,144(2):728-740.
[9] Bomati E K,Austin M B,Bowman M E,et al.Structural elucidation of chalcone reductase and implication for deoxychalcone biosynthesis [J].The Journal of Biological Chemistry,2005,280(23):30496-30503.
[10] 王邦俊,王 强,张志刚,等.利用RACE技术扩增大豆抗病基因同源cDNA 5′末端序列 [J].遗传,2003,25(4):425-427.
Wang B J,Wang Q,Zhang Z G,et al.Amplification of 5′cDNA end of a disease resistance gene homolog using race system from soybean [J].Genetic,2003,25(4):425-427.(in Chinese)
[11] 闻 静,李凤兰,赵 刚,等.大豆膨胀素GmEXPA4基因的克隆及表达分析 [J].中国油料作物学报,2012,34(4):343-347.
Wen J,Li F L,Zhao G,et al.Clone and expression of expansinGmEXPA4 gene in soybean [J].Chinese Oil Crop Sciences,2012,34(4):343-347.(in Chinese)
[12] 陈华涛,陈 新,顾和平,等.大豆铁蛋白(Ferritin)基因家族的克隆与表达 [J].中国油料作物学报,2012,34(6):586-591.
Chen H T,Chen X,Gu H P,et al.Cloning and expression analysis of soybean ferritin gene family [J].Chinese Oil Crop Sciences,2012,34(6):586-591.(in Chinese)
[13] 吴向阳,仰玲玲.RP-HPLC法同时测定野葛的根茎和叶中葛根素大豆苷和大豆苷元的含量 [J].食品科学,2009,30(14):248-252.
Wu X Y,Yang L L.Simultaneous RP-HPLC determination of Puerarin,daidzin and daidzein in roots,stem and ofPuerarialobata(wild) Ohwi [J].Food Science,2009,30(14):248-252.(in Chinese)
[14] 张君旗.SDS-PAGE与HPLC检测食品中大豆蛋白的方法研究 [D].郑州:河南工业大学,2011.
Zhang J Q.The determination of soy protein in foods by SDS-PAGE and HPLC [D].Zhengzhou:Henan University of Technology,2011.(in Chinese)
[15] Dixon R,Lahoucine Achnine,Parvathi Kota,et al.The phenylpropanoid pathway and plant defense:A genomics perspective [J].Molecular Plant Pathology,2002,3(5):371-390.
[16] Kulling S E,Honig D M,Metzler M.Oxidative metabolism of the soy isoflavones daidzein and genistein in humansinvitroandinvivo[J].ACS Publications,2001,49(6):3024-3033.
[17] Morita H,Mizuuchi Y,Ade T,et al.Cloning and functional analysis of a novel aldo-keto reductase from aloe arborescens [J].Biological and Pharmaceutical Bulletin,2007,30(12):2262-2267.
[18] 马 婧.金养麦[Fagopyrumdibotrys(D.Don) Hara]类黄酮生物合成途径重要功能基因的克隆、功能验证及表达特性分析 [D].重庆:西南大学,2012.
Ma J.Cloning,function identification and expression analysis of the important functional genes involved in the flavonoids biosynthsis pathway of Golden buckwheat [Fagopyrumdibotrys(D.Don) Hara] [D].Chongqing:Southwestern University,2012.(in Chinese)
[19] Hyndman D,Bauman D B,Heredia V V,et al.The aldo-keto reductase (AKR) superfamily:An update [J].Chem Biol Interact,2003,1:143-144.
[20] Park J H,Troxel A B,Harvey R G,et al.Polycyclic aromatic hydrocarbon (PAH) o-Quinones produced by the aldo-keto-rReductases (AKRs) generate abasic sites,oxidized pyrimidines,and 8-oxo-dGuo via reactive oxygen species [J].Chem Res Toxicol,2006,19(5):719-728.
[21] Yu O,Jung W,Shi J,et al.Production of the isoflavones geni-stein and daidzein in non-legume dicot and monocot tissues [J].Plant Physiology,2000,124(2):781-794.
[22] Liu C J,Dixon R A.Elicitor-Induced association of isoflavone o-methyltransferase with endomembranes prevents the formation and 7-O-Methylation of daidzein during isoflavonoid phytoalexin biosynthesis [J].The Plant Cell,2001,13(12):2643-2658.
[23] Kim M, Han J,Kim S-U.Isoflavone daizein:Chemistry and bacterial metabolism [J].Appl Biol Chem,2008,51(6):253-261.
Cloning of soybean chalcone reductase gene (chr3) and its expression inEscherichiacoli
ZHANG Chaoa,WANG Pi-wua,ZHANG Zhuob
(aCollegeofAgriculture,bCollegeofLifeScience,JilinAgriculturalUniversity,Changchun,Jilin130118,China)
【Objective】 Soybean chalcone reductase 3 (chr3) was cloned and its coding protein catalyticinvitrowas analyzed to provide references for synthesis and regulation of daidzein.【Method】 The soybeanchr3 was amplified by RACE technology and the domain and function of its coding protein was analyzed using soybean “Jinong 17” as material.The fragment was introduced into pET28a vector,which was transformed intoE.coliBL21.Recombinant vector BL21-pET28a-chr3 was constructed and recombinant protein was verified by high performance liquid chromatography (HPLC).【Result】 The geneschr3 (GenBank accession:KF927169) with full length of 1 416 bp was cloned successfully.The recombinant vector BL21-pET28a-chr3 was constructed successfully,and HPLC showed that synthesis of isoliquiritigenin catalyzed by recombinant protein increased the content of soybean isoliquiritigenin to 33.673 μmol/g.【Conclusion】 The soybeanchr3 genes was isolated and identified,which improved catalytic activities of coding protein CHR3.
soybean;daidzein;chalcone redutase 3 gene (chr3);gene cloning;Escherichiacoli
时间:2015-08-05 08:56
10.13207/j.cnki.jnwafu.2015.09.013
2014-01-20
国家自然科学基金面上项目“大豆查尔酮还原酶(CHR)在大豆苷元合成中的作用机理”(31171568)
张 超(1988-),男,吉林长春人, 在读硕士,主要从事生物技术在作物遗传育种中的应用研究。 E-mail:312703991@qq.com
王丕武(1958-),男,吉林长春人,教授,博士,博士生导师,主要从事生物技术与作物遗传育种研究。 E-mail:peiwuw@163.com
Q78;S565.103.3
A
1671-9387(2015)09-0089-08
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20150805.0856.026.html

