耐热β-甘露聚糖酶基因的克隆与表达及酶学性质
赵 梅, 魏喜换, 王春娟, 董运海, 李剑芳*, 邬敏辰
(1.江南大学 食品学院,江苏 无锡 214122;2.江南大学 无锡医学院,江苏 无锡 214122)
β-甘露聚糖酶 (EC 3.2.1.78) 是内切 β-1,4-甘露聚糖甘露糖苷水解酶的简称,能够随机催化甘露聚糖分子主链中β-1,4-D-甘露糖苷键的水解,是甘露聚糖降解酶系中最重要的组分,属于半纤维素酶类[1-2]。它广泛存在于多种生物体内,在食品、医药、饲料、纺织、纸浆漂白及能源开发等众多领域具有重要的应用价值[2]。基于一级结构同源性比对和疏水簇分析,绝大多数β-甘露聚糖酶归属糖苷水解酶 (GH)第5或26家族[3-4]。 目前,已有许多关于GH5家族β-甘露聚糖酶的报道,但其对极端环境耐受性差和催化活性低等缺点极大地限制了其在各领域的应用[5-6],因此有关新型酶基因的开发和酶的分子改造[7-8]方面的研究逐年增多。随着GH5家族β-甘露聚糖酶研究的成熟以及GH26家族β-甘露聚糖酶基因序列的公布,研究人员逐渐转向对GH26家族β-甘露聚糖酶的研究。研究发现两个糖苷水解酶家族的活性位点作用模式不尽相同[9],许多报道也显示GH26家族β-甘露聚糖酶具有不同于GH5家族的优良特性[10-11],这为GH26家族β-甘露聚糖酶的深入研究提供了有效的参考。
作者所在课题组在前期的研究中从Aspergillus usamii E001发酵产物中分离到了电泳纯的β-甘露聚糖酶,之后克隆出了A.usamii E001 GH5家族β-甘露聚糖酶(AuMan5A)的编码基因,并在毕赤酵母GS115中实现了表达[8]。结果显示,来源于A.usamii E001发酵产物的β-甘露聚糖酶的催化活性明显高于重组酶的活性,推测电泳纯的A.usamii的β-甘露聚糖酶可能由两种或两种以上表观相对分子质量相似的酶组成。对Aspergillus niger CBS 513.88的基因组序列分析[12]发现,A.niger中存在两种β-甘露聚糖酶,分别属于GH5家族和GH26家族,且两种酶的氨基酸残基数相近。另外,在前期的研究中作者发现,A.usamii和A.niger的GH5家族β-甘露聚糖酶序列具有高度的相似[1-8]。本研究基于NCBI上A.niger CBS 513.88菌株的基因组序列,参照推测出的黑曲霉GH26家族β-甘露聚糖酶序列,借助RT-PCR技术从A.usamii E001中克隆出AuMan26A的编码基因,并在毕赤酵母GS115中实现了异源表达。同时,对重组酶reAuMan26A的酶学性质进行了初步研究,其极好的热稳定性表明该酶具有较大的应用潜力。
1 材料与方法
1.1 菌株和质粒
宇佐美曲霉A.usamii E001,大肠杆菌Escherichia coli JM109和DH5α,便于实现蛋白质天然N端表达的质粒pPIC9KM,由作者所在实验室构建和保藏;克隆质粒pUCm-T,购自上海Sangon公司;表达宿主菌毕赤酵母Pichia pastoris GS115,购自Invitrogen公司。
1.2 主要试剂和培养基
各种限制性内切酶,rTaq DNA聚合酶,T4DNA连接酶,250 bp DNA Ladder Marker,低相对分子质量蛋白质Marker和RNA PCR Kit(AMV)Ver.3.0,购自大连TaKaRa公司;胰蛋白胨,酵母提取物,Trizol,YNB,SDS,G418,UNIQ-10 柱 式 Trizol 总RNA抽提试剂盒和聚丙烯酰胺,购自上海Sangon公司;标准甘露糖和角豆胶,Sigma公司产品;其他试剂均为国产或进口分析纯。
种子活化培养基(mg/mL):玉米粉20,豆饼粉 30,KH2PO430,CaCl210,MgSO410; 诱导培养基(mg/mL):NaNO39,KH2PO40.5,MgSO40.5, 酵母提取物 4,魔芋粉 5;LB,YPD,MD,BMGY 和 BMMY 培养基的配制,参照 Multi-Copy Pichia Expression Kit(Invitrogen公司)操作手册。
1.3 引物的设计与合成
基于宇佐美曲霉与黑曲霉基因序列的相似性,参照NCBI上公布的A.niger CBS 513.88菌株基因组中推测的Man26A的基因序列 (登录号:XM_001397260.1),结合生物信息学分析手段,设计出一对扩增编码AuMan26A成熟肽cDNA的特异性上下游引物:
上游引物(AuMan26A-F):5'-CTCGAGAAAAG AGCTTCCAACCAGACTCTGTCC-3',含 XhoⅠ位点;
下游引物 (AuMan26A-R):5'-GAATTCTTAAG CCCCCTCCCAGTTCAG-3',含 EcoRⅠ位点。
1.4 AuMan26A成熟肽编码基因的克隆
将A.usamii E001接种至种子活化培养基中,于30℃、220 r/min条件下振荡培养24 h,以体积分数3%的接种量转至诱导培养基中,于上述条件下振荡培养24 h后收集菌体,用UNIQ-10柱式Trizol总RNA抽提试剂盒提取A.usamii的总RNA。以提取的A.usamii总RNA为模板,Oligo dT-Adaptor为引物,逆转录合成cDNA的第一条链。以所合成的链为模板,AuMan26A-F和M13 primer M4为引物,按如下条件进行第一轮PCR扩增:94℃预变性4 min;94℃变性 30 s,52℃退火 30 s,72℃延伸90 s,30个循环;72℃充分延伸10 min。以第一轮PCR产物为模板,AuMan26A-F和AuMan26A-R为引物,按如下条件进行第二轮PCR扩增AuMan26A成熟肽的编码基因:94℃预变性4 min;94℃变性30 s,57 ℃退火 30 s,72 ℃延伸 90 s,30 个循环;72 ℃充分延伸10 min。PCR产物经琼脂糖凝胶电泳检测、割胶回收,与pUCm-T连接,转化E.coli JM109,蓝白斑筛选阳性转化子,送上海Sangon公司测序。测序正确的重组质粒pUCm-T-Auman26A经XhoⅠ和EcoRⅠ双酶切,将目的基因克隆至能够实现天然N端表达的质粒pPIC9KM,转化E.coli DH5α,筛选重组表达质粒pPIC9KM-Auman26A并进行测序鉴定。
1.5 Auman26A基因在毕赤酵母中的表达
测序正确的pPIC9KM-Auman26A质粒经SalⅠ线性化,电转化P.pastoris GS115感受态细胞。电转化及多拷贝转化子筛选方法参见Multi-Copy Pichia Expression Kit(Invitrogen公司)操作手册。重组毕赤酵母的鉴定及诱导表达参见文献 [1],整合有pPIC9KM质粒的GS115/9KM做阴性对照。将获得的重组P.pastoris GS115在YPD平板上划线传代培养6次,每次挑选3个单菌落按标准流程进行诱导培养,然后测定发酵上清液中的β-甘露聚糖酶活性,研究重组P.pastoris GS115的遗传稳定性。
1.6 重组酶reAuMan26A的分离纯化
将摇瓶发酵液8 000 r/min离心10 min,除去菌体即得粗酶液。取40 mL粗酶液,向其中加入硫酸铵至50%饱和度,4℃过夜放置后离心去除部分杂蛋白质和色素。在上清液中继续加入硫酸铵至80%饱和度,4℃过夜放置,离心收集沉淀。将所得沉淀溶于40 mL的柠檬酸-Na2HPO4缓冲液(20 mmol/L、pH 5.5),经透析,超滤浓缩(截留相对分子质量为10 kDa)至3 mL。上样到预先用上述缓冲液平衡好的Sephadex G-75凝胶层析柱(Φ 1.6 cm×100 cm),然后同一缓冲液淋洗 (洗脱体积流量0.3 mL/min),收集有酶活力的组分。
1.7 β-甘露聚糖酶活性的测定
采用改良的DNS法测定β-甘露聚糖酶活性[1]。取2.4 mL 5 mg/mL角豆胶溶液 (pH 5.5的柠檬酸-Na2HPO4缓冲液配制),于40℃预热5 min后加入0.1 mL适当稀释的酶液,40℃下准确反应10 min,加入2.5 mL DNS试剂,沸水浴中显色7 min,加入5 mL去离子水混匀后测定OD540。在上述反应条件下,每分钟产生1 μmol还原糖(以甘露糖计)所需的酶量定义为1个酶活性单位(U)。
1.8 reAuMan26A酶学性质的分析
1.8.1 温度对酶活性的影响 在不同温度 (30~60℃)下,按1.7的方法测定reAuMan26A的活性。最适温度Topt定义为最高酶活性 (以相对活性100%计)所对应的温度。将酶液置于不同温度保温0~60 min,按1.7中的方法测定残余酶活性,未处理酶液(0 min)的酶活性以100%计。酶的半衰期t1/2定义为残余酶活性为50%时的保温时间。
1.8.2 pH对酶活性的影响 用不同pH值(pH 2.5~8.0)的100 mmol/L柠檬酸-Na2HPO4缓冲液配制5 mg/mL角豆胶溶液,按1.7中的方法测定reAuMan26A的活性。最适pH定义为最高酶活性所对应的pH值。将酶液在不同pH值 (pH 2.5~8.0柠檬酸-Na2HPO4缓冲液)、40℃保温60 min,按1.7中的方法测定残余酶活性。酶的pH稳定性定义为残余酶活性在85%以上的pH范围。
1.8.3 金属离子和EDTA对酶活性的影响 将酶液与不同金属离子或EDTA溶液(终浓度2.0 mmol/L)混匀后于40℃保温1 h,按1.7中的方法测定残余酶活性。以未加金属离子和EDTA的酶活性计为100%。
1.8.4 reAuMan26A的底物谱及动力学参数的测定用 100 mmol/L、pH 5.5柠檬酸-Na2HPO4缓冲液分别配制质量浓度为5 mg/mL的角豆胶、瓜尔豆胶、魔芋粉、桦木木聚糖、羧甲基纤维素钠(CMC-Na)和可溶性淀粉溶液,按照1.7中的方法测定reAuMan26A对不同底物的活性。以不同质量浓度(1~10 mg/mL)的角豆胶溶液为底物,按照1.7的方法测定reAuMan26A的活性,采用Origin 8.0软件进行非线性拟合,计算出reAuMan26A的Km和Vmax值。
2 结果与讨论
2.1 Auman26A基因的克隆
按1.4的方法进行PCR反应,经质量分数1%琼脂糖凝胶电泳检测第一轮PCR产物,在1 200 bp处有一条明显的条带 (图1泳道1),将目的产物割胶回收纯化。以第一轮PCR产物为模板,进行第二轮PCR扩增。结果显示PCR产物在1 000 bp处有一条特异性的条带 (图1泳道2),PCR产物长度与预期相符,割胶回收产物连接至pUCm-T,获得重组质粒pUCm-T-Auman26A,经蓝白斑筛选和菌液PCR检测后进行测序。测序结果显示获得的基因片段为954 bp,编码317个氨基酸,Blast分析表明该基因序列与 A.niger CBS 513.88(XM_00139760.1)、Aspergillus flavus NRRL3357 (XM_002377295.1)和Aspergillus oryzae RIB40(XM_001825696.2)序列同源性分别为 98%、84%和 83%。将 pUCm-TAuman26A和pPIC9KM经XhoⅠ和EcoRⅠ双酶切及回收纯化后,相互连接得到重组表达质粒pPIC9KM-Auman26A并进行测序鉴定。测序结果表明目的基因没有发生突变,且读码框完全正确。
2.2 Auman26A基因在毕赤酵母中的表达
提取pPIC9KM-Auman26A质粒,经SalⅠ线性化后,电转P.pastoris GS115感受态细胞,经MD平板和含不同浓度G418的YPD平板筛选后获得高拷贝的GS115/Auman26A。随机挑选几个高拷贝的GS115/Auman26A重组子用引物5'-AOX和3'-AOX进行菌液PCR检测,结果如图2所示。可见,以GS115/Auman26A为模板的PCR反应能扩增出长度约为2 100 bp和1 500 bp的产物,2 100 bp的产物为P.pastoris GS115本身的AOX1基因,1 500 bp的产物包括约为1 000 bp的目的基因和500 bp pPIC9KM质粒上5′-AOX和3′-AOX引物序列之间的片段。结果表明,Auman26A基因已成功整合入P.pastoris GS115基因组中。
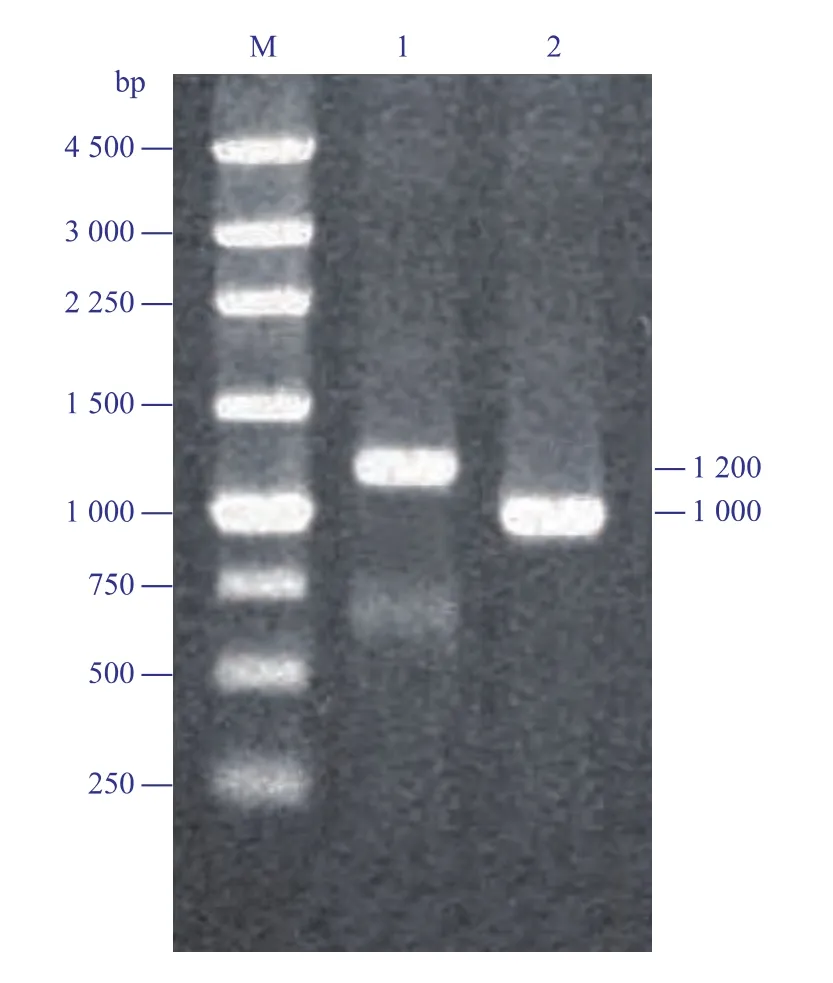
图1 Auman26A的PCR扩增Fig.1 PCR amplifications for Auman26A
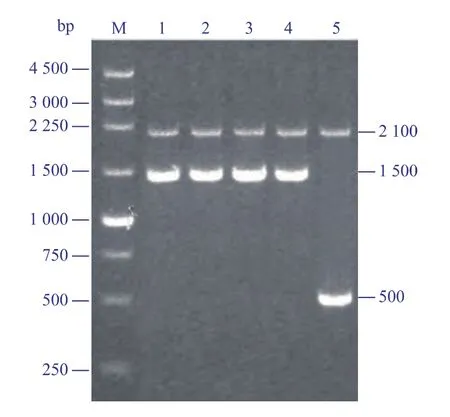
图2 重组毕赤酵母GS115/Auman26A的PCR检测Fig.2 PCR detections of recombinant P.pastoris GS115/Auman26A
挑取几个高拷贝的重组子按照Multi-Copy Pichia Expression Kit手册中的方法诱导表达,筛选到一个产酶活性最高的GS115/Auman26A重组子,其发酵上清液的酶活为281.9 U/mL,而GS115/9KM的发酵上清液中未检测到β-甘露聚糖酶的活性。SDS-PAGE的结果显示,reAuMan26A的表观相对分子质量约为48.3 kDa(图 3);而 GS115/9KM的发酵上清液在48.3 kDa处无特异性条带,与酶活性测定结果一致。遗传稳定性研究试验表明,P.pastoris GS115/Auman26A具有良好的遗传稳定性,经6次传代后表达水平无明显改变。
将A.usamii菌株诱导表达上清液分别经(NH4)2SO4盐析、透析、超滤和 Sephadex G-75 凝胶进行分离纯化。SDS-PAGE电泳分析显示,纯化后的酶呈现单一条带(图3),测得比酶活为2281.7U/mg。

图3 重组毕赤酵母表达上清液的SDS-PAGE分析Fig.3 SDS-PAGE analysis of the expressed supernatant of the recombinant P.Pastoris
2.3 reAuMan26A的酶学性质
按照1.7中的方法测定reAuMan26A的最适反应温度Topt,结果如图4(a)所示。由图可见,reAuMan26A在35~45℃范围内有较高的催化活性,其 Topt为 40℃。由图 4(b)可知,reAuMan26A 具有很好的热稳定性,80℃保温60 min可保留60%的酶活性;在90℃的半衰期t1/290为10 min,保温60 min仍可保留33%的酶活性。可见,该重组酶在高温下可长时间保存,恢复到40℃时仍可保留较高活性。不同来源的β-甘露聚糖酶在温度特性方面有着显著的差异,如源自海栖热袍菌(Thermotoga maritima)β-甘露聚糖酶的最适温度为95℃,85℃处理后酶活力保留50%以上[13];Songsiriritthigul等人[11]所研究的地衣芽胞杆菌 (Bacillus licheniformis)GH26 β-甘露聚糖酶重组酶的最适反应温度为50℃,50℃半衰期为80 h。而本研究中的reAuMan26A在温度特性上表现出不同于其它酶的特点,其最适反应温度为40℃,低于已报道过的β-甘露聚糖酶的值,而热稳定性却明显高于其它酶[1-7]。据报道,酶在一定温度范围内升温时的失活是可逆的,温度降低后酶活性就恢复正常。大多数酶在40℃以上催化活力趋于下降,60~70℃呈现可逆失活,70~80℃出现不可逆变性[14]。由图4可知,reAuMan26A在50℃时就开始出现可逆变性,90℃热处理1 h冷却后仍保留较高酶活性,可推测该酶经过90℃热处理恢复到40℃时发生了复性。从侧面说明,reAuMan26A的酶分子结构不同于之前报道过的其它酶,这为进一步探讨reAuMan26A的分子结构特点提供了线索。
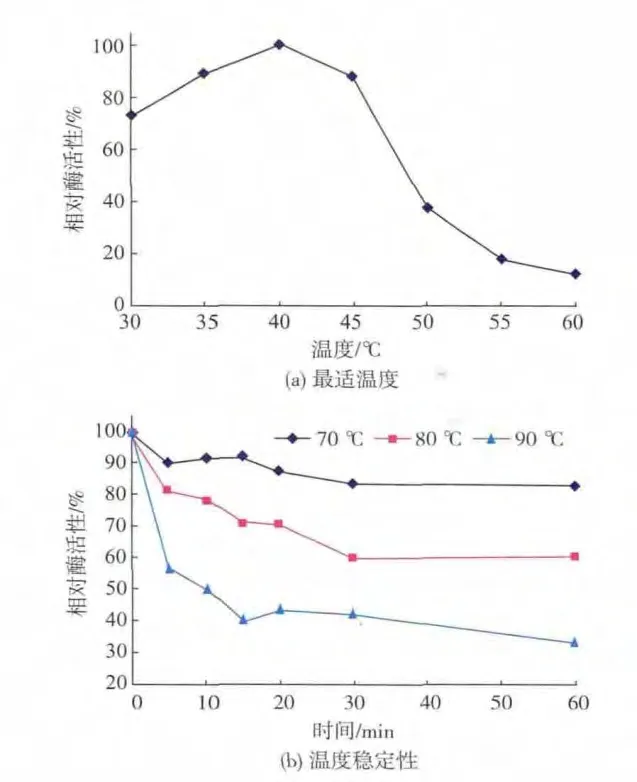
图4 reAuMan26A的最适温度及温度稳定性Fig.4 Optimal temperature (a) and thermostability (b)of reAuMan26A
reAuMan26A的最适反应pH和pH稳定性的研究结果如图5所示。可知,reAuMan26A在pH 3.5~5.5的范围内催化活性较高,其中最适pH为5.5;当pH低于3.5和高于5.5时,reAuMan26A的催化活性迅速下降。如图所见,reAuMan26A在pH 5.0~7.0的范围内具有较高的稳定性;当pH低于4.0时,酶活性丧失较快。细菌来源的甘露聚糖酶最适反应pH 多为 5.5~7.0,真菌来源的多为 3.0~5.5[2],该酶的最适pH与已有文献报道一致。

图5 reAuMan26A的最适pH及pH稳定性Fig.5 Optimal pH and stability of reAuMan26A
金属离子和EDTA对reAuMan26A酶活性的影响如图6所示。可见,不同的金属离子和EDTA对酶活性影响不明显,只有EDTA对reAuMan26A酶活性有轻微激活作用;而其他金属离子对reAuMan26A酶活性有一定的抑制作用,其中Fe2+和Fe3+的抑制作用比较明显。
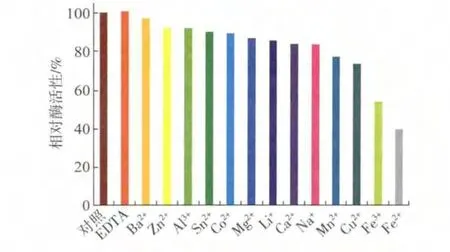
图6 金属离子和EDTA对reAuMan26A酶活性的影响Fig.6 Effects of metal ions and EDTA on the activity of reAuMan26A
reAuMan26A底物谱分析的结果显示,reAuMan26A对角豆胶的催化活性最高,将其对角豆胶的催化活性定为100%;而对魔芋粉和瓜尔豆胶的催化活性较低,相对酶活性分别为56.4%和28.5%。另外,reAuMan26A对可溶性淀粉、桦木木聚糖和羧甲基纤维素钠没有催化活性。由以上数据可知,该酶仅识别并催化水解含有甘露糖结构的多聚糖,更加表明其倾向于专一性水解主链中的β-1,4-糖苷键,侧链较多的瓜尔豆胶不易被催化水解。reAuMan26A对于角豆胶的Km和Vmax值分别为15.25 mg/mL和 7 841.9 U/mg。
3 结语
近年来,关于β-甘露聚糖酶的研究报道逐年增多。相对于GH5家族β-甘露聚糖酶,有关GH26家族β-甘露聚糖酶的研究较少。在少数的关于GH26家族β-甘露聚糖酶研究的报道中,均显示出其优良的酶学特性[10-11]。作者在前期研究的基础上,根据A.usamii与A.niger基因序列的相似性,通过生物信息学和基因组学分析,成功实现了AuMan26A编码基因的克隆表达。传统的通过基因或cDNA文库开发新基因的方法工作量较大,而且不易操作[15]。随着生物信息学技术的发展,挖掘新基因的手段也越来越多[16]。自然界微生物中具有丰富的酶资源,利用生物信息学和基因组学相结合的方法从微生物中挖掘具有优良性状的新型酶是一条获取优良酶的有效途径。
本研究中采用RT-PCR技术成功克隆了AuMan26A的编码基因并在毕赤酵母GS115中实现了异源表达,重组β-甘露聚糖酶 (reAuMan26A)的发酵上清液对角豆胶的酶活性为281.9 U/mL,表达水平明显高于GH5家族的A.usamii和A.niger LW-1 β-甘露聚糖酶 AnMan5A[1]和 AuMan5A[17]。 初步的酶学性质研究表明,该酶的最适反应温度Topt为40℃;在80℃处理60 min后残留酶活性为60%;90℃时的半衰期t1/290为10 min,处理60 min后残留酶活性为33%;其最适pH为5.5,在pH 5.0~7.0的范围内较稳定;Fe2+和Fe3+对其有明显的抑制作用,其它所测金属离子和EDTA对其没有明显的影响;所测酶的最适底物为角豆胶,其对角豆胶的Km和Vmax值分别为15.25 mg/mL和 7 841.9 U/mg。本研究成果为GH26家族β-甘露聚糖酶的深入研究奠定了理论基础,也为其它新酶基因的开发和进一步应用提供了新的思路和策略。
[1]Li J F,Zhao S G,Tang C D,et al.Cloning and functional expression of an acidophilic β-mannanase gene (Anman5A) from Aspergillus niger LW-1 in Pichia pastoris[J].Journal of Agricultural and Food Chemistry,2012,60(3):765-773.
[2]Moreira L R S,Filho E X F.An overview of mannan structure and mannan-degrading enzyme systems[J].Applied Microbiology Biotechnology,2008,79(2):165-178.
[3]Cantarel B L,Coutinho P M,Rancurel C,et al.The Carbohydrate-Active EnZymes database (CAZy):an expert resource for Glycogenomics[J].Nucleic Acids Research,2009,37(Database):233-238.
[4]Henrissat B,Bairoch A.New families in the classification of glycosyl hydrolases based on amino acid sequence similarities[J].The Biochemical Journal,1993,293(3):781-788.
[5]Chen X L,Cao Y H,Ding Y H,et al.Cloning,functional expression and characterization of Aspergillus sulphureus β-mannanase in Pichia pastoris[J].Journal of Biotechnology,2007,128(3):452-461.
[6]Tang C D,Guo J,Wu M C,et al.Cloning and bioinformatics analysis of a novel acidophilic β-mannanase gene,Auman5A,from Aspergillus usamii YL-01-78[J].World Journal of Microbiology and Biotechnology,2011,27(12):2921-2929.
[7]Luo H Y,Wang Y R,Wang H,et al.A novel highly acidic β-mannanase from the acidophilic fungus Bispora sp.MEY-1:gene cloning and overexpression in Pichia pastoris[J].Applied Microbiology and Biotechnology,2009,82(3):453-461.
[8]Hekmat O,Leggio L L,Rosengren A,et al.Rational engineering of mannosyl binding in the distal glycone subsites of Cellulomonas fimi endo-β -1,4-mannanase:mannosyl binding promoted at subsite-2 and demoted at subsite-3[J].Biochemistry,2010,49(23):4884-4896.
[9]Couturier M,Roussel A,Rosengren A,et al.Structural and biochemical analyses of glycoside hydrolase families 5 and 26 beta-(1,4) -mannanases from Podospora anserina reveal differences upon manno-oligosaccharide catalysis[J].The Journal of Biological Chemistry,2013,288(20):14624-14635.
[10]Politz O,Krah M,Thomsen K K,et al.A highly thermostable endo-(1,4) -β -mannanase from the marine bacterium Rhodothermus marinus[J].Applied Microbiology and Biotechnology,2000,53(6):715-721.
[11]Songsiriritthigul C,Buranabanyat B,Haltrich D,et al.Efficient recombinant expression and secretion of a thermostable GH26 mannan endo-1,4-β-mannosidase from Bacillus licheniformis in Escherichia coli[J].Microbial Cell Factories,2010,9(1):20-34.
[12]Pel H J,de Winde J H,Archer D B,et al.Genome sequencing and analysis of the versatile cell factory Aspergillus niger CBS 513.88[J].Nature Biotechnology,2007,25(2):221-231.
[13]张敏,江正强,李里特.重组海栖热袍菌极耐热甘露聚糖酶的纯化和性质研究[J].微生物学通报,2008,35(10):1565-1571.ZHANG Min,JIANG Zhengqiang,Li Lite.Purification and characterization of a recombinant thermostable β-mannanase from Thermotaga maritima[J].Microbiology,2008,35(10):1565-1571.(in Chinase)
[14]陈清西.酶学及其研究技术[M].厦门:厦门大学出版社,2010:138.
[15]Wang J Q,Zhang H M,Wu M C,et al.Cloning and sequence analysis of a novel xylanase gene,Auxyn10A,from Aspergillus usamii[J].Biotechnology Letters,2011,33(5):1029-1038.
[16]Shi H L,Yin X,Wu M C,et al.Cloning and bioinformatics analysis of an endoglucanase gene (Aucel12A) from Aspergillus usamii and its functional expression in Pichia pastoris[J].Journal of Industrial Microbiology and Biotechnology,2012,39(2):347-357.
[17]Tang C D,Li J F,Wei X H,et al.Fusing a carbohydrate-binding module into the Aspergillus usamii β-mannanase to improve its thermostability and cellulose-binding capacity by in silico design[J].Plos One,2013,8(5):e64766.

