水稻OsKAT2 基因的克隆及表达分析
高 南, 李俊林,2, 郝东利
(1.中国科学院南京土壤研究所土壤与农业可持续发展国家重点实验室,江苏 南京210008;2.南京林业大学森林资源与环境学院,江苏 南京210037)
水稻生产过程中时常遇到季节性和区域性的干旱、高盐等非生物逆境胁迫,严重影响水稻的产量和品质[1-3]。转基因是提高水稻抗非生物逆境能力的主要技术措施之一[1]。筛选合适的基因,是进行转基因水稻培育的前提。Shaker家族K+通道成员在植物体内的增强表达能提高植物的抗逆性,例如将水稻的K+通道基因OsKAT1转入水稻愈伤组织,转基因水稻愈伤组织的抗盐胁迫能力明显提高[4-5]。因此,Shaker家族K+通道成员是重要的候选基因。Shaker家族成员具有各自独立的 表 达 模 式[6-8]。拟 南 芥[Arabidopsis thaliana(L.)Heynh.]的KAT1基因在叶保卫细胞中表达,KAT2基因在叶保卫细胞、叶韧皮部和花中表达[6];玉米(Zea maysL.)的ZMK2.1基因和烟草(Nicotiana tabacumL.)的NKT6基因主要在叶中表达[7-8]。与无NaCl 胁迫的处理相比,NaCl 胁迫处理强烈抑制了拟南芥根中AKT1的表达量,但是没有改变地上部AKT1的表达量和拟南芥AKT2和SKOR的表达量[9]。在干旱胁迫下,烟草(Nicotiana tabacumL.)叶中NKT6的表达量低于对照[8]。在外源ABA 的胁迫下,KAT1、KAT2和NKT6在叶中的表达量降低[8,10]。水稻基因组测序已经完成,其K+通道的研究较少[4-5,11-12]。水稻生长在水中,可能具有其他旱地植物不具有的特性。因此,挖掘水稻自身的基因,研究其在胁迫下的响应特征,对进行水稻抗逆改良有重要意义。本研究通过电子克隆、生物信息学分析和实时荧光定量PCR(Real-time fluorescence quantitative PCR, RTqPCR)分析的方法,克隆水稻抵抗逆境胁迫的相关基因并进行分析。
1 材料与方法
1.1 材料
水稻(Orazy sativaL.cv Nipponbare)种子由中国科学院南京土壤研究所土壤-植物营养与肥料室保存。Trizol RNA 提取试剂盒(RNAisoTMPlus)、MMLV 反转录酶、DNase I、TaqDNA 聚合酶、DL2000 DNA Marker、大肠杆菌DH5α、pMD-18T 载体、质粒小提试剂盒、限制性内切酶和荧光定量PCR 用试剂盒购自宝生物(大连)有限公司(TaKaRa Co.Ltd.,Dalian,China)。胰蛋白胨和酵母提取物购自Oxoid公司(Oxoid Ltd,Basingstoke,Hampshire,England)。脱落酸(ABA)、甘露醇(Mannitol)和聚乙二醇-6000(PEG-6000)购自Sigma 公司(Sigma-Aldrich Co.LLC.,St.Louis,USA)。其他化学试剂均为国产分析纯。
NCBI(美国国立生物技术信息中心)中Gen-Bank 数据库中已经注册的蛋白质序列:拟南芥的AKT1(NP_180233.1)、KAT1(NP_199436.1)、KAT2(NP_193563.3)、AKT2/3(NP_567651.1)、AtKC1(P92960.1)KAT3(NP_194991.1)、AKT5(NP_194976.1)、AKT6(NP_180131.3)、GORK(NP_198566.2)、SKOR(NP_186934.1)、KCO1(Q8LBL1.2)、KCO2(CAB64717.1)、KCO3(CAB40380.1)、KCO4(Q9FWX6.2)、KCO5(CAB62162.1)和KCO6(AEE84005.1);玉米的KZM1(NP_001105240.1)、KZM2 (NP _ 001105161.1)和 ZMK2.1 (NP _001105240.1);烟草的NKT1(BAD81034.1)、NKT2(BAD81033.1)、NKT2 (BAD81033.1)、NKT3(ACI45550.2)和NKT5(ACJ05642.2);普通小麦(Triticum aestivumL.)的TaAKT1(AAF36832.1);雨树[Samanea saman(Jacq.) Merr.]的 SPICK1(AAD16278.1)和SPICK2(AAD39492.1);胡萝卜(Daucus carotaL.)的KDC1(CAB62555.1);水稻的OsKAT1(NP_001044290.1)。
1.2 方法
1.2.1 水稻幼苗培养和处理 水稻种子用自来水洗净,置于1%(质量体积比)的NaClO 溶液中剧烈振荡20 min,再用蒸馏水清洗6 ~8 次,最后在蒸馏水中吸胀24 h。将表面灭菌的水稻种子均匀铺在尼龙网上,放到人工气候箱中黑暗条件下萌发3 d,每天更换蒸馏水。将发芽的种子转到改良的木村营养液[13]中,继续在人工气候箱中生长。培养条件条件:光照16 h,黑暗8 h,温度为28 ℃,相对湿度为70%。改良后的木村营养液配方为0.36 mmol/L KCl,0.36 mmol/L (NH4)2SO4,0.18 mmol/L NaH2PO4,0.55 mmol/L MgSO4,0.36 mmol/L Ca(NO3)2,0.18 mmol/L NaNO3,0.50 μmol/L H2SiO3,30.00 μmol/L Na2Fe-EDTA,46.26 μmol/L H3BO3,9.15 μmol/L MnCl2,0.76 μmol/L ZnSO4,0.32 μmol/L CuSO4,0.32 μmol/L Na2MoO4,pH5.6。营养液每3 d 更换一次。选取长势一致健壮的14 d苗龄水稻幼苗,移至改良后的木村营养液中(对照)及分别含100 mmol/L NaCl、10 % (质量体积比)PEG-6000 和20 μmol/L ABA 的上述改良木村营养液中进行胁迫处理。处理3 h、6 h、24 h 和48 h 后,收获地上部或根部,迅速放入液氮中速冻,然后置于-80 ℃冰箱中保存。
1.2.2 水稻总RNA 的提取和cDNA 第一链的合成
取冻存的水稻样品,液氮中研磨成细末,取约100 mg 细末加入1 ml Trizol,然后按说明书操作步骤进行抽提。为除去基因组DNA 污染,得到的RNA 采用DNase I 于37 ℃酶解30 min,氯仿抽提,100 %乙醇沉淀。得到的沉淀用70 %乙醇洗涤后晾干,溶解于40 μl DEPC(焦炭酸二乙酯)水中。RNA 经电泳检测、紫外检测合格后,按说明书操作步骤用反转录酶M-MLV 反转录为cDNA。
1.2.3OsKAT2基因cDNA 序列的获得 在水稻数据库(Rice genome annotation project,RGAP)中,以拟南芥KAT1基因蛋白质序列为查询探针,进行BlastP 比对(http://rice.plantbiology.msu.edu/analyses_search_blast.shtml),得到1 条可能的钾离子通道序列(LOC_Os01g11250)。根据其CDS 两端序列,设计上游引物F1 (5'-ATGGCAGCTAGAAGCGAGCTTCTC-3')和下游引物R1(5'-CTAAAGATGACCGCTGACAAAAGCC-3')。 以地上部cDNA 为模板,进行50 μl 体系的PCR 扩增。反应条件为:第一步,94 ℃变性5 min;第二步,94 ℃变性1 min,55 ℃退火1 min,72 ℃延伸3 min,重复35 个循环;第三步,72 ℃延伸7 min。得到的PCR产物在质量浓度为1.0%的琼脂糖凝胶中电泳分离,回收目的片段并连接到pMD18 克隆载体,将连接产物转化大肠杆菌DH5α,筛选阳性克隆并送宝生物(大连)有限公司(TaKaRa Co.Ltd.,Dalian,China)测序。
1.2.4OsKAT2基因的生物信息学分析 用DNAMAN8.0 分析测序得到的cDNA 开放阅读框并推测其编码的氨基酸序列。用ProtParam (http://web.expasy.org/protparam/)在线分析Os-KAT2 蛋白质的理化性质。使用TMHMM (http://www.cbs.dtu.dk/services/TMHMM/)在 线分析蛋白质跨膜结构。使用ProtScale (http://web.expasy.org/protscale/)在线分析亲水性/疏水性预测。使用WoLF PSORT (http://wolfpsort.org/)在线分析蛋白质序列上潜在的结构域和功能位点。使用MotifScan (http://myhits.isb-sib.ch/cgi-bin/motif_scan)和CCD (http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)在 线 分析预测蛋白质亚细胞定位。使用NetPhos (http://www.cbs.dtu.dk/services/NetPhos/)在线分析蛋白质磷酸化位点。使用SignalP4.1 (http://www.cbs.dtu.dk/services/SignalP/)在线分析蛋白质信号序列。使用SOPMA (http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page = npsa_sopma.html)在线分析蛋白质二级结构。使用Phyre2(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id =index)在线预测蛋白质三级结构。利用MEGA6.05 软件,采用Neighbor-joining法构建系统进化树。
1.2.5 实时荧光定量PCR 分析 应用RT-qPCR技术分析OsKAT2在水稻不同部位(根部和地上部)和不同胁迫处理下的表达水平,每个处理3 个重复。目的基因所用上游引物为F3(5'-GAAAACAGCGATGACAGACGAGT-3'),下游引物为R3(5'-AAGCCATAGAATGACCATTATTAGC-3')。 用看家基因OsActin基因作为内标,上游引物为Os-ActinF464(5'-CATGTTTGAGACCTTCAACACCCCTGCTA-3'),下 游 引 物 为 OsActinR933 (5'-ATGTAGTCTCATGGATACCCGCAGCTT-3')。按试剂盒说明书进行(TaKaRa SYBR® PremixEx TaqTM)操作和数据分析。
2 结果
2.1 OsKAT2 基因的获得和序列分析
2.1.1 cDNA 序列分析 利用引物F1 和R1 进行cDNA 编码区序列的RT-PCR 扩增。测序结果显示,OsKAT2基因的ORF 区为1 806 bp。推导可知Os-KAT2 蛋白质含601 个氨基酸残基。除比RGAP 中推测的ORF 序列(1 707 bp)多104 bp(图1)外,其余碱基两者相同。
2.1.2 OsKAT2 蛋白质理化性质分析 用Prot-Param 软件分析,OsKAT2 蛋白质中含量较高的氨基酸残基包括亮氨酸残基(11.6%)、异亮氨酸残基(8.2%)和精氨酸残基(6.5%),含量较低的有色氨酸残基(1.7%)、组氨酸残基(2.5%)和蛋氨酸残基(2.5%)等。带负电荷的氨基酸(天冬氨酸+谷氨酸)残基为61 个,带正电荷的氨基酸(精氨酸+赖氨酸)残基为68 个。OsKAT2 蛋白质共有9 854个原子,分子量为6 980 639,蛋白质的分子式为C3164H4938N846O876S30,理论等电点为8.57。在哺乳动物的网织红细胞、酵母和大肠杆菌中的半衰期分别为30 h(体外)、>20 h(体内)和>10 h(体内);该蛋白质不稳定指数为37.22,为稳定蛋白质。
2.1.3 OsKAT2 蛋白质的亲水性/疏水性和亚细胞定位 用ProtScale 预测OsKAT2 蛋白质亲水性/疏水性。结果(图2)显示,氨基酸的最低分值为-3.011,最高分值为2.756。从整体来看,亲水氨基酸残基分布在整条肽链,且多于疏水性氨基酸残基。使用TMHMM 进行预测,参数设置为默认值,OsKAT2 蛋白质为跨膜蛋白质。WolfPsort 预测该蛋白质可能在细胞质膜上。

图1 水稻数据库(RGAP)中推测的OsKAT2 序列与RT-PCR 获得的OsKAT2 序列比对Fig.1 Alignment of OsKAT2 sequences achieved by rice genome annotation and RT-PCR

图2 OsAKT2 蛋白质的亲水性/疏水性预测Fig.2 Prediction of hydrophilicity /hydrophobicity of protein OsKAT2
2.1.4 OsKAT2 蛋白质结构域和功能位点预测用MotifScan 和CCD 预测OsKAT2 蛋白质序列上潜在的结构域和功能位点。结果表明,该蛋白质可能存在的翻译后修饰位点包括4 个N-糖基化位点、2个蛋白激酶磷酸化位点、3个酪蛋白激酶II位点、6个N-酰基化位点和7 个蛋白激酶C 磷酸化位点;发现有5 种结构域(图3),分别是环腺苷一磷酸结合结构域(Crp,CO G0664)、离子转运结构域(Ion_trans_2,pfam07885)、离子转运结构域(Ion_trans,pfam00520)、未知功能的DUF3354 超级家族结构域(DUF3354,pfam11834)和环核苷酸结合结构域(CAP_ED,cd00038)。该蛋白质具有Shaker基因家族的典型特征,即在蛋白质的N 末端包括6 个跨膜区(S1 ~S6),有电压感受区和高度保守的通道孔区。用磷酸化位点预测程序NetPhos 2.0 进行预测,发现OsKAT2 有15 个潜在的丝氨酸磷酸化位点、7个潜在的苏氨酸磷酸化位点和4 个潜在的酪氨酸磷酸化位点。利用SignalP 4.1 Server 在线分析Os-KAT2 蛋白质信号肽情况,结果显示,该蛋白质不含信号肽序列。推测OsKAT2 属于Shaker家族,可能具有吸收K+的功能。

图3 OsKAT2 蛋白质的结构域预测Fig.3 Prediction of conserved domain of protein OsKAT2
2.1.5 OsKAT2 蛋白质二级结构和三级结构分析
用SOPMA 软件预测OsKAT2 蛋白质的二级结构。结果显示,OsKAT2 蛋白质由54.08% 的α-螺旋、15.64%的折叠延伸、3.3%的β-转角和26.96%的无规则卷曲构成。折叠延伸和的β-转角不均匀地分布在整个蛋白质的多肽链上。依据模板c2ptmA,采用折叠识别法用Phyer 在线软件预测OsKAT2 蛋白质序列主链原子位置生成预测蛋白质三级结构模型(图4),最后依据能量最小化原理使侧链集团处于能量最小的位置。

图4 预测的OsKAT2 蛋白质三级结构Fig.4 Prediction of 3D structure of protein OsKAT2
2.1.6OsKAT2编码基因的系统进化分析 搜索与水稻K+通道蛋白质OsKAT2 同源的拟南芥及相关植物蛋白质序列,运用MEGA6.05 软件绘制构建系统进化树。结果(图5)显示,构建的系统进化树可分为6 组,其中I、II、III、IV、V 组为Shaker家族的K+通道蛋白质,第VI 组为KCO 家族的K+通道蛋白质,OsKAT2 位于第II 组中。从图5 可以看出,水稻OsKAT2 在进化上与单子叶植物玉米的KZM2 和ZMK2.1 的进化关系最近。可以推测,OsKAT2 可能是Shaker家族的内向整流蛋白质。
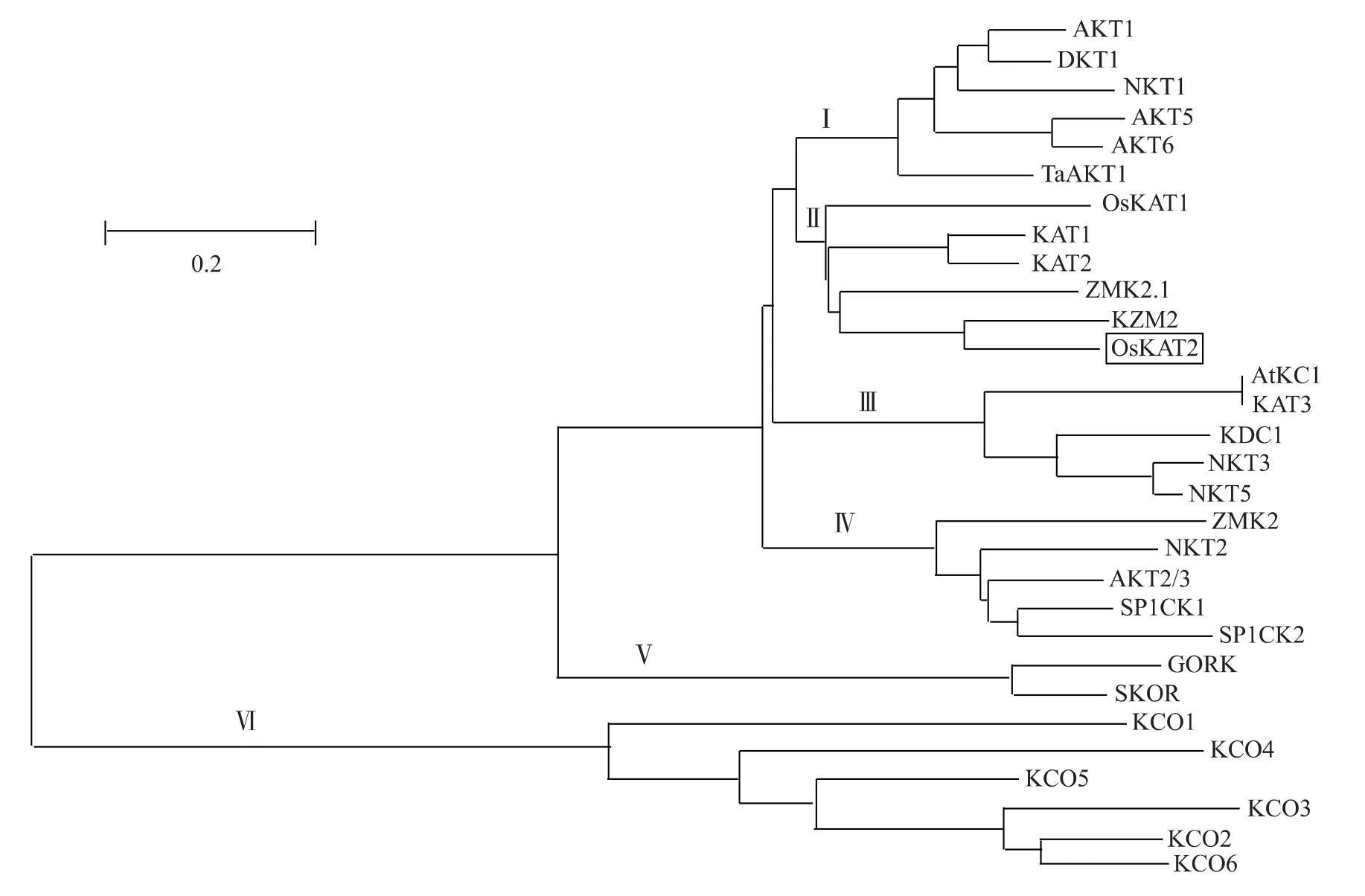
图5 部分植物钾离子通道的系统进化树Fig.5 Phylogenetic tree of potassium channels of some plants
2.2 OsKAT2 基因的表达特点
采用RT-qPCR 对水稻OsKAT2基因在非胁迫下的表达部位进行分析。结果(图6)表明,OsKAT2基因地上部的表达量比根的表达量多近10 倍。因此,在分析干旱、高盐和ABA 胁迫对水稻OsKAT2基因表达的影响时,以地上部为研究部位。由图7 可见,在正常对照条件下,水稻地上部OsKAT2基因有表达,随着培养时间的延长,其表达的丰度也有增减。在PEG 模拟干旱、NaCl 模拟高盐和ABA 的胁迫下,水稻地上部OsKAT2基因表达量明显下调,且不同处理响应的程度不同。在PEG-6000 处理3 h 时,水稻地上部OsKAT2基因相对表达量明显降低;当处理24 h 时,水稻地上部OsKAT2基因相对表达量降低最大,可达68.6%;当处理48 h 时,水稻地上部OsKAT2基因相对表达量降低略有下降,仍然可以达到36.2%。在NaCl 处理6 h 时,水稻地上部Os-KAT2基因相对表达量减少62.9%;当处理24 h 时,水稻地上部OsKAT2基因相对表达量降低最大,可达72.7%%;当处理48 h 时,水稻地上部OsKAT2基因相对表达量降低达到61.2%。在ABA 处理3 h时,水稻地上部OsKAT2基因相对表达量减少68.4%;当处理6 h 时,水稻地上部OsKAT2基因相对表达量的降低最大,可达73.3%;当处理24 h 时,水稻地上部OsKAT2基因相对表达量降低略有减少。总体上,水稻地上部OsKAT2基因在ABA 胁迫时下调表达最明显。

图6 OsKAT2 基因在水稻不同器官中的表达Fig.6 Expression levels of OsKAT2 in different organs of rice

图7 水稻地上部OsKAT2 基因在非生物胁迫下的诱导表达Fig.7 Inducible expression of OsKAT2 in rice shoot under abiotic stresses
3 讨论
同源克隆在获得植物新基因中有广泛的应用[8,14-15]。本研究通过电子克隆的方法,获得了一个可能的K+通道基因。应用生物信息学的方法和工具对OsKAT2基因进行了分析,发现OsKAT2 蛋白质为疏水性蛋白质,α-螺旋和折叠延伸是主要的构成原件,这些与大多数KAT1 类K+通道蛋白质的特征是一致的。OsKAT2 蛋白质含离子转运结构域(pfam07885 和pfam00520),功能分析结果说明该蛋白质属于阳离子转运蛋白质。OsKAT2 蛋白质与KZM2 和ZMK2.1 的同源性较高,含K+通道的标志性序列TxxTxGYGD[8,16-17]。OsKAT2 不存在锚定蛋白质结构域,这与大部分Shaker家族第II 组成员在C-末端胞内结构上存在一致性,说明OsKAT2 可能不参与通道蛋白质亚基的异聚化作用,而在与其他蛋白质的互作或细胞膜锚定的过程中发挥作用[17]。上述结果说明OsKAT2基因是一个K+通道基因。然而,来源不同的同源通道往往有其独特之处[7]。水稻长期生长在淹水环境中,其K+通道在长期适应物种生境的进化过程中,可能形成了有别于拟南芥和玉米等旱生植物的独特功能特征和作用机制[12]。OsKAT2的功能还需要进一步研究。
植物在适应非生物逆境胁迫过程中,许多基因的表达会发生变化,由此导致体内一些生物化学与生物物理过程的适应性调整,如离子含量的增加,脯氨酸的积累等,进而抗非生物逆境的能力增强[4-5]。其中,部分响应非生物逆境胁迫的基因能提高作物抵抗非生物逆境胁迫的能力。在本研究中,利用RT-qPCR 测定了OsKAT2在模拟干旱(PEG 处理)、高盐和外源ABA 胁迫下表达量的变化。结果表明,OsKAT2主要在地上部表达,受模拟干旱、高盐和外源ABA 胁迫诱导下调。ZMK2.1、KAT2等基因与OsKAT2高度同源,基因的表达部位主要在地上部[6-7],与本研究的结果一致。在系统进化树第II组的KAT1、KAT2和NKT6基因,在ABA 等逆境条件下,叶片中的表达量降低[6,8,10],这与本研究中Os-KAT2在胁迫时的表达特征基本一致。ZMK2.1、KAT2、KAT1、KAT2和NKT6等基因均参与保卫细胞的运动,在植物抵抗非生物逆境胁迫中发挥重要的作用[6-8,10]。因此,推测OsKAT2可能在水稻抵抗逆境胁迫中发挥作用,是一个能通过转基因技术提高水稻抗非生物逆境能力的候选基因。除转录水平外,OsKAT2 蛋白质存在翻译后修饰位点,即OsKAT2 与大多数Shaker家族K+通道一样也存在转录后水平的调控。对OsKAT2 在干旱、高盐和ABA 等逆境胁迫下的蛋白质水平调控还需要进一步研究。
本研究获得了一个Shaker家族K+通道基因OsKAT2。该基因编码产物OsKAT2 由601 个氨基酸组成,包含K+通道的保守结构域,属于Shaker家族第II 组。OsKAT2的mRNA 在水稻地上部大量表达;干旱、高盐和外源ABA 胁迫处理诱导OsKAT2的表达量下调。初步推测OsKAT2可能在水稻抵抗逆境胁迫中发挥作用,是一个水稻抗非生物逆境改良的候选基因。
[1] 朱德峰,程式华,张玉屏,等.全球水稻生产现状与制约因素分析[J].中国农业科学,2010,43(3):474-479.
[2] 陈 煜,杨燕凌,谢小芳,等.水稻耐盐相关基因的克隆与转化研究进展[J].中国农学通报,2010,26(11):23-27.
[3] 陈丽珍,叶剑秋,王荣香.水稻盐胁迫的研究进展[J].热带农业科学,2011,31(3):87-93.
[4] OBATA T,KITAMOTO H K,NAKAMURA A,et al.Rice shaker potassium channelOsKAT1confers tolerance to salinity stress on yeast and rice cells[J].Plant Physiology,2007,144(4):1978-1985.
[5] FUCHS I,STÖLZLE S,IVASHIKINA N,et al.Rice K+uptake channelOsAKT1is sensitive to salt stress[J].Planta,2005,221(2):212-221.
[6] PILOT G,LACOMBE B,GAYMARD F,et al.Guard cell inward K+channel activity inArabidopsisinvolves expression of the twin channel subunits KAT1 and KAT2[J].The Journal of Biological Chemistry,2001,276(5):3215-3221.
[7] SU Y H,NORTH H,GRIGNON C,et al.Regulation by external K+in a maize inward shaker channel targets transport activity in the high concentration range[J].The Plant Cell,2005,17(5):1532-1548.
[8] 靳义荣,宋毓峰,白 岩,等.林烟草钾离子通道基因NKT6的克隆与表达定位分析[J].作物学报,2013,39(9):1602-1611.
[9] KADDOUR R,NASRI N,M'RAH S,et al.Comparative effect of potassium on K and Na uptake and transport in two accessions ofArabidopsis thalianaduring salinity stress[J].Comptes Rendus Biologies,2009,332(9):784-794.
[10] KANG J Y,CHOI H I,IM M Y,et al.Arabidopsisbasic leucine zipper proteins that mediate stress-responsive abscisic acid signaling[J].The Plant Cell,2002,14(2):343-357
[11] GUPTA M,QIU X,WANG L,et al.KT/HAK/KUP potassium transporters gene family and their whole-life cycle expression profile in rice (Oryza sativa)[J].Molecular Genetics and Genomics,2008,280(5):437-452.
[12] 李俊林,高 南,刘春生,等.水稻钾离子通道基因OsKAT1.1的克隆、表达载体的构建及其电生理功能[J].基因组学与应用生物学,2011,30(4):346-351.
[13] MA J F,GOTO S,TAMAI K,et al.Role of root hairs and lateral roots in silicon uptake by rice[J].Plant Physiology,2001,127:1773-1780.
[14] 张 曼,羊杏平,王薇薇,等.西瓜ClPDF2.6基因的鉴定与表达谱分析[J].江苏农业学报,2013,29(4):831-836.
[15] 刘晓庆,徐照龙,许摇玲,等.大豆GmNAC8基因克隆与原核表达[J].江苏农业学报,2013,29(4):734-737.
[16] VÉRY A A,SENTENAC H.Molecular mechanisms and regulation of K+transport in higher plants [J].Annual Review of Plant Biology,2003,54:575-603.
[17] WANG Y,WU W H.Potassium transport and signaling in higher plants[J].Annual Review of Plant Biology,2013,64:451-476.

