豇豆分子遗传学研究进展
潘磊 ,李依 ,余晓露 ,李佳楠 ,陈禅友
(1.江汉大学生命科学学院,武汉,430056;2.湖北省豆类(蔬菜)植物工程技术研究中心)
豇豆[Vigna unguiculata(L.) Walp.]属于豆科蝶形花亚科豇豆属,为一年生草本植物,耐高温干旱。豇豆是一种重要的豆类作物,在世界范围内种植广泛,主要种植区域位于热带和亚热带的35°N和30°S之间,包括亚洲、大洋洲、中东、欧洲南部、非洲、美国南部和中南美洲(图1)。主产国为尼日利亚、尼日尔、埃塞俄比亚、突尼斯、中国、印度、菲律宾等。中国的豇豆种植面积常年维持在33万hm2以上,主要产区为河北、河南、江苏、浙江、安徽、四川、重庆、湖北、湖南、广西等省(自治区)。
豇豆属有3个栽培亚种,分别是:短豇豆(V.unguiculatassp.cylindrica)、 普 通 豇 豆 (V.unguiculatassp.unguiculata)和长豇豆(V.unguiculatassp.sesquipedalis)。普通豇豆主要分布在非洲的撒哈拉地区;长豇豆主要分布在中国和印度,而且中国是长豇豆的次生起源中心和多样性中心。豇豆可供食用的部位为嫩荚和种子,亚洲地区主要栽培以食用嫩荚为主的长豇豆,而非洲地区主要栽培以食用种子为主的普通豇豆。根据联合国粮农组织(Food and Agriculture Organization,FAO)的统计,全世界的豇豆种植面积一千多万公顷,种子常年产量约 600 万 t(http://faostat3.fao.org/faostat-gateway/go/to/download/Q/QC/E)。由于豇豆富含多种人体所需的植物性蛋白质以及膳食纤维,在人们的日常饮食和营养保健中发挥了重要作用。豇豆除可供鲜食外,还可以进行干制、腌制等产后加工,国内外市场需求旺盛。
与水稻、小麦、大豆等作物相比,豇豆的分子遗传学理论与应用基础研究比较滞后,被称为“孤儿作物”。可喜的是,随着现代生物学理论与技术的不断发展进步,近年来豇豆DNA分子水平上的研究愈来愈成为学者们关注的焦点和热点。下面本文分述豇豆分子遗传相关的研究进展。

图1 世界豇豆种植分布主要区域
1 豇豆的基因库资源
1.1 豇豆种质资源概况
豇豆是二倍体植物(2n=2x=22),基因组大小约为587 Mb[1];其栽培亚种之一长豇豆(V.unguiculatassp.sesquipedalis)的核型为 10m+1sm[2]。
当前,在世界范围内的豇豆种质资源的搜集和保存等研究方面已经卓有成效(表1)。位于尼日利亚的国际热带农业研究所(International Institute for Tropical Agriculture,IITA;http://old.iita.org)建立了世界上最大的豇豆种质资源库,搜集保存了来自89个国家和地区的1 507份野生豇豆和15 003份豇豆栽培种,其中对逾12 000份进行了28个农艺性状的评价,并构建了核型种质资源库,共筛选出1 701份地方品种、225份改良的栽培种(包括品种、品系或株系)和130份代表性种质材料[3]。美国农业部(United States Department of Agriculture,USDA;http://www.ars-grin.gov)保存了世界各地搜集的6 845份豇豆种质资源。位于我国台湾省的亚洲蔬菜研究开发中心(The Asian Vegetable Research and Development Center,AVRDC;http://avrdc.org/)保存了1 572份豇豆资源;中国农业科学院(Academy of Agricultural Sciences,CAAS)国家农作物种质资源保存中心(http://icgr.caas.net.cn/)保存有1 202份不同类型的豇豆种质材料。
1.2 豇豆及其相关的基因组数据库资源
已建立了豇豆分子生物学方面的专业数据库资源,且豆科中其他种属的植物基因组学数据库资源也可借鉴与利用(表2)。当前,豇豆的数据库主要有4个,包括NCBI数据库(http://archive-dtd.ncbi.nlm.nih.gov/),豇豆 EST 数据库 HarvEST(http://www.harvest-web.org/)、豇豆甲基化位点相关的数据库CGKB(http://cowpeagenomics.med.virginia.edu/CGKB)和豇豆物理图谱数据库Physical Map of Cowpea(http://phymap.ucdavis.edu/cowpea/)。此外,豆科模式植物大豆 (Glycine max)、蒺藜苜 蓿(Medicago truncatula)等建立的基因组学相关数据库资源,亦可作为豇豆基因组学研究的重要参考。
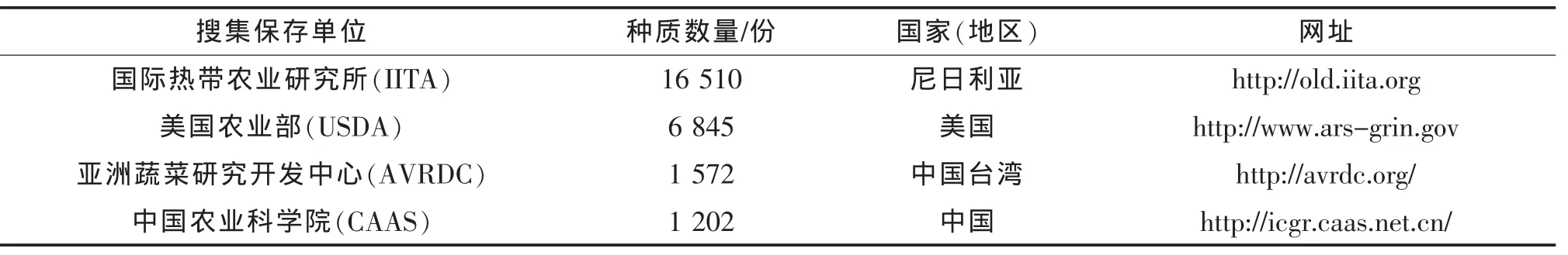
表1 世界范围内豇豆种质资源库概况
2 豇豆的DNA分子标记研究
2.1 豇豆DNA分子标记的发掘
当前,DNA分子标记已经广泛应用于植物种质资源和作物遗传育种研究。DNA分子标记是遗传标记的一种,是基因组上存在核酸变异的特异性DNA序列,能够反映个体或种群之间基因组中存在的某些差异。与形态标记、细胞标记和生化标记相比较而言,DNA分子标记存在诸多优点:①具有较高的遗传多态性;②直接以DNA的形式呈现,不受环境条件、发育时期的影响,也没组织器官特异性;③分布于整个基因组,而且数量多;④许多分子标记表现为共显性,能够区分基因型中的杂合子;⑤一般表现为选择中性;⑥经济方便而且易于检测分析。
在豇豆的分子标记研究中,大多采用基于PCR(Polymerase chain reaction,聚合酶链式反应)的DNA分子标记。利用显性分子标记,如RAPD、ISSR等,已经筛选获得一批重复性好、稳定可靠的分子标记引物[4,5]。近年来,SSR和SNP等共显性分子标记的应用备受青睐。Muchero等[6]2009年首次报道了基于EST序列的1 375个SNP位点。Li等[7]2001年率先开发出27个SSR标记应用于豇豆研究。Gupta等[8]2010年从NCBI数据库(http://archive-dtd.ncbi.nlm.nih.gov/)的豇豆unigenes序列中设计筛选出102个SSR标记。类似的研究中,Xu等[9]2010年则从豇豆基因组数据库HarvEST(http://www.harvest-web.org/)和 CGKB(http://cowpeagenomics.med.virginia.edu/CGKB)中发掘出172个多态性豇豆SSR分子标记(45个EST-SSR标记和127个gSSR标记)。

表2 豇豆及豆科植物基因组数据库资源
2.2 豇豆种质资源的DNA分子标记应用研究
探究豇豆种质资源DNA水平的遗传变异,特别是对野生资源和栽培种遗传多样性现状与亲缘关系的研究,是开展豇豆分子遗传改良及其应用研究的基础。因此,世界豇豆分布区的国家和地区对豇豆种质资源开展了不同分子标记方面的研究。
①豇豆野生资源的分子标记研究 一般认为豇豆起源于非洲,因为野生豇豆在非洲地区分布广泛,但是,豇豆在非洲的起源地点具体位于何处尚不清楚,学者们采用不同DNA分子标记进行了研究,认为非洲东部地区是野生豇豆起源地,而非洲南部地区可能是野生豇豆的多样性中心。Coulibaly等[10]2002年采用AFLP分子标记分析了117份豇豆材料,发现非洲地区野生豇豆比栽培豇豆的多样性水平高,推测非洲东部地区可能是野生豇豆的起源地。与此类似的研究中,Ba等[11]2004年通过对来自非洲东部、西部和南部地区的栽培豇豆和野生豇豆进行RAPD分子标记分析,也推测非洲东部可能是野生豇豆的起源地。Ogunkanmi等[12]2008年基于SSR分子标记分析非洲野生豇豆的遗传多样性,揭示出非洲南部地区野生豇豆的PIC(Polymorphic information content,多态信息含量)较高,推测非洲南部地区可能是野生豇豆的多样性中心。
②豇豆品种资源的分子标记研究 在豇豆品种资源的研究上,采用DNA分子标记技术探究了中国、泰国等国家的豇豆品种间的遗传多样性水平和遗传关系。徐雁鸿等[13]2007年对来自中国等亚洲国家和部分非洲地区的316份豇豆品种进行SSR分子标记分析,发现中国豇豆品种比外来品种的遗传多样性水平低,在中国的豇豆品种遗传分布不均,广西和湖北等省份的豇豆具有较高遗传多样性,而安徽、吉林、黑龙江和山西等省的则较低;而且基于RAPD分子标记的分析结果表明,我国不同豇豆品种之间遗传差异较大[4]。在泰国长豇豆遗传关系的研究中,通过综合农艺表型、SSR和ISSR分子标记等进行聚类分析,能够有效区分不同的豇豆材料[14]。
在豇豆地方品种的DNA分子标记研究中,非洲和亚洲地区的地方品种比较受关注,并且在DNA分子标记种类的选择上进行了比较分析。Tosti等[15]2002年发现在豇豆地方品种检测效率方面,AFLP比RAPD技术揭示的多样性指数更高,尤其是在分析遗传背景较狭窄的材料中,AFLP技术更加有效。在AFLP分子标记研究方面,刘永华等[16]于2007年构建了一套优化的豇豆AFLP分析技术体系。Fang等[17]2007年采用6个AFLP分子标记引物组合分析了来自非洲、亚洲和南美地区的27个豇豆地方品种,揭示出这些材料具有较高的遗传相似性,亚洲和美洲的豇豆可能有共同的起源,而与来自西非的材料不同。此外,在豇豆地方品种的RAPD分子标记研究中,研究者发现马拉维豇豆地方品种的RAPD标记聚类结果与其形态特征之间并不一致[18];而基于RAPD分子标记揭示出贝宁地区豇豆之间存在较大的遗传差异[19]。豇豆地方品种的SSR分子标记研究中,Badiane等[20]2012年利用44个EST-SSR分子标记将塞内加尔的22个豇豆地方品种聚类到一起。Lee等[21]2009年则采用6个SSR分子标记分析了492份韩国豇豆地方品种,揭示出较高的遗传多样性水平。
③豇豆种质材料的分子标记研究 除了豇豆野生种、品种、地方品种外,对不同国家地区的不同豇豆种质材料也开展了DNA分子标记的研究。Malviya等[22]2012年采用18个RAPD标记将10份印度豇豆材料分为2个类群;类似的研究中,Gajera等[23]2014年发现,基于RAPD和ISSR这两种分子标记能较好地将豇豆品种材料与优良基因型材料进行聚类区分。Gillaspie等[24]2005年的研究表明,采用3个AFLP引物组合和10对SSR引物能有效鉴定出豇豆亚种之间遗传关系较近的材料和杂合个体。Asare等[25]2010年采用SSR分子标记评估了来自加纳国内9个地区的141份豇豆材料,可以聚类为5个类群,每个类群与其地理来源存在一定的相关性。Sawadogo等[26]2010年通过SSR分子标记筛选出豇豆对寄生杂草(Striga gesnerioides)的抗性材料和敏感型材料。此外,鲁忠富等[27]2010年从研究长豇豆的SSR引物中筛选出10对诊断性引物,建立了基于SSR分子标记技术的长豇豆种子纯度快速鉴定方法。
近年来,以测序为基础的SNP分子标记也逐渐应用于豇豆研究中。Xu等[28]2012年采用1 127个基于EST序列的SNP标记,分析了包含有长豇豆和普通豇豆的99份核心材料,结果表明,多态性SNP的比例相对较低(39%),平均每个位点1.33个SNP,Bayesian群体结构分析揭示出2个亚群,大体上分别与SV亚群(Standard vegetable type)和NSV亚群(Non-standard vegetable type)一致;LD(r2)较高,且LD在亚群SV中比在亚群NSV中持续得更长,而LD衰减在2个亚群中均较快,LD衰减在不同染色体中存在差别,最长的是最短的约5倍。该研究首次进行了豇豆群体遗传结构分析,并证明了基于豇豆SNP的全基因组关联分析可以对复杂性状进行关联作图。
综上所述,不同类型DNA分子标记已经在豇豆种质资源研究中成功应用(表3),而且以基于PCR技术的DNA分子标记居多,近年来基于测序的SNP分子标记也越来越受到青睐。在基于DNA分子标记的研究中,认为非洲东部地区是野生豇豆起源地,而非洲南部地区可能是野生豇豆的多样性中心,为豇豆的起源和遗传多样性中心问题提供了分子依据。DNA分子标记能有效揭示豇豆品种、地方品种等现存豇豆种质资源中蕴含较为丰富的遗传变异,而且不同地区豇豆种质资源的遗传多样性水平不均一,存在地区差异,通过聚类可以揭示豇豆个体之间的遗传关系,通常情况下,基于分子标记的聚类与性状特征之间没有一致性,Schut等[29]认为其原因可能是所观测的性状、性状的遗传变异、性状相关的基因数量等偏少,也可能是基因的上位相互作用。

表3 豇豆研究中采用的几种主要分子标记

表4 成功构建的豇豆遗传连锁图谱
2.3 豇豆分子遗传图谱研究进展
豇豆中分子遗传图谱方面的研究虽然起步较晚,但是近年来发展迅速,已经成功构建了多个高密度连锁遗传图(表4),为豇豆的分子遗传学研究奠定了良好的基础。
Ouédraogo等[30]2002年采用133个 RAPD、36个RFLP和267个AFLP标记,利用F2群体,构建了包含11个连锁群的豇豆遗传图谱,总长2 670.0 cM。2009年,豇豆中第一张高密度SNP连锁遗传图谱成功构建[6],此遗传图谱将928个SNP标记整合到11个连锁群上,覆盖了680.0 cM,平均0.73 cM 1个SNP标记;同时,在与豆科模式植物的大豆和蒺藜苜蓿进行基因组同线性分析时发现,长豇豆与大豆和蒺藜苜蓿在连锁遗传图上的共线性比率分别为85%和82%。在国内的研究中,Xu等[31]2011年利用长豇豆杂交组合(ZN016×ZJ282),构建了 1个覆盖11个连锁群的375个标记位点(其中有191个SNP标记和184个SSR标记),覆盖基因组745.0 cM,每个标记平均为1.98 cM。Lucas等[32]2011年采用基于EST序列的SNP,对13个群体的1 293个个体用Illumina 1536 GoldenGate Assay分析时,构建了基于 1 107个 SNP的 11个连锁群(680.0 cM),并将其与大豆基因组进行了共线性比较分析,揭示两者基因组上存在较高的同源性。Kongjaimun等[33]2012年从豇豆属的红豆(V.angularis)和绿豆(V.radiata)基因组中开发SSR标记,利用长豇豆JP81610和野豇豆(V.unguiculatasubsp.unguiculatavar.spontanea)种质TVnu457的杂交后代,构建了包含226个SSR标记在11个连锁群上覆盖852.4 cM的连锁图谱,每个标记间平均3.96 cM。
3 豇豆中功能基因的发掘与鉴定研究
功能基因的发掘与鉴定是进行分子遗传改良的前提和基础。与水稻、玉米、小麦和棉花等农作物相比较而言,豇豆功能基因的发掘与鉴定研究十分薄弱,包括与高温干旱等非生物逆境抗性相关的基因,与抗炭腐病(茎枯病)、根腐病、枯萎病等生物逆境相关的基因,以及与叶片、豆荚、花期等生长发育相关的基因的研究。
3.1 豇豆转基因研究
由于当前豇豆转基因体系尚不成熟,对于细菌转化体系、电转化体系和基因枪等不同转基因手段的选择,目标组织的遴选等转基因技术体系中的关键问题仍需不断深入研究(表5)。近些年,豇豆中已经开展nptII基因、CPMV基因、gus基因、hpt基因、αAI-1基因、Cry1Ab基因和Atahas基因等转基因研究[34~50];最近还报道了一种利用6-磷酸甘露糖异构酶基因作选择标记筛选转基因豇豆的方法[51]。
3.2 耐旱、耐热相关基因研究
在耐旱相关基因的研究中,Franca等[52]2008年通过克隆表达豇豆耐旱相关基因PAP(Phosphatidic acid phosphatase,磷脂酸磷酸酶)的2个cDNA(Vu-PAP alpha和 VuPAP beta),对 VuPAP alpha启动子的生物信息学分析,鉴定出了一些与干旱相关的调节元件。另外,采用深度测序分析豇豆干旱胁迫下表达的miRNA,鉴定出44个与干旱相关的miRNA,其中30个在干旱中是上调作用,14个为下调作用[53]。Mondal等[54]2014年克隆了豇豆中的β碳酸酐酶基因VuCA1,该基因的表达具有明显的组织差异性,在花蕾、茎秆和根部表达较弱,而在叶片中表达较强,且在叶片中的基因表达水平因干旱和盐胁迫而上升。

表5 豇豆中转基因研究发展概况
在对豇豆耐旱性相关基因的QTL定位中,发掘了一些相关的QTL位点。采用AFLP分子标记,对豇豆耐旱型与敏感型的重组自交系群体进行QTL分析,揭示出与苗期干旱耐性和成熟性相关的12个QTL[55];进一步的研究中,发现了7个AFLP标记位于干旱或非生物胁迫诱导相关的同源区EST[56]。Lucas等[57]基于SNP分子标记,发掘出与豇豆耐热相关的5个QTL位点Cht-1~Cht-5(表6)。

表6 豇豆中QTL作图研究
3.3 豇豆抗虫、抗病相关基因的研究
在豇豆抗虫相关基因的研究方面,豇豆胰蛋白酶抑制剂(Cowpea trypsin inhibitor,CpTI)基因具有抗虫谱广而且昆虫不易对其产生耐受性等特点,Hilder等[58]1987年首先获得转cpti基因的抗虫烟草植株;目前,已经应用在水稻、棉花、红薯、油棕等多种作物中[59~64]。
Muchero等[65]2011年在豇豆中将抗炭腐病相关的9个 QTL(Mac-1~Mac-9)位点,定位到 5个连锁群 LG2、LG3、LG5、LG6、LG11 上。Pottorff等[66]2012年发现位于连锁群LG6上的1个QTL位点Fot3-1与抗镰刀菌根腐病相关。Pottorff等[67]2014年分析出豇豆中与抗镰刀霉菌枯萎病相关的2个QTL位点,Fot4-1和Fot4-2,分别定位到豇豆通用连锁遗传图的 LG5和 LG3上(表6)。
3.4 豇豆发育相关基因的研究
豇豆发育相关的功能基因研究鲜见报道,主要涉及戟形叶片、荚长、花期、结荚、豆荚柔嫩度和可溶性固形物总量、始花时间、盛花时间、种皮颜色等相关基因的QTL研究。
Pandey等[68]2004年研究发现,豇豆中退化的托叶(Rudimentary stipules,RS)由显性基因控制,隐性基因控制着叶状叶托(FS)表型。Pottorff等[69]2012年采用SNP标记,将1个与豇豆戟状叶片相关的QTL位点(即Hls基因位点),定位于豇豆第4号连锁群上,而且发现1个与Hls基因区域共分离的连锁标记SNP 1_0349,序列比对分析发现,Hls候选基因与拟南芥 EZA1/SWINGER(AT4G02020.1)基因同源性高(表6)。
此外,现有的研究表明,籽粒大小、豆荚大小、茎秆大小和叶片大小等驯化相关基因的QTL主要位于连锁群LG7[70]。荚长相关的7个QTL[33],始花期相关的1个QTL位点Qfld.zaas-11,始花节位相关的1个QTL位点Qnff.zaas-11,单株结荚数相关的1个QTL位点Qpn.zaas-3,叶片衰老相关的一个QTL位点Qls.zaas-11[71],豆荚柔嫩度相关的3个QTL位点Psn7.1,Psn8.1,Psn11.1,可溶性固形物总量相关的2个QTL位点Psw1.1和Psw3.1[72],始花时间相关 的 5 个 QTL 位 点qfot1.1、qfot1.2、qfot1.3、qfot2、qfot10,盛花时间相关的3个QTL位点qdtf1、qdtf2、qdtf7[73];豇豆种皮褐变相关的3个QTL位点Hbs-1、Hbs-2和Hbs-3[74](表6),上述研究报道的 QTL位点中,部分位点已经整合到豇豆通用遗传图谱[7],包括Hls基因位点,Fot3-1基因位点,Mac-1~Mac-9基因位点,Fot4-1~Fot4-2基因位点,Hbs-1~Hbs-3(图2)。
4 豇豆的分子进化研究
在豇豆的起源、进化与分类等的研究中,已经获得一些分子生物学证据,较多开展了基于ITS(Internal transcribed spacer,内部转录间隔区)、IGS(Intergenic spacer,基因间隔区)等序列变异的系统发生学研究,不论是在豇豆属的层次,或是在种属之内,ITS和IGS序列长度都存在较大遗传变异,多态性位点较多,能有效用于豇豆分子鉴定和遗传系谱分析。
在豇豆属的分类水平上,Goel等[75]测序比较了豇豆属29个物种和菜豆属9个物种的rDNA基因中ITS序列(18S-26S rDNA重复区),发现在豇豆属和菜豆属中的ITS序列存在差别,ITS1在豇豆属中的长度在 187~243 bp,而在菜豆属中为217~290 bp;ITS2在豇豆属中的长度变化为187~219 bp,而在菜豆属中为225~243 bp;进一步分析发现,ITS谱系图与基于形态学、生物化学、细胞学和孢粉学的分类基本一致,而且这些ITS序列可以用于区分V.mungo、V.radiata、V.umbellata和V.unguiculata的野生种。
在探究豇豆亚属各物种间的系统发生学关系的研究中,Vijaykumar等[76]2010年基于rRNA基因的ITS区域,发现356个多态性位点中的80%为简约信息位点,通过邻接法(Neighbor joining)和最大简约法(Maximum parsimony)进行遗传发育系统关系重构,可以将豇豆亚属的57个豇豆材料(属于15个种)聚类到5个主要分支。之后,Vijaykumar等[77]于2011年又分析了豇豆亚属不同种的豇豆rDNA基因间隔区(IGS)序列,结果表明,5S IGS在长度(189~237 bp)和序列(58%多态性位点)上存在极大变异,而且大多数的豇豆种都有一种单一类型的5S rRNA重复单元,但是V.unguiculata和V.reticulata之间是例外,因为它们之间表现出多种5S rRNA类型。此外,Doi等[78]研究了豇豆亚属Ceratotropis的系统发生学关系,比较了rDNA ITS与叶绿体DNA的atpB-rbcL基因间隔区,2种序列都约 700 bp,rDNA-ITS比atpB-rbcL的多态性位点多,是atpB-rbcL简约信息位点(Parsimony-informative sites)的5倍,atpB-rbcL间隔区更适于对豇豆属在物种水平上进行分析;基于rDNA-ITS聚类的3个类群,基本与豇豆亚属Aconitifoliae、Angulares和Ceratotropis相对应。
5 结语

图2 部分性状在豇豆通用遗传图谱上的QTL定位
当前,被称为“孤儿作物”的豇豆其分子遗传学研究发展迅速,极大地促进了豇豆的理论与应用基础研究。在全球范围内已经建立了类型丰富的豇豆种质资源库,总份数超过2.5万份,而且豇豆及一些豆科模式植物中已建立了专业数据库资源,为豇豆的基础理论与应用研究奠定了坚实基础;通过DNA分子标记技术的应用,世界豇豆资源的遗传多样性总体水平较高,但是存在地域性差异;在豇豆分子连锁遗传图谱研究方面,以往多采用AFLP和SSR分子标记,近年来基于测序的SNP分子标记越来越受青睐,虽然标记数量较少,不足2 000个分子标记[67],图谱标记密度不高,但是有效地加深了对目标性状的基因定位研究;当前,豇豆中仅开展了少数目的基因的转基因研究(nptII、CPMV、gus、hpt、αAI-1、Cry1Ab和Atahas等基因),获得了转基因的愈伤组织或者单株,但是豇豆的转基因体系仍不完善,尚未广泛应用,需要构建高效稳定的转基因体系;此外,豇豆的分子进化研究为传统的起源、进化与分类等提供了分子水平的佐证。
尽管在豇豆的分子遗传学研究方面取得了上述进展,加深了豇豆的分子基础理论与应用研究,但是仍然有许多问题值得我们持续关注,尤其是在现代生物技术大发展的背景下,豇豆分子水平的研究面临新的机遇与挑战:①虽然已在世界范围内开展豇豆种质资源的交流与合作研究,实现了豇豆资源的合理有效利用,进行了豇豆种质创新和遗传改良,但是由于我国豇豆种质资源的收集、鉴定和评价等研究发展缓慢,在国家种质资源库中的豇豆数量偏少,亟待充实更新,且需要建立健全豇豆公共数据库;②豇豆DNA分子标记需要大量的开发及应用,尤其是高密度分子遗传连锁图谱的构建,与生物逆境(虫害、病害等)、非生物逆境(高温、干旱、盐碱、湿涝等)以及与正常发育相关的功能基因鉴定与发掘;③完善豇豆的转基因系统,通过豇豆的转基因研究,阐明豇豆基因的功能与作用;④随着基因组学、转录组学、蛋白组学和代谢组学等蓬勃发展,“组学”将在多个层次上拓宽和加深豇豆的分子遗传机理阐释。
[1]Iwata A,Greenland C M,Jackson S A.Cytogenetics of legumes in the Phaseoloid clade[J].Plant Genome,2013,8.
[2]She C W,Jiang X H,Ou L J,et al.Molecular cytogenetic characterisation and phylogenetic analysis of the seven cultivatedVignaspecies(Fabaceae)[J].Plant Biology,2014.DOI:10.1111/plb.12174(online).
[3]Mahalakshmi V,Ng Q,Lawson M,et al.Cowpea[Vigna unguiculata(L.) Walp.]core collection defined by geographical,agronomical and botanical descriptors[J].Plant Genet Resour,2007,5:113-119.
[4]陈禅友,潘磊,胡志辉,等.长豇豆品种资源的RAPD分析[J].江汉大学学报:自然科学版,2008(36):77-83.
[5]Chen C Y,Peng H.Differentiation and phylogenetic relationship among different cultivars of asparagus bean[Vignaunguiculata(L.)ssp.sesquipedalis]assessed using ISSR markers[J].Nord J Bot,2010,28:251-256.
[6]Muchero W,Diop N N,Bhat P R,et al.A consensus genetic map of cowpea[Vigna unguiculata(L.)Walp.]and synteny based on EST-derived SNPs[J].Proc Natl Acad Sci USA,2009,106:18 159-18 164.
[7]Li C D,Fatokun C A,Ubi B,et al.Determining genetic similarities and relationships among cowpea breeding lines and cultivars by microsatellite markers[J].Crop Sci,2001,41:189-197.
[8]Gupta S K,Gopalakrishna T.Development of unigenederived SSR markers in cowpea (Vigna unguiculata)and their transferability to otherVignaspecies[J].Genome,2010,53:508-523.
[9]Xu P,Wu X,Wang B,et al.Development and polymorphism ofVigna unguiculatassp.unguiculatamicrosatellite markers used for phylogenetic analysis in asparagus bean[Vigna unguiculatassp.sesquipedialis(L.)Verdc.][J].Mol Breeding,2010,25:675-684.
[10]Coulibaly S,Pasquet R S,Papa R,et al.AFLP analysis of the phenetic organization and genetic diversity ofVigna unguiculataL.Walp.reveals extensive gene flow between wild and domesticated types[J].Theor Appl Genet,2002,104:358-366.
[11]Ba F S,Pasquet R S,Gepts P.Genetic diversity in cowpea[Vigna unguiculata(L.)Walp.]as revealed by RAPD markers[J].Genet Resour Crop Ev,2004,51:539-550.
[12]Ogunkanmi L A,Ogundipe O T,Ng N Q,et al.Genetic diversity in wild relatives of cowpea(Vigna unguiculata)as revealed by simple sequence repeats(SSR)markers[J].J Food Agric Environ,2008,6:263-268.
[13]徐雁鸿,关建平,宗绪晓.豇豆种质资源SSR标记遗传多样性分析[J].作物学报,2007(33):1 206-1 209.
[14]Tantasawat P,Trongchuen J,Prajongjai T,et al.Variety identification and comparative analysis of genetic diversity in yardlong bean (Vigna unguiculataspp.sesquipedalis)using morphological characters,SSR and ISSR analysis[J].Sci Hortic,2010,124:204-216.
[15]Tosti N,Negri V.Efficiency of three PCR-based markers in assessing genetic variation among cowpea (Vigna unguiculatasubsp.unguiculata)landraces[J].Genome,2002,45:268-275.
[16]刘永华,李国景,吴晓花,等.长豇豆AFLP技术体系的构建与优化[J].浙江农业学报,2007(19):156-159.
[17]Fang J G,Chao C T,Roberts P A,et al.Genetic diversity of cowpea[Vigna unguiculata(L.)Walp.]in four West African and USA breeding programs as determined by AFLP analysis[J].Genet Resour Crop Ev,2007,54:1 197-1 209.
[18]Nkongolo K K.Genetic characterization of Malawian cowpea[Vigna unguiculata(L.)Walp.]landraces:diversity and gene flow among accessions[J].Euphytica,2003,129:219-228.
[19]Zannou A,Kossou D K,Ahanchede A,et al.Genetic variability of cultivated cowpea in Benin assessed by random amplified polymorphic DNA[J].Afr J Biotechnol,2008,7:4 407-4 414.
[20]Badiane F A,Gowda B S,Cissé N,et al.Genetic relationship of cowpea (Vigna unguiculata)varieties from Senegal based on SSR markers[J].Genet Mol Res,2012,11:292-304.
[21]Lee J R,Back H J,Yoon M S,et al.Analysis of genetic diversity of cowpealandraces from Korea determined by simple sequence repeats and establishment of a core collection[J].Kor J Breed Sci,2009,41:369-376.
[22]Malviya N,Sarangi B K,Yadav M K,et al.Analysis of genetic diversity in cowpea (Vigna unguiculataL.Walp.)cultivars with random amplified polymorphic DNA markers[J].Plant Syst Evol,2012,298:523-526.
[23]Gajera H P,Domadiya R K,Patel S V,et al.Appraisal of RAPD and ISSR markers for genetic diversity analysis among cowpea(Vigna unguiculataL.)genotypes[J].J Crop Sci Biotech,2014,17:79-88.
[24]Gillaspie A G,Hopkins M S,Dean R E.Determining genetic diversity between lines ofVigna unguiculatasubspecies by AFLP and SSR markers[J].Genet Resour Crop Ev,2005,52:245-247.
[25]Asare A T,Gowda B S,Galyuon I K A,et al.Assessment of the geneticdiversity in cowpea (Vigna unguiculataL.Walp.)germplasm from Ghana using simple sequence repeat markers[J].Plant Genet Resour,2010,8:142-150.
[26]Sawadogo M,Ouedraogo J T,Gowda B S,et al.Genetic diversity of cowpea(Vigna unguiculataL.Walp.)cultivars in Burkina Faso resistant toStriga gesnerioide[J].Afr J Biotechnol,2010,9:8 146-8 153.
[27]鲁忠富,徐沛,汪宝根,等.基于SSR分子标记技术的长豇豆种子纯度快速鉴定技术[J].浙江农业学报,2010,22:727-730.
[28]Xu P,Wu X,Wang B,et al.Genome wide linkage disequilibrium in Chinese asparagus bean (Vigna unguiculatassp.sesquipedialis)germplasm implications for domestication history and genome wide association studies[J].Heredity,2012,109:34-40.
[29]Schut J W,Qi X,Stam P.Association between relationship measures based on AFLP markers,pedigree data and morphological traits in barley[J].Theor Appl Genet,1997,95:1 161-1 168.
[30]Ouédraogo J T,Gowda B S,Jean M,et al.An improved genetic linkage map for cowpea (Vigna unguiculataL.)combining AFLP,RFLP,RAPD,biochemical markers,and biological resistance traits[J].Genome,2002,45:175-188.
[31]Xu P,Wu X,Wang B,et al.A SNP and SSR based genetic map of asparagus bean (Vigna unguiculatassp.sesquipedialis)and comparison with the broader species[J].PLoS One,2011,6:e15952.
[32]Lucas M R,Diop N N,Wanamaker S,et al.Cowpea-soybean synteny clarified through an improved genetic map[J].Plant Genome,2011,4:218-225.
[33]Kongjaimun A,Kaga A,Tomooka N,et al.An SSR-based linkage map of yardlong bean(Vigna unguiculataL.Walp.subsp.unguiculataSesquipedalis group)and QTL analysis of pod length[J].Genome,2012,55:81-92.
[34]Garcia J A,Hille J,Goldbach R.Transformation of cowpea(Vigna unguiculata)cells with an antibiotic-resistance gene using a Ti-plasmid-derived vector[J].Plant Sci,1986,44:37-46.
[35]Garcia J A,Hille J,Vos P,et al.Transformation of cowpea(Vigna unguiculata)with a full-length DNA copy of cowpea mosaic virus M-RNA[J].Plant Sci,1987,48:89-98.
[36]Penza R,Lurquin P F,Filippone E.Gene transfer by cocultivation of mature embryos withAgrobacterium tumefaciens:application to cowpea(Vigna unguiculataL.Walp.)[J].J Plant Physiol,1991,138:39-43.
[37]Penza R,Akella V,Lurquin P F.Transient expression and histological localization of aguschimeric gene after direct transfer to mature cowpea embryos[J].Biotechniques,1992,13:576-580.
[38]Akella V,Lurquin P F.Expression in cowpea seedlings of chimeric transgenes after electroporation into seed derived embryos[J].Plant Cell Rep,1993,12:110-117.
[39]Muthukumar B,Mariamma M,Veluthambi K,et al.Genetic transformation of cotyledonexplants of cowpea (Vigna unguiculataL.Walp)usingAgrobacterium tumefaciens[J].Plant Cell Rep,1996,15:980-985.
[40]Ikea J,Ingelbrecht I,Uwaifo A,et al.Stablegene transformation in cowpea(Vigna unguiculataL.Walp.)using particle gun method[J].Afr J Biotechnol,2003,2:211-218.
[41]Popelka J C,Gollasch S,Moore A,et al.Genetic transformation of cowpea(Vigna unguiculataL.)and stable transmission of the transgenes to progeny[J].Plant Cell Rep,2006,25:304-312.
[42]Chaudhury D,Madanpotra S,Jaiwal R,et al.Agrobacterium tumefaciens-mediated high frequency genetic transformation of an Indian cowpea (Vigna unguiculataL.Walp.)cultivar and transmission of transgenes into progeny[J].Plant Sci,2007,172:692-700.
[43]Solleti S K,Bakshi S,Purkayastha J,et al.Transgenic cowpea (Vigna unguiculata)seeds expressing a bean αamylase inhibitor 1 confers resistance tostorage pests,bruchid beetles[J].Plant Cell Rep,2008,27:1 841-1 850.
[44]Ivo N L,Nascimento C P,Vieira L S,et al.Biolistic-mediated genetic transformation of cowpea(Vigna unguiculata)and stable Mendelian inheritance of transgenes[J].Plant Cell Rep,2008,27:1 475-1 483.
[45]Umaharan P,Ariyanayagan R P,Haque S Q.Resistance to cowpea severe mosaic virus,determined by three dosage dependent genes inVigna unguiculataL.Walp.[J].Euphytica,1997,95:49-55.
[46]Adesoye A,Machuka J,Togun A.CRY 1ABtransgenic cowpea obtained by nodal electroporation[J].Afr J Biotechnol,2008,7:3 200-3 210.
[47]Adesoye A,Machuka J,Togun A.Transformation of cowpea(Vigna unguiculataL.Walp.)byAgrobacteriuminfiltration[J].J Appl Biosci,2010,30:1 845-1 860.
[48]Raveendar S,Ignacimuthu S.ImprovedAgrobacteriummediated transformation in cowpeaVigna unguiculataL.Walp.[J].Asian J Plant Sci,2010,9:256-263.
[49]Citadin C T,Cruz A R R,Aragao F J L.Development of transgenic imazapyr-tolerant cowpea (Vigna unguiculata)[J].Plant Cell Rep,2013,32:537-543.
[50]Citadin C T,Ibrahim A B,Aragao F J L.Genetic engineering in cowpea (Vigna unguiculata):history,status and prospects[J].GM Crops,2011,2:144-149.
[51]Bakshi S,Saha B,Roy N K,et al.Successful recovery of transgenic cowpea (Vigna unguiculata)using the 6-phosphomannose isomerase gene as the selectable marker[J].Plant Cell Rep,2012,31:1 093-1 103.
[52]Franca M G C,Matos A R,D'Arcy-Lameta A,et al.Cloning and characterization of drought-stimulated phosphatidic acid phosphatase genes fromVigna unguiculata[J].Plant Physiol Biochem,2008,6:1 093-1 100.
[53]Barrera-Figueroa B E,Gao L,Diop N N,et al.Identification and comparative analysis of drought-associated microRNAs in two cowpea genotypes[J].BMC Plant Biol,2011,11:127.
[54]Mondal T K,Ganie S A,Niraj R R K,et al.Cloning and in silico analysis of a gene encoding a putative β-carbonic anhydrase from cowpea (Vigna unguiculataL.Walp.)[J].J Plant Interact,2014,9:504-513.
[55]Muchero W,Ehlers J D,Close T J,et al.Mapping QTL for drought stress-induced premature senescence and maturity in cowpea[Vigna unguiculata(L.)Walp.][J].Theor Appl Genet,2009,118:849-863.
[56]Muchero W,Ehlers J D,Roberts P A.Restriction site polymorphism-based candidate gene mapping for seedling drought tolerance in cowpea[Vigna unguiculata(L.)Walp.][J].Theor Appl Genet,2010,120:509-518.
[57]Lucas M R,Ehlers J D,Huynh B L,et al.Markers for breeding heat-tolerant cowpea[J].Mol Breeding,2013,31:529-536.
[58]Hilder V A,Gatehouse A M,Sheerman S E,et al.A novel mechanism of insect resistance engineered into tobacco[J].Nature,1987,330:160-163.
[59]刘春明,朱祯,周兆斓,等.豇豆胰蛋白酶抑制剂抗虫转基因烟草的获得[J].科学通报,1992(37):1 694-1 697.
[60]NewellC A,LoweJM,MerryweatherA,etal.Transformation of sweet potato(Ipomoea batatasL.Lam.)withAgrobacterium tumefaciensand regeneration of plants expressing cowpea trypsin inhibitor and snowdrop lectin[J].Plant Sci,1995,107:215-227.
[61]杨朝辉,宋洪元,何凤田.豇豆胰蛋白酶抑制剂基因转化芥菜及抗虫鉴定[J].中国生物化学与分子生物学报,2003(19):731-735.
[62]徐鸿林,翟红利,王锋,等.豇豆胰蛋白酶抑制剂基因(cpti)及其在抗虫转基因作物中的应用[J].中国农业科技导报,2008(10):18-27.
[63]Ismanizan I,Fong S L,Ruslan A,et al.Molecular and expression analysis of cowpea trypsin inhibitor(CpTI)gene in transgenicElaeis guineensisJacq leaves[J].Aust J Crop Sci,2010,4:37-48.
[64]Wu H,Zhang Y,Liu P,et al.Effects of transgenicCry1Ac+CpTIcotton on non-target mealybug pestFerrisia virgataand its predatorCryptolaemus montrouzieri[J].PloS One,2014,9:e95537.
[65]Muchero W,Ehlers J D,Close T J,et al.Genic SNP markers and legume synteny reveal candidate genes underlying QTL forMacrophomina phaseolinaresistance and maturity in cowpea[Vigna unguiculata(L.)Walp.][J].BMC Genomics,2011,12:8-21.
[66]Pottorff M,Wanamaker S,Ma Y Q,et al.Genetic and physical mapping of candidate genes for resistance toFusarium oxysporumf.sp.tracheiphilumrace 3 in cowpea[Vigna unguiculata(L.)Walp.][J].PloS One,2012,7:e41600.
[67]Pottorff M,Li G,Ehlers J D,et al.Genetic mapping,synteny,and physical location of two loci forFusarium oxysporumf.sp.tracheiphilumrace 4 resistance in cowpea[Vigna unguiculata(L.)Walp.][J].Mol Breeding,2014,33:779-791.
[68]Pandey R N,Dhanasekar P.Morphological features and inheritance of foliaceous stipules of primary leaves in cowpea(Vigna unguiculata)[J].Ann Bot,2004,94:469-471.
[69]Pottorff M,Ehlers J D,Fatokun C,et al.Leaf morphology in cowpea[Vigna unguiculata(L.)Walp.]:QTL analysis,physical mapping and identifying a candidate gene using synteny with model legume species[J].BMC Genomics,2012,13:234-245.
[70]Kongjaimun A,Kaga A,Tomooka N,et al.The genetics of domestication of yardlong bean,Vigna unguiculata(L.)Walp.ssp.unguiculatacv.-gr.sesquipedalis[J].Ann Bot,2012,109:1 185-1 200.
[71]Xu P,Wu X,Wang B,et al.QTL mapping and epistatic interaction analysis in asparagus bean for several characterized and novel horticulturally important traits[J].BMC Genet,2013,14:4-13.
[72]Kongjaimun A,Somta P,Tomooka N,et al.QTL mapping of pod tenderness and total soluble solid in yardlong bean[Vigna unguiculata(L.)Walp.subsp.unguiculatacv.-gr.sesquipedalis][J].Euphytica,2013,189:217-223.
[73]Andargie M,Pasquet R S,Muluvi G M,et al.Quantitative trait loci analysis of flowering time related traits identified in recombinant inbred lines of cowpea(Vigna unguiculata)[J].Genome,2013,56:289-294.
[74]Pottorff M,Roberts P A,Timothy J,et al.Identification of candidate genes and molecular markers for heat-induced brown discoloration of seed coats in cowpea[Vigna unguiculata(L.)Walp.][J].BMC Genomics,2014,15:328-338.
[75]Goel S,Raina S N,Ogihara Y.Molecular evolution and phylogenetic implications of internal transcribed spacer sequences of nuclear ribosomal DNA in thePhaseolus-Vignacomplex[J].Mol Phylogenet Evol,2002,22:1-19.
[76]Vijaykumar A,Saini A,Jawali N.Molecular characterization of intergenic spacer region of 5S ribosomal RNA genes in subgenusVigna:extensive hybridization amongV.unguiculatasubspecies[J].Plant Syst Evol,2011,294:39-55.
[77]Vijaykumar A,Saini A,Jawali N.Phylogenetic analysis of subgenusVignaspecies using nuclear ribosomal RNA ITS:evidence of hybridization amongVigna unguiculatasubspecies[J].J Hered,2010,101:177-188.
[78]Doi K,Kaga A,Tomooka N,et al.Molecular phylogeny of genusVignasubgenusCeratotropisbased on rDNA ITS andatpB-rbcL intergenic spacer of cpDNA sequences[J].Genetica,2002,114:129-145.

