大黄鱼cyclin B 1和cdc 2 cDNA序列特征及组织表达分析
蔡明夷,周 鹏,韩坤煌,谢芳靖,张子平,王艺磊*
(1.集美大学 水产学院,农业部东海海水健康养殖重点实验室,福建 厦门361021;2.福建省国家级大黄鱼原种场,宁德市富发水产有限公司,福建 宁德352103;3.美国西东大学生物学系,新泽西州 南奥兰治07090)
鱼类的卵子发生通常要经历减数分裂的中断、暂停与再启动等步骤,而精子发生则是一个减数分裂连续的过程.因而,鱼类的性腺被认为是研究减数分裂周期调控的理想模型[1].许多基因参与了鱼类配子发生减数分裂过程的调控.其中,成熟促进因子(maturation promoting factor,MPF)是诱导减数分裂暂停的卵子重新进入成熟过程的关键因子[2].MPF包括2个亚基:调节亚基细胞周期蛋白B(cyclin B,CB)和周期蛋白依赖性蛋白激酶(cyclin-dependent kinase 1,CDK1,由cdc2基因编码)[3].CB和CDK1结合形成 MPF前体,经过磷酸化/去磷酸化作用成为具有活性的MPF.活化的MPF可以催化一系列靶蛋白的磷酸化,诱导G2期向M期转换;在卵子发生过程中,则诱导发生卵泡破裂(GVBD)、停滞减数分裂启动等细胞行为的发生[4-6].现有研究资料表明,MPF的有序表达、活化及降解是保证两性生殖生物配子发生、成熟与分化等生物过程有序进行的重要条件[7].
大黄鱼(Larimichthys crocea)是我国特有海水鱼类,也是最重要的海水养殖鱼类之一[8].其生长具有明显的雌雄二态性,雌性生长速度明显快于雄性[9].研究大黄鱼性腺发育与性别分化分子机制,可为开发高效大黄鱼性别控制方法、提高大黄鱼生殖控制技术水平奠定基础.然而,目前关于大黄鱼性腺发育和配子发生的知识主要集中于细胞水平[10-13],分子水平的研究才刚刚起步,研究报道较少[14-15].因此,本研究利用cDNA 末端快速扩增法(rapid-amplification of cDNA ends,RACE)克隆了大黄鱼cyclin B1 基因(Lc-cb1)和cdc2 基因(Lccdc2)的cDNA序列,并利用实时荧光定量PCR(qRTPCR)技术分析了这2个基因在大黄鱼性腺及其他组织的mRNA水平,为解析MPF在大黄鱼性腺发育以及配子发生中的作用机理奠定基础.
1 材料与方法
1.1 材料收集
实验大黄鱼取自宁德市富发水产有限公司,雌雄各5尾,鱼体质量(524.7±118.2)g.活鱼尾椎取血后迅速解剖取精巢、卵巢、肌肉、肝脏、脑、肾脏等组织,立即放入液氮速冻,取出置-80℃冰箱中保存备用.
1.2 总RNA的提取和cDNA合成
按Zhang等[16]描述的方法从组织中提取总RNA,然后用DNA酶Ⅰ(DNaseⅠ)37℃处理30 min,去除可能混杂的基因组DNA,用琼脂糖凝胶电泳验证RNA的完整性.按照SMART-RACE cDNA扩增试剂盒说明书(Clontech,美国)所描述的方法,以3μg 总 RNA 为模板、Oligo(dT)为引物,并在SMARTⅡOligo的介导下,利用 M-MLV反转录酶(Promega,上海)在37℃下反应90min,合成RACE适用的cDNA第1链.合成产物在70℃下处理15 min灭活酶,然后贮存于-20℃冰箱中备用.
1.3 Lc-cb1 和Lc-cdc2 cDNA的克隆与测序
利用SMART-RACE方法获得Lc-cb1 和Lc-cdc2 cDNA 5′端和3′端扩增产物.第1轮扩增以cDNA第1条链为模板,正反向引物分别为基因特异外引物(GSP)和通用引物组合(UPM);第2轮扩增的模板为第1轮扩增产物,正反向引物为GSP内引物和巢式通用引物(NUP).其中,GSP内引物和外引物是基于前期实验构建的EST数据库中的Lc-cb1和Lc-cdc2 cDNA部 分 序 列[17]、采 用 Primer 5.0 软 件 设 计 的 (表1).UPM 和 NUP 序列参见SMARTer RACE cDNA扩增试剂盒说明书(Clontech,美国).25μL PCR反应体系包括:0.5μL 模板 DNA,0.5μL 10mmol/L GSP,2.5μL 10mmol/L UPM(或 NUP),2.5μL 10×PCR 缓冲液,0.5μL 10mmol/L dNTP 混合液,0.5μL Taq DNA聚合酶和18μL双蒸水.PCR反应条件为:95℃预变性3min;然后95℃ 变性30s,60~68℃(详见表1)退火30s,72℃90s,32个循环;72℃继续延伸10min.
上述第2轮PCR产物经质量分数为1%的琼脂糖凝胶电泳后,目标片段经回收纯化,克隆到pMD18-T质粒(Takara,日本),进一步转化大肠杆菌(Escherichia coli)JM109并增殖.从阳性克隆中分离质粒,送上海捷瑞生物工程有限公司测序.序列结果用VecScreen(http:∥www.ncbi.nlm.nih.gov/VecScreen/VecScreen.html)去除载体序列,再用 BLAST2(http:∥www.ncbi.nlm.nih.gov/blast/bl2seq/wblast2.cgi)比对拼接出cDNA全长序列.
1.4 qRT-PCR
大黄鱼精巢、卵巢、肌肉、血、脾、肝、肾和脑等8个组织的样品用于qRT-PCR,β-actin为内参基因,以检查Lc-cb1 和Lc-cdc2 mRNA 组织表达特征.反应体系包括:cDNA 第1链9μL,10mmol/L qRT-PCR正反向引物各0.5μL(表1),SYBR Green Realtime PCRMaster Mix(Life Technology,美国)10μL.反应体系混匀后,在ABI 7500PCR仪器上完成热循环反应和荧光检测.PCR反应条件为:50℃保温2min,95℃变性1min;95℃变性15s、60℃退火1min,重复循环40次.每种组织检测了取自不同个体的4个样品,每个样品做4次重复.

表1 本研究所用引物Tab.1 Primers used in this study
mRNA表达水平用相对定量(relative quantification,RQ)平均值±标准差来表示.mRNA表达量平均值的组织间差异的显著性,用SPSS 15.0统计软件的Bonferroni模型作多重比较进行单因素方差分析,p<0.05为差异显著.
1.5 基因序列的生物信息学分析
推导的氨基酸序列的比对和保守区域结构比较利用GenBank网站的BLAST算法(http:∥www.ncbi.nlm.nih.gov)在线完成.蛋白的分子质量和等电点预测利用 Compute pl/Mw tool(http:∥ca.expasy.org/tools/pi_tool.html)完成.用InterProScan软件4.8(ftp:∥ftp.ebi.ac.uk/pub/software/unix/iprscan/index.html)分析氨基酸序列的结构域.氨基酸序列的多重比较利用BioEdit软件(http:∥www.mbio.ncsu.edu/BioEdit/bioedit.html)完成.蛋白进化树利用 Mega 5.1软件(http:∥www.megasoftware.net/)的邻接算法构建.
2 结 果
2.1 Lc-cb1 和Lc-cdc2 cDNA序列特征
将3′RACE获得片段、5′RACE获得片段和文库插入cDNA片段以BLAST 2软件进行序列拼接后,获得Lc-cb1 基因的全长cDNA 序列1 882bp(Gen-Bank登录号FJ195327),如图1.其中,编码区(ORF)长度为1 194bp,可编码397个氨基酸的蛋白.5′-非翻译区(UTR)长度为63bp;3′-UTR的长度为580bp(去除poly A),含有2个加尾信号(AUUAAA),和4个多聚腺苷酸化元件(cytoplasmic polyadenylation elements,简称为 CPEs;A/UUUUUAU/A).推导的LC-CB1蛋白预测分子质量约为44.56ku,等电点约为8.76,序列中含有一个典型的周期蛋白盒(166~313aa),其中272~276aa为CB特有的蛋白酶K位点(RRxSK),N端附近有一个周期蛋白A和CB共有的毁坏盒(RxALGxIxN,32~40aa).氨基酸序列同源性比较结果显示,该序列与爪哇青鳉(Oryzias javan-icus,84%)、青 鳉 (Oryzias latipes,83%)、斑 马 鱼(Danio rerio,67%)、非 洲 爪 蟾 (Xenopus laevis,64%)、小鼠(Mus musculus,61%)、智人(Homo sapiens,63%)等动物的CB1均有较高的同源性.
将3′RACE获得片段、5′RACE获得片段和文库插入cDNA片段以BLAST 2软件进行序列拼接后,获得Lc-cdc2基因全长cDNA序列 1 151bp(GenBank登录号 FJ800565),如图2.其中,ORF长度为 912bp,5′-UTR长度为72bp,3′-UTR的长度为150bp(去除poly A).在poly A上游21bp处有一个典型的加尾信号(AATAAA ).Lc-cdc2 cDNA 可编码303个氨基酸的蛋白,预测分子质量约为34.58ku,等电点约为8.80.推导的蛋白序列中包含一个CDK1典型的丝氨酸/苏氨酸激酶催化结构域(4~287aa),内含丝氨酸/苏氨酸蛋白激酶活性位点(124~136aa)、与ATP结合相关的保守序列GxGxxGxV(11~17aa)、与周期蛋白结合相关的保守序列PSTAIRE(45~51aa)等.氨基酸序列同源性比较结果显示,该序列与龟壳攀鲈(Anabas testudineus,97%)、爪哇青鳉(95%)、虹鳟(Oncorhynchus mykiss,95%)、斑马鱼(90%)、大西洋鲑(Salmo salar,88%)、胡瓜鱼(Osmerus mordax,92%)、金鱼(Carassius auratus,90%)、非洲爪蟾(85%)、褐家鼠(Rattus norvegicus,81%)和智人(82%)等物种的CDK1均有较高的同源性.
2.2 同源性分析和系统发育分析
将推导的LC-CB1氨基酸序列与爪哇青鳉(登录号Q9DGA0)、斑马鱼(登录号 BAA92876)、青鳉(登录号Q9IBG1)、非洲爪蟾(登录号 AAH41302)、智人(登录号P14635)和小鼠(登录号CAA45968)等6个物种的CB1氨基酸序列进行多序列比对.结果表明,大黄鱼及其他6个物种都具有CB1特征结构域(详见图3).根据Lc-cb1 cDNA序列推导的氨基酸序列与上述6个物种的CB1氨基酸序列一致性在46%~84%之间,N端近中部的氨基酸序列变化较大,其他区域的保守性较高.
将推导的大黄鱼CDK1氨基酸序列与爪哇青鳉(登录号BAB17220)、龟壳攀鲈(登录号AAS59851)、胡瓜鱼(登录号 AC0008896)、大西洋鲑(登录号 NP 001134623)、虹鳟(登录号 NP 001118132)、金鱼 (登录号AFV07384)、斑马鱼(登录号 NP 997729)、非洲爪蟾(登 录 号 NP 001080093)、鸭 嘴 兽 (Ornithorhynchus anatinus,登 录 号 XP 001509674)、原 鸡 (Gallus gallus,登 录 号 NP 990645)、智 人 (登 录 号 NP 001777)、褐家鼠(登录号 NP 062169)和果蝇(Drosophila melanogaster,登录号 NP 476797)等13个物种CDK1氨基酸序列进行多序列比对.结果表明,大黄鱼CDK1与其他13个物种一样,都具有CDK1特征结构域(详见图4).根据Lc-cdc2 cDNA序列推导的氨基酸序列与上述的6种鱼类的CDK1氨基酸序列一致性在88%~95%之间,与果蝇的CDK1氨基酸序列一致性也有71%.

图1 Lc-cb1 cDNA全长及推导的氨基酸序列Fig.1 The nucleotide and deduced amino acid sequences of Lc-cb1 cDNA
通过邻接法构建的CB系统发育树(图5(a))显示,大黄鱼CB1与青鳉、爪哇青鳉和斑马鱼等鱼类的CB1首先聚在一个小枝上,再与非洲爪蟾、小鼠和智人等其他脊椎动物CB1所聚的一小枝聚为一个较大分枝;与斑马鱼、非洲爪蟾、小鼠和智人等物种的CB2关系较远,与丽新杆线虫(Caenorhabditis elegans)的CB3的关系更远.通过邻接法构建的CDK1系统发育树(图5(b))显示,大黄鱼首先与龟壳攀鲈聚为一枝,并与爪哇青鳉等其他6种鱼类聚为一个二级分枝;其他5种非鱼类脊椎动物聚为另一个二级分枝;果蝇这种无脊椎动物的CDK1与其他13种脊椎动物距离最远.

图2 Lc-cdc2基因cDNA全长及推导的氨基酸序列Fig.2 The nucleotide and deduced amino acid sequences of Lc-cdc2 cDNA
2.3 Lc-cb1 和Lc-cdc2 基因在大黄鱼各器官中的表达
qRT-PCR分析大黄鱼不同组织中Lc-cb1和Lccdc2基因mRNA水平如图6所示.Lc-cb1和Lc-cdc2基因在大黄鱼各组织均有表达.2个基因的表达组织谱具有相似特征,并呈现出较强的组织特异性:在卵巢的表达量均为最多,在精巢的表达量次之,其他组织仅有微量表达,表达量最少的是脑组织.Lc-cb1卵巢表达量约为脑表达量的2 145倍,Lc-cdc2卵巢表达量约为脑表达量的349倍.
3 讨 论
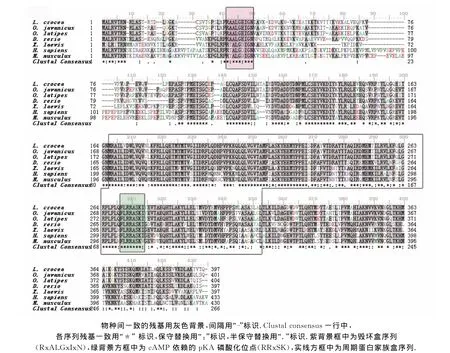
图3 大黄鱼及其他物种CB1氨基酸序列的多重比较Fig.3 Multiple alignment of the CB1amino acid sequence between L.croaker and other species
本研究获得了大黄鱼 Lc-cb1 和Lc-cdc2 cDNA全长,GenBank登录号分别为FJ195327和FJ800565.根据所获得的 Lc-cb1 cDNA 序列(FJ195327)推导的氨基酸序列与其他已登录GenBank的鱼类的CB1氨基酸序列有较高相似性(67%~84%),具有周期蛋白家族的特征,如周期蛋白盒和RxALGxIxN序列;周期蛋白A和CB所特有的毁坏盒以及CB特征的蛋白酶K位点.本研究获得的Lc-cdc2 cDNA (FJ800565)推测的氨基酸序列与其他已登录GenBank的鱼类的CB1氨基酸序列也有较高相似性(88%~97%),且具有CDK1的特征序列,如丝氨酸/苏氨酸激酶催化结构域、ATP结合相关的保守序列GxGxxGxV和与周期蛋白结合相关的保守序列PSTAIRE等.可见,生物信息学分析结果证明,本研究所获得的2条序列分别具备了cb1和cdc2基因cDNA的基本特征.
CB是MPF的调节亚基.脊椎动物中,涉及M期调控的周期蛋白主要有4种亚型.各亚型的分布和功能存在差异.例如,在智人培养细胞中,CB1定位在微管,CB2主要分布在高尔基器上[18].在非洲爪蟾卵子中,CB2在第一次减数分裂双极纺锤体的形成中发挥作用[19-20].小鼠中,CB1是胚胎发育和性腺发育的关键基因并能够补偿CB2的缺失[21].在鸡10日龄胚胎中,CB3兼具周期蛋白A和CB的特点,既能与CDK1也能与CDK2联合作用[22].本研究基于氨基酸序列比对,构建了CB进化树.结果表明,大黄鱼CB1与其他鱼类CB1关系最近;与亲缘关系较远的哺乳动物(如智人类和小鼠)CB1的关系要近于与近缘种(如斑马鱼)CB2的关系.这与虹鳟、青鳉、日本鳗鲡(Anguilla japonica)等其他鱼类中得到的结果一致[23-25].CB 的mRNA通常有一条较长的3′-UTR,例如智人(NM031966)cb1 mRNA 3′-UTR 的长度为615bp.3′-UTR可能通过影响mRNA的多聚腺苷化/去腺苷化、翻译效率、翻译定位和mRNA稳定性来实现基因表达调控.cb1 3′-UTR上含有参与mRNA翻译调控的顺式元件,其中研究最为深入的是CPE元件.CPE为富含U的序列(A/UUUUUAU/A),在卵子成熟过程中通过帮助联接加尾信号活化CB的翻译[26].在卵子中显微注射CPE能够诱导CB1蛋白合成[27].Lccb1 mRNA 3′-UTR长度为580bp,初步分析发现其中含有4个CPE元件、多个富U类的CPE序列和2个加尾信号,与斑马鱼、非洲爪蟾、中华绒螯蟹(Eriocheir sinensis)等生物cb1 3′-UTR 结构基本相似,但CPE元件的数目和分布在不同物种间有所变化.cb1 mRNA 3′-UTR的总体结构、CPE元件的数目和分布均可能影响翻译效率,并可能与生物的具体功能需求相关[28].
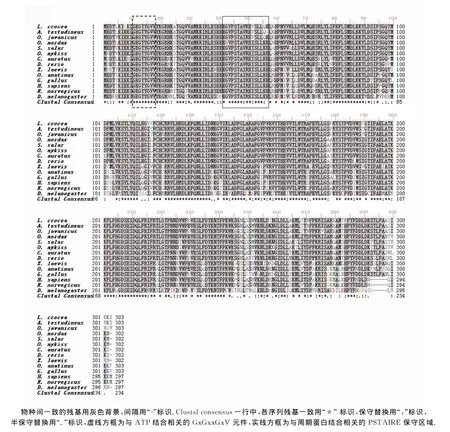
图4 大黄鱼及其他物种CDK1氨基酸序列的多重比较Fig.4 Multiple alignment of the CDK1amino acid sequence between L.croaker and other species
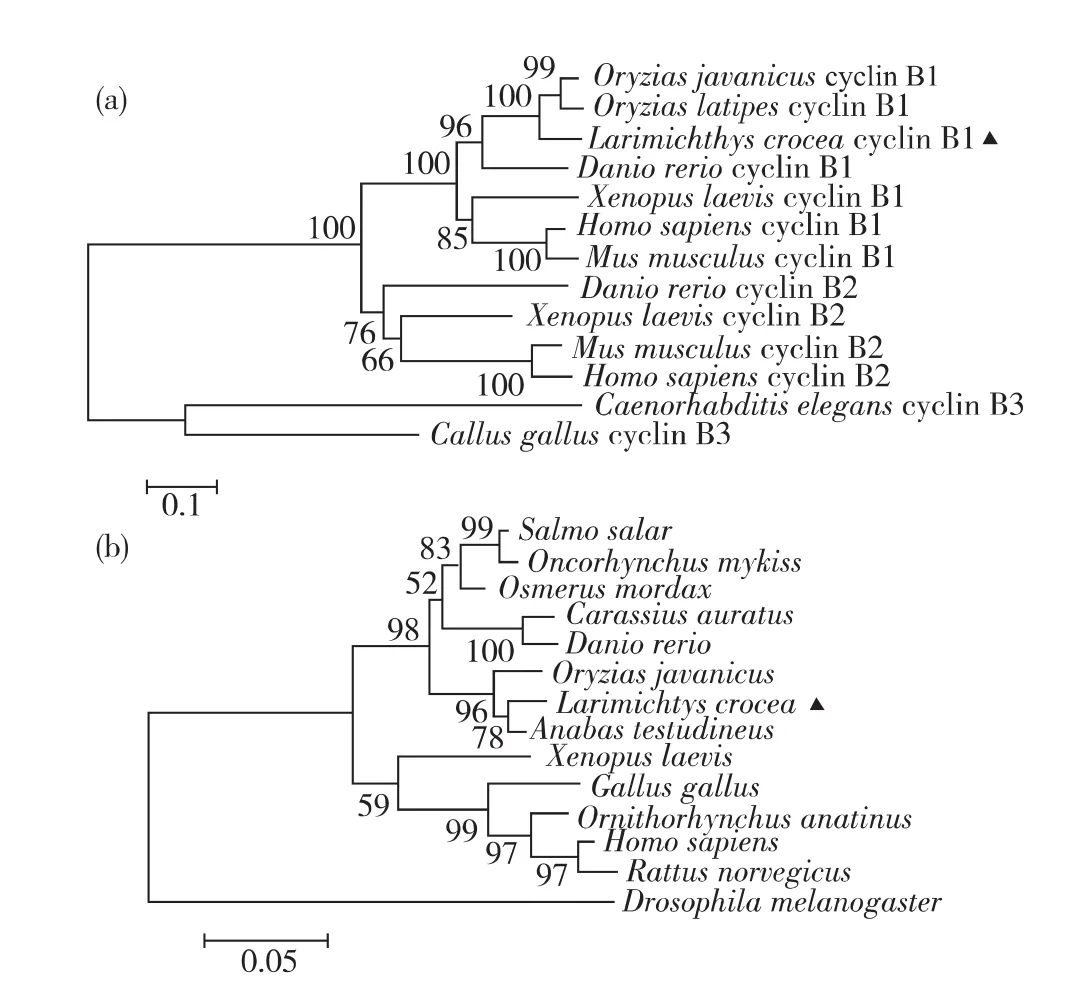
图5 基于大黄鱼和其他物种的CB1(a)和CDK1(b)氨基酸序列构建系统发育树Fig.5 Phylogenetic tree based on the amino acid sequences of CB1(a)and CDK1(b)between L.crocea and other species

图6 大黄点Lc-cb1(a)和Lc-cdc2(b)mRNA的组织表达图谱Fig.6 Tissue expression profiles of Lc-cb1(a)and Lc-cdc2(b)mRNA transcripts in L.crocea
CDK1是一个高度保守的小蛋白,分子质量约34 ku.CDK1参与了细胞周期的全部进程,如细胞周期启动、从G1期向S期转换、从S期向G2期转换,以及G2期向M期转换等.CDK1能够有序行使功能主要依靠三方面的调控:调节亚基周期蛋白的结合、保守残基的磷酸化、以及CDK抑制物(CKI)结合.调节亚基周期蛋白的类型,通过影响CDK1活性位点的位置与结构,决定周期蛋白-CDK复合物的适合底物和特殊功能[29].其中,CB1-CDK1复合物主要调控 G2期向M期转换过程.CB1和CDK1结合形成MPF前体,需要活化才能行使功能.在鱼类卵子成熟过程中,目前已发现2种形式的MPF活化机制.在第1种机制中,CB1在即将发生GVBD时,在激素作用下开始合成,并立即与CDK1结合形成MPF前体;之后通过CDK1上Thr161的磷酸化,即可完成MPF的活化,如金鱼[5].在第2种机制中,未成熟卵子已存在完整的MPF前体,MPF的活化既需要磷酸化,还需要CDC25催化的去磷酸化作用,如龟壳攀鲈[30].基于氨基酸序列的进化树显示,大黄鱼与龟壳攀鲈的CDK1相似性最高(图5(b)).那么,大黄鱼MPF的活化机制是否与龟壳攀鲈一致,MPF的活化机制的分化是否与物种的分化相符,尚需要进一步研究.
qRT-PCR分析结果显示,Lc-cb1 和Lc-cdc2 mRNA表达具有较强的组织特异性,性腺中Lc-cb1和Lc-cdc2 mRNA水平极显著地高于其他组织.这与前人在虹鳟、青鳉和锯缘青蟹等生物中观察到的结果一致[23-24,31].CB1和CDK1组成的MPF是细胞周期调控因子,Lc-cb1和Lc-cdc2在大黄鱼性腺中高水平表达,与性腺中有丝分裂和减数分裂活动活跃的特点相符合.
4 结 论
大黄鱼是我国最重要的海水鱼类.本研究克隆大黄鱼Lc-cb1和Lc-cdc2基因全长cDNA序列,并分析了这2条序列及推测蛋白序列特征,发现大黄鱼与龟壳攀鲈CDK1序列的相似性高达97%;同时,研究还揭示大黄鱼中Lc-cb1和Lc-cdc2 mRNA的表达具有不严格的组织特异性,在性腺中优势表达,表明MPF在大黄鱼性腺发育以及配子发生中具有重要作用.本文研究结果为进一步揭示MPF在大黄鱼性腺发育以及配子发生中的作用机理奠定了基础.
[1]Yamashita M. Molecular mechanisms of meiotic maturation and arrest in fish and amphibian oocytes[J].Semin Cell Dev Biol,1998,9(5):569-579.
[2]Masui Y,Markert C L.Cytoplasmic control of nuclear behavior duringmeiotic maturation of frog oocytes[J].J Exp Zool,1971,177(2):129-145.
[3]Lohka M J,Hayes M K,Maller J L.Purification of maturationpromoting factor,an intracellular regulator of early mitotic events[J].Proc Natl Acad Sci USA,1988(9),85:3009-3013.
[4]Kondo T,Yanagawa T,Yoshida N,et al.Introduction of cyclin B induces activation of the maturation-promoting factor and breakdown of germinal vesicle in growing ze-brafish oocytes unresponsive to the maturation-inducing hormone[J].Dev Bio,1997,190(1):142-152.
[5]Katsu Y,Yamashita M,Kajiura H,et al.Behavior of the components of maturation-promoting factor,cdc2kinase and cyclin B,during oocyte maturation of goldfish[J].Dev Biol,1993,160(1):99-107.
[6]Chesnel F,Bazile F,Pascal A,et al.Cyclin B2/cyclin-dependent kinase1dissociation precedes CDK1Thr-161dephosphorylation upon M-phase promoting factor inactivation in Xenopus laevis cell-free extract[J]Int J Dev Biol,2007,51:297-305.
[7]虞晋晋,叶海辉,黄辉洋.水生动物细胞周期蛋白研究进展[J].厦门大学学报:自然科学版,2006,45(增刊2):185-189.
[8]刘家富,刘招坤.福建闽东大黄鱼Larimichthys crocea (Richardson)产业展望[J].现代渔业信息,2008,23(12):3-5.
[9]王清印.海水养殖研究新进展[M].北京:海洋出版社,2008:123-130.
[10]林丹军,张健,骆嘉,等.人工养殖的大黄鱼性腺发育及性周期研究[J].福建师范大学学报:自然科学版,1992,8(3):81-87.
[11]尤永隆,林丹军,陈莲云.大黄鱼的精子发生[J].动物学研究,2001,22(6):461-466.
[12]方永强,翁幼竹,周晶,等.大黄鱼性早熟的机制:精巢中间质细胞和足细胞的显微与亚显微结构[J].台湾海峡,2002,2(3):276-279.
[13]游秀容,蔡明夷,姜永华,等.大黄鱼性腺性别分化的组织学观察[J].水产学报,2012,36(7):1057-1064.
[14]Pu L L,Han K H,Xie F J,et al.Molecular cloning,characterization,and gene expression of the androgen receptor in the large yellow croaker,Larimichthys crocea[J].Fish Physiol Biochem,2013,39(2):309-324.
[15]周鹏,张子平,王艺磊,等.大黄鱼ubc9基因的克隆和组织表达[J].生物技术通报,2009(8):76-82.
[16]Zhang Z,Wu R S,Mok H O,et al.Isolation,characterization and expression analysis of a hypoxia-responsive glucose transporter gene from the grass carp,Ctenopharyngodon idellus[J].Eur J Biochem,2003,270(14):3010-3017.
[17]Zhou P,Zhang Z P,Wang Y L,et al.EST analysis and identification of gonad-related genes from the normalized cDNA library of large yellow croaker,Larimichthys crocea[J].Comp Biochem Physiol Part D Genomics Proteomics,2010,5(2):89-97.
[18]Jackman M,Firth M,Pines J.Human cyclins B1and B2 are localized to strikingly different structures:B1to microtubules,B2primarily to the Golgi apparatus[J].EMBO J,1995,14(8):1646-1654.
[19]Ihara J,Yoshida N,Tanaka T,et al.Either cyclin B1or B2is necessary and sufficient for inducing germinal vesicle breakdown during frog (Rana japonica)oocyte maturation[J].Mol Reprod Dev,1998,50(4):499-509.
[20]Kotani T,Yoshida N,Mita K,et al.Requirement of cyclin B2,but not cyclin B1,for bipolar spindle formation in frog(Rana japonica)oocytes[J].Mol Reprod Dev,2001,59(2):199-208.
[21]Brandeis M,Rosewell I,Carrington M,et al.Cyclin B2-null mice develop normally and are fertile whereas cyclin B1-null mice die in utero[J].Proc Natl Acad Sci USA,1998,95(8):4344-4349.
[22]Gallant P,Nigg E A.Identification of a novel vertebrate cyclin:cyclin B3shares properties with both A-and B-type cyclins[J].EMBO J,1994,13(3):595-605.
[23]Qiu G F,Ramachandra R K,Rexroad C E 3rd,et al.Molecular characterization and expression profiles of cyclin B1,B2and CDC2kinase during oogenesis and spermatogenesis in rainbow trout (Oncorhynchus mykiss)[J].Anim Reprod Sci,2008,105(3/4):209-225.
[24]Mita K,Ohbayashi T,Tomita K,et al.Differential expression of cyclins B1and B2during medaka (Oryzias latipes)spermatogenesis[J].Zool Sci,2000,17(3):365-374.
[25]Kajiura-Kobayashi H,Kobayashi T,Nagahama Y.The cloning of cyclin B3and its gene expression during hormonally induced spermatogenesis in the teleost,Anguilla japonica [J].Biochem Biophys Res Commun,2004,323:288-292.
[26]Huarte J,Stutz A,O′Connell M L,et al.Transient translational silencing by reversible mRNA deadenylation[J].Cell,1992 69:1021-1030.
[27]de MoorC H,Richter J D.Cytoplasmic polyadenylation elements mediate masking and unmasking of cyclin B1 mRNA[J].EMBO J,1999,18:2294-2303.
[28]Zhang Y,Sheets M D.Analyses of zebrafish and Xenopus oocyte maturation reveal conserved and diverged features of translational regulation of maternal cyclin B1mRNA[J].BMC Dev Biol,2009,9:7.
[29]Nigg E A.Cyclin-dependent protein kinases:key regulators of the eukaryotic cell cycle[J].Bioessays,1995,17(6):471-480.
[30]Basu D,Navneet A K,Dasgupta S,et al.Cdc2-cyclin B-induced G2to M transition in perch oocyte is dependent on Cdc25[J].Biol Reprod,2004,71(3):894-900.
[31]Han K,Dai Y,Zou Z,et al.Molecular characterization and expression profiles of cdc2 and cyclin Bduring oogenesis and spermatogenesis in green mud crab (Scylla paramamosain)[J].Comp Biochem Physiol B Biochemmol Biol,2012,163(3/4):292-302.

