蛋白质界面网络中模体和模块的探测
胡尊胜,林锦贤,吕暾
(1.福州大学 数学与计算机科学学院,福建 福州350108;2.福州大学 生物科学与工程学院,福建 福州350108)
自从Duncan等[1]和Barabási等[2]提出了小世界网络模型和无标度网络模型等概念以来,复杂网络已经有了很大的发展.Albert等[3]研究了复杂网络的统计动力学,Ernesto[4]研究了蛋白质残基网络的拓扑特征.Andrea[5]分析了信息网络、科技网络、社会网络和生物网络,发现同一类网络具有相似的结构特征.由Milo等[6]首次提出的网络模体是一种重要的结构性质,Shen-Orr等[7]研究了基因调控网络,发现一些网络模体是基本的信息处理模块.模块是网络中的社团结构,模块内部联系紧密,模块间联系稀疏,不同的网络有不同的模块特征[8].模体和模块的研究是分析网络结构和性质之间关系的重要方法.为了对不同网络中的模体和模块加以分析,根据Milo提出的网络模体的定义,Kashtan等[9]提出了ESA子图抽样算法,Wernicke等[10]提出了ESU算法来发现网络中的模体;对于加权网络,Sarvenaz等[11]提出了一种考虑边权的模体挖掘方法.对于模块,主要有图形分割方法和分级聚类算法,如Kernighan-Lin算法和派系过滤算法等[12-13].在生物网络中,模体和模块被用来理解一些功能机制.Billur等[14]提出了一种基于蛋白质-蛋白质界面模体的策略来帮助识别药物靶标;Zhang等[15]分析蛋白质-蛋白质网络中的功能模块,促进对脊柱炎发病机理的理解.在蛋白质界面网络中也存在这样的模体和模块,但是这些模体和模块的功能以及与网络的拓扑性质的关系还不清楚.因此,本文在构建蛋白质网络的基础上,研究蛋白质界面残基网络中的模体和模块.
1 材料和方法
1.1 数据集
蛋白质数据取自PDB数据库中非同源的蛋白质复合物,每个复合物中至少有一条链的氨基酸序列长度大于85个.数据集中选取了11个蛋白质复合物的20条肽链,分别是1buw_A,1buw_B,1agr_A,1agr_E,1e96_B,1cgi_E,1avw_A,1avw_B,1ycs_A,1ycs_B,1a4y_B,1cse_I,1d09_B,1stf_E,1stf_I,1a4y_A,1cse_E,1d09_A,1e96_B,1fss_A.这些肽链分α,β,α+β,α/β共4种结构类型,每种类型包括5条肽链.
1.2 构建蛋白质网络
对数据集中的20条肽链构建无向网络模型.将蛋白质肽链中每个残基的CA 原子定义为一个节点,节点集合为V,且|V|=N.计算各个CA原子之间的欧氏距离,定义一个截断距离R,当原子之间的距离小于R时则这两个原子间有边连接;否则,没有边连接.另定义边的集合为E,且|E|=M,得到一个邻接矩阵A.
1.3 构建蛋白质界面网络
蛋白质复合物中肽链之间的相互作用,使蛋白质表现出不同的结构和功能,因此研究肽链之间相互作用的界面特征尤为重要.对于数据集中蛋白质,首先确定两条相互作用的肽链,然后定义一个截断距离S,当分别来自两条肽链间的残基的CA原子之间的距离小于S时,定义这个两个残基之间有边相连,称为界面残基,并对每条肽链的界面残基构建网络.这里定义S为0.7nm时得到的界面残基较合理.以1buw_A链为例,使用RasMol软件显示的界面残基如图1所示.蓝色丝带链为A链,蓝色骨架模式的链为B链.A链中红色球体包括30,31,34和35号残基(右),黄色球体包括106,107,110,111,112和114号残基(中),绿色球体包括119,120,122和123号残基(左).

图1 RasMol软件显示的1buw_A的界面残基Fig.1 Interface residues of 1buw_A in RasMol
1.4 网络的模体和模块的定义
按照Milo提出的精确网络模体的定义来识别模体[6],Palla等的模块定义是基于模块内部节点连接很多,但又不必与模块内部其他所有节点都连接的思想.称一个完全子图为一个k-派系,k是子图的节点数,如果一个k-派系可以通过一系列相邻的k-派系到达另一个k-派系,则它们是连通的,定义k-派系-模块为一系列连通的k-派系的集合.
采用Wernicke提出的ESU和Rand-ESU算法来搜索蛋白质界面网络中的模体.这个算法首先产生一组与真实蛋白质界面网络具有相同度序列的随机网络,再用ESU和Rand-ESU算法搜索子图;然后基于这些子图计算模体的统计意义,可以使用FANMOD软件搜索蛋白质界面网络的模体[16].文中产生1 000个与真实网络有相同度序列的随机网络,令U=5,|Z-score|>2,P=0.05.
采用Palla提出的派系过滤(CPM)算法分析蛋白质界面网络中的模块.在CPM算法中,采用由大到小、迭代回归的算法来寻找网络中的派系,文中使用Cfinder软件搜索蛋白质界面网络中的派系-模块[17].
2 讨论
2.1 蛋白质肽链网络与其界面网络的拓扑性质
首先计算蛋白质肽链网络及其界面网络的度K、聚类系数C、特征路径长度L、介数中心度Bet和接近中心度Clo等拓扑参数[18].当截断距离R=0.7nm时,数据集中蛋白质肽链网络及界面网络的统计性质,如表1所示.表1中:差异比r表示两类网络的参数值之差与肽链网络参数值的比.
从表1可知:界面网络中节点的平均度要小于肽链网络的平均度,而界面网络的平均聚类系数要比肽链网络的大,且网络的特征路径长度差别最明显.这是因为肽链网络中残基要远远多于界面网络中的残基,而边数也要远多于界面网络中的边数,但界面网络中的平均边密度为0.32,要远大于整个肽链网络中的平均边密度0.05.此外,肽链网络中节点的介数中心度都较小,而界面网络中除其中4个外,其他界面网络中节点的介数中心度都小于相应的肽链网络.这是因为界面网络中的节点大部分都在蛋白质界面上.蛋白质肽链网络与其界面网络的接近度中心性差别较小,可知残基是否在界面上对其影响不大.

表1 蛋白质肽链网络及界面残基网络的统计性质Tab.1 Statistical properties of protein peptide networks and protein interface networks
2.2 蛋白质界面网络的模体
对于蛋白质界面网络,数据集中蛋白质界面残基数范围为4~18,可见界面残基数目较少,故模体的阶数取3,4,5,6阶(阶数即模体中的节点数).当不同截断距离R的界面网络中的模体分布,如图2所示.图2中:nM,nN分别为模体和网络数.图3为使用FANMOD软件显示的模体图,其中“↔”表示无向边.

图2 不同截断距离的界面网络模体分布Fig.2 Distributions of motifs in interface networks

图3 使用FANMOD软件显示的模体图Fig.3 Motifs shown by FANMOD
当R=0.5nm时,只有1buw_B,1avw_A,1cse_I,1cse_E,1d09_A界面网络中可以探测到模体.其中:1buw_B,1avw_A,1cse_I,1d09_A界面网络的模体均为3阶模体,如图3(a);而1cse_E界面网络的模体是6阶的,如图3(b)所示.
当R=0.7nm时,有14个蛋白质界面网络中存在模体,其中3阶模体均是3-派系,如图3(c)所示.4阶模体中出现了3种类型(图3(d)),它们出现的频数依次为2,10,4,可见第二种类型的4阶模体出现最多,在界面网络结构中较重要.9个界面网络中存在5阶模体,且它们有一个共同模体(图3(e)),表明这种模体在几个界面网络中较普遍.8个界面网络中存在6阶模体,但它们没有共同模体.
当R=1.2nm时,有15个蛋白质界面网络中存在模体,且每个界面网络中都包含一个4-派系模体(图3(f)),表明4-派系模体普遍存在于界面网络中.此时,界面网络中3阶模体均是3-派系,1cgi_E,1avw_A,1stf_E,1stf_I,1cse_E,1d09_A,1buw_A和1fss_A界面网络中都有3个4阶模体且前6个网络的4阶模体相同,最后两个的4阶模体相同;而1buw_B,1agr_A和1agr_E界面网络中都有4个4阶模体且3个界面网络的4阶模体相同.有13个界面网络中存在5阶模体,且它们有2个共同的模体(图3(g));有14个界面网络中存在6阶模体,且模体个数相对较多,但它们没有共同模体.
当R=2.4nm时,1agr_E,1e96_B,1avw_A,1avw_B,1cse_I,1d09_B,1e96_A链界面网络的各阶模体数均为1,且各阶模体为各阶的k-派系(图3(h));而1d09_A的界面网络中只有一个6阶模体且为6-派系.这是因为这些界面网络中边密度很大,倾向于形成规则网络,只有1buw_B的界面网络中模体较多,其他蛋白质界面网络均无模体.
从上面分析可知,蛋白质界面网络中的模体受截断距离R影响较大,R值不同,则网络中的模体类型和数量都有较大差别.
2.3 蛋白质界面网络的模块
为了给蛋白质界面网络合理的模块划分,要找出合理的截断距离R,并用Cfinder探测蛋白质界面网络里的模块.当R=0.5nm时,只有1stf_I和1cse_E的界面网络中各存在一个3-派系-模块.对比发现这不符合其真实界面,而其他蛋白质界面网络中都没有模块,显然不符合实际.当R=0.7nm时,除1a4y_B外,其他蛋白质界面网络中都可以发现3-派系-模块.
以1buw_A界面网络为例,使用CFinder软件测得其3-派系-模块如图4所示.图4中:1,2,3,4号节点模块对应图1中的红色球体即30,31,34和35号残基;5,6,7,8,9,10号节点模块对应图1中的黄色球体即106,107,110,111,112和114号残基;11,12,13,14号节点模块对应图1中的绿色球体即119,120,122和123号残基.显然,从图4可知此模块划分与实际界面网络中的模块一致.

图4 1buw_A界面网络中的模块划分Fig.4 Modules partition in the interface network of 1buw_A
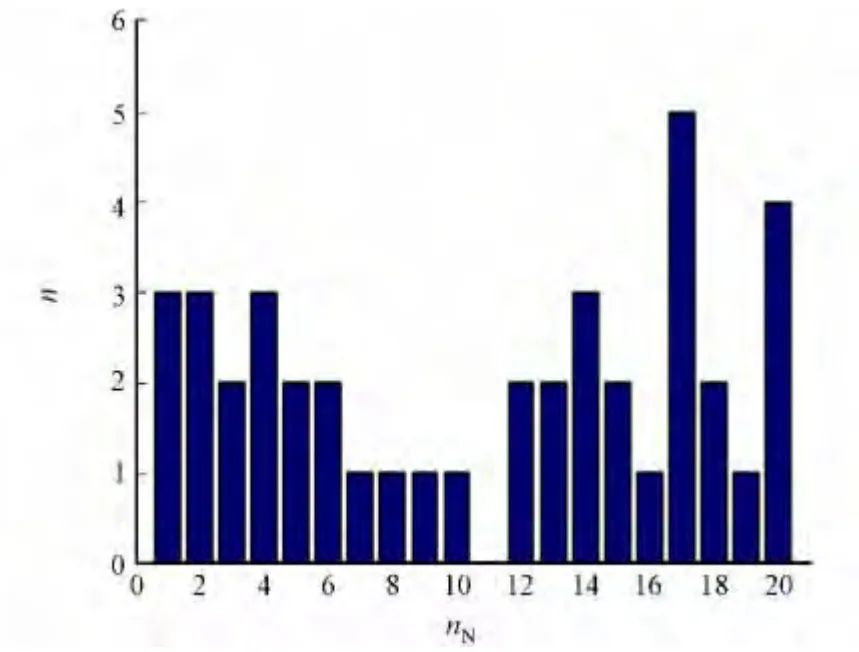
图5 20个蛋白质界面网络的3-派系-模块分布Fig.5 Distribution of 3-clique-modules in 20protein interface networks
此时,20个蛋白质界面网络的3-派系-模块分布,如图5所示.从图5可知:除1a4y_B的界面网络无模块外,其他蛋白质界面网络中均有3-派系-模块.与蛋白质界面对比发现这个划分较合理.
当R=1.2nm时,除1a4y_B的界面网络无模块及1buw_B界面网络有2个3-派系-模块外,其他蛋白质界面网络中只有1个3-派系-模块.这是因为此时的R较大,界面网络中的边密度较大,3-派系均是连通的,故只有1个模块.当R=2.4nm时,所有蛋白质界面网络中都只有一个3-派系-模块,这是因为R较大,网络中的节点倾向于形成规则网络.
由上面的分析可知,当R=0.7nm时的蛋白质界面网络模块划分比较符合实际.
2.4 界面网络模块与其拓扑性质的关系
当R=0.7nm时,分析此时界面网络中的模块与其拓扑性质的关系.蛋白质界面网络中的残基数与3-派系-模块数的关系,如图6所示.由图6可知:二者呈线性关系,拟合直线为y=0.2x-0.15,其中,x为残基数量,y为模块数.显然,界面网络中残基越多,模块越多.

图6 R为0.7nm的蛋白质界面网络残基数(节点数)与3-派系-模块数的关系图Fig.6 Relationship between the number of residues(nodes)and 3-clique-modules(R=0.7nm)
2.5 结果比较分析
基于复杂网络理论研究了20条蛋白质肽链相互作用界面网络与蛋白质肽链网络的拓扑性质的差异,发现网络的平均路径长度和介数中心度差别明显,而二者的介数中心度则差别较小.当截断距离R为0.7,1.2nm时,存在模体的界面网络较多,且多个网络中包含共同的模体.这与韩华等[19]使用FANMOD软件对8个不同规模网络进行模体检测分析所得结论,即验证了网络中模体的存在性相一致.文献[19]重点分析了Karate网络和Dophin网络,得到编号为78和238的3阶模体和编号为13278,8598,4958,31710的模体,而这些模体与本文蛋白质界面网络中部分模体相同,分别为图3的(a),(c),(d)和(f),这证明了本文方法的有效性以及蛋白质界面网络中模体的存在性.
以往的研究多侧重于对蛋白质相互作用网络统计研究,一些算法并未应用到蛋白质界面网络这一层次.梅娟等[20]采用基于模块度优化的图聚类算法,从具有2 617个节点,11 855个相互作用的酵母蛋白相互作用网络中探测出68个模块,但其并未从更深层次分析其作用特征.本文对蛋白质界面网络模块的分析恰好是更深层次的细化,同时,对拓扑性质与模块关系的分析也有利于进一步研究蛋白质界面网络的形成机制等问题.
3 结束语
文中基于复杂网络研究了蛋白质复合物中肽链相互作用界面网络的拓扑性质,讨论了其与蛋白质肽链网络的拓扑性质的差异.对蛋白质界面网络中的模体和模块特征研究发现,蛋白质界面网络中的模体类型和数量受截断距离R影响较大.这些特征分析有利于进一步理解界面网络的结构特征,有助于研究蛋白质分子相互结合的机制,促进蛋白质结合界面预测理论的发展.
然而,如何将蛋白质界面网络中的模体和模块特征应用到蛋白质单体的结合界面残基预测中,即把蛋白质界面网络中的模体和模块特征与打分函数相结合,从而提高预测的准确率,将是今后研究的一个重要课题.
[1]DUNCAN J W,STEVEN H S.Collective dynamics of“small-world”networks[J].Nature,1998,393(6684):440-442.
[2]BARABÁSI A L,ALBERT R.Emergence of scaling in random networks[J].Science,1999,286(5439):509-512.
[3]ALBERT R,BARABÁSI A L.Statistical mechanics of complex networks[J].Review of Modern Physics,2002,74(1):47-97.
[4]ESTRADA E.Universality in protein residue networks[J].Biophysical Journal,2010,98(5):890-900.
[5]LANCICHINETTI A,KIVELA M,SARAMÄKI J,et al.Charaterizing the community structure of complex networks[J].PLOS ONE,2010,5(8):e11976.
[6]MILO R,SHEN-ORR S S,ITZKOVITZ S.Network motifs:Simple building blocks of complex networks[J].Science,2002,298(5594):824-827.
[7]SHEN-ORR S S,MILO R,MANGAN S,et al.Network motifs in the transcriptional regulation network ofEscherichiancoli[J].Nature Genetics,2002,31(1):64-68.
[8]KELLER I,VIENNET E.A characterization of the modular structure of complex networks based on consensual communities[C]∥Eighth International Conference on Signal Image Technology and Internet Based Systems.Naples:IEEE Press,2012:717-724.
[9]KASHTAN N,ITZKOVITZ S,MILO R,et al.Efficient sampling algorithm for estimating subgraph concentrations and detecting network motifs[J].Bioinformatics,2004,20(11):1746-1758.
[10]WERNICKE S.A faster algorithm for detecting network motifs[C]∥Proceedings of the 5th Workshop on Algorithms in Bioinformatics.Spain:Springer Verlag,2005:165-177.
[11]CHOOBDAR S,RIBEIRO P,SILVA F.Motif mining in weighted networks[C]∥IEEE 12th International Conference on Data Mining Workshops.Brussels:IEEE Press,2012:210-217.
[12]KERNIGHAN B W,LIN S.A efficient heuristic procedure for partitioning graphs[J].Bell System Technical Journal,1970,49(2):292-307.
[13]PALLA G,DERÉNYI I,FARKAS I,et al.Uncovering the overlapping community structure of complex networks in nature and society[J].Nature,2005,435(7043):814-818.
[14]ENGIN H B,KESKIN O,NUSSINOV R,et al.A strategy based on protein-protein interface motifs may help in identifying drug off-targets[J].Journal of Chemical Information and Modeling,2012,52(8):2273-2286.
[15]ZHANG C,SHEN L.Functional modules analysis based on protein-protein network analysis in ankylosing spondylitis[J].European Review for Medical and Pharmacological Sciences,2012,16(13):1821-1827.
[16]WERNICKE S,RASCHE F.FANMOD:A tool for fast network motif detection[J].Bioinformatics,2006,22(9):1152-1153.
[17]ADAMCSEK B,PALLA G,FARKAS I,et al.CFinder:Locating cliques and overlapping modules in biological networks[J].Bioinformatics,2006,22(8):1021-1023.
[18]de NOOY W,MRVAR A,BATAGELJ V.Exploratory social network analysis with Pajek[M].Cambridge:Cambridge University Press,2005:1-362.
[19]韩华,刘婉璐,吴翎燕.基于模体的复杂网络测度量研究[J].物理学报,2013,62(16):168904(1-9).
[20]梅娟,纪志成.基于模块度优化的蛋白质网络集团探测与分析[J].计算机与应用化学,2012,29(5):591-596.

