马铃薯Y病毒福建分离物P1基因的分子变异和结构特征
史凤阳,高芳銮,沈建国,常飞,詹家绥
1.福建农林大学植物病毒研究所,福建省植物病毒学重点实验室,福州 350002;2.福建出入境检验检疫局检验检疫技术中心,福州 350001
病原物群体遗传变异及其演化机制直接影响植物病害的发生、发展及控制。在寄主、病原和环境的病害三角中,病原物承受着来自寄主和环境的双重选择。为了增加对新的抗病寄主和新环境的适应性,病原物通过突变、基因流和基因重组不断引进和产生新的变异。在自然选择下,有益变异得到积累,有害变异则被淘汰[1]。由于寄主和病原物本身都是由复杂的、动态结构的个体组成的,随着病原物群体结构的时空变化,这种特定的寄主——病原物关系也随之发生改变。马铃薯Y病毒(Potato virus Y,PVY)是目前感染马铃薯并造成最严重经济损失的病毒之一。PVY寄主范围广,在全球马铃薯种植区广泛流行,在我国马铃薯种植区及亚洲地区周边国家也普遍发生,并呈上升发展趋势[2,3]。PVY是马铃薯Y病毒科(Potyviridae)马铃薯Y病毒属(Potyvirus)的重要成员之一[4],其基因组由正单链RNA分子组成,大小约 10 kb,包含一个大的开放阅读框(Open reading frame,ORF)。PVY基因组采用多聚蛋白切割(Polyprotein cleavage)和移码翻译(Read frame shift)方式最终生成 11个成熟的多功能蛋白[5,6],使该病毒呈现不同程度的遗传多样性。根据是否发生基因重组,PVY株系分为两大类:一类是非重组株系,包括PVYO、PVYC和 PVYN株系; 另一类是PVYO和PVYN在不同基因区段发生重组的株系,即 N×O重组株系,如:PVYNTN、PVYN-Wi(北美称 PVYN:O)、PVYNTN-NW[7,8]。由于N×O重组株系具备了非重组株系的其他适应性优势,表现出比PVYO株系和PVYN株系更强的致病力,如:PVYNTN株系侵染马铃薯后,可以引发马铃薯块茎环斑坏死病(Potato tuber necrotic ringspot disease,PTNRD),导致马铃薯种质退化、产量降低。目前,PVY重组株系已在全球的不同地区蔓延[9]。由于 PVY群体遗传多样性高,株系分化严重,具有相对强的生存和进化优势,可以快速适应新寄主(或生存环境)。随着近年来农产品贸易的全球化,PVY变异频繁,不断有新的重组株系产生,对马铃薯生产造成极大损失[10],严重时减产高达80%以上[11]。因此,开展PVY功能基因的分子多样性研究有利于探索变异、重组等遗传机制在PVY进化中的作用,也有助于了解植物病毒的发生、流行和防控特点,具有重要的理论和现实意义。
对于P1基因的研究侧重于蛋白质组学、生物化学和系统发育学方面[12],在群体遗传学领域,P1基因的研究多局限于对遗传多样性的描述,对该基因分子变异、P1蛋白3D结构的影响及遗传多样性形成的遗传机制研究较少。如:吴兴泉等[13]比较了 1个福建分离物与其他已发表的33个分离物的P1蛋白氨基酸序列同源性,发现氨基酸序列在不同分离物之间存在明显差异; Hu 等[14]研究表明,PVY全基因组存在5个显著的重组热点,其中P1基因可能是重组的热点区之一; Wang等[15]通过分析烟草上的两个山东PVY分离物AQ4和FZ10的分子特征,发现两个分离物的P1基因均为N×O重组型; 张俊祺等[16]对烟草上一个贵州PVY分离物的P1基因进行了克隆和序列分析,表明该分离物的变异来自碱基位点的突变而非基因重组。以上发现虽然为了解PVY进化奠定了基础,但许多结果还没有得到其他数据的有效验证。
福建省是中国最早种植马铃薯的地区之一,也是南方马铃薯冬种的优势区和主产区。对于福建省PVYP1基因遗传多样性及其形成的遗传机制与我国乃至世界其他地区PVYP1基因的异同性尚未有系统的报道。为此,本文在前期检测的53份福建省PVY阳性样品中[3],对采自福州、宁德、龙岩、漳州4个地区各3个样品,通过克隆获得的12个PVY分离物P1基因的全长 cDNA序列,并应用生物信息学方法对其核苷酸序列、氨基酸序列的分子变异及结构特征等开展系列分析,旨在探索福建PVYP1基因的遗传变异特征以及明确突变和重组等遗传机制在该基因进化的作用,为PVY病毒病的流行、变异趋势及其有效防控奠定基础。
1 材料和方法
1.1 材料
马铃薯病株于2011年~2012年之间采自福建省马铃薯主要种植区,在前期研究共检测到的 53份PVY阳性样品中[3],分别从福州、宁德、龙岩、漳州 4个地区采用随机抽样法各抽取 3个样品,共计12个福建分离物用于本研究。
1.2 方法
1.2.1 RNA提取及P1基因克隆
采用 Trizol 试剂法从感染 PVY的马铃薯病叶中提取总 RNA,提取方法参照 RNA Simple Total RNA 试剂盒(TianGen)说明书进行。取2 μL 总RNA用 oligo(dT)18为引物按照操作说明书进行反转录,获得 PVY全长的 cDNA。根据 GenBank已报道的PVY常见株系P1基因序列保守区,设计一对用于扩增该基因的简并引物 P1-F(5′-CVATGGCAAYYTACAYGTCAAC-3′)和 P1-R(5′-AGGRTATCTCADYHGTGCCC-3′),预期片段大小为 915 bp,引物由南京金斯瑞生物技术有限公司合成。
PCR 扩增采用 25 μL 反应体系:10×TransTaqHiFi Buffer II 2.5 μL,dNTPs(2.5 mmol/L) 2 μL,P1-F(10 μmol/L)1 μL,P1-R(10 μmol/L)1 μL,ddH2O 34.5 μL,TransTaqHiFi Polymerase(5 U/μL) 0.5 μL,cDNA 2 μL。PCR反应条件为:94℃预变性4 min;94℃变性30 s,53℃复性30 s,72℃延伸1 min,共30个循环; 最后一轮循环后72℃延伸5 min。
PCR反应结束后,取产物5 µL用1%琼脂糖凝胶电泳进行检测。PCR产物经胶回收试剂盒纯化后与 pEASY-T5 Zero 克隆载体连接,并转化到Trans1-T1感受态细胞中。筛选得到阳性克隆子,随机选择其中3~6个由南京金斯瑞生物技术有限公司测序列,并根据测序峰图及序列比对分析排除由PCR扩增引起的突变。
1.2.2P1基因的分子变异
测序获得的序列使用 DNAMAN等软件进行处理,P1基因的核苷酸使用 MEGA软件中的 Muscle(Codons) 子程序进行多重比对,并根据对应的编码氨基酸序列进行校正。序列同源性使用BLAST(http://blast.ncbi.nlm.nih.gov/)工具在线比对,分子变异通过DnaSP、MEGA5等软件进行分析。
1.2.3P1基因的重组分析
以文献报道的PVY 分离物 Oz(EF026074) 和Mont(AY884983)作为参考亲本株系[14],并以分离物N-605 作为备选参考株系[2]。使用SimPlot 3.5进行潜在重组事件的相似性作图(Similarity plot)和Bootscanning分析[17]。使用遗传算法重组检测法(Genetic Algorithm Recombination Detection,GARD)进行重组位点的检测并评估重组点的可靠性[18]。
1.2.4P1基因的系统发育分析
为进一步了解 PVYP1基因的分子进化,从GenBank中选取20个已知株系的PVYP1基因序列作为参考(表1),使用贝叶斯法(Bayesian inference,BI)进行PVYP1基因的系统发育分析。建树前,使用 MAFFT[19]软件对建树的 32条P1基因序列进行多重序列比对,利用Mrmodeltest[20]选择最优化的进化模型—GTR+I模型。使用 Mrbayes3.04 b[21]进行BI法分析时,根据 Mrmodeltest的 AIC(akaike information criterion)标准,替换模型为nst = 6,位点间变异速率设置为rates = propinv,建立4个马尔可夫链,以随机树为起始树,共运行2 000 000代。每100代抽样1次,舍弃25%老化样本后,根据剩余的样本构建一致树,并计算后验概率(Posterior probability)。
1.2.5 P1蛋白的序列特征
通过 ExPASy的 ProSITE数据库(http:// prosite.expasy.org/)搜索P1蛋白中可能存在的Motif。为确保重要信息不被遗漏,避免发生单一数据库中不能被检测到的错误,Motif搜索时,联网至Profile数据库(http://www.biochem.ucl.ac.uk/bsm/dbbrowser/jj/pfscan2.html)进行蛋白质序列轮廓搜索(Profile Search); 通过 COILS Server(http://www.ch.embnet.org/software/COILS_form.html)分析P1蛋白是否含有卷曲螺旋(Coiled-coil,CC)结构; P1蛋白的结构功能域通过SMART4.0服务器搜索; P1蛋白的三级结构及功能位点通过 I-TASSER在线服务器(http://zhanglab.ccmb.med.umich.edu)进行预测。

表1 PVY参考序列及其GenBank登录号
2 结果与分析
2.1 P1基因的序列分析
使用简并引物P1-F/P1-R扩增到P1基因的PCR产物与预计的目的片段大小一致,约 915 bp(图1),12个分离物及阳性对照均扩增到目的片段,阴性(健康马铃薯叶片)和空白对照均未扩增到相应的片段。
RT-PCR扩增到的目的片段克隆、测序后经DNAMAN、BLAST等分析,确定目的基因片段大小为 915 bp,其中1~825 bp为PVY P1蛋白(First protein)的编码基因序列(GenBank登录号分别为 KF722800、KF722802、KF722806、KF722808、KF722809、KF722811、KF722814、KF722819、KF722826、KF722828、KF722831和KF722833),编码275个氨基酸的P1蛋白。序列同源性分析显示,福建 12个 PVY分离物P1基因与8个PVY不同株系参考核苷酸序列一致性为 73%~99%(表 2),其中分离物 XT04、LH14、LH21、LY08、LY35、XQ04、ZL02与Oz的序列一致性最低(73%~74%),与 Mont、HN1、Mb112、Wilga5、HN2的序列一致性最高(98%~99%); 分离物 QK44、XT02、XT08、LH05与 Wilga5的序列一致性最高(95%~99%),而与其他分离物的一致性为77%~90%;分离物LY30与Oz的序列一致性最高(99%),而与其他分离物序列一致性均不高(74%~84%)。通过DNAMAN分析可知,福建分离物P1序列之间核苷酸序列一致性为 73%~99%。一致性分析结果表明,P1基因在福建不同分离物之间存在较大差异。

图1 福建分离物P1基因RT-PCR扩增
翻译后的 P1蛋白存在 85个(占 31%)变异的氨基酸位点,其中 LY08、LY35、XT02和 XT08分离物各有一个特异性的氨基酸变异位点,ZL02分离物含有两个特异性的氨基酸变异位点,LH05分离物有4个特异性的氨基酸变异位点,而LY30分离物则有30个特异性的氨基酸变异位点,显示出与其他分离物显著的差异(表3)。

表2 PVY不同分离物P1基因核苷酸序列一致性(%)

表3 福建省PVY分离物P1基因特异氨基酸突变位点
2.2 P1基因的重组分析
通过Simplot软件分析(图2:A和B),发现分离物 QK44、XT02、XT08、LH05中均检测一个潜在的重组信号,表明分离物QK44、XT02、XT08、LH05的P1基因序列可能是PVYN和 PVYO的重组而成。进一步通过GARD 确认这4个分离物存在两个重组位点(图2C):(1)309位核苷酸的重组位点平均模型支持率达99.32%,且KH检验显示P= 0.0040 < 0.01;(2)423位核苷酸的重组位点的平均模型支持率为36.52%,且KH检验显示P= 0.0040 < 0.01。
2.3 P1基因的系统发育分析
重建的系统发育树结果显示(图3A),在PVY基因组的P1基因区段上,相同株系型的分离物以较高的置信值优先相聚成簇,福建分离物12个分离物形成3个簇,LY30分离物与O株系型(O Clade)的Oz、SASA110分离物聚为一簇(Cluster 3),QK44、XT02、XT08、LH05分离物与 N×O 重组型(N×O Clade)的SYR-II-2-8、3401、Wilga5等分离物聚为一簇(Cluster 2),而其他的7个分离物与N株系型(N Clade)的分离物聚为一簇(Cluster 1)。
2.4 P1蛋白的结构特征
COILS Server分析结果显示,12个PVY福建分离物中,XT04、LH14、LH21、LY08、LY35、XQ04、ZL02分离物P1蛋白的70~98位氨基酸之间均检测到一个明显的卷曲螺旋 CC domain(图 3B),而其他分离物也在相似位置检测到 CC domain,但置信值不高。
通过 SMART服务器搜索,发现 P1蛋白第41~275氨基酸是一个高度保守的结构功能域—Peptidase_S30,为PotyvirusP1 protease家族成员共有的典型结构域。P1蛋白的C′端区域是一类催化裂解 P1/HC-pro蛋白的丝氨酸蛋白酶(Serine-type protease),Petpidase_S30主要由多个Potyvirus的P1蛋白的C′端区域组成。虽然PVY不同分离物的P1蛋白氨基酸序列差异较大,进一步通过 Motif搜索发现 P1蛋白内存在 7个保守的 Motif:I-x-F-G、V-E-L-I、I-S-I-x-G-G、F-L-x-G、H-x(8)-D/E、G-x-S-G和R-G,其中Motif H-x(8)-D/E和G-x-S-G包含丝氨酸蛋白酶Peptidase_S30的3个活性位点(H192、D201、S235)(图 4:A 和 C)。

图2 重组的检测和验证

图3 分支间的P1基因的特征比较
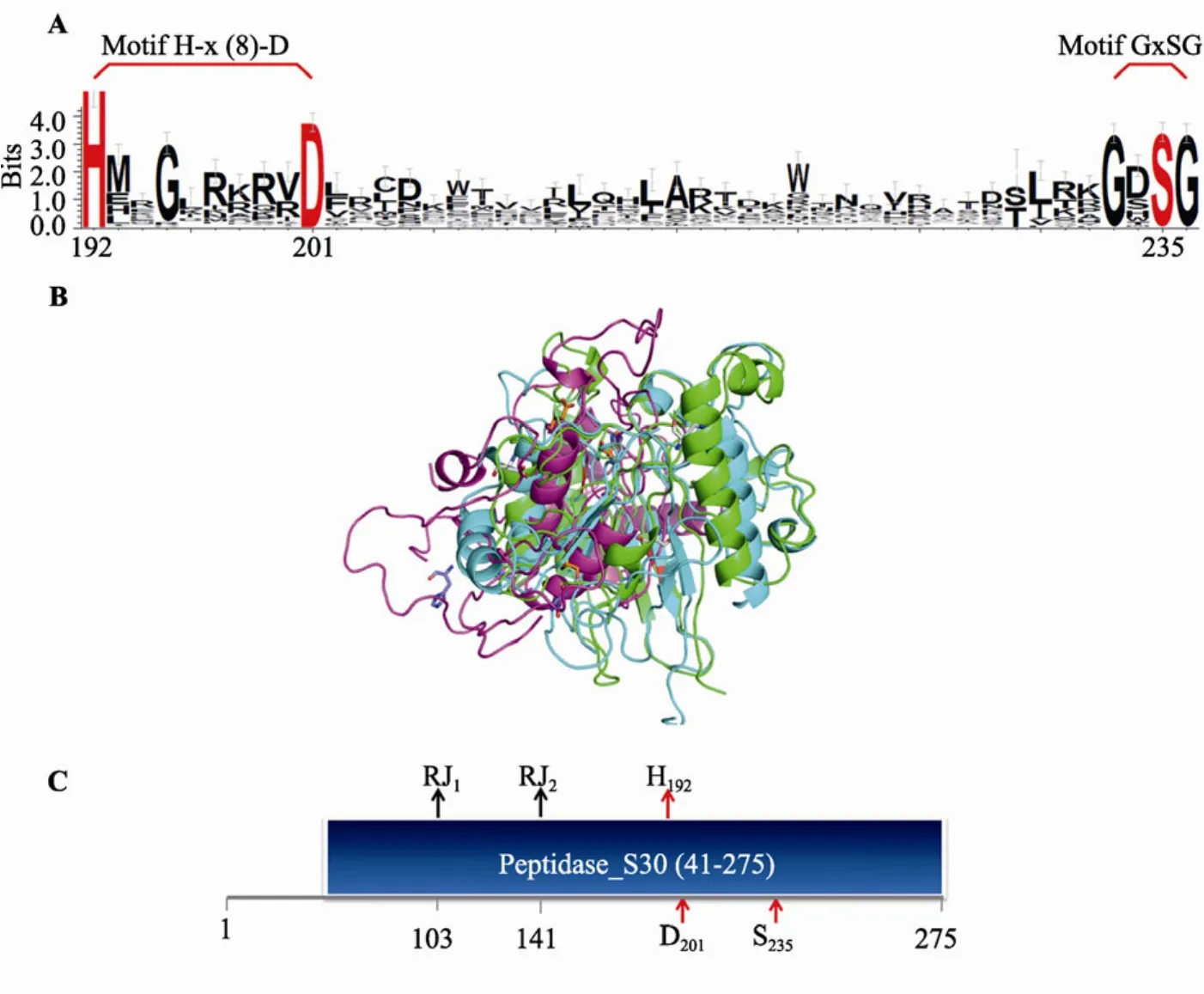
图4 P1蛋白结构预测
I-TASSER服务器返回 P1蛋白三级结构,结果显示12个福建分离物的P1蛋白存在3种不同的空间结构,经PyMOL渲染后如图3C所示,其中分离物 XT04、LH14、LH21、LY08、LY35、XQ04、ZL02的P1蛋白三级结构为N Clade型; 分离物QK44、XT02、XT08、LH05的P1蛋白三级结构为N×O Clade型; LY30分离物的P1蛋白三级结构为O Clade型。N Clade型和O Clade 型的P1蛋白三级结构较为接近,但是N×O Clade型的P1蛋白三级结构与前两者差别大(图4B),且P1蛋白的3个活性位点(H192、D201和S235)在空间上相距较远。
3 讨 论
3.1 P1基因的遗传变异及分子进化
高度变异性是RNA病毒的典型特征之一,主要由于依赖 RNA 的 RNA 聚合酶(RdRp)或复制酶缺乏校正功能,使得 RNA病毒具有非常高的突变率(约为 10-4个核苷酸/复制循环),这也是 RNA 病毒的一种进化策略[22,23]。前人研究表明,P1蛋白不论在长度及氨基酸序列上是所有Potyvirus中分化最明显的一个蛋白[24]。ICTV报告规定同一种病毒的全基因组核苷酸序列的同源性不低于85%,3′端非编码区序列同源性不低于75%,CP蛋白的氨基酸序列同源性不低于80%[4]。PVY福建分离物P1基因具有高度的变异性,12个不同分离物P1基因之间以及与已知株系的其他分离物核苷酸序列一致性均在 73%~99%之间(表 2),已跨越了Potyvirus划分种的序列一致性阈值[4],Valli等[24]认为PotyvirusP1基因的高度变异性与其适应不同的寄主密切相关。
在Potyvirus中,基因重组是病毒进化的一个重要机制[25],通过基因重组可以产生大量的遗传变异,这远比仅仅由突变造成的变异来得更快[26]。除了高突变率外,基因重组在 PVY 基因组中也频繁发生。Simplot和 GARD重组分析结果,显示福建分离物QK44、XT02、XT08、LH05分离物的P1基因存在明显的基因重组现象,即由PVYN和PVYO株系在P1基因区段不同位置发生重组(图2)。在4个分离物中,GARD检测到两个重组位点(309 nt和423 nt),其中 309 nt的重组位点平均模型支持率达99.32%,经KH检验极其显著(P< 0.01),表明该位点为福建分离物P1基因重组位点的置信度非常高。综合P1基因的分子变异分析结果,表明福建分离物P1基因的变异主要源自碱基位点的突变和基因重组,说明两者在 PVY进化中起着重要的作用,是 PVY新株系不断形成的主要因素。
系统发育分析显示(图3A),PVYP1基因分化明显,12个福建分离物形成3种不同的大簇,故而单独使用P1基因序列为分子标记进行系统发育分析,无法对 PVY 重组株系进行准确鉴定,如图 3A 中HN1分离物(PVYNTN株系)和 HN2分离物(PVYNTN-NW株系),两者分属不同的株系,但因在P1基因区段上均为 N株系型(N Clade)而聚为一簇(Cluster 1)。当笔者联合P1和CP基因分析显示,12个福建分离物中仅 LY30为 PVYO株系,而其他11个福建分离物均为 N×O重组株系(未发表数据),表明重组株系已成为福建PVY的主流。因此,生产中需要密切关注这些分离物的发展动态。
3.2 P1蛋白的结构特征
CC domain是蛋白质中一类特殊的α螺旋链互相缠绕形成的平行或反平行同寡聚体或异寡聚体结构的总称,是控制蛋白质寡聚化的元件[27]。前人研究表明,CC domain 在病毒复制、致病性以及增强病毒RNA沉默抑制子(Viral suppressor of RNA silencing,VSR)活性中起着重要的作用[28~30]。P1基因位于PVY基因组的起始位置,编码丝氨酸蛋白酶(P1 peptidase),是病毒基因复制中的一个辅助因子,在致病性和增强VSR等方面也起着重要的作用[31~33]。通过Coiled-coil server分析结果显示,N Clade型的分离物含有典型的CC domain,而O Clade型和N×O Clade型分离物的CC domain则不明显,这可能是不同株系侵染寄主时产生表现不同病症的原因。
虽然 P1蛋白在氨基酸序列上存在高度的变异性(表3),但还是存在多个保守的 Motif(图 4A),且行使P1蛋白功能密切相关的3个活性位点所在的氨基酸残基均保持不变(图 4A),从而保证该蛋白酶发挥正常的功能。比较N Clade、N×O Clade和O Clade 3种类型的P1蛋白活性位点空间位置(图4B),显示3个活性位点在空间位置相距较远,可能影响P1蛋白酶的活性强弱。蛋白质的生物学功能在很大程度上取决于蛋白质的空间结构,三级结构是蛋白质结构预的最终目的[34]。弄清PVY P1蛋白质的结构特征,对于了解P1蛋白结构与其功能的关系具有重要的意义。
本文通过扩增、克隆12个PVY福建分离物P1基因的cDNA全长序列,并通过系列分析表明PVYP1基因的变异源主要来自碱基突变,但部分分离物也存在明显的基因重组现象。同时,重组株系已成为福建马铃薯主栽区的主流。虽然P1基因高度变异,但行使 P1 proteinase 活性位点所在的 3个氨基酸(H192、D201和 V235)却高度保守,从而保证该蛋白酶发挥正常的功能。
[1]祝雯,詹家绥.植物病原物的群体遗传学.遗传,2012,34(2):157–166.
[2]Ali MC,Karasev AV,Furutani N,Taniguchi M,Kano Y,Sato M,Natsuaki T,Maoka T.Occurrence ofPotato virus Ystrain PVYNTNin foundation seed potatoes in Japan,and screening for symptoms in Japanese potato cultivars.Plant Pathol,2013,62(5):1157–1165.
[3]高芳銮,沈建国,史凤阳,方治国,谢联辉,詹家绥.中国马铃薯Y病毒的检测鉴定及CP基因的分子变异.中国农业科学,2013,46(15):3125–3133.
[4]King AMQ,Lefkowitz E,Adams MJ,Carstens EB.Virus Taxonomy:ninth report of the international committee on taxonomy of viruses.Amsterdam:Elsevier Academic Press,2011.
[5]Chung BY,Miller WA,Atkins JF,Firth AE.An overlapping essential gene in thePotyviridae.Proc Natl Acad Sci USA,2008,105(15):5897–5902.
[6]高芳銮,沈建国,史凤阳,常飞,谢联辉,詹家绥.马铃薯Y病毒pipo基因的分子变异及结构特征分析.遗传,2013,35(9):1125–1134.
[7]Singh RP,Valkonen JPT,Gray SM,Boonham N,Jones RAC,Kerlan C,Schubert J.Discussion paper:the naming ofPotato virusYstrains infecting potato.Arch Virol,2008,153(1):1–13.
[8]Ali MC,Maoka T,Natsuaki T,Natsuaki KT.PVYNTN-NW,a novel recombinant strain ofPotato virus Ypredominating in potato fields in Syria.Plant Pathol,2010,59(1):31–41.
[9]Quenouille J,Vassilakos N,Moury B.Potato virus Y:a major crop pathogen that has provided major insights into the evolution of viral pathogenicity.Mol Plant Pathol,2013,14(5):439–452.
[10]Rahman MS,Akanda AK.Performance of seed potato produced from sprout cutting,stem cutting and conventional tuber against PVY and PLRV.Bangladesh J Agril Res,2009,34(4):609–622.
[11]Whitworth JL,Nolte P,McIntosh C,Davidson R.Effect ofPotato virus Yon yield of three potato cultivars grown under different nitrogen levels.Plant Dis,2006,90(1):73–76.
[12]Rohožková J,Navrátil M.P1 peptidase–a mysterious protein of family Potyviridae.J Biosci,2011,36(1):189–200.
[13]吴兴泉,陈士华,吴祖建,林奇英,谢联辉.马铃薯 Y病毒 P1基因的克隆与序列分析.中国病毒学,2003,18(4):376–380.
[14]Hu X,Karasev AV,Brown CJ,Lorenzen JH.Sequence characteristics ofPotato virus Yrecombinants.J General Virol,2009,90(12):3033–3041.
[15]Wang B,Jia JL,Wang XQ,Wang ZY,Yang BH,Li XD,Zhu XP.Molecular characterization of two recombinantPotato virus Yisolates from China.Arch Virol,2012,157(2):401–403.
[16]张俊祺,代昌明,杨军,宋纪真,罗朝鹏,王燃,林福呈.马铃薯 Y病毒贵州黔西烟草分离物 P1基因序列分析.植物病理学报,2012,42(1):88–92.
[17]Lole KS,Bollinger RC,Paranjape RS,Gadkari D,Kulkarni SS,Novak NG,Ingersoll R,Sheppard HW,Ray SC.Full-length human immunodeficiency virus type 1 genomes from subtype C-infected seroconverters in India,with evidence of intersubtype recombination.J Virol,1999,73(1):152–160.
[18]Kosakovsky PSL,Posada D,Gravenor MB,Woelk CH,Frost SD.GARD:a genetic algorithm for recombination detection.Bioinformatics,2006,22(24):3096–3098.
[19]Katoh K,Standley DM.MAFFT multiple sequence alignment software version 7:improvements in performance and usability.Mol Biol Evol,2013,30(4):772–780.
[20]Nylander JAA.MrModeltest v2.3.Program distributed by the author.Evolutionary Biology Centre,Uppsala University,2008.
[21]Ronquist F,Teslenko M,van der Mark P,Ayres DL,Darling A,Höhna S,Larget B,Liu L,Suchard MA,Huelsenbeck JP.MrBayes 3.2:Efficient Bayesian phylogenetic inference and model choice across a large model space.Syst Biol,2012,61(3):539–542.
[22]Malpica JM,Fraile A,Moreno I,Obies CI,Drake JW,García-Arenal F.The rate and character of spontaneous mutation in an RNA virus.Genetics,2002,162(4):1505–1511.
[23]Gibbs A,Ohshima K.Potyvirusesand the digital revolution.Annu Rev Phytopathol,2010,48(1):205–223.
[24]Valli A,López-Moya JJ,García JA.Recombination and gene duplication in the evolutionary diversification of P1 proteins in the familyPotyviridae.J Gen Virol,2007,88(Pt3):1016–1028.
[25]Tugume AK,Mukasa SB,Kalkkinen N,Valkonen JP.Recombination and selection pressure in the ipomovirus sweet potato mild mottle virus(Potyviridae) in wild spe-cies and cultivated sweetpotato in the centre of evolution in East Africa.J Gen Virol,2010,91(Pt4):1092–1108.
[26]Chao L.Fitness of RNA virus decreased by Muller's ratchet.Nature,1990,348(6300):454–455.
[27]Alminaite A,Halttunen V,Kumar V,Vaheri A,Holm L,Plyusnin A.Oligomerization of hantavirus nucleocapsid protein:analysis of the N-terminal coiled-coil domain.J Virol,2006,80(18):9073–9081.
[28]Du YM,Zhao JP,Chen TY,Liu Q,Zhang HL,Wang Y,Hong YG,Xiao FM,Zhang L,Shen QH,Liu YL.Type I J-domain NBMIP1 proteins are required for both tobacco mosaic virus infection and plant innate immunity.PLoS Pathog,2013,9(10):e1003659.
[29]van WR,Dong XL,Liu HT,Tien P,Stanley J,Hong YG.Mutation of three cysteine residues inTomato yellow leaf curl virus-China C2 protein causes dysfunction in pathogenesis and posttranscriptional gene-silencing suppression.Mol Plant Microbe Interact,2002,15(3):203–208.
[30]van WR,Liu HT,Wu ZR,Stanley J,Hong YG.Contribution of the zinc finger to zinc and DNA binding by a suppressor of posttranscriptional gene silencing.J Virol,2003,77(1):696–700.
[31]Verchot J,Carrington JC.Evidence that the potyvirus P1 proteinase functions in trans as an accessory factor for genome amplification.J Virol,1995,69(6):3668–3674.
[32]Pruss G,Ge X,Shi XM,Carrington JC,Bowman Vance V.Plant viral synergism:the potyviral genome encodes a broad-range pathogenicity enhancer that transactivates replication of heterologous viruses.Plant Cell,1997,9(6):859–868.
[33]Brigneti G,Voinnet O,Li WX,Ji LH,Ding SW,Baulcombe DC.Viral pathogenicity determinants are suppressors of transgene silencing inNicotiana benthamiana.EMBO J,1998,17(22):6739–6746.
[34]吴祖建,高芳銮,沈建国.生物信息学分析实践.北京:科学出版社,2010:129–138.