玉米苗期SR蛋白基因家族的干旱胁迫应答
李娇, 郭予琦, 崔伟玲, 许爱华, 田曾元
郑州大学生命科学学院, 郑州 450001
玉米苗期SR蛋白基因家族的干旱胁迫应答
李娇, 郭予琦, 崔伟玲, 许爱华, 田曾元
郑州大学生命科学学院, 郑州 450001
基因表达的选择性剪接(Alternative splicing, AS)调控与植物对逆境胁迫应答密切相关, SR蛋白(Serine/ arginine-rich proteins)是其中关键的调节因子。文章对玉米B73参考基因组进行分析显示: 多数SR蛋白家族基因成员启动子区域含有3~8种与发育或胁迫相关的顺式调控元件; 27个基因成员编码碱性蛋白, 其中23个成员的编码蛋白依照其N′端的首个RRM(RNA recognition motif)结构域特征大体上可划分为5个亚组。利用双向分级聚类方法, 对三叶期干旱胁迫下玉米杂交种郑单958及其亲本郑58和昌7-2的SR蛋白基因家族的分析显示,该基因家族的表达模式具有明显的组织表达特异性和基因型依赖性特征; 其中在干旱胁迫下地下组织以下调表达模式为主, 而地上组织中以上调表达模式为主。在重度干旱胁迫后的3个不同时段复水过程中, 地上和地下组织中SR蛋白基因家族的表达皆以下调表达模式为主。另外, 尽管不同基因成员的表达模式在干旱胁迫及其后的复水过程中存在明显差异, 但普遍存在自身选择性剪接现象。SR蛋白基因家族在玉米干旱胁迫的应答规律, 为从AS-network视角解析玉米的抗逆分子机制提供了新思路。
玉米干旱胁迫; SR蛋白基因; 表达模式; 选择性剪接
选择性剪接(Alternative splicing, AS)又叫可变剪接, 指某真核生物基因转录出的 pre-mRNA通过不同的剪接方式和剪接位点的选择调节, 形成不同的成熟mRNA的过程。以不同的成熟mRNA为模板翻译出不同的蛋白质变体形式, 能够丰富蛋白质组的多样性和基因表达调控的灵活性[1]。研究表明, 植物中选择性剪接的调节与逆境胁迫的适应机制密切相关[2,3]: 拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、大麦(Hordeumvulgare)和小麦(Triticumaestivum)等物种的抗逆基因在环境胁迫条件下会发生选择性剪接; 细胞信号转导相关基因可以通过选择性剪接方式的改变使植物适应干旱、高盐、涝渍和低温等逆境胁迫。干旱是严重影响玉米(Zea mays)生长发育的主要逆境因素。玉米耐旱性作为一种数量性状, 不仅涉及生理、遗传、农艺等众多因素, 也涉及胁迫响应的大量转录因子基因及其下游受控基因的选择性剪接调节[4]。
选择性剪接机制受多种顺式调控元件和反式作用因子互作的调节, 包括 SR蛋白(Serine/argininerich proteins)家族和 hnRNP 蛋白(heterogeneous nuclear RNP)家族在内的多种剪接因子参与的拮抗作用。SR蛋白成员都具有一个富含丝氨酸/精氨酸(S/R)重复序列的RS结构域(RS domain), 在RNA剪接体的组装和选择性剪接的调控过程中具有重要的作用。绝大多数SR蛋白是生存的必需因子, 通过其RS结构域和特有的RRM(RNA recognition motif)结构域, 实现与前体mRNA的特异性序列或其他剪接因子的相互作用, 协同完成剪接位点的正确选择或促进剪接体的形成。SR蛋白往往作为正调控因子与外元剪接增强子(Exon splicing enhancer, ESE)结合,而 hnRNP则作为负调控因子与外元剪接沉默子(Exon splicing silencer, ESS)结合, 共同构筑了真核生物基因表达的AS调控网络(AS-network)[5]。研究表明, 拟南芥中19个SR蛋白基因家族成员的表达具有发育和组织特异性, 并受非生物胁迫和植物激素影响, 其中的15个成员自身也存在选择性剪接现象[6]。尽管Richardson等[7]对27个真核生物(包括玉米)的 SR蛋白基因家族成员进行了分类和比较, 但目前对玉米 SR蛋白基因家族不同成员在干旱胁迫下的差异表达模式及其自身选择性剪接与下游基因胁迫应答尚缺乏系统分析。玉米B73自交系基因组测序工作的完成, 为利用生物信息学手段开展玉米抗逆分子机制研究提供了契机[8]。郑单958是近20年来全国推广面积最大的高产、稳产和广适的玉米单交种。本文在系统分析玉米B73的SR蛋白基因家族成员的系统进化和启动子区域顺式调控元件特征的基础上, 以玉米杂交种郑单958及其亲本(郑58和昌7-2)为实验材料, 分析SR蛋白基因家族在干旱胁迫下玉米杂种与亲本中的应答模式及其自身选择性剪接规律, 丰富 AS调控网络视角下的玉米干旱胁迫分子机理。
1 材料和方法
1.1 玉米SR蛋白基因家族的生物信息学分析
玉米SR蛋白基因家族成员序列: 通过2009年11月20日完成的玉米B73自交系参考基因组序列数 据 库 (http://www.maizesequence.org/index.html)获取。
玉米 SR蛋白基因成员的序列特征分析: 利用SWTSS-PROT数据库中的ExPASy Compute pI/Mw tool(http://web.expasy.org/compute_pi/)[9~11]在线分析工具, 对 SR蛋白的分子量及等电点(pI)等理化性质进行分析。利用 Sanger-Pfam HMM Search (http:// pfam.sanger.ac.uk/search)、Profile Scan (http: // prosite.expasy.org/scanprosite/)[12~15]以及 predict protein(http://www.predictprotein.org/)在线分析工具对 SR蛋白的结构和功能进行预测。
玉米 SR蛋白基因家族系统发育树构建: 玉米 28个SR蛋白基因家族成员中, 有5个基因(GRMZM2G-019738、GRMZM2G010754、GRMZM2G097593、GRMZM2G139085和GRMZM2G383967)的编码蛋白在N′-端没有RRM。在23个编码蛋白含有RRM的基因成员中, 有 16个成员的编码蛋白含单个 RRM, 有 7个成员(GRMZM2G021223、GRMZM2G052166、GRMZM2G064612、GRMZM2G121269、GRMZM2G-170365、GRMZM2G331811和GRMZM2G436092)的编码蛋白拥有2个 RRM, 选取其位于N′-端的第 1个RRM, 进行基于RRM保守结构域的SR蛋白基因家族系统发育树构建。利用MEGA4.1软件, 采用邻接法构建系统进化树, Bootstrap 值设置为500。
玉米SR基因启动子顺式调控元件分析: 从B73参考基因组数据库中提取所选的 SR蛋白基因启动区域——转录起始位点(Transcription start site, TSS)上游 1 kb序列, 应用 PLACE(http://www.dna.affrc. go.jp/PLACE/)[16,17]对发育、植物激素和胁迫响应相关的主要顺式作用元件进行量化分析和双向分级聚类分析。
1.2 玉米SR基因选择性剪接分析
1.2.1 实验材料与处理
以郑单958及其亲本郑58和昌7-2为材料, 选饱满无菌种子种植于沙盒中, 于光照培养箱中进行培养。培养条件: 30℃ 16 h光照/26℃ 8h 黑暗, 相对湿度50%。在三叶期开始进行10 d控水处理, 通过测定叶片相对含水量(Relative water content, RWC)确定干旱胁迫程度, 分为中度干旱组(Moderate drought stress, MS), 相对含水量(67±3)%和重度干旱组(Severe drought stress, SS), 相对含水量(411±7)%,并对重度干旱胁迫处理组的植株进行4、24和48 h复水处理。实验设3个重复, 同时设立对照组。对处理组和对照组的倒二叶进行生理指标测定, 叶片RWC参考Larbi和Mekliche[18]方法测定。
1.2.2 RNA提取与cDNA合成
对上述对照组、处理组和复水组玉米植株随机各取出 5株分别进行地上和地下混合取样, 液氮研磨后用按 Total RNA Purification Kit(LC Sciences, Cat. TRK-1001)用户手册进行总RNA提取, 紫外分光光度计测量A260和A280比值用以检验RNA提取质量, -80℃贮存备用。按照 EasyScript First-Strand cDNASynthesis Super Mix(TaKaRa, Cat.: AE301)说明书, 以总RNA为模板, 以Oligo(dT)18为引物, 反转录合成第一链cDNA。
1.2.3 引物设计
对玉米SR蛋白基因家族成员进行qRT-PCR, 采用各基因成员不同剪接体共同外显子区域的保守片段设计特异引物, 以检测不同成员的差异表达(附表1); 对发生选择性剪接事件的SR蛋白基因的AS变体进行RT-PCR, 采用不同剪接体的特异片段设计引物(附表2)。
1.2.4 基因表达分析与数据处理及荧光定量PCR
利用Bio-Rad PCR system进行基因表达定量分析。PCR反应体系(20 μL): 模板(第一链cDNA) 1 μL,引物(Forward primer/Reverse primer)各0.5 μL, 2× MixTransStart®Top Green qPCRSuperMix(TransGen Biotech, Cat. AQ131-02) 10 μL, Passive Reference Dye II(50×) 0.4 μL, ddH2O 7.6 μL。PCR扩增程序: 95℃预变性30 s; 95°C变性5 s, 60°C复性30 s, 40轮循环。以玉米β-actin作为内参基因, 实验设3个重复。对荧光定量实验结果用BIO-RAD RT-PCR读取工具Opticon Monitor 3进行读取, 采用-ΔΔCt法进行计算。SR蛋白基因家族成员表达谱, 用MeV v4.8 (http://mev.tm4.org/)[19]软件进行量化分析和双向分级聚类作图。对发生选择性剪接事件的SR基因AS变体进行半定量分析, PCR扩增循环数参考对应基因qRT-PCR的Ct值。对半定量实验的PCR扩增产物进行琼脂糖凝胶电泳, 统计扩增出的不同 AS变体期望条带(分子量见附表2)和新条带(代表新的AS变体), 分析SR蛋白基因自身的选择性剪接事件。
2 结果与分析
2.1 玉米SR蛋白家族基因的分子生物学特征分析
玉米 B73基因组数据库中明确功能注释的 SR蛋白基因家族共有28个成员(附表3), 在10条染色体上均有分布: 其中在4号染色体上有SR2E、SR7B、SR7E、SR10和RSP41C共5个成员分布, 在5和9号染色体上分别有 4个成员分布(SR7A、SR12B、RSZp22、RSP41D和SR2D、SR7F、SRP32、RSP41B),在10号染色体上有SRP1D、SR12A和 RSP41A共3个成员分布, 其余染色体上分别都有 2个成员分布(1号染色体的SR2B和SR2C, 2号染色体的SR2A和SC35, 3号染色体的SR1和SR30, 6号染色体的SR7D和SRP31, 7号染色体的SR4和SR7C)。该家族基因编码的蛋白质分子量最大(77.6 kDa)、氨基酸最长(712aa)的为 GRMZM2G474658_P01(SR6), 而编码的蛋白质分子量最小(6.5 kDa)、氨基酸序列长度最短(60aa)的为GRMZM2G383967_P01(SRP1D)。其中有27个基因成员的编码蛋白为碱性(pH8.93~12.33),只有 GRMZM2G383967_P01(SRP1D)蛋白呈酸性(pH4.37)。对SRP1D的结构分析表明其为SR1变体蛋白4, 但其基因序列并不具备5′-UTR区域和完整的ORF框, 也没有RRM结构域, 可能是没有功能活性的假基因。
2.2 玉米SR蛋白基因家族的系统发育分析
由28个成员组成的SR蛋白基因家族中, 对编码蛋白中含有 RRM的 23个基因成员进行的基于RRM保守结构域构建的系统发育树分析(图1)表明,玉米SR蛋白家族的23个成员划分为5个亚组。第I亚组包括5个成员: 4个SR7成员(SR7A/B/E/F)和RSZP22。第II亚组包括4个成员: 3个SRP41成员(SRP41A/B/C)和RS2A。第III亚组包括5个成员: SR6、SR7D、SR30、SRP31和SRP32。第IV亚组包括4个成员: SR2D、SR4、SR41D和SR45。第V亚组包括5个成员: 3个SR2成员(SR2B/C/E)、SR10和SR35。

图1 基于RRM(RNA recognition motif)结构域构建的玉米SR家族系统发育树
2.3 玉米SR家族基因启动子顺式调控元件分析
对玉米B73参考基因组数据库中SR蛋白基因的TSS上游1 kb序列进行分析(图2)表明: 除核心启动子元件(CAAT-Box, TATA-Box)外, 28个SR蛋白基因的启动子区域中含有3~8种与发育、植物激素和胁迫响应相关的顺式调控元件; 90%以上的SR蛋白基因家族成员都含有MYB、MYC、GATA-BOX和WBOX调控元件, 57%(16/28)的成员含有ABRE调控元件, 36%(10/28)的成员含有LTR调控元件, 29% (8/28)的成员含有 T/G-BOX调控元件, 14%(4/28)的成员含有ARF或ERE调控元件, 7%(2/28)的成员含有 DRE调控元件。另外, 同一注释名称下的基因,如SR7有6个成员(SR7A/B/C/D/E/F), SR2有5个成员(SR2A/B/C/D/E),分别聚类于不同亚群中; 而不同的SR蛋白基因成员又能够同时聚类于同一亚群。根据其原因可以推测: 即使相同注释成员下的 SR蛋白基因不同成员, 也可能由于启动子区域中顺式调控元件的种类和数目的不同而具有差异的表达模式; 而不同的 SR蛋白基因也可能由于启动子区域中顺式调控元件的种类和数目的相似性而具有相似或相近的表达模式。
2.4 玉米SR蛋白家族基因与干旱胁迫应答
植物叶片相对含水量(RWC)是植物组织水分状况的重要指标, 对抗旱育种的选择具有特殊的意义。本研究对郑单958及其亲本郑58和昌7-2在三叶期中度和重度干旱胁迫处理后进行RWC测定(表1)。在中度干旱胁迫下, 叶片RWC及其与对照组的变化量在郑单958及其亲本郑58和昌7-2间没有显著差异。而在重度干旱胁迫下, 叶片RWC及其与对照组的变化量在郑单958及其亲本郑58和昌7-2间存在显著差异, 其中郑单958在水分胁迫下RWC变化较小, 表现对干旱胁迫较强的耐受性; 而昌7-2在干旱胁迫下水分变化量最大, 表现较强的水分胁迫敏感性。
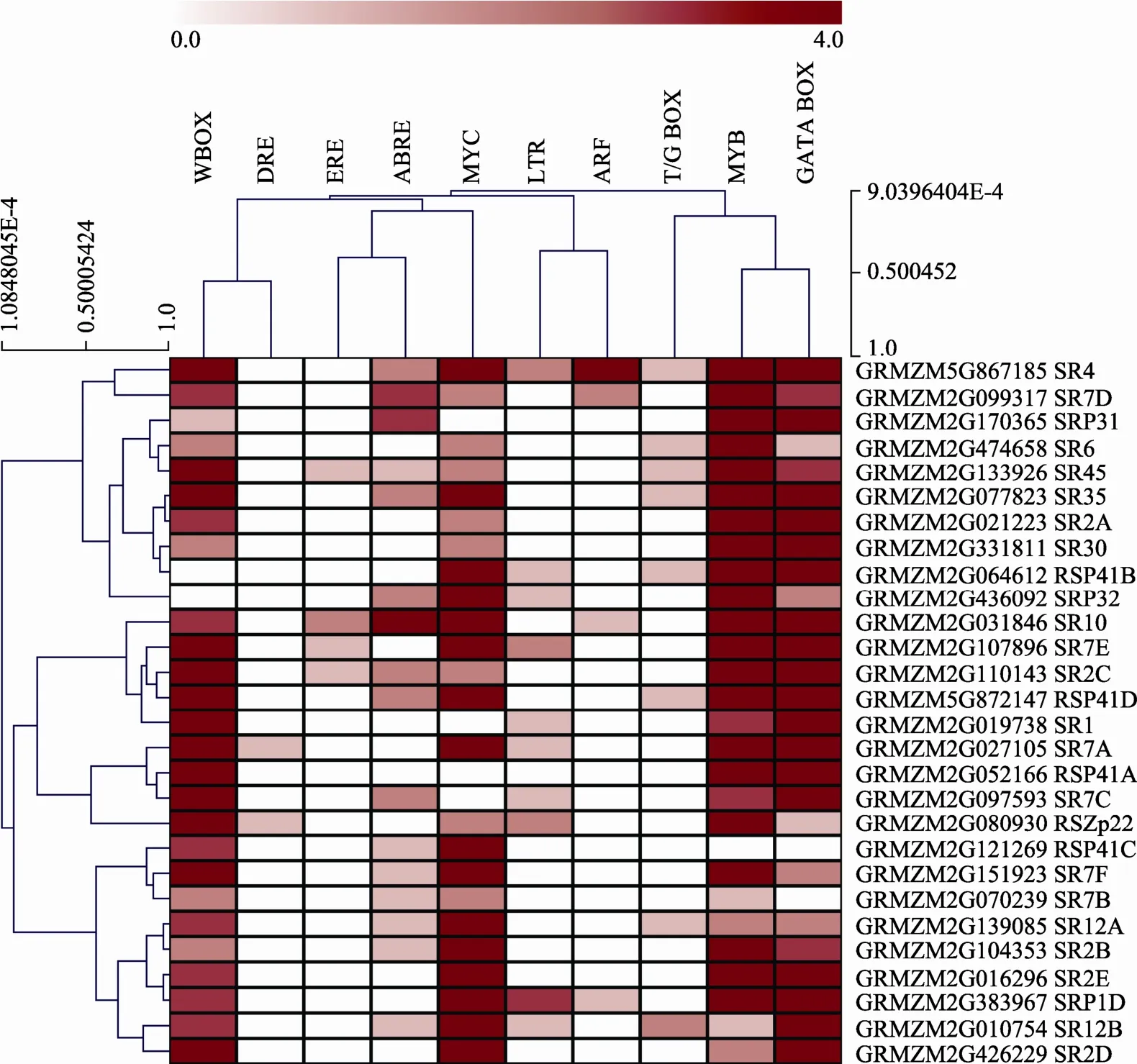
图2 玉米SR蛋白基因启动子区域顺式调控元件分析方格中红色深浅表示启动子区域顺式调控元件拷贝数多少。
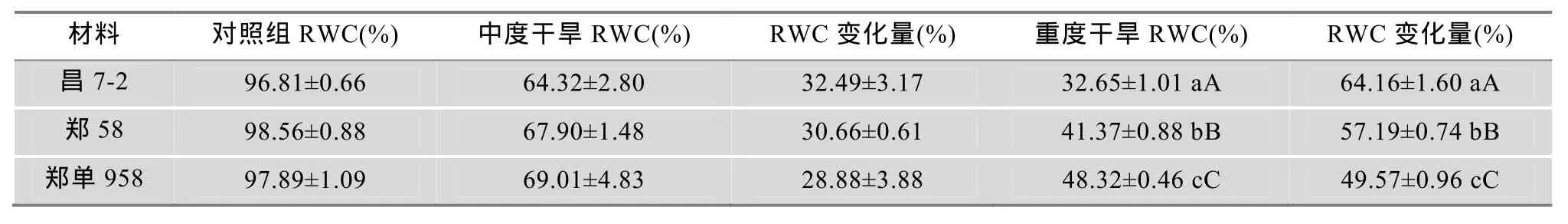
表1 郑单958及其亲本干旱胁迫下叶片RWC变化比较
2.4.1 干旱胁迫下SR蛋白基因差异表达
根据前期玉米自交系旱21和掖478的SR蛋白基因家族干旱胁迫下基因数字化表达谱结果(未发表结果), 选取其中 13个显著差异表达的基因成员,对郑单 958及其亲本在干旱胁迫下基因差异表达进行 qRT-PCR检测(图 3)。基因差异表达谱经双向分级聚类分析表明: 无论地上组织或地下组织, 同一基因型材料的中度干旱组和重度干旱组多聚类于同一亚群中(如ZD958-MS-S和ZD958-SS-S、Z58-MS-R和Z58-SS-S、C7-2-MS-R和C7-2-SS-R分别聚类于最小亚群中; C7-2-MS-S和C7-2-SS-S、ZD958-MS-R和 ZD958-SS-R分别聚类于较小亚群中); 对同一组织而言, 同一基因型材料随干旱胁迫程度加剧, SR蛋白基因家族的整体表达模式仍具有调控的连续性和较强的基因型依赖性(如 ZD958-MS-S和ZD958-SS-S、C7-2-MS-R和C7-2-SS-R分别聚类于最小亚群中; C7-2-MS-S和C7-2-SS-S、ZD958-MS-R和ZD958-SS-R分别聚类于较小亚群中)。
对地下组织而言, 显著性上调(Log2处理/对照>1)和显著性下调(Log2处理/对照<-1)的样品分别为 21和 44,表明 SR蛋白基因家族在干旱胁迫下整体表达模式以下调为主; 对地上组织而言, 显著性上调和显著性下调的样品分别为27和17, 表明SR蛋白基因家族在干旱胁迫下整体表达模式以上调为主。除郑58中度干旱的根(Z58-MS-R)和重度干旱地上组织(Z58-SS-S)外, 郑单958和昌7-2的地上组织和地下组织有规律地聚类于两个不同亚群中, 表明 SR蛋白基因家族整体表达模式在不同遗传材料中具有较一致的组织特异性。
对同一基因型材料的不同组织而言, 中度干旱处理下 SR蛋白家族基因显著性反向调节的有23.1%(9/39), 重度干旱处理下 SR蛋白家族基因显著性反向调节的有35.9%(14/39), 表明SR蛋白基因家族组织特异性调节的差异随干旱胁迫程度加剧而更明显。另外, 在干旱胁迫下SR基因家族不同成员的表达模式有明显差异: SR2B和SC35在50%以上的胁迫样品中显著下调表达, 而 SR7D则在 50%以上的胁迫样品中显著上调表达。
2.4.2 复水处理后SR蛋白基因差异表达
由于只在重度干旱胁迫下郑单 958及其亲本郑58和昌7-2之间的叶片RWC才表现出显著差异, 因此对重度干旱胁迫处理组的植株进行了 3个不同时间段(4 h、24 h和48 h)的复水处理。对选取的13个玉米 SR蛋白基因家族成员在不同时间段复水处理的差异表达谱分析(图4)表明: 与干旱组处理情形规律相似, 除郑58复水4 h地上组织(Z58-4h-S)外, 郑单958和昌7-2的地上组织和地下组织的SR蛋白基因表达模式都有规律的聚类于各自不同亚群中; 无论地上组织或地下组织, 同一基因型材料的不同复水时段 SR蛋白基因表达模式基本聚于同一亚群中;表明对同一组织而言, 同一基因型材料随复水时段的延长, SR蛋白基因家族的整体表达模式具有调控的连续性。
对地下组织而言, 显著性上调和显著性下调的样品分别为29和56, 表明SR蛋白基因家族在干旱胁迫后的复水处理过程中整体表达模式以下调为主;对地上组织而言, 显著性上调和显著性下调的样品分别为29和50, 表明SR蛋白基因家族在干旱胁迫后的复水处理过程中整体表达模式也以下调为主。以上结果表明, 在干旱胁迫后的复水处理过程中SR蛋白基因家族整体表达模式以下调为主。
复水处理过程中 SR蛋白家族不同成员的表达模式表现出不同趋势: 其中成员 SR45、SR12A、SR12B、SC35和SR7A在50%以上的复水样品中显著下调表达; 而成员SR7B和RSP41A则在50%以上的复水样品中显著上调表达。
2.4.3 干旱胁迫下SR蛋白自身的选择性剪接
SR基因是一类对选择性剪接起重要调控作用的剪接因子[20,21]。Richardson等[7]研究表明, 在其统计的22个玉米SR基因中有21个能够发生自身选择性剪接, 而且主要以3′选择性剪接方式为主。

图3 玉米SR基因干旱胁迫差异表达分析ZD958、Z58和C7-2分别表示郑单958及其亲本郑58和昌7-2; MS和SS分别表示中度和重度干旱胁迫处理组; S和R分别表示中度地上组织(Shoot)和地下组织(Root)。图中所示处理组/对照组的相对表达量(-ΔΔCt)变化趋势(上/下调)用不同颜色(红/绿)的深浅程度表示, 其中绿色越深代表下调表达越明显, 红色越深代表上调表达越显著。下图同。

图4 玉米SR基因复水处理组差异表达分析ZD958、Z58和C7-2分别表示郑单958及其亲本郑58和昌7-2; 4 h、24 h和48 h分别表示复水4 h、24 h和48 h; S和R分别表示地上组织(Shoot)和地下组织(Root)。
本研究对选取SR蛋白家族的13个成员, 根据各基因不同 AS变体特异性片段设计引物, 进行半定量PCR扩增, 对扩增产物的琼脂糖凝胶电泳预期条带和新AS变体产生的新条带进行统计(附表4)。结果表明: (1)53.8%(7/13)的SR成员AS变体数目在对照组多于干旱胁迫组, 而 46.2%(6/13)的 SR成员AS变体数目在对照组少于干旱胁迫组; (2)同一 SR基因的 AS变体数目分布: 郑 58>昌 7-2>郑单 958; (3)有12个SR基因成员中检测到有新的AS变体形式。图 5 A显示 GRMZM2G099317(SR7D)基因中在C7-2-DS-R样品中检测到有新的AS变体(Novel1) 形式。

图5 GRMZM2G099317(SR7D)和ß-actin基因半定量PCR扩增的凝胶电泳图ZD958、Z58和C7-2分别表示郑单958及其亲本郑58和昌7-2; CK和DS分别表示对照组和干旱胁迫处理组; S和R分别表示地上组织(Shoot)和地下组织(Root)。A 图中箭头所指 T02/T03/T04/ T05为玉米 B73参考基因组序列数据库(http://www.maizesequence.org/index.html)中GRMZM2G099317注释的AS变体编号(T02/03/04/05), Novel1为新的AS变体条带。
3 讨 论
SR蛋白基因家族作为一类重要的选择性剪接因子, 通过对转录本ESE的识别与结合参与剪接体的组装, 进而调节基因转录后的选择性剪接。对玉米B73参考基因组测序结果的分析表明, 玉米SR蛋白基因家族的28个成员在玉米10条染色体上均有分布, 启动子区域中含有 3~8种与发育、植物激素和胁迫响应相关的顺式调控元件; 27个基因编码碱性蛋白质, 另外1个基因可能为假基因。其中的23个基因编码蛋白依其N′-端的首个RRM结构域大体上划分为 5个亚组。郑单 958及其亲本郑 58和昌7-2在三叶期进行的干旱胁迫及其后的复水过程中, SR蛋白基因家族整体表达模式不但具有组织表达特异性和基因型依赖性特点, 而且不同成员的表达模式也存在明显差异, 并普遍存在自身选择性剪接现象。
3.1 SR蛋白基因参与胁迫响应的分子基础
玉米的SR蛋白基因启动子含有多种与发育、植物激素和胁迫响应相关顺式调控元件: 90%以上启动子含有MYB、MYC、GATA -BOX和WBOX应答元件, 部分成员还含有ABRE、LTR、T/GBOX、ARF和DRE应答元件。其中LTR元件能够对低温产生应答; GATA-BOX对光调节产生应答; WBOX能够通过与WRKY转录因子结合, 对植物的抗性和发育产生响应和调节。植物基因启动子中的ABRE、DRE和MYC能够直接或间接影响ABA两种相互交叉途径形成的调控网络。玉米不同SR蛋白基因家族成员启动子区域存在多种胁迫响应元件, 表明 SR蛋白基因的表达有其参与胁迫响应基因选择性剪接调控的分子基础; 而顺式调控元件的种类、拷贝数和组合方式的差异, 则预示 SR蛋白基因表达模式的多样性和应对不同胁迫的灵活性。
3.2 SR蛋白家族基因表达与胁迫响应
在郑单958及其亲本郑58和昌7-2苗期干旱胁迫及其后复水过程中, SR蛋白基因家族虽然整体上表现出组织表达特异性和基因型依赖性特点, 但不同成员的表达模式又呈明显差异: SC35在50%以上的胁迫处理样品和复水处理样品中显著下调表达,而SR7B和RSP41A则在50%以上的复水处理样品中显著上调表达。拟南芥中多个SR蛋白家族的基因成员在高温、高盐和强光条件下表达量显著改变[22,23]。SCL33能够影响盐分和低温胁迫下 ABA响应基因的表达[6,24]。SR45是SC35like类SR蛋白, 在剪接过程中常与U1-70K互作[25], 影响5′-端和3′-端剪接位点的选择[26]。苗期SR45通过下调ABA信号通路相关基因的表达来调节ABA和葡萄糖信号途径[27]。在玉米中ZmSRp32的过量表达能够导致pre-mRNA加工过程中 5'剪接位点的增强[28]。由此看来, 筛选和鉴定玉米干旱胁迫响应的 SR蛋白基因成员, 并分析其表达模式对转录组所涉基因的具体 AS调节方式的影响, 是解析 AS调控机制在玉米抗逆过程的关键工作之一。
3.3 SR蛋白基因的AS调节与胁迫响应
植物SR蛋白家族基因自身的AS调节具有普遍性。对玉米SR蛋白家族28个成员自身的AS统计表明, 大多数SR蛋白家族基因成员存在自身AS方式, 且以3'选择性剪接方式为主(结果未发表); 本文选取的13个SR蛋白家族基因成员中有12个能够检测到有新的AS变体形式。SR蛋白基因的自身选择性剪接能够调节下游基因的选择性剪接事件。SR45基因在拟南芥中有多种剪接变体, 并在多种逆境胁迫下上调表达, 揭示其为干旱胁迫相关基因[21,29];其中两个剪接变体分别控制花和根的形态发育[25]。高光胁迫下, atSR45a基因能够通过自身选择性剪接机制精细调节2种变体(atSR45a-1和atSR45a-2)的比率来影响剪接体的组装效率[22,30], 进而显著改变下游217个基因的选择性剪接模式[20]。这些下游基因的表达主要涉及信号转导、细胞周期、DNA加工、蛋白质命运和转录活动等。上述研究提示, 玉米胁迫响应的SR蛋白基因成员有可能通过自身AS变体的精细调节, 影响转录组组分及其相对表达丰度,进而多维地调整植物体对逆境胁迫的适应性。
本文系统分析了玉米 SR蛋白基因家族在干旱胁迫下的基因表达规律和自身选择性剪接现象。与一般的编码蛋白基因表达情形类似, 其表达模式具有组织特异性和基因型依赖性特点, 预示着 AS介导的基因表达调控机制能够影响干旱胁迫下植物体转录组可塑性的复杂程度。另外, SR蛋白基因家族的表达主要以剂量效应和 RS结构域中丝氨酸残基的磷酸化状态与程度, 作为正调控因子参与基因表达的选择性剪接。在其活性调节过程中, RS结构域的磷酸化/去磷酸化是参与选择性剪接的主要条件, 参与SR蛋白磷酸化的激酶主要有SRPK1和Clk/Sty[31]。因此, 筛选通用型的干旱胁迫相关的 SR基因成员并鉴定其主要的AS变体形式, 厘清SR蛋白基因表达—下游基因 AS调节—玉米抗旱性三者的关联程度, 研究其 RS结构域中丝氨酸残基的磷酸化状态、克隆涉及的激酶基因并揭示其与玉米干旱胁迫响应的内在规律, 将深化从AS-network视角探索玉米的抗逆分子机制研究。
[1] Weiss C, Zeng Y, Huang J, Sobocka MB, Rushbrook JI. Bovine NAD+-dependent isocitrate dehydrogenase: alternative splicing and tissue-dependent expression of subunit 1. Biochemistry, 2000, 39(7): 1807-1816.
[2] Kazan K. Alternative splicing and proteome diversity in plants: the tip of the iceberg has just emerged. Trends Plant Sci, 2003, 8(10): 468-471.
[3] Barbazuk WB, Fu Y, McGinnis KM. Genome-wide analyses of alternative splicing in plants: opportunities and challenges. Genome Res, 2008, 18(9): 1381-1392.
[4] Kakumanu A, Ambavaram MMR, Klumas C, Krishnan A, Batlang U, Myers E, Grene R, Pereira A. Effects of drought on gene expression in maize reproductive and leaf meristem tissue revealed by RNA-Seq. Plant Physiol, 2012, 160(2): 846-867.
[5] Streitner C, Koster T, Simpson CG, Shaw P, Danisman S, Brown JWS, Staiger D. An hnRNP-like RNA-binding protein affects alternative splicing by in vivo interaction with transcripts in Arabidopsis thaliana. Nucleic Acids Res, 2012, 40(22): 11240-11255.
[6] Palusa SG, Ali GS, Reddy AS. Alternative splicing of pre-mRNAs of Arabidopsis serine/arginine-rich proteins: regulation by hormones and stresses. Plant J, 2007, 49(6): 1091-1107.
[7] Richardson DN, Rogers MF, Labadorf A, Ben-Hur A, Guo H, Paterson AH, Reddy ASN. Comparative analysis of serine/arginine-rich proteins across 27 eukaryotes: insights into sub-family classification and extent of alternative splicing. PLoS ONE, 2011, 6(9): e24542.
[8] Okagaki RJ, Phillips RL. Maize DNA-sequencing strategies and genome organization. Genome Biol, 2004, 5(5): 223.
[9] Bjellqvist B, Hughes GJ, Pasquali C, Paquet N, Ravier F, Sanchez JC, Frutiger S, Hochstrasser D. The focusing positions of polypeptides in immobilized pH gradients can be predicted from their amino acid sequences. Electrophoresis, 1993, 14(10): 1023-1031.
[10] Bjellqvist B, Basse B, Olsen E, Celis JE. Reference points for comparisons of two-dimensional maps of proteins from different human cell types defined in a pH scale where isoelectric points correlate with polypeptide compositions. Electrophoresis, 1994, 15(3-4): 529-539.
[11] Wilkins MR, Gasteiger E, Bairoch A, Sanchez JC, Williams KL, Appel RD, Hochstrasser DF. Protein identification and analysis tools in the ExPASy server. Methods Mol Biol, 1999, 112: 531-552.
[12] Sigrist CJA, De Castro E, Cerutti L, Cuche BA, Hulo N, Bridge A, Bougueleret L, Xenarios L. New and continuing developments at PROSITE. Nucleic Acids Res, 2013, 41(Database issue): D344-D347.
[13] Sigrist CJA, Cerutti L, Hulo N, Gattiker A, Falquet L, Pagni M. PROSITE: a documented database using patterns and profiles as motif descriptors. Brief Bioinform, 2002, 3(3): 265-274.
[14] De Castro E, Sigrist CJ, Gattiker A, Bulliard V, Langendijk-Genevaux PS, Gasteiger E, Bairoch A, Hulo N. ScanProsite: detection of PROSITE signature matches and ProRule-associated functional and structural residues in proteins. Nucleic Acids Res, 2006, 34(Web Server issue): W362-W365.
[15] Sigrist CJ, De Castro E, Langendijk-Genevaux PS, Le Saux V, Bairoch A, Hulo N. ProRule: a new database containing functional and structural information on PROSITE profiles. Bioinformatics, 2005, 21(21): 4060-4066.
[16] Higo K, Ugawa Y, Iwamoto M, Korenaga T. Plant cis-acting regulatory DNA elements (PLACE) database: 1999. Nucleic Acids Res, 1999, 27(1): 297-300.
[17] Prestridge DS. SIGNAL SCAN: a computer program that scans DNA sequences for eukaryotic transcriptional elements. Compute Appl Biosci: CABIOS, 1991, 7(2): 203-206.
[18] LarbiA, Mekliche A. Relative water content (RWC) and leaf senescence as screening tools for drought tolerance in wheat. Options Méditerranéennes: Série A, Séminaires Méditerranéens, 2004, (60): 193-196.
[19] Saeed AI, Sharov V, Whie J, Li J, Liang W, Bhagabati N, Braisted J, Klapa M, Currier T, Thiagarajan M, Sturn A, Snuffin M, Rezantsev A, Popov D, Rylsov A, Kostukovich E, Borisovsky I, Liu Z, Vinszvich A, Trush V, Quackenbush J. TM4: a free, open-source system for microarray data management and analysis. Biotechniques, 2003, 34(2): 374-378.
[20] Yoshimura K, Mori T, Yokoyama K, Koike Y, Tanabe N, Sato N, Takahashi H, Maruta T, Shigeoka S. Identification of alternative splicing events regulated by an Arabidopsis serine/arginine-like protein, atSR45a, in response to high-light stress using a tiling array. Plant Cell Physiol, 2011, 52(10): 1786-1805.
[21] Tanabe N, Yoshimura K, Kimura A, Yabuta Y, Shigeoka S. Differential expression of alternatively spliced mRNAs of Arabidopsis SR protein homologs, atSR30 and atSR45a, in response to environmental stress. Plant Cell Physiol, 2007, 48(7): 1036-1049.
[22] Tanabe N, Kimura A, Yoshimura K, Shigeoka S. Plant-specific SR-related protein atSR45a interacts with spliceosomal proteins in plant nucleus. Plant Mol Biol, 2009, 70(3): 241-252.
[23] Ward AJ, Cooper TA. The pathobiology of splicing. J Pathol, 2010, 220(2): 152-163.
[24] Reddy ASN, Shad Ali G. Plant serine/arginine-rich proteins: roles in precursor messenger RNA splicing, plant development, and stress responses. Wiley Interdiscip Rev RNA, 2011, 2(6): 875-889.
[25] Zhang XN, Mount SM. Two alternatively spliced isoforms of the Arabidopsis SR45 protein have distinct roles during normal plant development. Plant Physiol, 2009, 150(3): 1450-1458.
[26] Day IS, Golovkin M, Palusa SG, Link A, Ali GS, Thomas J, Richardson DN, Reddy ASN. Interactions of SR45, an SR-like protein, with spliceosomal proteins and an intronic sequence: insights into regulated splicing. Plant J, 2012, 71(6): 936-947.
[27] Carvalho RF, Carvalho SD, Duque P. The plant-specific SR45 protein negatively regulates glucose and ABA signaling during early seedling development in Arabidopsis. Plant Physiol, 2010, 154(2): 772-783.
[28] Gao H, Gordon-Kamm WJ, Lyznik LA. ASF/SF2-like maize pre-mRNA splicing factors affect splice site utilization and their transcripts are alternatively spliced. Gene, 2004, 339: 25-37.
[29] Golovkin M, Reddy AS. An SC35-like protein and a novel serine/arginine-rich protein interact with Arabidopsis U1-70K protein. J Biol Chem, 1999, 274(51): 36428-36438.
[30] Ali GS, Palusa SG, Golovkin M, Prasad J, Manley JL, Reddy ASN. Regulation of plant developmental processes by a novel splicing factor. PLoS ONE, 2007, 2(5): e471.
[31] Colwill K, Feng LL, Yeakley JM, Gish GD, Cáceres JF, Pawson T, Fu X. SRPK1 and Clk/Sty protein kinases show distinct substrate specificities for serine/arginine-rich splicing factors. J Biol Chem, 1996, 271(40): 24569-24575.
(责任编委: 郝东云)
Response of maize serine/arginine-rich protein gene family in seedlings to drought stress
Jiao Li, Yuqi Guo, Weiling Cui, Aihua Xu, Zengyuan Tian
College of Life Science, Zhengzhou University, Zhengzhou 450001, China
Alternative splicing (AS) in eukaryotic organisms is closely related to the gene regulation in plant abiotic stress responses, in which serine/arginine-rich proteins (SR proteins) act as key regulators. The genome sequence of maize inbred line B73 was analyzed, showing that the promoter regions of SR genes possess about three to eight kinds of cis-acting regulatory elements. Twenty-seven SR genes encode alkaline proteins, and 23 of which are divided into five subgroups in terms of the first RNA recognition motif (RRM) at the amino terminal. The expression of SR genes showed tis-sue-specific and genotype-dependent features under drought stress in the hybrid Zhengdan-958 and its parents, Zheng-58 and Chang-7-2 via bidirectional hierarchical clustering. SR genes were down-regulated in roots while they were up-regulated in shoots under drought stress. However, SR genes were down-regulated in both roots and shoots in three different rehydration stages after severe drought stress. Additionally, a widespread alternative splicing exists in all SR genes although SR genes showed differential expression tendency under drought stress and/or during rehydration stages. Results above will deepen our understanding of the molecular mechanisms of plant response to abiotic stress from the perspective of AS-network.
maize under drought stress; SR genes; expressional pattern; alternative splicing
2013-12-12;
2014-03-07
基金农业部948项目(编号:2012-Z38)资助
李娇, 硕士研究生, 专业方向:植物分子生物学。E-mail: li_jiao_2006@126.com
郭予琦, 博士, 副教授, 研究方向:植物抗逆分子机理。E-mail: guoyuqi@zzu.edu.cn
李娇和郭予琦同为第一作者。
田曾元, 博士, 副教授, 研究方向:玉米逆境胁迫分子机制。E-mail: tianzengyuan@zzu.edu.cn
10.3724/SP.J.1005.2014.0697
时间: 2014-6-16 17:06:11
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140616.1706.001.html