定向进化技术在蛋白质开发中的应用进展
张志来,陈建华
定向进化技术在蛋白质开发中的应用进展
张志来,陈建华
210009 南京,中国药科大学生命科学与技术学院
定向进化技术是一种利用实验室手段通过反复改造遗传多样性,结合文库高通量筛选而获得理想性状生物体的过程,现已成为在基础生物学和应用生物学领域应用最广泛和最有效的技术之一。目前已成功应用于蛋白质的研究和开发。蛋白质在生物催化、生物医药等领域具有广泛的应用。如何提高目的蛋白的产量、改善蛋白质的理化特性以及构建具有新催化活性的蛋白质以满足日益增长的需求是亟待解决的问题。而定向进化技术的发展为实现这样的目的提供了强有力的技术支持。本文就定向进化技术在蛋白质开发中的应用进行了综述。
1 定向进化技术
与理性突变方法相比,定向进化技术具有独特的优势:突变体的构建无需受蛋白质结构信息的限制。早先的定向进化技术的靶标多数是单个蛋白,通过随机或定点插入突变,获得稳定性更好或催化活性更高的突变体以适应工业化要求。而近年来的趋势则转向于对代谢途径甚至是基因组水平进行改造,以创造新型全细胞催化剂用于合成有价值的生物产品[1]。
目前,比较经典的定向进化技术有易错 PCR 技术和 DNA 改组技术等,而新兴的技术则是在这两种技术的基础上改进发展起来的。
1.1 易错 PCR 技术
易错 PCR 是利用 Taq DNA 聚合酶,或改变 PCR 反应体系的条件,在 DNA 聚合过程中随机引入错配碱基,其突变位点发生在分子内部,是应用最早、最成熟的一种定向进化方法[2]。易错 PCR 无需改变基因的长度,突变频率可以根据反应条件进行相应的控制,并且能有效地获得理想突变体,在蛋白质定向进化中得到了广泛的应用。但是易错 PCR 只能发生点突变,而且获得活性突变体的效率比较低,因此其应用受到了一定的限制。在易错 PCR 基础上,Gratz 和 Jose[3]发明了重叠延伸蛋白域文库法(PDLGO),它克服了易错 PCR 突变率低的缺陷,可以在预期的区域内进行随机突变。
1.2 DNA 改组
DNA 改组,又称有性 PCR 技术,是由 Stemmer[4]于 1994 年首先提出,后经不断改进,现已成为比较完善的定向进化技术。它将单个基因或同源性较高的多个基因通过 DNase I 随机片段化,藉由小片段之间的同源性,互为模板,互为引物重新组装成大片段。然后,加入特异性引物进行有引物 PCR 扩增全长嵌合体基因,结合高通量筛选方法选择具有功能改进或全新功能的突变体。目前,DNA 改组技术已成功应用于蛋白质工程、生物医药等领域,是基因定向进化的最有效的方式,在蛋白生产和研究中是应用最成功的技术之一[5]。
近年来,随着对 DNA 改组的不断完善和改进,新的依赖于同源重组的定向进化技术不断涌现,如交叉延伸 PCR(StEP)技术[6]、随机链交换法(RAISE)[7]、随机引物体外重组(RPR)技术[8]、临时模板随机嵌合(RACHITT)技术[9]等(图 1)。而基于同源重组的定向进化技术,需要待改造的基因具有一定的同源性,无法有效地在非同源性或同源性低的亲本中应用,同时文库的多样性往往无法达到理想的需求。因此,一些基于非同源重组实现随机突变的技术应运而生,如 SCRATCHY 文库[10]、随机多重 PCR[11]、外显子改组[12]等。
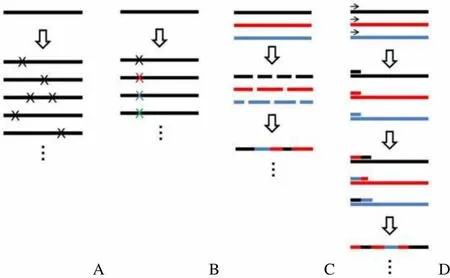
ABC D
不断更新的新技术致力于提高突变率,克服现有定向进化技术的限制因素。定向进化技术在生物医药、蛋白工业化生产等领域得到了广泛的应用。随着技术的发展,单纯的技术已不足以获得理想的突变体,越来越多的研究则是趋向于结合多种技术对生物个体或是大分子进行改造,以期获得理想的成果。
2 定向进化技术在蛋白质开发中的应用
近年来,由于工业化用酶及药用蛋白的需求急剧增加,如何提高蛋白的产量、改善蛋白的特性以及构建新功能蛋白以适应日益增长的需求,是人们亟待解决的问题。定向进化技术的发展为实现这样的目标提供了基础。
2.1 提高蛋白产量
2.1.1 目的蛋白过表达 启动子是基因表达调控的重要顺式作用元件,位于基因上游,是由 RNA 聚合酶特异性识别的一段 DNA 序列。启动子对外源基因的表达水平影响很大,其强弱直接影响到外源基因表达的有效性,因此筛选强启动子对于增强蛋白产量具有一定的促进作用。定向进化技术促进了高效启动子的筛选。Alper 等[13]采用易错 PCR 结合核苷酸类似物诱变的技术对 TEF1 启动子进行改造获得一个长度不同的启动子突变体库,从中筛选出不同特性的启动子突变体以提高特定产物的表达量。
除了单纯的对启动子进行改造以外,提高启动子结合蛋白,即 RNA 聚合酶的活性,对于增强目的基因的转录也具有重要的影响。Esvelt 等[14]采用 PACE(噬菌体辅助的持续进化)技术对启动子结合蛋白 T7 聚合酶进行定向进化研究,经过 200 轮的蛋白质进化,获得了一株比野生型 T7 RNA 聚合酶蛋白活性提高几百倍的突变体,使得报告基因产物的产量提高了 100 倍。
对基因本身进行改造,使其更适于在相应宿主内表达,也是提高蛋白质产量的一种方式。Egloff 等[15]采用易错 PCR 技术,结合适当的选择压力对神经降压素(NTR1)进行改造,使得其在大肠杆菌内的表达量提高,同时提高了蛋白的稳定性。
2.1.2 蛋白质生物合成途径的调控 目前,细菌和酵母是蛋白外源表达最常用的宿主。通过在宿主中构建或插入异源代谢途径实现目的蛋白的大规模生产具有一定的应用前景。然而,异源途径的代谢通量需要进行优化以避免因过表达特定基因引起代谢负荷,同时还得避免因相关产物积累导致宿主细胞的生长抑制。平衡代谢通量的常用方法是确认代谢途径的限速步骤,进而对限速酶进行修饰改造。最常用的解除瓶颈的方法是过表达代谢途径中的关键基因、删除竞争性途径、利用定向进化技术提高限速酶的活性等[16]。
Dong 等[17]采用易错 PCR 技术对 CpcA(藻青蛋白α 亚基)生物合成途径中的限速酶进行了诱变,使得 CpcA 的产量比对照组提高了 29.7%。李晓萍等[18]为了解除代谢产物对芳香族氨基酸生物合成途径中关键酶的反馈抑制作用,利用 DNA 改组技术对 aroG 基因进行改造,在一定程度上解除了氟苯丙氨酸对关键酶的抑制作用,从而使得目的产物得到积累。Gu 等[19]采用易错 PCR 和定点突变技术对色氨酸代谢途径中的启动子和 trpE、aeoG 基因进行改造,同时对 L-色氨酸合成途径中的弱化子和分支途径进行敲除,最终使得改造菌株发酵 48 h 的 L-色氨酸产量达到 10.15 g/L。这些研究证实,对代谢通路中的酶进行改造可以作为蛋白质合成和提高产量的有效策略。
近年来,越来越多的系统性诱变技术发展迅速。Wang 等[20]通过在大肠杆菌中反复进行单链寡核苷酸介导的等位替换对基因组进行改造,发明了 MAGE(多重自动基因工程)技术。寡核苷酸含有简并碱基,可以对靶基因引入插入或缺失突变,该研究对 1-脱氧-D-木酮糖-5-磷酸(DXP)途径中的 24 个基因组分同时修饰,使得番茄红素的产量提高了 5 倍。而将这一技术应用于蛋白质产量的提高具有一定的实践前景。
为了获得理想的目的产物,有些研究则是按照自己的意愿构建代谢途径。而新代谢途径的构建费时费力,而且多重基因的同时调控往往成为其应用瓶颈。Wingler 和 Cornish[21]则发明了一种快速构建代谢途径的迭代重组技术,该技术可以在进化过程中调整代谢途径中所需基因的比例,从而获得目的产物的高表达。因此人们可以运用这些技术按照自己的意愿高效、快速构建蛋白合成途径,从而提高蛋白的产量。
2.2 蛋白质性质的改良
为了适应药用蛋白及蛋白工业化生产的要求,许多研究致力于通过定向进化技术获得催化活性高、稳定性好、蛋白质相关性质得到改善的突变体。目前定向进化技术已在提高酶的催化活性、稳定性、底物特异性、选择性等方面得到成功的应用(表 1)。
2.3 修饰蛋白,赋予新的功能
目前,大部分定向进化的研究集中于对蛋白质现有功能的改善,但是如何对天然蛋白质进行改造以获得新功能仍是研究的一大难题。蛋白质新功能的产生往往需要对原始序列进行一系列突变、重组的大幅度改造,所得的突变体库常含有大量的无活性蛋白,增加了筛选难度。
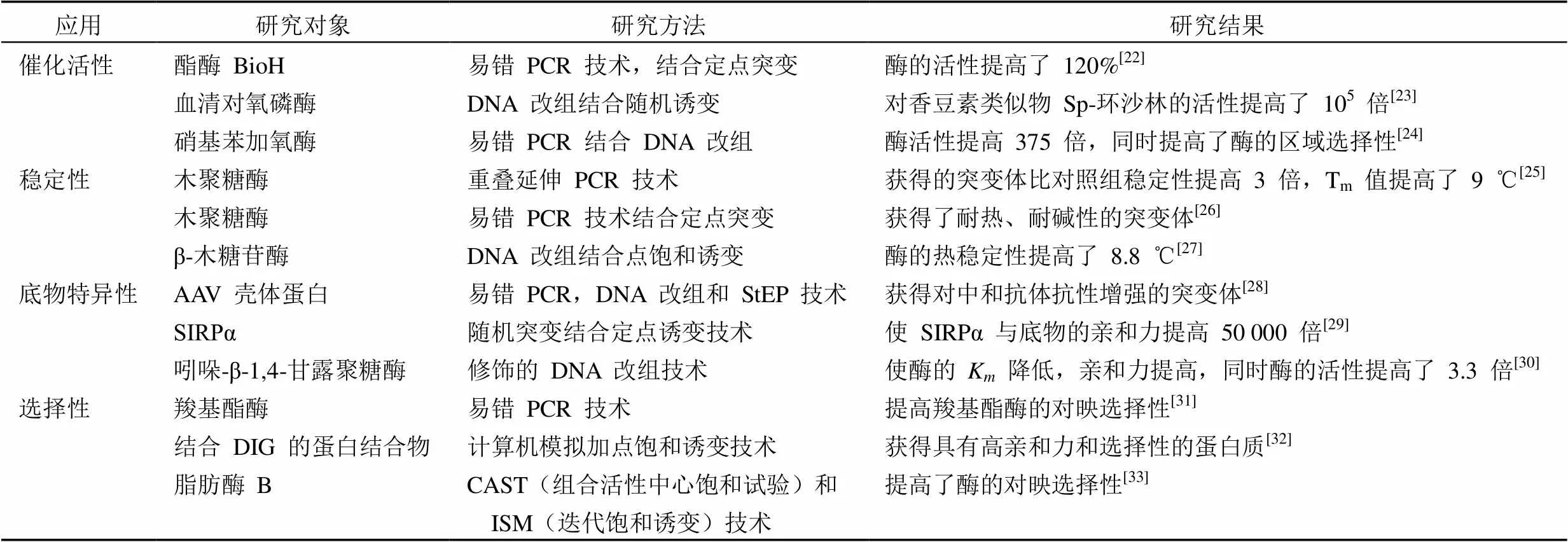
表 1 定向进化技术在改善蛋白质性质方面的应用
定向进化技术的发展,为筛选和改造新功能的蛋白提供了契机。Li 等[34]通过人工设计并结合 DNA 改组技术对 IFN-α 进行改造,获得了具有抗肿瘤活性的新型抗生素 Novaferon。Chao 等[35]则是采用计算机模拟结合定向进化技术在非催化活性支架蛋白的基础上构建获得了一种新型的 RNA 连接酶,该酶能将 5-三磷酸 RNA 连接到另一 RNA 的 3-羟基上,完成了自然界酶不能完成的反应。Sabile 等[36]则以转氨酶脚手架蛋白为基础,结合点饱和诱变技术获得了一种能够催化合成手性胺的新型酶,提高了 II 型糖尿病治疗药物西他列汀的产量。
3 小结
运用定向进化技术获得人们期望催化功能和活性的蛋白,对于加速蛋白工业化生产、蛋白药物研发及蛋白质基础研究具有重要的作用。除了上述几个方面以外,定向进化技术在研究蛋白质的结构与功能关系、研究生物反应机制、合成生物学等方面也具有广泛的应用。随着科技产业的发展,人们要求的提高,蛋白质理性设计手段已难以满足人们的实际需求。而现有的定向进化技术往往存在操作复杂、突变效率低等缺陷。因此,开发快捷、方便、高效的定向进化技术,对于蛋白质的开发应用具有促进作用。
[1] Cobb RE, Chao R, Zhao HM. Directed evolution: Past, present and future. AIChE J, 2013, 59(5):1432-1440.
[2] Leung DW, Chen E, Goeddel DV, et al. A method of random mutagenesis of a defined DNA segment using a modified polymerase chain reaction. Technique, 1989, 1(1):11-15.
[3] Gratz A, Jose J. Protein domain library generation by overlap extension (PDLGO): a tool for enzyme engineering. Anal Biochem, 2008, 378(2):171-176.
[4] Stemmer PC. DNA shuffling by random fragmentation and reassembly: in vitro recombination for molecular evolution. Proc Natl Acad Sci U S A, 1994, 91(22):10747-10751.
[5] Wang XF, Li QZ, Bao TW, et al. In vitro rapid evolution of fungal immunomodulatory proteins by DNA family shuffling. Appl Microbiol Biotechnol, 2012, 97(6):2455-2465.
[6] Zhao H, Giver L, Shao Z, et al. Molecular evolution by staggered extension process (StEP) in vitro recombination. Nat Biotechnol, 1998, 16(3):258-261.
[7] Fujii R, Kitaoka M, Hayashi K. RAISE: a simple and novel method of generating random insertion and deletion mutation. Nucleic Acid Res, 2006, 34(4):e30.
[8] Shao Z, Zhao H, Giver L, et al. Random-primering in vitro recombination: an effective tool for directed evolution. Nucleic Acid Res, 1998, 26(2):681-683.
[9] Coco WM, Levison WE, Crist MJ, et al. DNA shuffling method for generating highly recombined genes and evolved enzymes. Nat Biotechnol, 2001, 19(4):354-359.
[10] Lutz S, Ostermeier M, Moore GL, et al. Creating multiple-crossover DNA libraries independent of sequence identity. Proc Natl Acad Sci U S A, 2001, 98(20):11248-11253.
[11] Tsuji T, Onimaru M, Yanagawa H. Random multi-recombinant PCR for the construction of combinational protein libraries. Nucleic Acid Res, 2001, 29(20):E97.
[12] Kolkman JA, Stemmer WP. Directed evolution of proteins by exon shuffling. Nat Biotechnol, 2001, 19(5):423-428.
[13] Alper H, Fiscer C, Nevoigt E, et al. Tuning genetic control through promoter engineering. Proc Natl Acad Sci U S A, 2005, 102(36): 12678-12683.
[14] Esvelt KM, Carlson JC, Liu DR. A system for the continuous directed evolution of biomolecules. Nature, 2011, 472(7344):499-503.
[15] Egloff P, Hillenbrand M, Klenk C, et al. Structure of signaling-competent neurotensin receptor 1 obtained by directed evolution in Escherichia coli. Proc Natl Acad Sci U S A, 2013, 111(6): E655-E662.
[16] Du J, Yuan YB, Si T, et al. Customized optimization of metabolic pathways by combinatorial transcriptional engineering. Nucleic Acids Res, 2012, 40(18):e142.
[17] Dong D, Pan H, Yu P. Directed evolution of the CpcA biosynthetic pathway and optimization of conditions for CpcA production and its properties. Appl Microbiol Biotechnol, 2014, 98(11):4995-5007.
[18] Li XP, Bian YN, Hao RX, et al. Using DNA shuffling to construct the aroG mutant relieved the feedback inhibition of para-fluoro- phenylalanine. J Fudan Univ (Nat Sci), 2010, 49(5):568-574. (in Chinese)
李晓萍, 边英男, 郝瑞昕, 等. 利用DNA shuffling构建部分解除对氟苯丙氨酸反馈抑制的aroG. 复旦学报(自然科学版), 2010, 49(5): 568-574.
[19] Gu P, Yang F, Kang J, et al. One-step of tryptophan attenuator inactivation and promoter swapping to improve the production of L-tryptophan in Escherichia coli. Microb Cell Fact, 2012, 11:30.
[20] Wang HH, Isaacs FJ, Carr PA, et al. Programming cells by multiplex genome engineering and accelerated evolution. Nature, 2009, 460(7257):894-898.
[21] Wingler LM, Cornish VW. Reiterative recombination for the in vivo assembly of libraries of multigene pathways. Proc Natl Acad Sci U S A, 2011, 108(37):15135-15140.
[22] Ye LD, Xu HN, Yu HW. Enhanced acylation activity of esterase BioH from Escherichia coli by directed evolution towards improved hydrolysis activity. Biochem Eng J, 2013, 79:182-186.
[23] Gupta RD, Goldsmith M, Ashani Y, et al. Directer evolution of hydrolases for prevention of G-type nerve agent intoxication. Nat Chem Biol, 2011, 7(2):120-125.
[24] Bernath-Levin K, Shainsky J, Sigawi L, et al. Directed evolution of nitrobenzene dioxygenase for the synthesis of the antioxidant hydroxytyrosol. Appl Microbiol Biotechnol, 2014, 98(11):4975-4985.
[25] Zhang S, He Y, Yu H, et al. Seven N-terminal residues of a thermophilic xylanase are sufficient to confer hyperthermostability on its mesophilic counterpart. PLos One, 2014, 9(1):e87632.
[26] Zheng H, Liu Y, Sun M, et al. Improvement of alkali stability and thermostability of Paenibacillus campinasensis Family-11 xylanase by directed evolution and site directed mutagenesis. J Ind Microbiol Biotechnol, 2014, 41(1):153-162.
[27] Singh SK, Heng C, Braker JD, et al. Directed evolution of GH43 β-xylosidase XylBH43 thermal stability and L186 saturation mutagenesis. J Ind Microbiol Biotechnol, 2014, 41(3):489-498.
[28] Louis Jeune V, Joergensen JA, Hajjar RJ, et al. Pre-existing anti-adeno-associated virus antibodies as a challenge in AAV gene therapy. Hum Gene Ther Methods, 2013, 24(2):59-67.
[29] Weiskopf K, Ring AM, Ho CC, et al. Engineered SIRPα variants as immunotherapeutic adjuvants to anticancer antibodies. Science, 2013, 341(6141):88-91.
[30] Wang J, Zhang Q, Huang Z, et al. Directed evolution of a family 26 glycoside hydrolase: endo-β-1, 4-mannanase from Pantoea agglomerans A021. J Biotechnol, 2013, 167(3):350-356.
[31] Ma J, Wu L, Guo F, et al. Enhanced enantioselectivity of a carboxyl esterase from Rhodobacter sphaeroides by directed evolution. Appl Microbiol Biotechnol, 2013, 97(11):4897-4906.
[32] Tinberg CE, Khare SD, Dou J, et al. Computational design of ligand-binding proteins with high affinity and selectivity. Nature, 2013, 501(7466):212-216.
[33] Qin B, Liang P, Jia X, et al. Directed evolution of Candida antarctica lipase B for kinetic resolution of profen esters. Catalysis Communications, 2013, 38:1-5.
[34] Li M, Rao C, Pei D, et al. Ovaferon, a novel recombinant protein produced by DNA-shuffling of IFN-α, shows antitumor effect in vitro and in vivo. Cancer Cell Int, 2014, 14(1):8.
[35] Chao FA, Morelli A, Haugner JC 3rd, et al. Structure and dynamics of a primordial catalytic fold generated by in vitro evolution. Nat Chem Biol, 2013, 9(2):81-83.
[36] Sabile CK, Janey JM, Mundorff EC, et al. Biocatalytic asymmetric synthesis of chiral amines from ketones applied to sitagliptin manufacture. Science, 2010, 329(5989):305-309.
陈建华,Email:jhchen@cpu.edu.cn.
2014-04-16
10.3969/cmba.j.issn.1673-713X.2014.06.010

