一个新型耐热普鲁兰酶的结构与功能
甄杰,胡政,李树芳,徐健勇,宋诙
1 天津科技大学,天津 300457
2 中国科学院天津工业生物技术研究所,天津 300308
淀粉 (Starch)是植物中普遍存在的储藏多糖,是植物体内能量的储备物质。淀粉有两种结构形式,一种是直链淀粉 (Amylose),另一种是支链淀粉 (Amylopectin)。直链淀粉是由葡萄糖通过α-1,4-糖苷键连接组成的,是不分支类型的淀粉;支链淀粉中除有α-1,4-糖苷键外,还有4%–5%的α-1,6-糖苷键。工业淀粉制糖生产中,淀粉首先经过高温液化步骤,经过 α-淀粉酶(α-Amylase)水解,将高聚糖水解为低聚糖,这一过程的反应条件为pH 6.0、温度95 ℃−105 ℃。液化产生的低聚糖随后在 β-淀粉酶(β-Amylase)、葡萄糖淀粉酶 (Glucoamylase)和普鲁兰酶 (Pullulanase)、以及异淀粉酶(Isoamylase)等作用下,残留的直链α-1,4-糖苷键和支链α-1,6-糖苷键被进一步水解,最终形成葡萄糖浆,该过程称为糖化步骤,其最适酶促条件为 pH 4.5、温度 60 ℃−62 ℃[1-3]。
普鲁兰酶 (Pullulanase,EC 3.2.1.41),也称去分支酶,是一种专一性水解支链淀粉分支点的α-1,6-糖苷键的葡萄糖苷水解酶,可特异性地将支链淀粉水解形成长度不同直链淀粉,是糖苷水解酶13家族成员之一[4]。在淀粉加工工业中,普鲁兰酶与糖化酶等的联合应用可有效地提高淀粉的水解效率和葡萄糖的产量,广泛地应用于高葡萄糖浆、高麦芽糖浆和干啤酒生产等应用中,是一种重要的工业酶。
本论文从我国云南轮马热泉下游淤泥中筛选获得一株耐热普鲁兰酶产生菌 Anoxybacillus sp.LM18-11,对其所产的耐热普鲁兰酶的结构和功能进行了系统全面的研究,并在枯草芽胞杆菌中进行了高效异源表达,为研发具有自主知识产权的高效耐高温的普鲁兰酶制剂奠定了良好的基础。
1 材料与方法
1.1 材料
1.1.1 菌株、质粒及培养基
菌株Anoxybacillus sp.LM18-11由本实验室分离,大肠杆菌DH5a、大肠杆菌BL21(DE3)、枯草芽胞杆菌 WB600为本实验室收藏。质粒pET32a用于在大肠杆菌中表达外源基因,大肠杆菌-芽胞杆菌穿梭质粒pBE980B (芽胞杆菌质粒pWB980的EcoRⅠ位点处插入来源于pUC18的大肠杆菌复制子,同时去除了原有的SacB信号肽序列),本实验室构建,用于在枯草芽胞杆菌WB600中表达外源基因。
Thermus培养基 (1 L):1 g 蛋白胨, 1 g 酵母粉,100 mg 氨三乙酸, 60 mg CaSO4·2H2O,100 mg Mg2SO4·7H2O, 8 mg NaCl, 100 mg KNO3, 700 mg NaNO3, 140 mg Na2HPO4·2H2O,0.47 mg FeCl3·6H2O, 2.2 mg MnSO4·H2O, 0.5 mg ZnSO4·7H2O, 0.5 mg H3BO3,25 μg CuSO4·5H2O,25 μg Na2MoO4·2H2O, 46 μg CoCl2·6H2O, 1.5%琼脂粉, pH 7.8。普鲁兰酶筛选培养基 (1 L):5 g 蛋白胨, 1 g 酵母粉, 0.7 g NaNO3, 0.1 g Na2HPO4, 0.2 g MgSO4·7H2O, 0.1S g CaCl2, 5 g普鲁兰糖, 1.5% 琼脂粉。鲁格式碘液(300 mL):1 g I2,2 g KI。常规LB培养基配制参见文献[5]。
1.1.2 酶及相关试剂
实验所用到的各种限制性内切酶、T4 DNA连接酶、Taq酶等购自Thermo Fisher公司。肠激酶购自NEB公司。各种抗生素、普鲁兰糖、IPTG等各种化学试剂购自Sigma公司。蛋白质纯化用HisTrap柱购自GE公司。引物由北京六合华大基因科技股份有限公司合成。基因组DNA提取及基因组步移试剂盒购自TaKaRa公司。
1.2 方法
1.2.1 普鲁兰酶产生菌的分离
称取1 g土样加入100 mL无菌水,经充分混匀静置30 min后,取上清液1 mL,做适当的梯度稀释,涂布于 Thermus固体平板,置于恒温培养箱60 ℃培养48−72 h。待长出1 mm左右大小的菌落后,再分别转接至普鲁兰酶筛选培养基,60 ℃培养48 h后,在平板表面滴加鲁格式碘液,挑选有水解圈的菌株进行进一步的分析。
1.2.2 常规实验操作
常规大肠杆菌的培养、质粒提取、转化和DNA基本操作参见文献[5],枯草芽胞杆菌的培养、转化参见文献[6]。DNA序列测定由天津华大基因完成。
1.2.3 普鲁兰酶在大肠杆菌中表达及纯化
PCR扩增的全长普鲁兰酶基因插入到质粒pET32a的EcoRⅤ-XhoⅠ位点,重组质粒转化大肠杆菌 BL21(DE3),转化子生长到 OD600达到0.6–0.8时,加入终浓度0.05 mmol/L的IPTG进行诱导,在16 ℃下培养16 h,离心收集菌体,超声破碎。表达的重组蛋白经HisTrap柱纯化,SDS-PAGE检测纯度。N1端缺失的普鲁兰酶基因的表达纯化方法同上。
1.2.4 蛋白晶体的制备及X-ray衍射
8 mg/mL的蛋白溶液 (25 mmol/L Tris-HCl,150 mmol/L NaCl,pH 7.5)经过一系列晶体生长条件优化,最终在 7% PEG8000、10% 乙二醇和10% 4-羟乙基哌嗪乙磺酸(pH 7.5)条件下,室温静置48 h获得普鲁兰酶晶体,并进行了X-ray衍射。蛋白-底物 (产物)复合物的晶体 X-ray衍射实验采用浸泡法,10 mmol/L麦芽三糖 (麦芽四糖)与蛋白晶体浸泡3 h后收集X-ray衍射数据。晶体结构图像的绘制利用 PyMOL软件(http://www.pymol.org/)。
1.2.5 普鲁兰酶的活性分析
酶活分析采用二硝基水杨酸 (DNS)法[7],在相应的温度、pH条件下,以0.5%普鲁兰糖为底物,加入0.1 μg普鲁兰酶反应10 min,加入DNS液终止反应,100 ℃水浴10 min,测定OD540光吸收值。以葡萄糖的DNS显色OD540光吸收值绘制标准曲线进行标定。酶活力单位定义为:在特定的反应条件下,每分钟水解普鲁兰糖产生相当于1 μmol葡萄糖的还原糖,所需的酶量为1活力单位 (U)。

表1 本研究中用到的引物Table 1 Primers used for this work
2 结果及分析
2.1 耐热普鲁兰酶菌株的分离及鉴定
土壤样品采自中国云南腾冲地区轮马温泉下游的淤泥,采样点位于北纬 25°25.357、东经98°16.442。经60 ℃培养共分离获得4株可水解普鲁兰糖的菌株,挑选出其中一株有明显普鲁兰水解圈的菌株LM 18-11进行进一步的分析、鉴定。提取该菌株的基因组DNA,合成16S rDNA扩增引物P1和P2,进行PCR反应,扩增得到一个约1.4 kb的DNA片段,对该PCR产物进行了序列测定 (GenBank Accession No. JF272543),经序列比对、系统进化树分析 (图1),确定该菌株为厌氧芽胞杆菌属菌株,将其命名为Anoxybacillus sp. LM 18-11。同时对该菌株所产普鲁兰酶进行了分析,对该酶水解普鲁兰糖的产物的进行了HPLC分析,结果显示该酶水解普鲁兰糖的主要产物为麦芽三糖 (图2),这一结果证实该酶为Ⅰ型普鲁兰酶。
2.2 耐热普鲁兰酶编码基因的克隆及在大肠杆菌中的表达

图1 菌株LM18-11的16S rDNA系统进化树Fig. 1 Phylogenetic tree of 16S rRNA of the LM18-11.

图2 普鲁兰糖水解产物的HPLC分析Fig. 2 HPLC analysis of catalytic products of PulA. (A)The HPLC spectrum of the standards. The peaks of G1,G2 and G3 represent glucose, maltose, respectively. (B)The HPLC spectrum of the pullulan after hydrolysis.
通过在 GenBank数据库中检索,获知在Anoxybacillus属中,菌株好热黄无氧芽胞杆菌Anoxybacillus flavithermus WK1已完成了全基因组的测序 (GenBank Accession No. CP000922),基因组注释查询显示 Anoxybacillus flavithermus WK1编码一个Ⅰ型的普鲁兰酶,该基因座位被命名为"Aflv_0438"。根据Ⅰ型普鲁兰酶保守区进行同源比对分析,同时参照该序列,合成引物P3和P4,以Anoxybacillus sp. LM 18-11基因组DNA为模板,进行PCR反应,扩增得到一个0.6 kb的DNA片段,并对该片段进行了测序分析。以该片段为基础,采用基因组步移技术,最终扩增得到包含一个完整阅读框序列的DNA片段 (GenBank Accession No. HQ844266),分析显示其中 140–2 263 bp为编码区,编码一个由707个氨基酸组成的蛋白质,与Aflv_0438一致性达84%,相似性达 91%,该基因被命名为 pulA。合成引物 P5和P6,PCR扩增获得完整的基因编码区,经酶切后,插入到诱导表达质粒 pET32a的EcoRⅤ-XhoⅠ位点,重组质粒转化大肠杆菌BL21(DE3),筛选正确的转化子,经IPTG诱导表达,SDS-PAGE分析 (图3),证实基因pulA获得了正确的表达,测定酶活力为23 U/mL。利用引物P6和P7扩增获得的N1端缺失的普鲁兰酶基因也如上述插入到质粒pET32a的EcoRⅤ-XhoⅠ位点,在大肠杆菌中实现了有效的表达。

图3 普鲁兰酶PulA诱导表达分析Fig. 3 SDS-PAGE analysis of PulA. M: marker; 1:before induction; 2: after induction.
2.3 普鲁兰酶PulA的酶学性质分析
pulA的大肠杆菌表达产物通过HisTrap柱纯化、肠激酶消化去除N端融合肽后,经SDS-PAGE检测,获得纯度在95%以上的酶蛋白PulA,被用于进行酶活性分析。
2.3.1 普鲁兰酶PulA最适反应温度、最适pH的分析
普鲁兰酶PulA反应最适pH的分析是通过测定 PulA 在一系列缓冲液 (乙酸-乙酸钠缓冲液(0.1 mol/L,pH 4.0–5.5);磷酸氢二钠-磷酸二氢钾缓冲液 (0.1 mol/L,pH 5.5–7.5);Tris-盐酸缓冲液(0.1 mol/L,pH 7.5–9.0)中,温度在55 ℃的条件下的催化活性完成的 (图 4),分析结果显示,普鲁兰酶PulA的最适pH为6.0,在pH 5.6–6.4的范围内,酶活力在90%以上。最适反应温度的测定是在pH 6.0的条件下,测定一系列温度范围下的催化活性,分析表明,该酶的最适反应温度为55–60 ℃ (图 5)。
2.3.2 普鲁兰酶PulA的酶动力学常数分析

图4 pH对普鲁兰酶PulA活性的影响Fig. 4 Relative activity of PulA at different pH.

图5 温度对普鲁兰酶PulA活性的影响Fig. 5 Relative activity of PulA at different temperature.

图6 普鲁兰酶PulA的双倒数曲线Fig. 6 Lineweaver–Burk plot of PulA.
在60 ℃、pH 6.0的条件下,通过测定0.1 µg普鲁兰酶PulA在一系列普鲁兰糖浓度(1.0、1.25、1.33、2.0、2.5、3.33和5.0 mg/mL)的条件下的表观催化速度,绘制 1/V-1/[S]双倒数曲线 (图 6),通过米氏方程计算出普鲁兰酶PulA的Vmax和Km分别为750 U/mg和1.47 mg/mL。
2.3.3 普鲁兰酶PulA的热稳定性分析
将普鲁兰酶PulA分别置于55 ℃、60 ℃、65 ℃条件下温育不同的时间后,取出0.1 µg普鲁兰酶PulA在pH 6.0、60 ℃条件下测定残余酶活性。结果显示PulA具有良好的热稳定性,经过60 ℃下热处理48 h,仍可保持50%以上的活性 (图7)。
2.3.4 金属离子、化学试剂等对普鲁兰酶PulA的影响
在反应液中分别加入不同的离子 (试剂),测定酶活的改变。结果显示Mg2+对普鲁兰酶PulA活性有一定的促进作用,Li+、K+、Ca2+、Ba2+、二硫苏糖醇 (DTT)等对酶活没有显著影响;Mn2+、Ni2+和Fe3+抑制普鲁兰酶PulA的活性;1 mmol/L Cu2+可使PulA的活性完全消失;EDTA也在一定程度上抑制PulA的活性,添加50 mmol/L Ca2+或Mg2+可部分恢复普鲁兰酶的活性 (表2)。
2.4 普鲁兰酶晶体的获得及X-ray分析
纯化后的普鲁兰酶在7% PEG8000, 10% 乙二醇, 10% HEPES (pH 7.5)的条件下获得了晶体 (图8A),并经X-ray衍射分析,蛋白-产物复合物的结构数据采用浸泡法收集,分辨率达到2.2 Å (图8B)。晶体结构分析显示该酶具有4个相对独立的结构域,其中催化中心结构域(A domain)具有糖苷水解酶13家族中典型的催化中心活性结构域[4],位于412位天冬氨酸、422位的谷氨酸和 526位的天冬氨酸是与催化重要相关的氨基酸;C端具有一个与催化结构域紧密作用的结构域 (C domain),PCR缺失实验表明该结构域的缺失将导致酶功能的完全丧失;在N端具有一个独立的结合域 (N1 domain),与底物的结合有关,N1结构域缺失了的普鲁兰酶PulA-N1的酶促反应动力学分析显示Vmax和Km分别为324 U/mg和1.95 mg/mL (图略),这一结果证实该结合域的缺失可导致酶活力和底物结合力的降低;N2结构域对于A结构域的稳定具有重要的作用,N2域缺失后表达的蛋白形成包涵体,上清液SDS-PAGE检测不到可溶性蛋白,同时也检测不到酶活性。此外,在该酶的催化活性域中还具有一个 Ca2+结合区,与 Ca2+紧密结合。EDTA螯合实验表明Ca2+的去除对该酶的催化活力没有显著的影响 (表 2),但额外添加Ca2+可提高酶的热稳定性,在65 ℃下,普鲁兰酶PulA的半衰期由2 h提高到12 h,表明Ca2+可能在酶的结构稳定性上发挥重要作用。

表2 金属离子、化学试剂等对普鲁兰酶PulA的影响Table 2 Effect of metal ions and reagents on PulA activity
2.5 普鲁兰酶基因在枯草芽胞杆菌中的异源表达

图7 普鲁兰酶PulA的热稳定性Fig. 7 Thermostability of PulA.
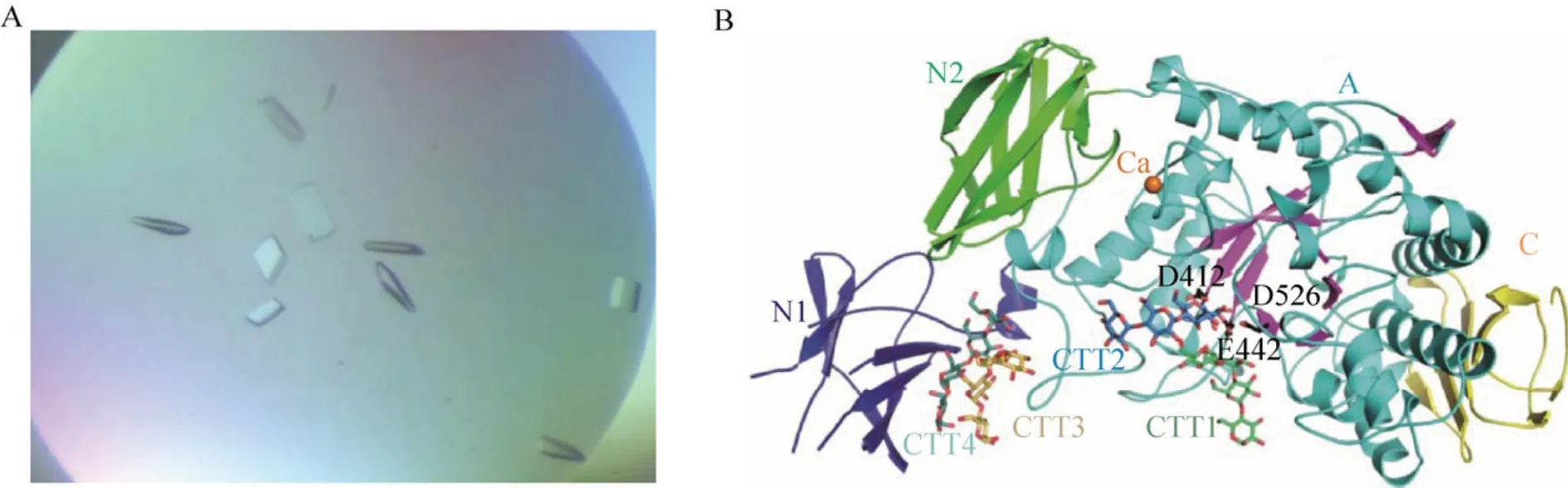
图8 普鲁兰酶PulA的晶体 (A)及酶蛋白-麦芽三糖复合物的结构 (B)Fig. 8 Crystal of PulA (A)and overall structure of PulA-maltotetraose complex (B). The secondary structural elements of PulA was shown and each domain was indicated: N1 (blue), N2 (green), A (cyan for α-helix, purple for β-strand), and C (yellow), CTT represent maltotriose.
利用引物P8和P9,扩增全长的普鲁兰酶基因pulA,经XhoⅠ-PstⅠ酶切后,插入到经过同样酶切的质粒 pBE980B位点上,重组质粒中pulA位于芽胞杆菌组成型强启动子P43的下游。该质粒转化大肠杆菌DH5α,筛选验证正确的转化子后,提取重组质粒,转化枯草芽胞杆菌WB600,转化子 WB600(pBE980B-pulA)在 LB培养基中振荡培养24 h,上清液的酶活达到42 U/mL (pH 6.0、60℃),相比原始菌株Anoxybacillus sp. LM 18-11 (1.02 U/mL),表达量提高了40倍以上。
3 讨论
自上世纪70年代起,酶制剂工业逐渐成为一个重要的产业,目前世界酶制剂总产值达数百亿美元,淀粉水解酶类作为第二大类酶制剂,尤其是近年来随着生物质能源开发的兴起,淀粉已成为生产乙醇的重要原料,淀粉水解相关的酶类更加显示出巨大的市场前景。自然界中的淀粉大多为支链淀粉,其中含有 4%−5%的α-1,6-糖苷键,为提高淀粉的水解作用,提高淀粉利用率,降低生产成本,提高效益,世界各国纷纷研究开发普鲁兰酶。目前,普鲁兰酶的生产商主要是诺维信和杰能科,我国国内的普鲁兰酶还基本依赖进口。
近些年,普鲁兰酶的研究还主要集中在耐热、耐酸 (耐碱)高产普鲁兰酶菌株的筛选及基因的克隆与表达[9-20]。本研究从我国云南轮马热泉土壤中筛选获得一株耐热普鲁兰酶产生菌Anoxybacillus sp.LM 18-11,并从中克隆表达了耐热普鲁兰酶的编码基因,并对其结构和功能进行了细致的研究,研究表明该普鲁兰酶具有极高的催化效率,比活高达750 U/mg,是目前已有报道中比活力最高的普鲁兰酶[14-28]。其中诺维信普鲁兰酶 Promozyme 200 L的比活为350 U/mg[8],广西大学李美蓉等从腊样芽胞杆菌中克隆的普鲁兰酶PulB和PulC的最大比活分别为435 U/mg和400 U/mg[15]。晶体结构分析的结果首次发现在该酶的N端有一个底物结合域,对酶的催化功能有重要的作用。同时该酶还具有良好的热稳定性,在 60 ℃条件下处理48 h,仍可保持50%以上的活力,能很好的适应淀粉加工工艺的需要。此外,为了进一步提高酶的产量和活性,降低生产成本,真正实现产业化,我们利用枯草芽胞杆菌作为表达系统,使普鲁兰酶在枯草杆菌组成型启动子的控制下得到了有效的表达,胞外分泌表达量达到了42 U/mL,比原始菌株的表达量提高了40倍以上,进一步的培养基、发酵条件优化研究正在进行中。同时,在细致的晶体结构、蛋白-底物作用方式了解的基础上,通过理性设计、定向进化等方式,能够进一步提高该酶的热稳定性、催化活力,耐酸、耐碱等特性,使之能够更好地满足淀粉加工、洗涤、纺织等工业生产多方面的需求。本研究为改变我国普鲁兰酶依赖进口的局面,获得具有自主知识产权的新型耐热普鲁兰酶,具有重要的意义,有望创造巨大的经济效益。
致谢 本论文的完成要特别感谢中国科学院天津工业生物技术研究所郭瑞庭研究员、黄俊祥副研究员、任飞飞博士在蛋白晶体X-ray数据收集、结构分析等给予的帮助。
[1]Horváthová V, Godány A, Sturdík E, et al.α-Amylase from Thermococcus hydrothermalis:Re-cloning aimed at the improved expression and hydrolysis of corn starch. Enzyme Microb Technol,2006, 39(6): 1300–1305.
[2]Wang S, Lu Z, Lu M, et al. Identification of archaeon-producing hyperthermophilic α-amylase and characterization of the α-amylase. Appl Microbiol Biotechnol, 2008, 80 (4): 605–614.
[3]Taniguchi H, Honnda Y. Amylases, Encyclopedia of Microbiology. 3rd ed. San Diego, USA:Academic Press, 2009: 159–173.
[4]Monika DP, Jacek B. Pullulan degrading enzyme of bacterial origin. Crit Rev Microbiol, 2004, 30:107–121.
[5]Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning: A Laboratory Manual. 2nd ed. New York:Cold Spring Harbor Laboratory Press, 1989.
[6]Anagnostopoulus C, Spizizen J. Requirements for transformation in Bacillus subtilis. J Bacteriol,1961, 81 (5): 741–746.
[7]Bernfeld P. Amylases, alpha and beta. Methods Enzymol, 1955, 1: 149–158.
[8]Chen C. Promozyme 200L and its production strain.Food Fermn Ind, 1992, 6: 72–76 (in Chinese).程池. 普鲁兰酶Promozyme 200L及其生产菌种.食品与发酵工业, 1992, 6: 72–76.
[9]Lu J, Jin C. Pullulanase and its production strains.Liquor Making, 1998, 5: 1–6 (in Chinese).陆健, 金冲. 普鲁兰酶及其生产菌种. 酿酒,1998, 5: 1–6.
[10]Suzuki Y, Hatagaki K, Oda H. A hyperthermostable pullulanase produced by an extreme thermophile, Bacillus flavocaldarius KP1228, and evidence for the proline theory of increasing protein thermostability. Appl Microbiol Biotechnol, 1991, 34(6): 7–14.
[11]Tang BY, Zhu XH, Liu J. Selection of the acid heat-resistant pullulanase-producing strain and its fermentation conditions. Microbiol China, 2001,28(1): 39–43 (in Chinese).唐宝英, 朱晓慧, 刘佳. 耐酸耐热普鲁兰酶菌株的筛选及发酵条件的研究. 微生物学通报, 2001,28(1): 39–43.
[12]Ma XJ, Zhang XJ, Wang R, et al. Studies on screening and breeding of alkaline pullulanase producing bacterium and regulation of nutrition.Acta Bot Boreal-Occident Sin, 2002, 22(4): 3–8 (in Chinese).马晓军, 张晓君, 王锐, 等.碱性普鲁兰酶产生菌选育和发酵条件的研究. 西北植物学报, 2002,22(4): 3–8.
[13]Yan W, Nie R, Xu Y. Expression and secretion regulation of Bacillus naganoensis pullulanase in recombinant Escherichia coli. Acta Microbiol Sin,2013, 53(2): 145–153 (in Chinese).严伟, 聂尧, 徐岩. 长野芽胞杆菌 (Bacillus naganoensis)普鲁兰酶在大肠杆菌中的活性表达与分泌调控. 微生物学报, 2013, 53(2): 145–153.
[14]Han P, Zhou P, Yan QJ, et al. Expression of a pullulanase gene from the thermophilic Bacillus subtilis and characterization of the recombinant enzyme. Microbiol China, 2011, 38(12):1755–1761 (in Chinese).韩鹏, 周鹏, 闫巧娟, 等. 嗜热枯草芽胞杆菌普鲁兰酶基因的表达与重组酶的性质. 微生物学通报, 2011, 38(12): 1755–1761.
[15]Li MR, Wang XB, Huang Y, et al. Gene expression and characterisation of three pullulanases from Bacillus cereus GXBC-3. Chin J Biotech, 2012,28(4): 466−475 (in Chinese).李美蓉, 汪小波, 黄英, 等. 蜡样芽胞杆菌GXBC-3三个普鲁兰酶基因的表达及其酶学特性. 生物工程学报, 2012, 28(4): 466–475.
[16]Zhang Y, Liu YH, Li Y, et al. Extracellular expression of pullulanase from Bacillus naganoensis in Escherichia coli. Ann Microbiol,2013, 63(1): 289–294.
[17]Li YR, Zhang L, Niu DD, et al. Cloning, expression,characterization, and biocatalytic investigation of a novel bacilli thermostable typeⅠ pullulanase from Bacillus sp. CICIM 263. J Agric Food Chem, 2012,60(44): 11164–11172.
[18]Wasko A, Polak-Berecka M,Targonski Z.Purification and characterization of pullulanase from Lactococcus lactis. Prep Biochem Biotechnol,2011, 41(3): 252–261.
[19]Moubasher H, Wahsh SS, El-Kassem NA.Purification of pullulanase from Aureobasidium pullulans. Microbiology, 2010, 79(6): 759–766.
[20]Kang J, Park KM, Choi KH, et al. Molecular cloning and biochemical characterization of a heat-stable type Ⅰpullulanase from Thermotoga neapolitana.Enzyme Microb Technol, 2011, 48(3): 260–266.
[21]Rudiger A, Jorgensen PL, Antranikian G. Isolation and characterization of a heat-stable pullulanase from the hyperthermophilic archaeon Pyrococcus woesei after cloning and expression of its gene in Escherichia coli. Appl Environ Microbiol, 1995,61(2): 567–575.
[22]Kim CH, Nashiru O, Ko JH. Purification and biochemical characterization of pullulanase type Ⅰfrom Thermuscaldophilus GK-24. FEMS Microbiol Lett, 1996, 138(2/3): 147–152.
[23]Koch R, Canganella F, Hippe H, et al. Purification and properties of a thermostable pullulanase from a newly isolated thermophilic anaerobic bacterium,Fervidobacterium pennavorans Ven5. Appl Environ Microbiol, 1997, 63(3): 1088–1094.
[24]Kuriki T, Park JH, Okada S, et al. Purification and characterization of thermostable pullulanase from Bacillus stearothermophilus and molecular cloning and expression of the gene in Bacillus subtilis. Appl Environ Microbiol, 1998, 54(11):2881–2883.
[25]Bertoldo C, Duffner F, Jorgensen PL, et al.Pullulanase type Ⅰ from Fervidobacterium pennavorans Ven5: cloning, sequencing, and expression of the gene and biochemical characterization of the recombinant enzyme. Appl Environ Microbiol, 1999, 65(5): 2084–2091.
[26]Niehaus F, Peters A, Groudieva T, et al. Cloning,expression and biochemical characterisation of a unique thermostable pullulan-hydrolysing enzyme from the hyperthermophilic archaeon Thermococcus aggregans, FEMS Microbiol Lett, 2000, 190(2):223–229.
[27]Bertoldo C, Armbrecht M, Becker F, et al. Cloning,sequencing, and characterization of a heat- and alkali-stable type Ⅰpullulanase from Anaerobranca gottschalkii. Appl Environ Microbiol, 2004, 70(6): 3407–3416.
[28]Malle D, Itoh T, Hashimoto W, et al.Overexpression, purification and preliminary X-ray analysis of pullulanase from Bacillus subtilis strain 168. Acta Cryst, 2006, 62: 381–384.

