西沙群岛海域海洋放线菌的分离及其抗菌活性
房耀维,王淑军,刘 姝,吕明生,焦豫良,陈国强,潘建梅
(淮海工学院海洋学院,江苏 连云港 222005)
抗生素的长期广泛使用导致耐药性病原细菌种类增加,甚至出现“超级细菌”,新的感染性疾病也不断涌现,致使新型抗生素的开发迫在眉睫[1-2]。放线菌可以产生丰富的抗生素,约70%的抗生素由放线菌产生[3]。由于陆源放线菌已经被长期筛选和研究,从陆源放线菌中发现新抗生素的几率已逐步降低[4]。海洋占地球面积的70%,且环境独特,不仅造就了海洋放线菌种类丰富多样,而且可以产生比陆源放线菌结构和活性更加丰富多样的代谢产物,为新型抗生素的开发提供了丰富的资源[5]。近十几年来,日本、美国、德国、英国等一些国家和国内的研究者逐步把新型抗生素的筛选方向转向海洋[6]。特别是2005年首个专性海洋放线菌属盐孢菌属 (Salinospora sp.)的报道,带来新一轮海洋放线菌抗生素的研究和开发高潮,该属放线菌可以产生丰富的新型抗生素[7]。至2010年,各国研究者在海洋环境中发现的放线菌属有50个,其中在海洋环境首次描述的新属有12个,大部分新属可以产生新型抗生素[8]。分子生物学技术快速发展,通过构建宏基因组文库筛选新型抗生素可以避开微生物的分离过程,扩大了抗生素筛选范围。但是,目前环境大片段DNA的提取困难、宏基因在宿主细胞中不表达或表达量低、无法进行高通量的活性物质筛选等问题制约了宏基因组文库的实用性[9-10]。通过化学修饰或全化学合成可以获得新颖抗生素,但是合成过程复杂,且造成环境污染[11]。可见,对海洋放线菌进行调查,筛选分泌抗生素放线菌菌株,仍然是一个重要的开发新型抗生素的途径。本研究对我国地处亚热带的西沙群岛海洋沉积物样品的放线菌进行分离,比较了不同培养基及样品处理方式对菌株分离的影响,考察分离放线菌对海水的依赖性,并考察了放线菌的抗菌活性,为新型抗生素的发现和研究提供菌种来源。
1 材料与方法
1.1 材料
1.1.1 样品 取样于西沙群岛海域的永兴岛(16°50'N,112°19'E)、石岛(16°50'N,112°19'E)、七连屿(16°50'N,112°20'E)、西沙州(16°59'N,112°13'E)和鸭公岛(16°30'N,111°40'E),每个岛周围取样点20个,水深10~15 m,样品置入无菌玻璃瓶,立即放于冰盒保存,并尽快送回实验室进行试验处理。
1.1.2 供试菌株 金黄色葡萄球菌 (Staphylococcus aureus AS1.2465)、大肠埃希菌 (Escherichia coli AS1.487)、啤酒酵母 (Saccharomyces cerevisiae AS2.114)、扩展青霉 (Penicillium expansum AS3.3703),来源于中国普通微生物菌种保藏中心。
1.1.3 培养基 分离培养基见表1,pH均为7.2,1×105Pa灭菌30 min,降温后分别添加放线菌酮、萘啶酮酸至终浓度分别为100和25 mg/L。发酵培养基为黄豆粉培养基;指示菌细菌用培养基为营养琼脂培养基,真菌为PDA培养基[12]。

表1 海洋沉积物放线菌分离培养基Table 1 Media for isolation marine actinomycetes
1.2 方法
1.2.1 样品预处理 样品预处理具体方法见表2[17]。

表2 海洋沉积物样品预处理方法Table 2 Sampling processing strategy for marine sediment
1.2.2 菌株的计数与分离 称取每个岛的20份海泥样品5 g(湿重),混匀,作为这个岛的总样品,进行放线菌的分离。用不同方式对样品预处理后,用灭菌海水梯度稀释,选择适当稀释度涂布不同培养基平板,每个稀释度10个重复,用Parafilm封口膜密封琼脂平板,28℃培养箱中倒置培养4~8周。观察琼脂平板,根据菌落特征和简单染色后显微镜观察对不同放线菌和细菌进行区分,菌落计数。随机抽取3个重复进行数据统计分析。将分离菌株点种于M1平板,28℃培养箱中倒置培养4~8周,用玻璃纸法显微镜观察放线菌的气生菌丝和基内菌丝,用印片法显微镜观察放线菌孢子丝,结合样品的不同以及菌落的大小、色泽、形态、生长速度等差异,区分不同放线菌。如果在原培养基平板生长,但是点种到M1平板后菌株不生长,则记为不同种类,并用原平板点种,用上述同样方法区分不同菌株。
1.2.3 放线菌生长的海水依赖性判断 根据Jensen的方法判断菌株生长的海水依赖性[13]。将能在M1平板上生长的菌株点种于M1平板,不能生长于M1平板的菌株点种于菌株相应的筛选平板。28℃培养箱中倒置培养至肉眼可见菌落后,用接种环同时点种于用去离子水和海水配置的相应平板各一个。28℃培养箱中倒置培养6周,观察菌落生长情况。
1.2.4 菌株的鉴定 根据放线菌菌株不同培养基上气丝有无、菌丝颜色、可溶性色素有无等形态培养特征初步归类,从每个类群中选取代表菌株,根据于素亚等[1]报道的方法进一步鉴定放线菌到属。
1.2.5 发酵样品制备 将纯化后的单菌落接种至M1培养基,培养至细胞压积(packed cell volume,PCV)2%后,取0.2 mL接种于装有25 mL发酵培养基的100 mL三角瓶中发酵10 d,8000×g,离心10 min,上清液过0.22 μm微孔滤膜后为待测样品。
1.2.6 抗菌活性的测定 采用管碟法测定抑菌活性[18]。每个牛津杯中加入200 μL发酵样品,采用含金黄色葡萄球菌、大肠埃希菌、啤酒酵母、扩展青霉的指示平板,于指示菌适合的温度培养后观察,并用游标卡尺测定其抑菌圈大小。所有试验设3个重复。
1.2.7 数据统计分析 采用 SPSS 17.0(SPSS Inc,Chicago,USA)进行数据统计分析。
2 结果与分析
2.1 放线菌菌株的鉴定
根据放线菌菌株的形态培养特征归类后选取代表菌株93株,进行菌株鉴定。表明93株放线菌属于放线菌9个科,13个属,分别为链霉菌科(Streptomycetaceae)链霉菌属(Streptomyces);诺卡菌科(Nocardiaceae)诺卡氏菌属(Nocardia);拟诺卡菌科(Nocardiopsaceae)拟诺卡菌属(Nocardiopsis);微球菌科(Micrococcaceae)节杆菌属(Arthrobacter);束丝放线菌科(Actinosynnemataceae)束丝放线菌属(Alloactinosynnema);小单孢菌科(Micromonosporaceae)小单孢菌属(Micromonospora)、疣孢菌属(Verrucosispora)和盐孢菌属(Salinispora);假诺卡菌科(Pseudonocardiaceae)糖单孢菌属(Saccharomonospora)和无枝菌酸菌属(Amycolatopsis);高温单孢菌科(Thermomonosporaceae)马杜拉菌属(Actinomadura);微球菌科(Micrococcaceae)微球菌属(Micrococcus)。其中,链霉菌属菌株最多,占总菌数的40%。姜怡等[3]对波罗的海放线菌多样性进行分析,也发现链霉菌属菌株占培养菌株的大多数,达到50%以上。取样时间、地点及海水深度等条件对放线菌的种类影响较大[7-8]。
2.2 不同样品预处理方法对放线菌分离的影响
对样品进行预处理的目的是在提高放线菌孢子萌发,或者在最小程度杀死或抑制样品中放线菌生长的条件下,最大程度地杀死或抑制除放线菌以外其他微生物的生长,从而利于样品中放线菌的分离。采用11种物理及化学方法对样品进行预处理,利用10种培养基对海泥样品中的放线菌进行分离,共获得放线菌菌株383株。样品预处理后分离得到的放线菌菌株及筛选得率见表3。样品预处理方式对放线菌分离有较大影响。样品不进行预处理,细菌菌落较多,细菌生长较放线菌快,较快的覆盖满平板,造成放线菌难以分离。真菌有时亦能生长,较快即可布满平板,影响放线菌分离。通过加热、干燥、辐射、化学药剂及冷冻等方式对样品进行预处理,都不同程度地降低了细菌的数量,利于放线菌的分离。通过H2和H3处理后,不但降低了细菌的数量,并使放线菌数量有所增加,分离效果较好。其原因可能是55~60℃加热激发了放线菌孢子的萌发。常显波等[15]也发现55℃加热增加了海泥样品放线菌的分离数量。王海雁等[19]研究表明干燥可以显著降低海泥样品中细菌的数量,利于放线菌的分离。不同处理方式的放线菌分离率不同,表明不同放线菌对不同处理方式的抵抗性不同,从而造成放线菌得率的差异[16]。可见,采用不同预处理方式对同一个样品进行预处理,可以提高放线菌分离得率。

表3 不同样品预处理方式对放线菌分离的影响Table 3 The effect of sampling processing strategy on the isolation of marine actinomycetes
2.3 不同培养基对放线菌分离的影响
采用10种培养基对海泥样品中的放线菌进行分离,共分离获得383株放线菌,其中专性海洋放线菌23株,表4显示了不同培养基分离到的放线菌数量。不同培养基分离到的放线菌菌株数目和种类都有较大差异。其中,培养基M8分离获得的放线菌菌株数量最多,其次为M7培养基。Ward等[20]用20种培养基分离Mariana海沟处底泥样品中的放线菌,也发现棉籽糖和组氨酸培养基的分离效果最好。M3、M4和M5分离获得的菌株数量较少。这表明海洋放线菌对营养物的要求有一定偏好性,根据海洋放线菌的营养需求偏好性设计培养基成分,有望提高海洋放线菌的分离效率和数量。不同培养基筛选获得的海水依赖性放线菌的数量差异也较大。虽然培养基M3、M4和M5分离的放线菌菌株数量较少,但是其中海水依赖性放线菌即专性海洋放线菌的比例较高,M5 最高,达到25.76%。Jensen 研究组[16]利用培养基M1至M5筛选深海样品放线菌,分离得到了高比例的专性海洋放线菌,其中M5和M10分离得到的专性海洋放线菌比率分别为48%和82%。分析其原因,Jensen研究组取海泥样品的位置较深,海水越深受到陆地淡水及气候等因素的影响越小,因此专性海洋放线菌的比率较离岛屿或陆地较近或海水较浅的海泥样品高。
不同培养基分离到的放线菌种类不同,其中M10分离的放线菌的属的数量最多,达到9个属;M5分离的放线菌属只有2个,为盐孢菌属和链霉菌属;其他培养基分离到4~5个属的放线菌。尽管M6到M9分离得到的菌株数量较大,分离放线菌同样分布于4~5个属。这可能是由于添加了不同碳源、金属离子等营养物质,使得偏好这些营养条件的放线菌快速生长;但是这些快速生长的放线菌可能会抑制不偏好这些营养物的放线菌,减少放线菌分离的种类。M10分离的放线菌的属的数量最多,可能是由于NRS提取物能满足大多数放线菌的营养需求,且没有添加外源营养物质,不会加大放线菌之间的拮抗作用,因此能分离得到更多种类的放线菌。
2005年,Jensen研究组首次报道了专性海洋放线菌—盐孢菌属(Salinispora sp.)。目前,已经有超过12个属的专性海洋放线菌被分离报道[3]。我国的研究者Tian等[21]从南中国海筛选获得了专性海洋放线菌新种Streptomyces oceani。虽然专性放线菌的报道较晚,但是由于专性放线菌可以产生结构和生物活性丰富新颖的次级代谢产物,成为海洋微生物研究热点的明星类群,也引领更多研究者进行海洋专性微生物的研究。本研究分离的专性海洋放线菌分别属于盐孢菌属和小单孢菌属。
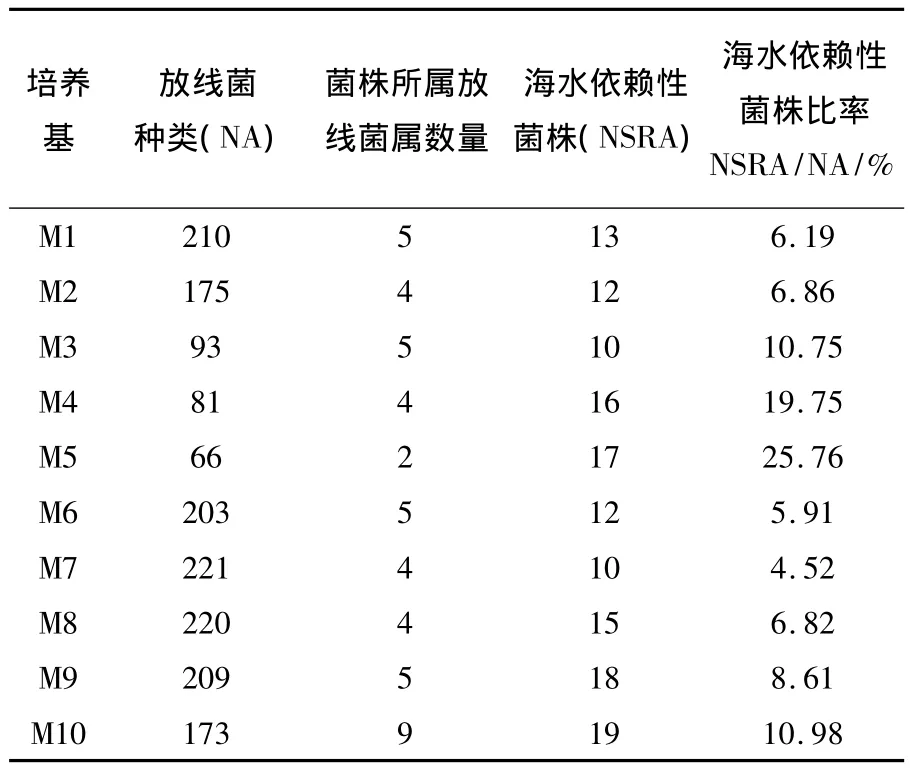
表4 不同培养基对放线菌分离的影响Table 4 The effect of media on the isolation of actinomycetes
2.4 分离菌株的抑菌活性
用管碟法测定383株放线菌的抗菌活性(图1,表5)。对于所有分离菌株而言,抑菌活性以抗革兰阳性菌Staphylococcus aureus为主,达到总菌数的 19.06%,抗 Saccharomyces cerevisiae和抗Penicillium expansum的较少;抗Escherichia coli最少,仅为4.96%。专性海洋放线菌中具有抑菌活性的菌株数量显著高于非专性海洋放线菌。姜怡等[3]对波罗的海放线菌进行了分离和抗菌活性筛选,抗Staphylococcus aureus的菌株为20%,抗真菌的为10%,抗 Escherichia coli的为2.5%。肖静等[22]对红树林样品中的放线菌进行了分离和抗菌活性筛选,发现对 Staphylococcus aureus、Escherichia coli、Candidaalb icans和水稻纹枯病原菌Rhizoctonia solani的抑菌活性的比率分别为19%、1.2%、8%和15%。由此可见,样品不同,样品中的放线菌组成不同,拮抗不同类型微生物的放线菌比率不同。但是总体上抗革兰阳性菌Staphylococcus aureus的比例较高,抗革兰阴性菌Escherichia coli的最少。

表5 383株放线菌的抗菌活性Table 5 The antimicrobial activities of 383 actinomycetes

图1 抗菌活性筛选代表图片Fig.1 The representative photo of antimicrobial activity strains
在所有产抗菌物质的菌株中,有16株菌株同时抗革兰阳性与阴性细菌,有13株可同时抗革兰阳性与阴性细菌和酵母,有9株可同时抗革兰阳性与阴性细菌和霉菌,有6株可同时抗革兰阳性与阴性细菌、酵母和霉菌,而这6株菌中有4株为专性海洋放线菌。6株放线菌对供试菌的抑菌直径见表6,CMG091属于链霉菌属,CMS053为小单孢菌属,CMS039为小单孢菌属,CMQ131、CMZ005和CMZ023均为盐孢菌属。CMZ005和CMZ023的抑菌圈直径较大。专性海洋放线菌多生长缓慢,在抢占营养物及生存空间方面较生长较快的其他微生物明显处于劣势,因此,这些微生物为了能在海洋环境中占有一席之地,更容易进化合成广谱抑菌物质,抑制其他微生物的生长,从而抢占营养及生存空间[23]。
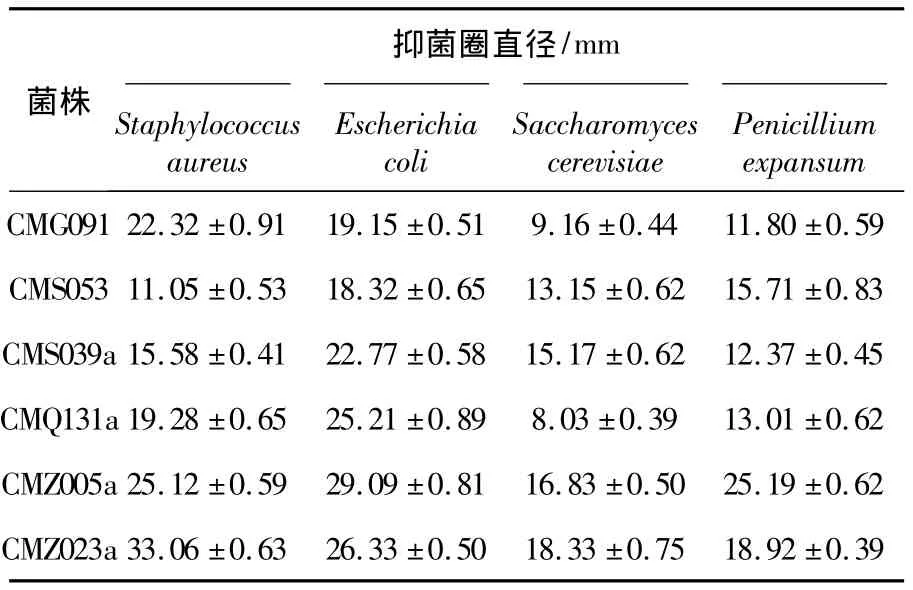
表6 6株抗菌活性菌株的抗菌效果Table 6 The antimicrobial activities of 6 actinomycetes
3 讨论
利用10种培养基、11种样品预处理方式对南中国海域西沙群岛的海洋沉积物样品中的放线菌进行分离,获得了包括23株专性海洋放线菌在内的383株放线菌。选择代表性菌株93株进行鉴定,表明菌株隶属于9个科,11个属。以Staphylococcus aureus、Escherichia coli、Saccharomyces cerevisiae和Penicillium expansum为指示菌考察分离放线菌的抑菌活性,发现抗Staphylococcus aureus最多,抗Escherichia coli最少,专性海洋放线菌中有抑菌活性的菌株比率显著高于其他菌株。并获得包含4株专性海洋放线菌在内的6株放线菌对4种供试菌都有抗性,可以作为抗菌物质开发的菌株来源。对于具有抗菌活性的放线菌产抗菌物质的纯化、结构鉴定,以及合成基因簇信息等研究正在进行中。
[1]于素亚,潘华奇,胡江春,等.深海放线菌08A4的鉴定及其抗真菌活性产物研究[J].微生物学杂志,2012,32(5):1-5.
[2]吴少杰,王淑军,李富超,等.胶州湾海洋放线菌的分布及其抗微生物活性[J].中国海洋药物杂志,2006,25(5):10-13.
[3]姜怡,曹艳茹,王茜,等.波罗的海放线菌的多样性[J].微生物学报,2011,51(11):1461-1467.
[4]Meena B,Rajan LA,Vinithkumar NV,et al.Novel marine actinobacteria from emerald Andaman&Nicobar Is-lands:a prospective source for industrial and pharmaceutical byproducts[J].BMC Microbiology,2013,13:145.
[5]王海雁,刘健,赵淑江.南麂岛海域沉积物中海洋放线菌的分离研究[J].海洋科学,2010,34(1):48-51.
[6]Goodfellow M,Fiedler HP.A guide to successful bioprospecting:informed by actinobacterial systematics[J].Antonie van Leeuwenhoek,2010,98(2):119-142.
[7]Mincer TJ,Fenical W,Jensen PR.Culture-dependent and culture-independent diversity within the obligate marine actinomycete genus Salinispora[J].Applied and Environmental Microbiology,2005,71(11):7019-7028.
[8]田新朋,张偲,李文均.海洋放线菌研究进展[J].微生物学报,2011,51(2):161-169.
[9]印蕾,高向东,顾觉奋.宏基因组技术研究进展[J].中国医药生物技术,2012,7(3):216-219.
[10]杨建,洪葵.宏基因组文库技术获得聚酮化合物[J].遗传,2006,28(10):1330-1336.
[11]Lane AL,Moore BS.A sea of biosynthesis:marine natural products meet the molecular age[J].Natural Product Reports,2011,28(2):411-28.
[12]林永成.海洋微生物及其代谢产物[M].北京:化学工业出版社,2003:36.
[13]Mincer TJ,Jensen PR,Kauffman CA,et al.Widespread and persistent populations of a major new marine actinomycete Taxon in ocean sediments[J].Applied and Environmental Microbiology,2002,68(10):5005-5011.
[14]梁效伟,陈名洪,林如,等.台湾海峡海洋沉积物放线菌的多样性[J].中国抗生素杂志,2012,37(4):269-275.
[15]常显波,刘文正,尹琦,等.海洋放线菌不同分离方法的比较研究[J].海洋科学,2012,36(8):35-39.
[16]Jensen PR,Gontang E,Mafnas C,et al.Culturable marine actinomycete diversity from tropical Pacific Ocean sediments[J].Environmental Microbiology,2005,7(7):1039-1048.
[17]Hames-kocabas EE,Uzel A.Isolation strategies of marine-derived actinomycetes from sponge and sediment samples[J].Journal of Microbiological Methods,2012,88(3):342-347.
[18]刘姝,陆颖健,陆兆新,等.海洋链霉菌GB-2发酵产物的抗细菌活性及性质研究[J].生物工程学报,2007,23(6):1077-1081.
[19]王海雁,刘健,赵淑江.南麂岛海域沉积物中海洋放线菌的分离研究[J].海洋科学,2010,34(1):48-51.
[20]Ward AC,Horikoshi K,Bull AT.Diversity of actinomycetes isolated from Challenger Deep sediment(10,898 m)from the Mariana Trench[J].Extremophiles,2006,10(3):181-189.
[21]Tian XP,Xu Y,Zhang J,et al.Streptomyces oceani sp.nov.,a new obligate marine actinomycete isolated from a deep-sea sample of seep authigenic carbonate nodule in South China Sea[J].Antonie van Leeuwenhoek,2012,102(2):335-343.
[22]肖静,许静,谢庶洁,等.红树林放线菌的分离及其抗菌和抗肿瘤细胞活性[J].应用与环境生物学报,2008,14(2):244-248.
[23]Penn K,Jensen PR.Comparative genomics reveals evidence of marine adaptation in Salinispora species[J].BMC Genomics,2012,13(1):86-97.

