基于拟靶向液相色谱-质谱联用的胃癌患者血清代谢组分析
杨太忠,罗 萍,李艳丽,华 瑞,尹沛源*,许国旺
(1.吉林大学第一医院,吉林 长春 130021;2.中国科学院大连化学物理研究所,辽宁 大连 116023)
胃癌(gastric cancer,GC)是全球最普遍和致命的癌症之一,每年确诊的新发病例大约有100万人,是癌症相关死亡的第二大病因[1]。由于早期胃癌通常无症状,由此带来的诊断滞后是目前癌症患者高死亡率的一个主要原因。迄今为止,侵入性的内窥镜检查仍然是胃癌诊断最可靠和准确的方法。尽管内镜筛查胃癌在日本广泛实施,但是在其他绝大部分国家,这种侵入性的检查方法的可行性和经济性仍受到质疑[2]。就这一点而言,目前临床上缺乏一种简单而可靠的用于胃癌诊断的血清标志物[3]来实现对这一致命癌症的无创筛检。
代谢组学(metabolomics)是目前癌症早期诊断和预后判断的相关分子标志物发现和筛选[4-6]的一种高效的技术手段。在代谢组学研究中,代谢组分析常采用靶向分析[7]及非靶向分析[8]的策略,而非靶向分析是其中的主流,尤其适用于代谢标志物发现的相关研究[9]。飞行时间(time of flight,TOF)质谱因其扫描速度快,分辨率高,是非靶向代谢组分析最常用的仪器之一。基于TOF的非靶向方法[10]具有分析通量高、数据信息丰富等优点,但其在数据的稳定性、重复性以及定量的线性范围等方面仍存在一定的缺陷。以三重四极杆(triple-quadrupole,TQ)质谱多反应监测(multiple reaction monitoring,MRM)技术为代表的靶向分析[11]方法虽然能够弥补非靶向方法的上述不足,但是其自身也存在着同时分析的靶标数目有限、建立方法依赖于标准品等不足,对于生物样本代谢组信息的覆盖仍有局限,并不适用于大规模的代谢轮廓分析。
考虑到非靶向及靶向分析方法的特征及优势,本实验室提出了“拟靶向”的概念[12]。拟靶向技术方案的核心思想是将非靶向分析的高通量、无偏向的代谢物信息获取和靶向方法的高特异性检测和准确定量相结合[13],从而实现对待测样本中已知及未知的多个代谢物离子靶标进行同时检测。该方法在保证对代谢组信息覆盖及检测灵敏度的同时,显著提高了数据的线性范围以及重复性等指标,保证了后续的基于代谢组数据的标志物发现及验证。基于这一概念,我们先后建立了基于气相色谱-质谱联用、液相色谱-质谱联用的拟靶向方法,并应用于血清肝癌标志物的筛选[13]、植物代谢组学等研究中[12,14]。
为了发现胃癌的早期诊断标志物,基于上述拟靶向分析策略,本研究采用超高效液相色谱-质谱(UPLC-MS)分析了20例胃癌患者与40例正常人群的血清代谢谱,通过代谢组数据分析获得胃癌患者系统代谢特征,研究胃癌患者的代谢分型,以期发现胃癌诊断的潜在代谢标志物。
1 实验部分
1.1 临床样本采集
本实验所采集的血液样本均具有病人的知情同意书,并得到吉林大学第一医院伦理委员会批准,其中胃癌患者均来自于吉林大学白求恩第一医院的接诊病人,不患有糖尿病、甲状腺功能亢进症等代谢性疾病;胃癌的肿瘤分级(tumor node metastasis,TNM)根据美国癌症联合委员会以及国际抗癌联盟的标准。正常对照组为吉林大学白求恩第一医院临床体检样本,样本信息与胃癌患者的年龄、性别等因素相匹配。所有样本年龄、丙氨酸氨基转移酶(alanine aminotransferase,ALT)、天门冬氨酸氨基转移酶(aspartate amino transferase,AST)信息见表1。其中按肿瘤TNM分期统计,Ⅰ期有0人,Ⅱ期有10人,Ⅲ期有8人,无法评级(not determined,ND)的有2人。所有采样对象采样前一天避免剧烈运动,避免饮用酒、咖啡、浓茶,清晨空腹使用真空无菌采血管按临床常规方法采血,获得的血清样本1 h内即保存于-80℃冰箱中。血清样本的运输全程使用干冰保证冷链完整。
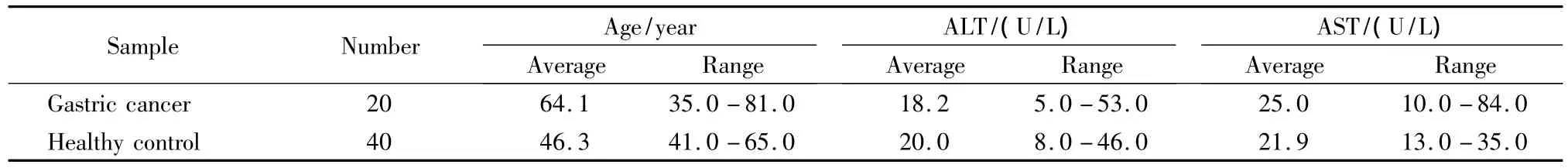
表1 胃癌及对照样本的临床信息Table 1 Clinical information of gastric cancers and healthy controls
1.2 仪器与试剂
采用Agilent 1290液相色谱仪(美国Agilent公司)联合QTRAP 5500质谱(美国AB Sciex公司)系统进行拟靶向分析。正离子模式分析使用的色谱柱是ACQUITY UPLC BEH C8(100 mm×2.1 mm,1.7 μm),负离子模式分析使用的色谱柱是ACQUITY UPLC HSS T3(100 mm ×2.1 mm,1.7 μm)(美国Agilent公司)。
乙腈(色谱纯,美国Merck公司),甲酸、碳酸氢铵(色谱纯)、肉碱(carnitine)-D3、肉碱 C2∶0-D3、肉碱 C10∶0-D3、肉碱 C16∶0-D3、磷脂酰胆碱(phosphatidylcholine,PC)38∶0、溶 血 磷 脂 酰 胆 碱(LysoPC)19∶0、脂肪酸(free fatty acid,FFA)18∶0-D3、色氨酸(tryptophan,Trp)-D5、苯丙氨酸(phenylalanine,Phe)-D5、缬氨酸(valine,Val)-D8、胆酸(cholic acid,CA)-D5、鹅脱氧胆酸(chenodeoxycholic acid,CDCA)-D4,亮氨酸(leucine,Leu)-D3(美国Sigma-Aldrich公司);去离子水经Milli-Q系统(美国Millipore公司)纯化后使用。
1.3 样品预处理
配制同位素内标的乙腈溶液,内标物的质量浓度:肉碱 C10∶0-D3 为0.1 μg/mL;肉碱 C16∶0-D3 为0.15 μg/mL;肉碱-D3 为 0.18 μg/mL;肉碱 C2∶0-D3 为 0.15 μg/mL;LPC-19∶0 为 0.75 μg/mL;PC(38∶0)为 0.625 μg/mL;CA-D5 为 0.25 μg/mL;CDCA-D4 为 1.25 μg/mL;FFA 18∶0-D3 为 2.5 μg/mL;Phe-D5 为 3.61 μg/mL;Trp-D5 为 4.25 μg/mL;VAL-D8 为 3 μg/mL;Leu-D3 为 0.267 μg/mL。
血清样本的预处理:分别吸取待测血清80 μL,加入320 μL 乙腈,涡旋30 s,室温下静置15 min,在4 ℃、14000×g条件下离心10 min,取150 μL 的上清液冻干,最后用 50 μL 水/乙腈(3∶1,v/v)复溶,取上清用于TQ-MRM靶向分析。为保证代谢轮廓分析的数据质量,质量控制(quality control,QC)样本穿插在实验分析过程中。本次实验QC样本来自于本实验室备用的健康人混合血清,预处理方法同上。在待测样本进行LC-MS分析前,QC样本先连续进样5次,开始分析程序后,每隔5个测试样本插入一个QC样本,样本分析结束后连续分析QC样本5次。
1.4 基于UPLC/Triple Q MRM MS的靶向分析
1.4.1 正离子模式的UPLC-MS条件
UPLC条件:流动相A为含甲酸0.1%(v/v)的水,B相为含甲酸0.1%(v/v)的乙腈;梯度洗脱程序:初始为10%B,3 min时上升到40%B,15 min时再上升到100%B并维持5 min,20.1 min时回到10%B,平衡3 min;流速为0.35 mL/min;柱温为50℃,进样量为 5 μL。
MS条件:气帘气压力为0.241 MPa,Gas 1压力为0.276 MPa,Gas 2压力为0.276 MPa,电喷雾离子源(ESI)温度为550℃,喷雾电压为5500 V。监测模式为多反应监测(MRM)。待检测的离子对及相关检测电压碰撞能信息按照文献[13]所述拟靶向策略获取。
1.4.2 负离子模式的UPLC-MS条件
UPLC条件:流动相A为5 mmol/L碳酸氢铵的水溶液,B相为5 mmol/L碳酸氢铵的甲醇溶液;梯度洗脱程序:初始为2%B,3 min时上升到40%B,12 min时再上升到100%B并维持4 min,16.1 min时回到10%B,平衡4 min;流速为0.35 mL/min;柱温为50℃;进样量为5 μL。
MS条件:气帘气压力为0.241 MPa,Gas 1压力为0.276 MPa,Gas 2压力为0.276 MPa,ESI离子源温度为450℃,喷雾电压为-4500 V。待检测的离子对及相关检测电压碰撞能信息按照文献[13]所述拟靶向策略获取。
1.5 数据处理及统计分析
采集到的原始数据使用Analyst中的Quantitation软件(美国AB Sciex公司)进行峰识别和匹配,并导出峰表。导出的数据经过内标校正后,利用SIMCA-P 11.0软件(瑞典Umetrics AB公司)对数据进行多变量分析,数据经过Pareto variance(Par变换)后,将正常组和胃癌组构建主成分分析(PCA)和PCA-偏最小二乘法(PLS-DA)模型,观察实验的稳定性和样品分型,响应置换检验用来判断模型是否存在过拟合现象;使用 SPSS 18.0软件(美国SPSS公司)进行非参数检验,判断离子在正常组和胃癌组之间是否有显著性差异。联合变量重要性因子值(variable importance in the projection,VIP)>1和p-检验(p<0.05)筛选差异离子。
2 结果与讨论
2.1 代谢组数据分析
本研究所采用的拟靶向分析方法的建立过程参考文献[13]。基于拟靶向分析方法对20例胃癌患者及40例健康对照者的血清代谢物进行了分析。典型的正、负离子模式的UPLC/Triple Q MS的MRM提取离子热图如图1所示。正离子模式可以检测373个代谢物离子,负离子模式可以检测247个代谢物离子。

图1 典型的ESI正、负离子模式下UPLC/Triple Q MRM MS的提取离子热图Fig.1 Typical heat maps of extracted ions by UPLC/Triple Q MRM MS in ESI positive-ion mode and ESI negative-ion mode
我们对该方法的稳定性进行了考察。整个实验过程中,QC样本中所测得的代谢物整体表现稳定。在正离子模式下84.6%的离子的峰面积的RSD<30%,占总峰面积的 94.4%;在负离子模式下90.4%的离子的峰面积的RSD<30%,占总峰面积的99.2%。结果表明该方法测定的代谢组数据稳定可靠,完全满足后续代谢组分析的要求。
2.2 差异离子的发现
实验得到的正离子模式下的373个变量经过Par变换后,建立了PCA模型。由得分图(图2a和c)可见所有QC紧密聚集,也说明了该方法的重复性良好。同时,胃癌患者和正常对照组样本的区别明显。为了进一步寻找对分类贡献大的变量,建立了PLS-DA模型。由PLS-DA得分图(图2b和d)可见正常组和胃癌组得到了很好的分离,同时响应置换检验(permutation test)参数:R2截距=0.076,Q2截距=-0.16,说明该模型没有过拟合。
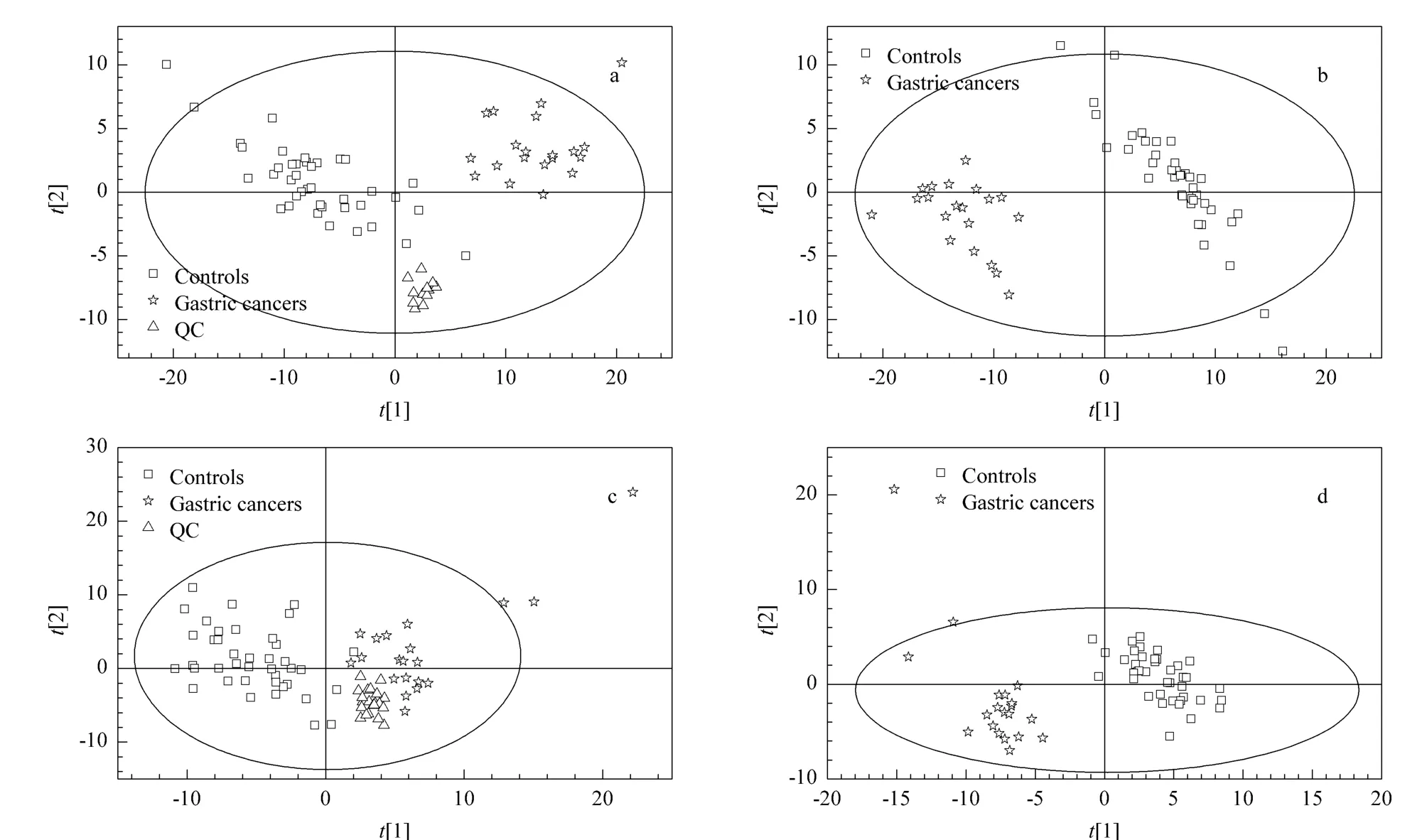
图2 胃癌和健康对照组的PCA和PLS-DA得分图Fig.2 Score plots of PCA and PLS-DA of gastric cancers and healthy controls
PLS-DA模型中149个变量的VIP>1,对于胃癌患者及健康组分类贡献明显。经进一步对变量进行非参数检验,发现有140个变量在两组间存在显著性差异(p<0.05)。其中41个代谢物的结构参考我们前期发表文章中已鉴定的结果[15-17]得到了鉴定。鉴定采用我们报道的基本策略[18],针对未知代谢物,在确定其分子离子峰的基础上,采用高分辨质谱测定其精确相对分子质量,分析其元素组成并参考二级质谱等信息,在网络数据库中查询可能的鉴定结果,最后以标准品对结果进行确证。负离子模式数据也经过上述处理步骤,从247个离子中最终发现87个离子在两组间存在显著差异,其中16个代谢物得到了鉴定。上述经结构鉴定的差异代谢物中,胃癌患者组相对于对照组的变化倍率大于1.5倍的44个差异代谢物见表2。
2.3 差异离子的变化趋势及生理意义
如表2所示,在所筛选出的差异离子中,磷脂类及鞘脂类代谢物(包括溶血磷脂酰乙醇胺(LPE 18∶2、LPE 18∶1)和溶血磷脂酰胆碱(LPC)(LPC 22∶5、LPC 20∶2、LPC 16∶1、LPC 15∶0、LPC 12∶0、LPC 14∶0、LPC 18∶2、LPC 20∶3、LPC 18∶1、LPC 20∶4、LPC 16∶0)和鞘脂类代谢物(SM d18∶1/15∶0、SM d18∶1、SM d18∶1/18∶1))在胃癌患者中的水平明显低于正常人。LPC在许多恶性肿瘤中会显著降低,赵素敏等[19]在液相色谱-质谱用于卵巢肿瘤的磷脂轮廓的代谢组学分析中发现LPC在卵巢癌患者外周血清中显著降低;Dong等[20]发现在肺癌患者血清中LPC的含量也明显降低。一方面,在自分泌运动因子(ATX)或溶血磷脂酶D(lyso-PLD)作用下,LPC会转变成溶血磷脂酸(LPA)[21]。而LPA可以促进细胞增殖,被普遍认为与癌症的发生有关[22]。作为细胞膜的基本组成成分,在肿瘤发生、发展过程中,过快的细胞增殖速度带来的磷脂消耗也是导致血清 磷脂水平下降的主要原因。
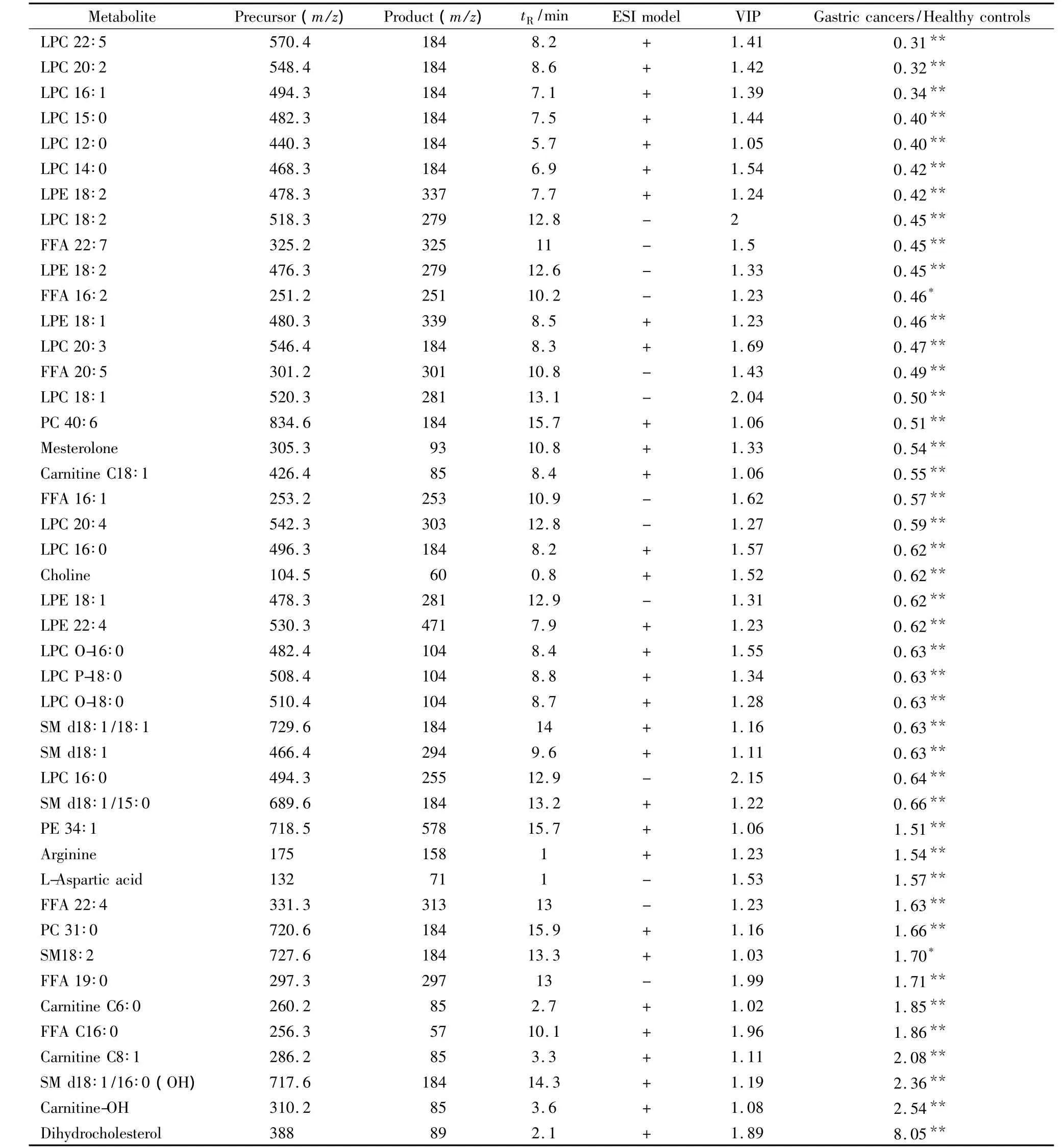
表2 正、负离子模式UPLC-MS发现的胃癌患者组和对照组的已知差异变量Table 2 Known difference variables detected by UPLC-MS in positive and negative ion modes from gastric cancers and healthy controls
此外,研究结果表明,长链肉碱(carnitine C18∶1)在胃癌患者血清中下降,而短链肉碱(carnitine C6∶0、carnitine-OH、carnitine C6∶0)在胃癌患者中水平显著升高。酰基肉碱是脂肪酸β-氧化过程的重要参与者,其血清浓度变化反映了参与脂肪酸β-氧化过程的酶活力的改变[23]。
除了上述两大类主要的代谢差异之外,胃癌患者与健康对照组之间也存在着其他的代谢差异,包括甲氢睾酮(mesterolone)、胆碱(choline)、精氨酸(arginine)、天门冬氨酸(L-aspartic acid)等代谢物都发生了显著的变化,提示了胃癌患者体内在激素代谢、胆碱代谢以及氨基酸代谢等通路上均有代谢紊乱的状况。
2.4 重要差异离子的验证
表2列出的差异代谢物中,升高最显著的代谢物为二氢胆固醇(dihydrocholesterol),它的变化倍率达到了8.05倍,降低最明显的代谢物(LPC 22∶5)的变化倍率也达到了3.23倍,这些代谢物具有成为潜在胃癌代谢标志物的潜力。如图3a和b所示,在发现组中,两组代谢物在胃癌及对照组间均存在显著差异(p<0.001),其中二氢胆固醇能够将正常人及胃癌患者进行很好(100%)的区分(cutoff:0.25),而LPC 22∶5也能够达到97.5%的特异性以及85%的灵敏度(cutoff:0.5)。为了进一步对上述代谢标志物进行验证,我们又以相同的方法测定了另外一组同样来自吉林大学第一医院的胃癌患者(20例)以及对照组(39例)血清中二氢胆固醇以及LPC 22∶5,结果如图3c和d。在验证组中,这两种代谢物仍然在两组患者间存在显著性差异(p<0.001),但是两种潜在代谢标志物对胃癌患者与对照组的区分能力存在明显不同。二氢胆固醇仍然能够将正常人及胃癌患者进行很好(100%)的区分(cutoff:0.25),而LPC 22∶5在确保灵敏度与发现组相同(85%)的情况下,其特异性下降到了70%(cutoff:1.8)。综合以上结果,二氢胆固醇能够作为一种更好的胃癌诊断的潜在代谢标志物。考虑到目前验证组的样本规模较小,后续仍需要扩大样本量进行验证。同时,其他消化道恶性肿瘤以及胃肠道的慢性炎症等疾病对该代谢物的影响以及对诊断结果的干扰也需详细考察。
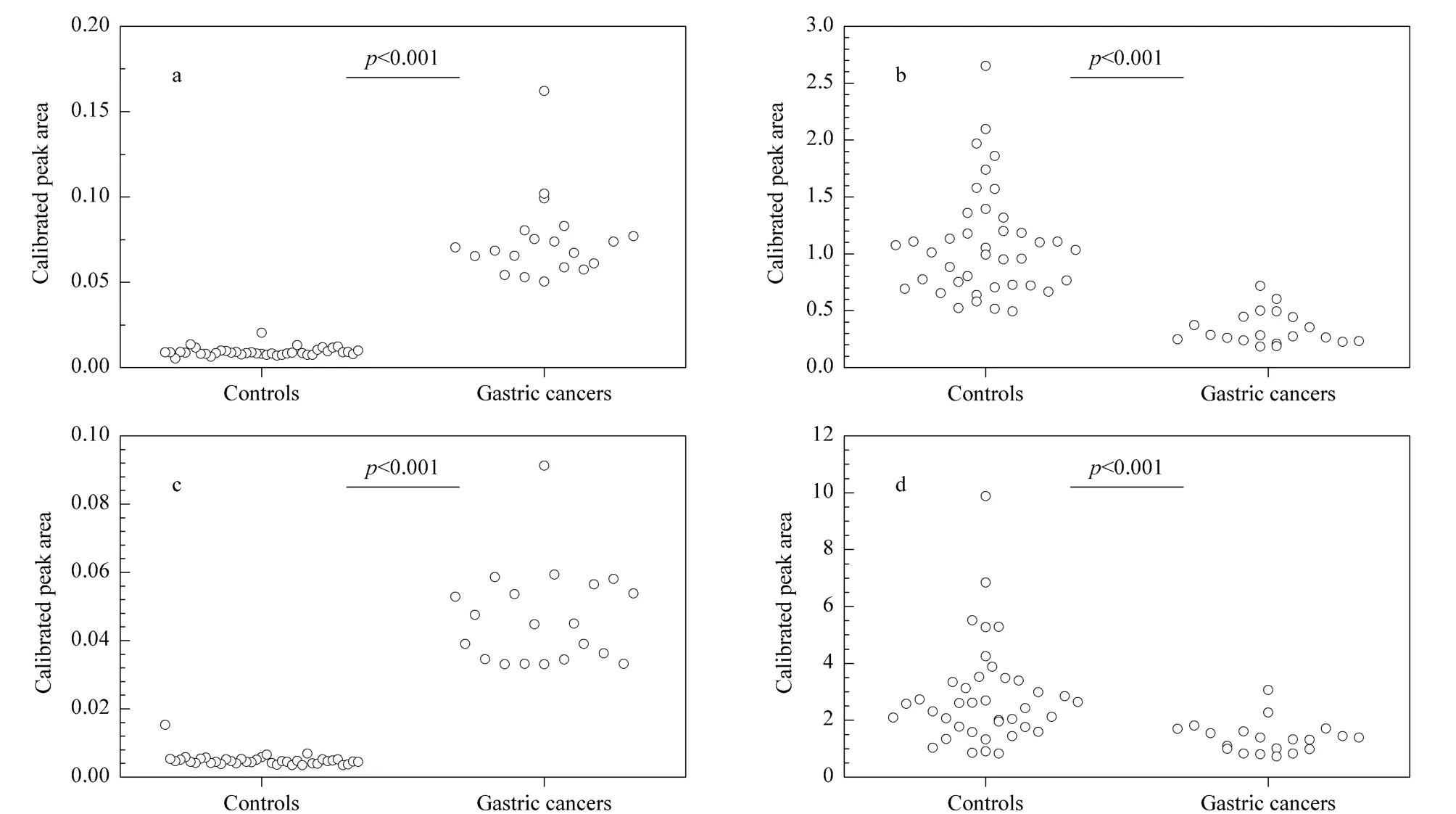
图3 血清中的胃癌潜在代谢标志物Fig.3 Serum potential biomarkers for gastric cancers
3 结论
本研究基于液相色谱-质谱联用的拟靶向代谢组学方法,分析了胃癌患者与正常人血清的代谢组特征以及两组人群间的代谢组差异。发现了胃癌患者包括磷脂、氨基酸、游离脂肪酸、肉碱等代谢物的代谢紊乱状态,其中二氢胆固醇在发现组及验证组中均能够对胃癌与健康对照组进行很好的区分,具有成为潜在胃癌诊断标志物的可能。
[1]Parkin D M,Bray F,Ferlay J,et al.CA Cancer J Clin,2005,55(2):74
[2]Genta R M.Aliment Pharmacol Ther,2004,20(2):42
[3]Marrelli D,Roviello F,De Stefano A,et al.Oncology,1999,57(1):55
[4]Karl-Heinz O,Nelly A,Singh B,et al.Phytochemistry,2003,62(6):971
[5]Nicholson J K,Connelly J,Lindon J C,et al.Nat Rev Drug Discov,2002,1(2):153
[6]Holmes E,Wilson I D,Nicholson J K.Cell,2008,135(5):714
[7]Zhu Y Z,Feng Y N,Jin Z H.Chinese Journal of Chromatography(朱永哲,冯雅男,金正汉,等.色谱),2013,31(9):850
[8]Dai W D,Zhang F X,Jia Z H,et al.Chinese Journal of Chromatography(戴伟东,张凤霞,贾振华,等.色谱),2011,29(11):1049
[9]Yin P,Xu G.Expert Rev Mol Diagn,2013,13(4):339
[10]Rainer L,Zhao X,Cora W,et al.Plos One,2010,5(7):e11519
[11]Anderson L,Hunter C L.Mol Cell Proteomics,2006,5(4):573
[12]Li Y,Ruan Q,Li Y L,et al.J Chromatogr A,2012,1255:228
[13]Chen S L,Kong H W,Lu X,et al.Anal Chem,2013,85(17):8326
[14]Zhao Y,Zhao C,Lu X,et al.J Proteome Res,2013,12(11):5072
[15]Zhou L N,Ding L L,Yin P Y,et al.J Proteome Res,2012,11(11):5433
[16]Yin P Y,Wan D F,Zhao C X,et al.Mol Biosyst,2009,5(8):868
[17]Zhao X,Fritsche J,Wang J,et al.Metabolomics,2010,6(3):362
[18]Chen J,Zhao X,Fritsche J,et al.Anal Chem,2008,80(4):1280
[19]Zhao S M,Wang Y S,Dou A B,et al.Chinese Journal of Chromatography(赵素敏,王宜生,窦阿波,等.色谱),2011,29(9):843
[20]Dong J,Cai X M,Zhao L L,et al.Metabolomics,2010,6(4):478
[21]Zhao Z,Xiao Y,Elson P,et al.J Clin Oncol,2007,25(19):2696
[22]Mills G B,Moolenaar W H.Nat Rev Cancer,2003,3(8):582
[23]Zhou L,Wang Q,Yin P,et al.Anal Bioanal Chem,2012,403(1):203

