海洋石油降解菌的筛选鉴定及其功能基因研究*
吴福顺,王 龙,黄泽瑜,史晓翀,张晓华
(中国海洋大学海洋生命学院 ,山东 青岛266003)
海洋约占地球表面积的3/4,为人类提供了丰富的生产、生活和空间资源。然而,随着人类对海洋资源的不合理开发利用,海洋污染日益严重,对人类的危害也愈发明显。作为当今及未来几十年内世界的主要能源,石油及其制品是主要的海洋污染源之一[1]。石油成分复杂,含量最多的是饱和链烷烃,在石油中的含量为50%~60%[2]。由于饱和烷烃极性官能基团的缺乏和C-H键的高稳定性,导致其具有低亲水性和低化学活性,进入海洋后,会在海面形成稳定油膜,从而对海洋环境和海洋生态系统造成持久的、严重的危害[3-5]。
生物修复由于无污染、花费低和操作简单,是目前治理海洋石油污染的最有前景的方法之一,其中高效石油降解菌在生物修复中发挥着举足轻重的作用[5]。据报道,一共有79个属的细菌可以把石油烃作为唯一的碳源和能源,海洋石油降解菌主要包括食烷菌属(Alcanivorax)、解环菌属(Cycloclasticus)、嗜油菌属(Oleiphilus)、油螺旋菌属(Oleispira)、深海弯菌属(Thalassolituus)、海杆菌属(Marinobacter)、芽孢杆菌属(Bacillus)等[6-8]。编码石油烃降解酶的基因是目前石油降解领域的研究热点之一。细菌中的石油烃降解酶类能够将石油中的惰性烃类催化降解成为小分子物质,并以此作为生长所需的碳源和能源。烷烃羟化酶AlkB[9-10]、细胞色素 P450[11-13],以及最近发现的黄素结合蛋白AlmA[14-16],都能够催化烷烃降解的起始反应,参与烷烃氧化最关键的一环,对石油污染的生物修复至关重要。
2011年渤海湾蓬莱19-3油田溢油事故,共造成超过840km2的海域水质达劣四类(劣于国家海水水质标准中4类海水水质),对渤海生态系统造成了极大危害。本研究以此为契机,利用石油原油为唯一碳源,从实验室海洋微生物菌种库中筛选鉴定具有石油降解活性的细菌,对筛选得到的菌株的石油烃降解酶基因进行研究,为海洋石油污染的生物降解、工业上生物催化剂的开发利用,提供菌株和基因资源。
1 材料与方法
1.1 实验材料和仪器
石油原油:胜利油田原油,由中国海洋大学化学化工学院包木太教授惠赠。
实验菌株:中国海洋大学海洋微生物菌种保藏中心(Marine Culture Collection,Ocean University of China,MCCO)菌株。其中,DSM14913和DSM6294购自德国生物资源中心(Deutsche Sammlung von Mikroorganismen und Zellkulturen),ZJ028分离自中国黄海 冷 水 团 区 域,ZXM008、ZXM018、ZXM112 和ZXM183分离自中国青岛汇泉湾浒苔爆发期近岸表层海水,WH069、WH072和 WH303分离自中国青岛海水,COW02和COW10分离自中国莱阳患病奶牛,Fyb20和H61分离自中国青岛文昌鱼繁殖海域海水,SW100分离自南太平洋环流区表层海水。
2216E培养基:蛋白胨5g,酵母膏1g,FePO40.01g,陈海水1L,调pH=7.6,121℃高压蒸汽灭菌20min(下同)。
高盐LB培养基:NaCl 30g,酵母膏5g,蛋白胨10g,去离子水1L,调pH=7.0,配固体培养基加琼脂粉15g/L。
MMC 培养基:NaCl 24g,MgSO4·7H2O 7g,NH4NO31g,KCl 0.7g,KH2PO42g,Na2HPO43g,去离子水1L,调pH=7.4,高压蒸汽灭菌后补加微量元素混合液5mL/L。
MMC微量元素混合液:CaCl22mg,FeCl3·6H2O 50mg,CuSO40.5mg,MnCl2·4H2O 0.5mg,ZnSO4·7H2O 10mg,去离子水1L。0.22μm滤膜过滤除菌。
无机盐原油培养基:50mL MMC培养基中加入250mg的原油。
主要仪 器:2720Termal Cycler PCR 仪 (Applied Biosystems);台式高速冷冻离心机(美国科峻仪器公司);紫外分光光度计(北京普析通用仪器有限责任公司)。
1.2 实验方法
1.2.1 海洋石油降解菌株的粗筛 菌株石油降解性能的测定采用紫外分光光度法[17]。将实验室菌种库中菌株活化后分别接种至高盐LB液体培养基中,28℃、200r/min过夜培养。将过夜富集培养的种子液于4℃、5 000r/min条件下离心10min,收集沉淀的菌体。用无菌MMC培养基按上述操作洗涤菌体2次后制成菌悬液,按2%(V/V)接种量接种于50mL无机盐原油培养基中,于28℃、200r/min培养2周后,参照徐冯楠等萃取步骤[27],将培养液用石油醚(60~90℃沸程)萃取至50mL比色管中,定容,并摇匀。以石油醚为参比测定OD225。然后根据标准曲线算出吸光度对应的残油浓度。
石油降解率η的计算公式:

式中:C为空白对照萃取液的残油浓度;S为样品萃取液的残油浓度。
1.2.2 高效石油降解菌的鉴定和系统发育树的构建
从筛选出的高效石油降解菌株中提取DNA,用通用引物B8F和B1510R(见表1),按相应反应体系和程序[22]进行16SrRNA基因的PCR扩增,PCR产物送往深圳华大基因科技有限公司测序,获得的序列在EzTaxon-e Database核酸数据库中进行比对鉴定;选择相似性较高的代表性序列,用 MEGA 5.05软件构建Neighborjoining系统进化树,并用自举法(Bootstrap method)1 000次重复测试进化树的可靠性。
1.2.3 石油烃降解酶基因的检测和分析 克隆石油烃降解酶基因(alkB、P450和almA)所用的简并引物见表1。其中烷烃羟化酶基因alkB用2对引物alkBwf/r(目的片段长约550bp)和monf/r(目的片段长约420 bp),细胞色素P450单氧酶P450基因用2对引物P450F/R(目的片段长约800bp)和 CYP153-F1/R2(目的片段长约820bp),黄素结合单氧酶almA基因用一对引物 AlmAdf/r(目的片段长约1 100bp),然后按照相应反应体系和程 序进行 PCR 扩增[16,19-21]。克隆测序具体步骤参照严书林[22],测序结果在NCBI上进行序列比对,并用MEGA5.05做系统发育树。
1.2.4 耐盐芽孢杆菌 WH072石油烃降解酶基因全长的克隆与分析 根据石油降解菌石油降解率初筛结果,选取未被报道的高效石油降解菌株耐盐芽孢杆菌(Bacillus halodurans)WH072利用高效热不对称性交错PCR(hiTAIL-PCR)[23]进行功能基因全长的克隆,引物见表1。将各功能基因翻译的氨基酸序列在NCBI上进行BLAST比对,然后用ClustalW比对其氨基酸序列和与之相似度高的蛋白序列。
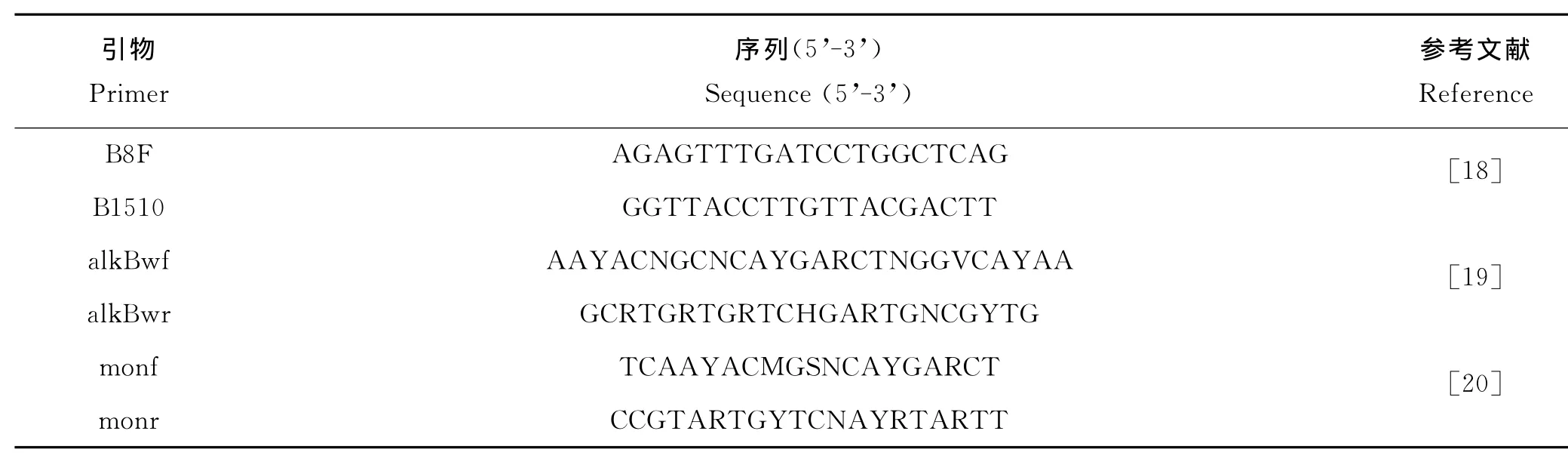
表1 实验所用引物Table 1 The primers used in experiments

续表1
2 实验结果
2.1 海洋石油降解菌株的粗筛
以原油为碳源,采用紫外分光光度法测定实验室菌种库菌株的石油降解率,以筛选出高效石油降解菌。各菌株的石油降解率结果见图1。从图1可以看出,WH072、WH069、WH303、ZXM008、ZXM183这5个菌株的石油降解效果较好,尤其是WH072和WH069,其降解率分别为(36.4±1.0)%和(20.0±0.2)%。
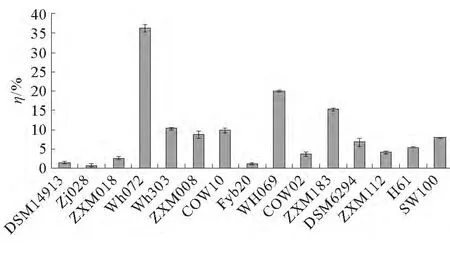
图1 各菌株的石油降解率Fig.1 Oil degradation rate of bacteria
2.2 高效石油降解菌的鉴定和系统发育树的构建
将测序获得的16SrRNA基因序列在EzTaxon public数据库中进行同源性比对,相似度比较结果见表2。结果显示,这5株菌与数据库中标准菌株的相似度都高于97%。WH072基本可确定为耐盐芽孢杆菌,且目前该种未有石油降解功能的报道。

表2 石油降解菌的种属鉴定Table 2 Identification of oil degradation bacteria
石油降解菌株的系统发育进化树见图2。这5株菌大体上可以分为2簇,第一簇包括 WH069、ZXM183和ZXM008,属于γ-变形菌纲(γ-Proteobacteria);WH303和 WH072组成第二簇,属于厚壁菌门(Firmicutes)。已有较多的报道发现芽孢杆菌属、海杆菌属及假单胞菌属的菌株参与石油烃的降解过程[8]。此外,曾在多个石油污染环境中发现Bacillus属的菌株[29-31],说明将该属细菌是一类分布较为广泛的石油降解菌。
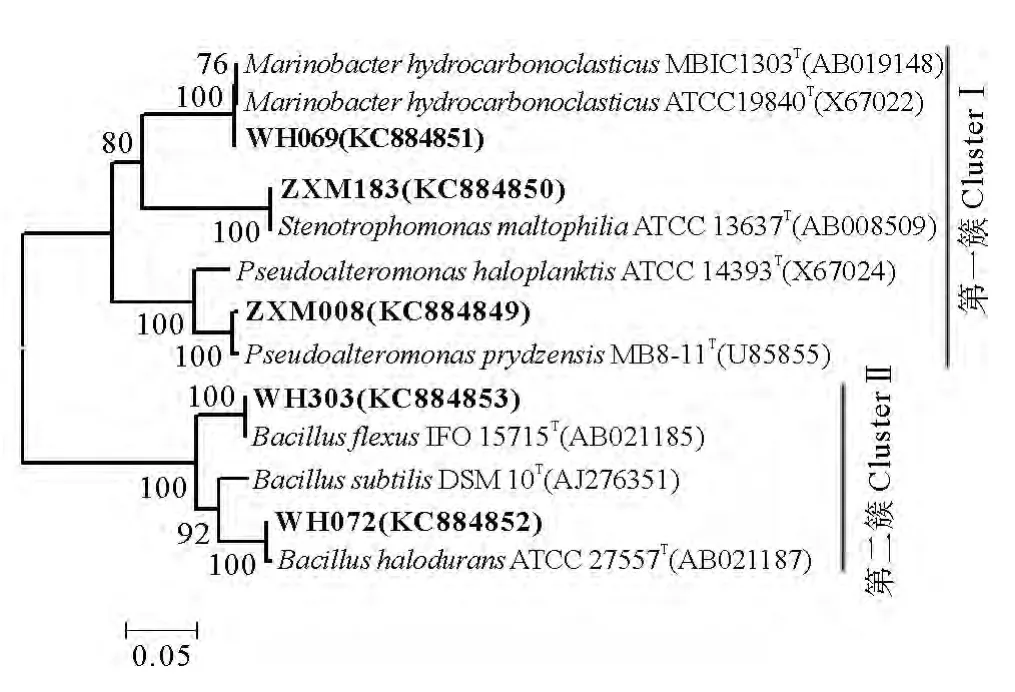
图2 石油降解菌株的16SrRNA系统发育树Fig.2 Phylogenetic tree based on 16SrRNA gene sequence of the oil degradation bacteria
2.3 石油烃降解酶基因的检测和分析
2.3.1 石油降解菌烃降解酶基因的PCR检测 采用简并引物alkBf/r和 monf/r、P450F/R 和 CYP-F1/R2、AlmAdf/r(见表1)分别对石油降解菌的alkB、P450和almA基因进行PCR扩增并进行凝胶电泳。结果显示,对于前2种基因,WH072用引物monf/r获得了长约420bp的预期目的产物,用引物CYP-F1/R2获得了长约820bp的目的产物;WH069用引物alkBf/r获得了长约550bp的目的产物,用引物P450F/R获得了长约800bp的目的产物。利用引物AlmAdf/r从WH072和WH069中都扩增到了长约1 100bp的目的产物。另外3株菌WH303、ZXM183和ZXM008PCR扩增后都没有发现这3种基因的目的条带。将所获得的目的条带切胶回收,克隆测序。
2.3.2 石油烃降解酶基因alkB、P450和almA的系统发育分析 将石油烃降解酶基因alkB、P450和almA的保守序列翻译成氨基酸序列,然后将其在GenBank数据库中进行BLAST比对,选取GenBank中有代表性的AlkB、P450和AlmA蛋白全长序列进行比对,用MEGA 5.05软件中NJ法构建系统发育树研究所获得的功能基因之间的进化关系,设置自展值(Bootstrap)为1 000次来评估进化树的可信度[19]。结果见图3~5。

图3 石油降解菌株烷烃羟化酶AlkB部分氨基酸序列的系统发育树Fig.3 Phylogenetic tree of the partial amino acid sequences of the(putative)alkane hydroxylases(AlkBs)
由图3可知,AlkB序列大致可以分为2支。其中WH069和 M.aquaeolei VT8AlkB(YP_960105)(相似度为94%)、M.hydrocarbonoclasticus AlkB (YP_005430622)(相似度为95%)聚为一簇,它们之间的alkB基因同源性很高,因此基因功能应该比较一致,底物范围接近。对于WH072来说,其AlkB与A.hongdengensis A-11-3AlkB (ZP_11171736)最为接近,两者的相似度仅为89%,与其他菌的AlkB亲缘关系较远,所以推测其alkB基因是一个相对较新的烷烃羟化酶基因,其表达产物的结构、功能和降解底物范围有待深入研究。

图4 石油降解菌株细胞色素P450的系统发育树Fig.4 Phylogenetic tree of the partial amino acid sequences of the(putative)cytochrome P450
从图4可以看出,这2株菌的P450序列可以聚为2簇。M.hydrocarbonoclasticus的细胞色素P450是单氧酶的典型成员,WH069的P450蛋白与其有很高的同源性(高达100%)。WH072与B.licheniformis P450 (ACP39677)、Alcanivorax sp.S14-2P450(ACP39680)的相似度分别为100%和99%,说明不同种属石油降解菌的P450有很高的相似性。

图5 石油降解菌株黄素结合蛋白AlmA的系统发育树Fig.5 Phylogenetic tree of the partial amino acid sequences of the(putative)flavin-binding protein AlmAs
图5 显示,WH069的黄素结合蛋白AlmA蛋白与M. hydrocarbonoclasticus ATCC 49840AlmA(CCG97212、YP_005431635)、M.aquaeolei VT8AlmA(YP_961086)可聚为一簇,序列相似度分别高达100%、99%。WH072的AlmA蛋白和A.borkumensis SK2 AlmA (YP_692002)(92%)、A.jadensis AlmA(ACO56905)(100%)也聚成了一个相对独立的分支,其与食烷菌属的AlmA序列相似度很高,功能可据此推测。
2.4 耐盐芽孢杆菌WH072石油烃降解酶基因全长的克隆与分析
为获得未被报道的高效石油降解菌株耐盐芽孢杆菌WH072的石油烃降解酶基因的全长序列,用hi-TAIL方法进行基因片段两端序列的扩增,然后进行比对拼接,从而得到了其基因的全长序列。WH072石油烃降解酶基因全长序列比对结果见表3。

表3 WH072石油烃降解酶基因全长序列分析Table 3 Analysis of full-length genes encoding hydrocarbon-degrading enzymes of WH072
经过BLASTP比对,WH072的AlkB蛋白与红灯食烷菌(A.hongdengensis)A-11-3的AlkB1和AlkB2的相似度分别为77%和76%,与泊库岛食烷菌(A.borkumensis)SK2AlkB的相似度为74%;P450蛋白与红灯食烷菌(A.hongdengensis)A-11-3P450和Alcanivoraxsp.DG881P450的相似度均为95%,与柴油食烷菌(A.dieselolei)P450有92%的相似度;AlmA蛋白与亚德食烷菌(A.jadensis)的AlmA有99%的相似度,但该菌的AlmA只是部分蛋白片段,不是完整蛋白,与泊库岛食烷菌(A.borkumensis)SK2 AlmA和红灯食烷菌(A.hongdengensis)A-11-3AlmA分别具有90%和86%的相似度。
3 讨论
目前已报道能够降解海洋石油污染物的微生物包括细菌、真菌和藻类,分属于200多个属,其中79个属的细菌可以把石油烃作为唯一的碳源和能源[6-8]。
为海洋石油污染的生物修复提供菌株和基因材料,本研究从实验室菌种库中的15株细菌中筛选出5株对石油原油降解效果较好的菌株,其中石油降解率最高的菌株为耐盐芽孢杆菌WH072,且该菌的石油降解特性为首次报道。芽孢杆菌属是石油污染区域的优势菌群之一,本研究中具有一定石油降解特性的WH303也属于芽孢杆菌属。Said等和Chikere等均从海洋沉积物中分离到了可以降解石油烃的芽孢杆菌[29-30]。王海峰等分离到的一株枯草芽孢杆菌(B.subtilis)对石油的降解率达到34.3%[31],而本研究中WH072对于原油的降解率达到36.4%,该菌株在石油污染生物修复领域具有较好的开发前景。
海杆菌属也是分离最多的石油降解菌属之一,具有较高的出现频率和多样性。除烃海杆菌在1992年首次从地中海炼油厂附近的海水中被分离描述,其后又有14种海杆菌被从不同的环境中分离鉴定[32-33]。WH069与除烃海杆菌(M.hydrocarbonoclasticus MBIC1303T)的相似度是98.0%。该菌株对于石油的降解率为20.0%,因此该菌株对于石油污染防治也具有重要应用价值。
烷烃羟化酶AlkB家族和细胞色素P450家族在石油烃降解酶基因中是被研究的最为广泛和深入的,两者主要负责短中链烷烃的降解,AlmA为最近才报道的专门降解长链烷烃的黄素结合蛋白,其研究刚刚起步。很多参与石油降解过程的高效石油降解菌都具有多种石油烃降解酶基因,如高效烷烃降解菌泊库岛食烷菌(A.borkumensis)SK2含有2个alkB基因和1个P450基因[34]。这种多个烷烃羟化酶基因存在于同一菌株中的现象可能有助于提高菌株的降解效率[35]。鉴于这5株菌具有较高的石油降解率,对这5株菌进行石油烃降解酶基因alkB、P450和almA的检测。本研究在WH069和WH072中都获得了alkB、P450和almA基因片段,说明这2种菌对长、中、短链的烷烃均有降解能力,底物范围较广。这与紫外分光光度法所测得的这两株菌的石油降解率都很高(>20%)相吻合,可能是这两株菌具有较强石油降解能力的原因。综合3种石油烃降解酶基因的系统发育分析及其氨基酸序列分析结果,WH072所含有的alkB基因与其他菌株的相似性相对较低,其结构和功能可能与现有菌株有差异。
值得注意的是,与革兰氏阳性菌之间alkB基因差异较小的情况不同,革兰氏阴性菌的alkB序列变化多样。16SrRNA基因系统发育树的分析能够比较准确的反映分离的不同石油降解菌在分类学上的地位,但是基于alkB基因的氨基酸序列所构建的系统发育树,不同菌株所处的系统发育位置发生了明显改变。这是因为微生物的16SrRNA基因在不同菌种之间相对保守,而alkB基因往往定位于染色体可转移元件和/或质粒上,能够在不同菌株间发生转移。即使基因整合在菌株的染色体上,由于其受到环境因素的影响而不断发生变异,从而产生该基因的多样性[24]。即使在同一水域,表层海水和深层海水中同一菌种不同菌株之间的alkB基因也存在很大差异[19]。对于石油烃降解微生物群落,现在研究比较广泛的是烷烃羟化酶家族基因,而alkB基因是该家族中编码关键酶的基因之一,对菌种所在环境的生态学功能分析具有极其重要的意义。
对比16SrRNA基因系统发育树可以发现,P450基因与细菌菌种有一定的相关性,WH069和WH072均与自身所在属的菌种有高度同源性的P450基因。相比较而言,alkB和almA在种间的分布规律性较低,文献表明alkB和P450基因在食烷菌属(Alcanivorax)的细菌中广泛存在[19]。almA基因的研究刚起步,有关报道较少,是当前研究的热点[16]。耐盐芽孢杆菌WH072的3种石油烃降解酶基因全长氨基酸序列比对结果表明其与食烷菌属的石油降解相关功能蛋白具有最高的相似度,推测其功能基因可能来自于食烷菌属。但是耐盐芽孢杆菌WH072 alkB基因编码的蛋白与最相近的食烷菌属相关蛋白相似度不高(77%),这可能是由基因可转移元件在菌株间水平转移过程中发生了结构的改变导致的[24]。在获得的菌株中,耐盐芽孢杆菌WH072是本研究新发现的具有石油降解特性的菌种,其石油降解率很高,且功能基因也很新颖,具有极大的研究价值。
石油降解菌广泛存在于海洋中,它们中很多生存在营养极为缺乏的“海洋沙漠”中,各种与石油降解相关的功能基因保证其能够适应极端环境,使它们能够利用石油烃类物质作为营养成分。然而,本文中克隆的基因只是其功能基因的很小一部分,蕴含着巨大遗传密码的宝库正等待着开发。
[1] Speight J G.Fossil Energy[M].New York:Springer-Verlag,2013:61-97.
[2] Hall C A S,Ramírez-Pascualli C A.The First Half of the Age of Oil[M].New York:Springer-Verlag,2013:19-27.
[3] Schühle J H K.Anaerobic biodegradation of hydrocarbons including methane[J].The Prokaryotes:Prokaryotic Physiology and Biochemistry,2013,17:605-634.
[4] 方曦,杨文.海洋石油污染研究现状及防治 [J].环境科学与管理,2007,32(9):78-80.
[5] 刘金雷,夏文香,赵亮,等.海洋石油污染及其生物修复 [J].海洋湖沼通报,2006,3:48-53.
[6] Head I M,Jones D M,Roling W F M.Marine microorganisms make a meal of oil[J].Nature Reviews Microbiology,2006,4(3):173-182.
[7] Adebusoye S A,Ilori M O,Amund O O,et al.Microbial degradation of petroleum hydrocarbons in a polluted tropical stream [J].World Journal of Microbiology and Biotechnology,2007,23(8):1149-1159.
[8] Yakimov M M,Timmis K N,Golyshin P N.Obligate oil-degrading marine bacteria[J].Current Opinion in Biotechnology,2007,18(3):257-266.
[9] van Beilen J B,Panke S,Lucchini S,et al.Analysis of Pseudomonas putida alkane degradation gene clusters and flanking insertion sequences:evolution and regulation of the alk-genes[J].Microbiology,2001,147:1621-1630.
[10] Smits T H M,Witholt B,van Beilen J B.Functional characterization of genes involved in alkane oxidation by Pseudomonas aeruginosa [J].Antonie Van Leeuwenhoek,2003,84:193-200.
[11] Grogan G,Roberts G A,Parsons S,et al.P450(camr),a cytochrome P450catalysing the stereospecific 6-endo-hydroxylation of(1R)-(+)-camphor[J].Applied Microbiology and Biotechnology,2002,59:449-454.
[12] Matson R S,Hare R S,Fulco A J.Characteristics of a cytochrome P450-dependent fatty acid omega-2hydroxylase fromBacillus megaterium [J].Biochimica et Biophysica Acta,1977,487:487-494.
[13] van Beilen J B,Funhoff E G,van Loon A,et al.Cytochrome P450alkane hydroxylases of the CYP153family are common in alkane-degrading eubacteria lacking integral membrane alkane hydroxylases [J].Applied and Environmental Microbiology,2006,72:59-65.
[14] Throne-Holst M,Wentzel A,Ellingsen T,et al.Identification of novel genes involved in long-chain n-alkane degradation by Acinetobacter sp.strain DSM 17874 [J].Applied and Environmental Microbiology,2007,73:3327-3332.
[15] Wentzel A,Ellingsen T E,Kotlar H K,et al.Bacterial metabolism of long-chain n-alkanes[J].Applied Microbiology and Biotechnology,2007,76:1209-1221.
[16] Wang W P,Shao Z Z.Diversity of flavin-binding monooxygenase genes(almA)in marine bacteria capable of degradation long-chain alkanes[J].FEMS Microbiology Ecology,2012,80(3):523-533.
[17] 大庆石油管理局油田建设设计研究院.SY/T 0530-93中华人民共和国石油天然气行业标准:油田污水中含油量测定方法——分光光度法[S].北京:华兴科标图书有限公司,1994.
[18] Wang Y,Yu M,Austin B.Oleispiralentasp.nov.,a novel marine bacterium isolated from Yellow sea coastal seawater in Qingdao,China[J].Antonie van Leeuwenhoek,2012,101(4):787-794.
[19] Wang L P,Wang W P,Lai Q L,Shao Z Z.Gene diversity of CYP153Aand AlkB alkane hydroxylases in oil-degrading bacteria isolated from the Atlantic Ocean[J].Environmental Microbiology,2010,12(5):1230-1242.
[20] Wang W,Wang L,Shao Z.Diversity and abundance of oil-degrading bacteria and alkane hydroxylase(alkB)genes in the subtropical seawater of Xiamen Island[J].Microbial Ecology,2010,60(2):429-439.
[21] Wang X B,Chi C Q,Nie Y.Degradation of petroleum hydrocarbons(C6-C40)and crude oil by a novel Dietziastrain[J].Bioresource Technology,2011,102(17):7755-7761.
[22] 严书林.3株新菌的分类鉴定及新型耐热胶琼酶基因的克隆表达[D].青岛:中国海洋大学,2011.
[23] Liu Y G,Chen Y.High-efficiency thermal asymmetric interlaced PCR for amplification of unknown flanking sequences[J].Bio-Techniques,2007,43:649-656.
[24] 于寒颖,杨慧.石油烃降解酶及其基因的结构,功能和表达调控[J].应用与环境生物学报,2012,18(6):1066-1074.
[25] Atlas R M.石油微生物学 [M]//Huang D F,Tan S,Yang W K,等译.北京:石油工业出版社,1991.
[26] van Beilen J B,Li Z,Duetz W A,et al.Diversity of alkane hydroxylase systems in the environment[J].Oil & Gas Science and Technology-Revue De L Institut Francais Du Petrole,2003,58(4):427-440.
[27] 徐冯楠,冯贵颖,马雯,等.高效石油降解菌的筛选及其降解性能研究 [J].生物技术通报,2010,7:221-226.
[28] Ramos J L,Marqués S.Transcriptional control of the Pseudomonas Tol plasmid catabolic operons is achieved through an interplay of host factors and plasmid-encoded regulators[J].Annual Review of Microbiology,1997,51:341-373.
[29] Ben Said O,Goni-Urriza M S,El Bour M,et al.Characterization of aerobic polycyclic aromatic hydrocarbon-degrading bacteria from Bizerte lagoon sediments,Tunisia[J].Journal of Applied Microbiology,2008,104(4):987-997.
[30] Chikere C B,Okpokwasili G C,Ichiakor O.Characterization of hydrocarbon utilizing bacteria in tropical marine sediments[J].African Journal of Biotechnology,2009,8(11):2541-2544.
[31] 王海峰,包木太,韩红,等.一株枯草芽孢杆菌分离鉴定及其降解稠油特性 [J].深圳大学学报:理工版,2009,26(3):221-227.
[32] Gauthier M J,Lafay B,Christen R,et al.Marinobacter hydrocarbonoclasticus gen.nov.,sp.nov.,a new,extremely halotolerant,hydrocarbon-degrading marine bacterium [J].International Journal of Systematic Bacteriology,1992,42(4):568-576.
[33] Green D H,Bowman J P,Smith E A,et al.Marinobacter algicola sp.nov.,isolated from laboratory cultures of paralytic shellfish toxin-producing dinoflagellates[J].International Journal of Systematic and Evolutionary Microbiology,2006,56(3):523-527.
[34] Schneiker S,dos Santos V A P M,Bartels D,et al.Genome sequence of the ubiquitous hydrocarbon-degrading marine bacterium Alcanivorax borkumensis [J].Nature Biotechnology,2006,24(8):997-1004.
[35] 孙敏,沈先荣,侯登勇,等.高效柴油降解菌Acinetobacter sp.W3分离鉴定及降解酶基因扩增分析 [J].生物技术通报,2012,6:159-165.

