基于 DNA 组装的纳米光电技术新进展
武春雷 粟 武
(中国科学院深圳先进技术研究院生物医药与技术研究所 深圳 518055)
基于 DNA 组装的纳米光电技术新进展
武春雷 粟 武
(中国科学院深圳先进技术研究院生物医药与技术研究所 深圳 518055)
新型纳米光电器件需要在纳米尺度实现对光学元件进行精确和可控的空间排列。DNA 具有独特的碱基配对识别机制和双螺旋结构,以及制备简单,形状和尺寸均可程序化设计,并可精确定点修饰等优势,因此已经作为模板组装各类超分子广泛应用于纳米光电和纳米医学等领域。DNA 组装的光子阵列是将一系列的发色官能团根据设计需要精确定位在 DNA 模板上,通过多步荧光共振能量转移,完成光子的传递过程。文章主要综述了近年来基于 DNA 组装的光子阵列取得的新进展,并对以后的发展进行展望。
DNA 纳米技术;光子阵列;荧光共振能量转移;吡咯—咪唑聚酰胺;量子点;光捕获
1 引 言
纳米技术的诞生为光电器件的设计与研制带来了新的发展前景。因此新一代的光电器件需要在纳米水平对光学元件进行精确、可控的空间排列,这是传统方法难以实现的。分子自组装技术通过静电作用、范德华力、疏水作用力、氢键等非共价键作用能够在分子水平实现有序的定位、定向排列,为纳米光电器件的研制提出了新的方法——自下而上(Bottom to Up)的组装方式。在自然界中,DNA 具有高度选择性的碱基配对作用(腺嘌呤 A 与胸腺嘧啶 T 配对,鸟嘌呤 G 与胞嘧啶 C 配对)和双螺旋结构,通过对 DNA 序列的精密设计,DNA 分子可以自组装成二维和三维等可控的纳米结构。而且 DNA 的合成方法简单、成本低,易于在特定位置进行化学修饰,有利于功能分子沿着 DNA 链精确排列。这些特点使 DNA 在功能纳米器件的组装和构建中展现出巨大的优势[1-4]。
在 DNA 组装的众多纳米光电器件中,各种类型的光子阵列的组装与设计备受关注[5-7],它们在光处理和光开关器件[8,9]和生物探针[10]等领域都具有潜在的应用价值。关于 DNA 组装的光子阵列本研究组曾做过相关的综述[5],而本文将主要对近年来 DNA 组装的光子阵列的研究发展前沿做一综述,包括 DNA 折纸术(DNA origami)组装的光子阵列,DNA 结合具有特异识别功能的聚酰胺、纳米材料组装光子阵列,以及 DNA用于光捕获天线的组装。
2 基于 DNA 组装的光子阵列
DNA 组装的光子阵列是以 DNA 分子作为模板,一系列的发色团以一定的模式排列在 DNA链上,这些发色团的吸收和发射光谱有部分重叠,并依次红移形成能量层。当输入发色团被激发后,激发态能量通过荧光共振能量转移(Fluorescence Resonance Energy Transfer,FRET)依次转移到输出发色团,完成光子的传递过程。FRET 有两种模式,即非均相(hetero-FRET)和均相(homo-FRET)。其中,hetero-FRET 指具有能量差的不同的荧光染料间,能量沿着能量差单向传递,并伴随着光谱红移(图1(a))。homo-FRET发生在同一种染料间,由于染料的斯托克斯位移太小,导致吸收与发射光谱重叠较多,能量在染料间无方向的扩散性传递(图1(b))。
2.1 基于 DNA origami 组装的光子阵列
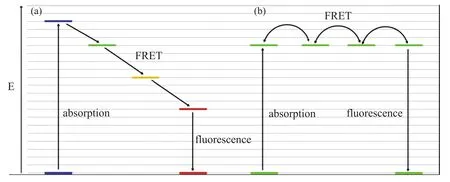
图1 多步 hetero-FRET (a) 和 homo-FRET (b) 示意图(箭头表示能量传递方向)Fig.1. Schematic diagram of multi-step hetero-FRET (a) and homo-FRET (b)(Arrows indicate the direction of energy transfer)
DNA origami 是利用 DNA 分子的特殊结构和碱基互补配对的规则,将 DNA 长链的特定区域进行折叠,并用短链加以固定,构造出预期的结构[11]。利用 DNA origami 得到的结构作为模板组装荧光分子,可以得到多种模式的光子阵列,并能控制能量传递方向。Stein 等[12]基于 DNA origami 自组装技术,利用长链 DNA 模板和修饰荧光基团的短链 DNA 构建了二维的光子阵列。修饰绿色“跳跃”染料 ATTO565 的短链 DNA 的位置选择性地控制能量从蓝色染料 ATTO488 转移到红色染料 ATTO647N 或者近红外染料 Alexa 750(图2(a))。与此类似,Hannestad 等[13]用“Y”型 DNA 链作为构建单元组装成了六边形的 DNA 网状结构,并在特定位置修饰 Pacific Blue(PB)、fluorescein、Cyanine3(Cy3)三种荧光发色基团。如图2(b)所示,没有 YO-PRO-1(YO)嵌入 DNA 双链时,PB 的激发能会转移到输出染料 fluorescein,转移效率达到 57%,随后少部分能量会继续从 fl uorescein 转移到 Cy3,但能量转移效率只有 29%。加入 YO 时,PB 的激发能经 YO 转移到 Cy3,能量转移效率显著增加,达到 50%。可以看出,YO 作为中间染料选择性地控制了能量的传递和输出。
Stein 等[12]和 Hannestad 等[13]利用 DNA origami 的多维纳米结构在中间染料的作用下控制了能量传递方向。Graugnard 等[14]则利用 DNA origami 为多步 FRET 设置了开关功能,动态控制能量传递。Graugnard 等[14]设计了两个开关,即开关 1 和开关 2,图2(c) 所示为开关 2。开关2 包含一条长链的支架 DNA 和三条用于固定的短链 DNA,其中三条短链 DNA 分别修饰输入染料 FAM(蓝色)、中间染料 TAMRA(绿色)和输出染料 Cy5(红色),第四条控制链(深红)在其靠近Cy5 的一端修饰淬灭基团 IBRQ,用于控制能量传递。当五条 DNA 链杂交后,通过多步 FRET能量由 FAM 经 TAMRA 传递到 Cy5,由于 IBRQ的存在,Cy5 发出的荧光被 IBRQ 淬灭,开关 2处于“OFF”的状态。当加入去除链(粉色)时,去除链与修饰 IBRQ 的控制链杂交,控制链脱离DNA 支架,Cy5 荧光恢复,开关 2 处于“ON”的状态。当继续加入恢复链(黄色),恢复链与去除链杂交,控制链被释放,重新与 DNA 支架杂交,Cy5 的荧光又被 IBRQ 淬灭,开关 2 回到“OFF”的状态。
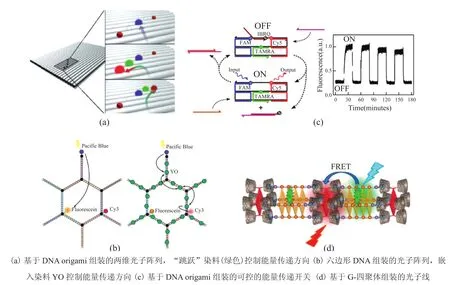
图2 DNA origami 组装的多样化光子阵列Fig.2. Various photonic array assemblies based on DNA origami
开关 1 的设计与开关 2 类似,不同的是TAMRA 代替 IBRQ 修饰在控制链的中间位置,之前修饰 TAMRA 的短链 DNA 不做任何修饰,只起固定作用。开关 1 的“ON”、“OFF”状态与开关 2 刚好相反,两个开关是互补的。原则上,两个开关结合使用能执行任何布尔函数,Cy5 荧光信号的存在或者消失对应逻辑“1”或者“0”。
利用 DNA origami 组装光子阵列多采用较长的 DNA 链,而且发色团一般通过共价作用连接在 DNA 链上,或者共价作用与非共价作用相结合。Zhang 等[15]打破了这一传统的方法,利用只有 6 个碱基长度的 DNA,不需要任何发色团的共价修饰,结合金属离子螯合作用、环糊精与金刚烷主客体作用等超分子组装模式,构建了 DNA 纳米光子线。这 6 个碱基长度的 DNA 包含四个连续的鸟嘌呤 G,两端修饰金刚烷分子。当 K+存在时,四条短链 DNA 基于鸟嘌呤的堆积作用形成四个 G 共面的 G-四聚体。当加入 β-环糊精修饰的卟啉环时,G-四聚体两端的金刚烷基于主客体作用嵌入 β-环糊精,形成数个 G-四聚体相连的 DNA 纳米线。此时 SYBR Green 1 以1∶1 的模式嵌入 G-四聚体,由于 SYBR Green 1 的发射光谱与卟啉的吸收光谱相重叠,因此从SYBR Green 1 到卟啉发生 FRET,能量传递效率达到 75%(图2(d))。
2.2 基于分子特异性识别 DNA 模板组装光子阵列
吡咯—咪唑聚酰胺(Pyrrole-Imidazole Polyamides,Py-Im PA)是一类能够特异性识别双链 DNA 的发夹型芳香杂环化合物,与 DNA 结合具有高度的选择性和亲和力,DNA 识别长度可以达到 6~10 个碱基。吡咯—咪唑聚酰胺中的Py、Im 单体与双链 DNA 的碱基发生氢键作用,产生特异性结合,其中 Py/Im 特异性识别 C/G,Py/Hp(N-甲基-3 羟基-吡咯)特异性识别 A/T,结合模式如图3(a) 所示[16]。
目前吡咯—咪唑聚酰胺已广泛应用于调控基因表达、抑制肿瘤活性和 DNA 检测等领域[17],很少有研究小组将吡咯—咪唑聚酰胺应用到纳米光子线的组装中。作者所在研究组基于吡咯—咪唑聚酰胺对 DNA 的识别作用,首次构建了吡咯—咪唑聚酰胺组装的 DNA 纳米光子线[18]。首先,一条 DNA 链的 5' 端修饰 PB 作为能量供体,与其互补的 DNA 链的 5' 端修饰 Cy3 作为能量受体,YO 作为内嵌染料修饰在聚酰胺末端。聚酰胺特异性识别杂交的双链 DNA 后,PB 的激发能就会经 YO 传递到 Cy3。与只用 YO 嵌入DNA 双链组装的光子线相比,借助聚酰胺组装的 DNA 光子线 FRET 效率大大提高,从 15% 增加到 49%。并且这种组装方法使光子线的长度能够达到 80 个碱基,约为 27 nm,超越了之前各类DNA 纳米组装方法所能实现的能量传递距离。
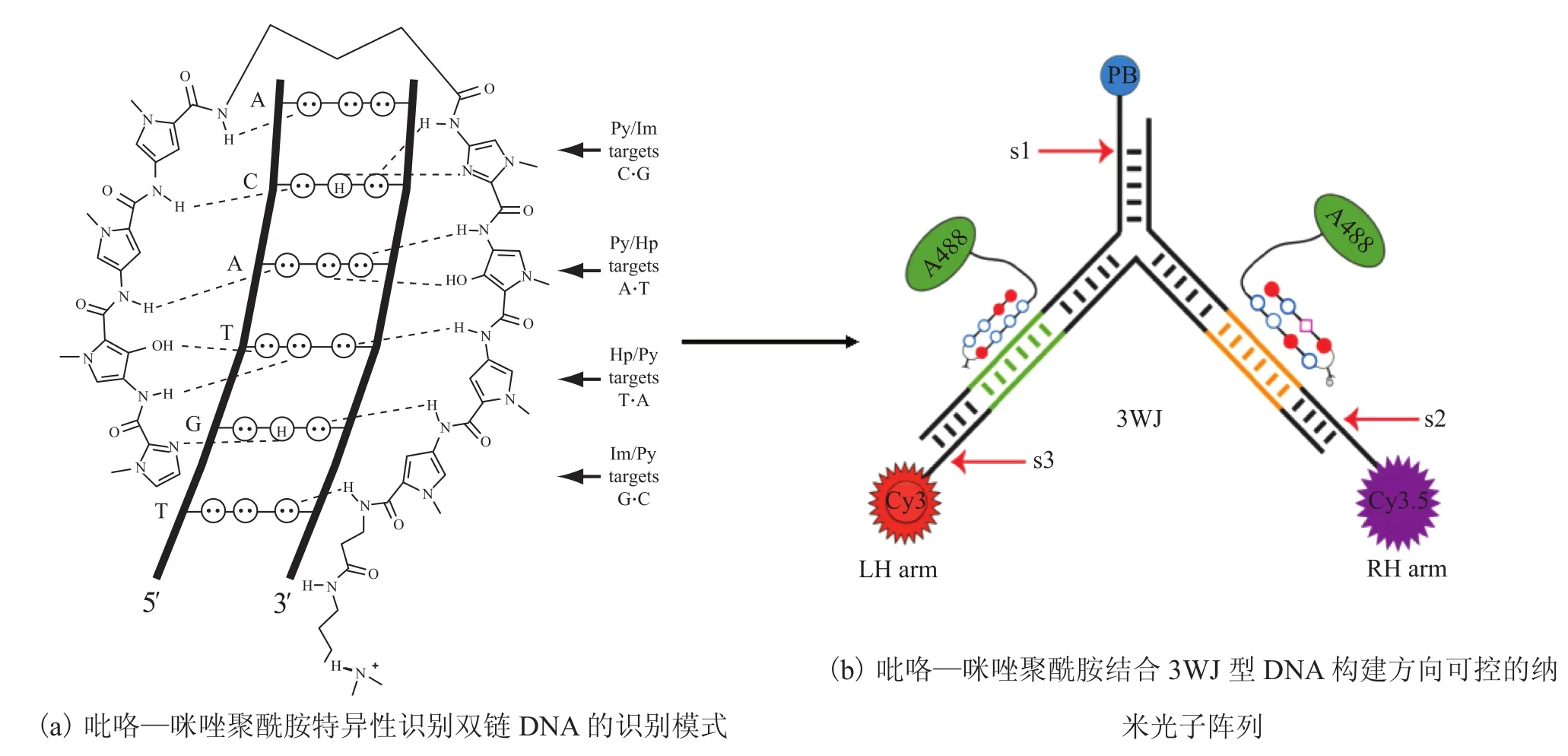
图3 基于特异性识别 DNA 模板组装光子阵列Fig.3. Photonic array assemblies based on the speci fi c recognition of DNA templates
虽然该组装方法的能量传递效率有所提高,但是在这种方式组装的纳米光纤结构中,YO 间的能量转移是双向共振式的 homo-FRET,因而许多能量在均相共振过程中损失,而不能有效定向地转移到 Cy3 受体,导致能量转移效率随着 YO-YO 间转移次数的增加而减少。为了设计更巧妙的、方向可控的基于聚酰胺组装的 DNA纳米光子阵列,本研究组又以“Y”型 DNA (Three-Way Junction,3WJ)为支架发展了控制能量传递方向的非均相的 DNA 纳米光子传递系统[19]。如图3(b) 所示,PB-DNA20、Cy3-DNA30 和 Cy3.5-DNA20 (20、30 表示 DNA 碱基个数,PB、Cy3、Cy3.5 表示修饰在 DNA 5' 端的染料)构成 3WJ,其中 3WJ 的左臂含有 PA1 的识别序列 5' -WGGWCW-3' (W 为 A 或者 T),右臂含有 PA3 的识别序列 5' -WGWCGW-3',PA1、PA3 末端均修饰中间染料 A488。在三维的 3WJ结构中,PA1、PA3 仍然能够有效识别靶序列,并控制 FRET 沿着 3WJ 的左臂从 PB 经 A488 到Cy3,或者沿着其右臂从 PB 经 A488 到 Cy3.5,或者两者同时发生。这是首次将聚酰胺应用到多维的 DNA 纳米光子阵列中,证明了具有高特异识别能力的聚酰胺成为一种新的强有力的工具用于 DNA 光子阵列的组装。
2.3 基于量子点感光的 DNA 光子阵列
DNA 组装的多发色团的纳米光子线,依赖于排列在 DNA 支架上的发色团的光物理性能。这些发色团多是普通的有机染料。然而有机染料的吸收与发射光谱较宽,在发生 FRET 时容易产生光谱干扰,并且光稳定性弱、易被光漂白,这些不足影响发色团间的能量传递效率,阻碍了纳米光子线的进一步发展。因此研究者努力寻找合适的发色团用于构建纳米光子线,将目光转向了光性能优越的荧光纳米材料。量子点(Quantum Dots,QDs)是一种尺寸介于 l~20 nm 的半导体荧光纳米颗粒,具有尺寸依赖的吸收和发射光谱,量子产率高,光稳定性强等独特的光学性能[20]。因此量子点成为 FRET 供体的最佳选择。
Boeneman 等[21]将能够发生连续 FRET 的荧光染料 Cy3、Cy5、Cy5.5、Cy7 依次标记在DNA 链上,当其与互补链杂交后修饰在 QDs 表面时,构成了以 QDs 为中心供体的 DNA 纳米光子线(图4)。这个体系充分利用了 QDs 的光学优势,并且一个量子点表面可以连接 4 条 DNA光子线,形成以 QDs 为中心的树枝状结构。然而该体系的能量传递效率并不高,四步连续的FRET 之后只有 0.1%。在后续的实验中,该研究小组为提高 FRET 效率,从四个方面进行优化:(1)优化 DNA 修饰化学使其更小和更易操作;(2)优化供体—受体染料对,去掉光不敏感的 Cy7,增加 Cy3.5,使供体、受体光谱重叠更充分;(3)根据斯托克斯位移 R0优化 DNA 链上荧光染料之间的距离,依次为 1.5 R0、1.0 R0和 0.5 R0;(4)增加 QDs 表面 DNA 光子线的数目。结果表明,当供体、受体之间的距离为 0.5 R0时,能量传递效率最高。与第一代 QDs-DNA 混合结构相比,第二代结构能量传递效率提高了 2 个数量级,达到 10%[22]。
QDs-DNA 混合光子结构的组装才刚刚开始探索,还有很多的条件需要优化以提高能量传递效率。例如避免染料的光降解或者采用光性能更强更稳定的染料,在结构设计方面将会朝着更复杂的结构发展,如 QDs 与多维 DNA 纳米结构相结合,或者多个 QDs 排列在 DNA 链上,构建多个能量供体少数受体的体系。
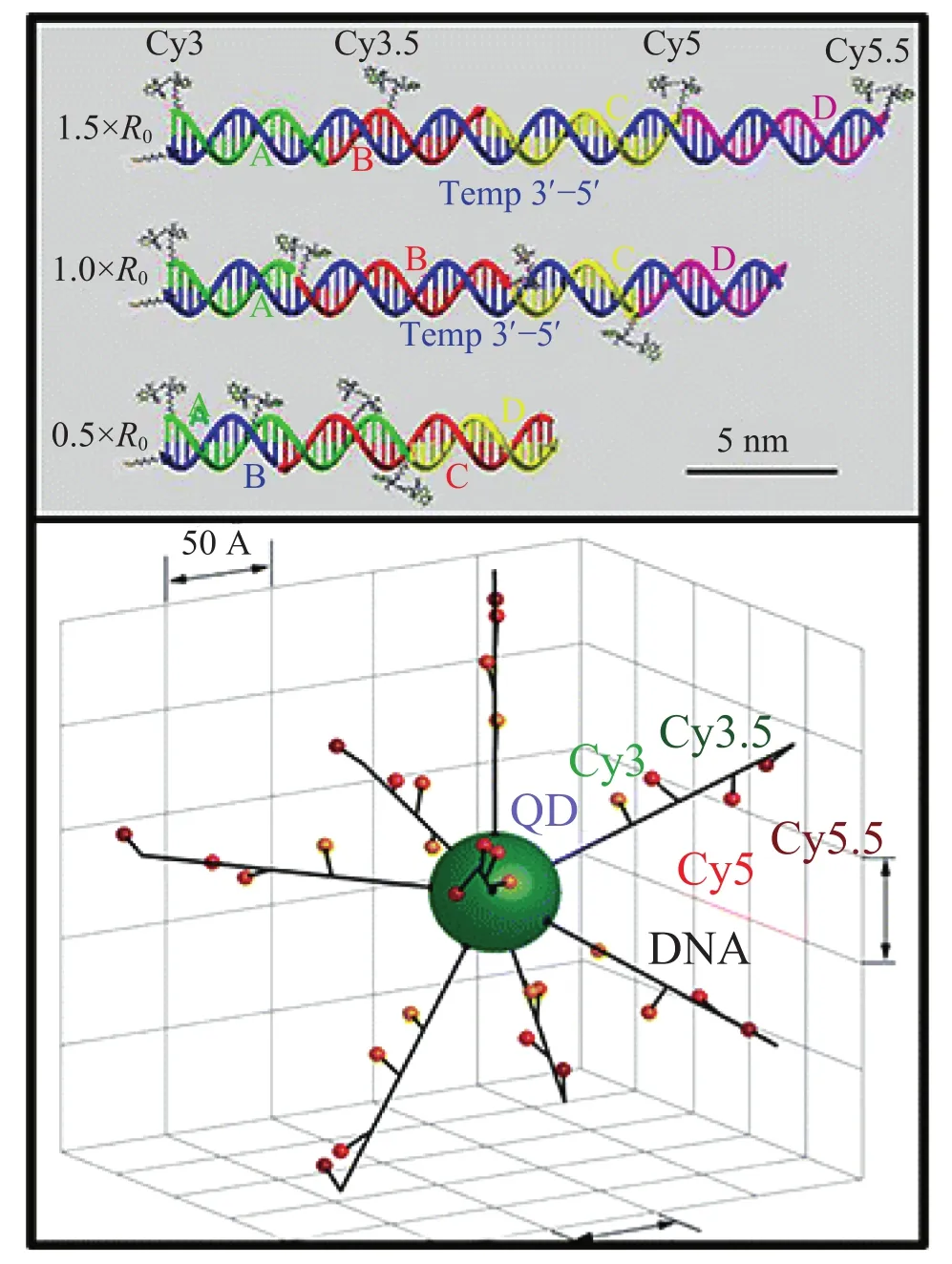
图4 基于 QDs 感光的 DNA 纳米光子线Fig.4. DNA programmed photonic arrays taking QDs as central donor
2.4 DNA 组装的光捕获天线
光捕获天线起源于生物有机体光合作用中的光捕获复合物[23]。光捕获天线通过多步的 homo-FRET 或者 hetero-FRET,将排列在分子支架上的多个光吸收分子的激发能转移到单个或者少数的能量受体,如图5(a)所示。类似于 DNA 组装的光子线,DNA 组装的光捕获天线也可以利用DNA orgami 组装成二维、三维等多种复杂结构。Dut
ta 等[24]以七条螺旋 DNA 束组装成柱形阵列构建了光捕获天线。如图5(b)所示,柱形阵列中周围的六条螺旋 DNA 修饰供体染料乙炔基芘(Py)、Cy3,中心的螺旋 DNA 修饰受体 Alexa Fluor 647 (AF)。其中 Py 作为一级供体,Cy3 作为二级供体,能量从 Py 转移到 Cy3,再从 Cy3转移到 AF。三种染料配比不同,能量传递效率也会受之影响。在比例为 6∶6∶1、6∶3∶1、3∶6∶1、1∶1∶1 时,天线效率 AE(激发供体时受体的发射强度与直接激发受体时受体的发射强度的比值)分别为 85%、43%、47%、16%。这是由于 Py 的减少会使光吸收能力减弱,而 Cy3 的减少会使 Py 到 Cy3 的能量传递效率降低,从而使 AE 有所降低。
Garo 等[25]将多个菲分子(Phenanthrene)代替DNA 碱基交错修饰在两条互补的 DNA 链的中间位置,一个芘(Pyrene)分子连接在其中一条堆积的菲分子的末端(图5(c))。当 320 nm 的光激发菲时,最末端的菲和芘形成激态复合物,发出450 nm 的荧光,荧光强度随着菲分子数目的增加而增强。因此在这个结构中,多个堆积的菲分子作为光吸收天线,菲—芘激态复合物作为能量收集中心。在此基础上,该研究小组对此构建方法进行改进,将芘修饰在另外一条链的中间位置(绿色),让其与中间位置修饰有多个菲分子的两条 DNA 链(红色和蓝色)形成 3WJ 型 DNA 纳米结构(图5(d))[26]。如果以二萘嵌苯或者 Cy 染料代替芘构建 3WJ 结构,菲的激发能就会通过能量转移
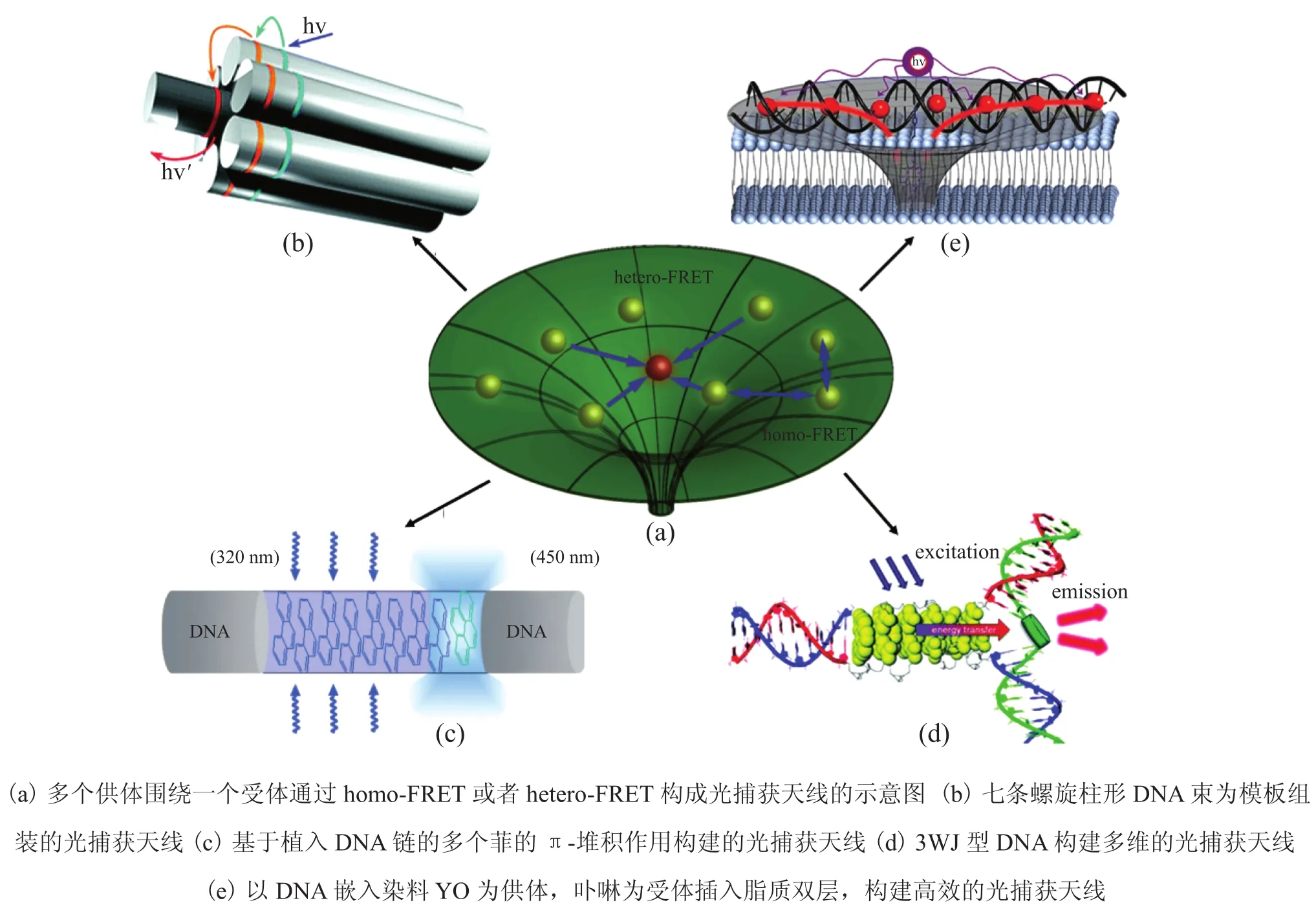
图5 DNA 作为模板组装的光捕获天线Fig.5. Light-harvesting antennas using DNA as the template
被二萘嵌苯淬灭或者使 Cy 发射荧光。如前所述,Dutta 和 Garo 等都是将供体分子共价连接在 DNA 链上,这就限制了光捕获复合物中供体分子的数目(Garo 等[25]设计的结构中能够连接 8 到 10 个菲分子),并且随着供体数目的增加,DNA 合成难度会逐渐增大。Benvin 等以嵌入染料 YO-PRO-1 作为能量供体,Cy3 作为能量受体,利用 3WJ 型、四面体型 DNA 构建多维的光捕获复合物时表明,每两个碱基对就能嵌入一个染料分子[27,28],可见 DNA 嵌入染料作为光捕获剂的巨大优势。因此 Woller 等[29]以嵌入染料 YO-PRO-1 作为供体,卟啉分子作为受体连接在 39 个碱基长的双链 DNA 的中间位置,并嵌入脂质双层,形成水油两相体系,如图5(e)所示。这种光捕获天线,最大供体/受体比能够达到20∶1,光吸收系数达到 260000 M—1·cm—1,与直接激发卟啉相比,光吸收能力增大了 31 倍,天线效应 AE 提高了 12 倍,充分说明这种体系强大的光捕获能力以及高效的能量转移。
3 展 望
DNA 通过严格的碱基互补配对规则,凭借其精确的纳米级结构特征(双螺旋直径 2 nm,碱基对间的距离 0.34 nm),以及可任意添加、剪裁,可定点修饰的独特优势,能够实现光敏元件在纳米水平高密度化、高精确化的组装,为新一代的光电器件超越微型化的技术极限提供了有力保障。随着 DNA 自组装技术的发展,纳米光电器件的组装与设计越来越多样化、复杂化、功能化。通过对 DNA 模板的巧妙设计,纳米光电器件有望在分子水平得以控制和操作,应用于光化学反应。在未来的研究中,光稳定性弱的有机染料有可能会被光性能优越的纳米材料、金属复合物代替,或采用可编辑的蛋白质-DNA 混合结构使光敏分子排列更紧密[2,30],以构建更加复杂的稳定性更好的光电器件,满足纳米光电、太阳能转换、医学诊断等对新材料的更高要求。
[1] Meredith P, Bettinger CJ, Irimia-Vladu M, et al. Electronic and optoelectronic materials and devices inspired by nature [J]. Reports on Progress in Physics, 2013, 76(3): 034501. doi: 10.1088/0034-4885/76/3/034501.
[2] Pan K, Boulais E, Yang L, et al. Structure-based model for light-harvesting properties of nucleic acid nanostructures [J]. Nucleic Acids Research, 2014, 42(4): 2159-2170.
[3] Wang ZG, Song C, Ding BQ. Functional DNA nanostructures for photonic and biomedical applications [J]. Small, 2013, 9(13): 2210-2222.
[4] Lu CH, Cecconello A, Elbaz J, et al. A threestation DNA catenane rotary motor with controlled directionality [J]. Nano Letters, 2013, 13(5): 2303-2308.
[5] Su W, Bonnard V, Burley GA. DNA-templated photonic arrays and assemblies: design principles and future opportunities [J]. Chemistry-A European Journal, 2011, 17(29): 7982-7991.
[6] Albinsson B, Hannestad JK, Boerjesson K. Functionalized DNA nanostructures for light harvesting and charge separation [J]. Coordination Chemistry Reviews, 2012, 256(21-22): 2399-2413.
[7] Teo YN, Kool ET. DNA-multichromophore systems [J]. Chemical Reviews, 2012, 112(7): 4221-4245.
[8] Claussen JC, Hildebrandt N, Susumu K, et al. Complex logic functions implemented with quantum dot bionanophotonic circuits [J]. ACS Applied Materials & Interfaces, 2014, 6(6): 3771-3778.
[9] Nishimura T, Ogura Y, Tanida J. Fluorescence resonance energy transfer-based molecular logic circuit using a DNA scaffold [J]. Applied Physics Letters, 2012, 101(23): 233703. doi: 10.1063/1.4769812.
[10] Kienzler A, Flehr R, Kramer RA, et al. Novel threecolor FRET tool box for advanced protein and DNA analysis [J]. Bioconjugate Chemistry, 2011, 22(9): 1852-1863.
[11] Rothemund PWK. Folding DNA to create nanoscale shapes and patterns [J]. Nature, 2006, 440(7082): 297-302.
[12] Stein IH, Steinhauer C, Tinnefeld P. Singlemolecule four-color FRET visualizes energytransfer paths on DNA origami [J]. Journal of the American Chemical Society, 2011, 133(12): 4193-4195.
[13] Hannestad JK, Gerrard SR, Brown T, et al. Selfassembled DNA-based fluorescence waveguide with selectable output [J]. Small, 2011, 7(22): 3178-3185.
[14] Graugnard E, Kellis DL, Bui H, et al. DNA-controlled excitonic switches [J]. Nano Letters, 2012, 12(4): 2117-2122.
[15] Zhang N, Chu XZ, Fathalla M, et al. Photonic DNA-chromophore nanowire networks: harnessing multiple supramolecular assembly modes [J]. Langmuir, 2013, 29(34): 10796-10806.
[16] Dervan PB, Edelson BS. Recognition of the DNA minor groove by pyrrole-imidazole polyamides [J]. Current Opinion in Structural Biology, 2003, 13(3): 284-299.
[17] Blackledge MS, Melander C. Programmable DNA-binding small molecules [J]. Bioorganic & Medicinal Chemistry, 2013, 21(20): 6101-6114.
[18] Su W, Schuster M, Bagshaw CR, et al. Sitespeci fi c assembly of DNA-based photonic wires by using programmable polyamides [J]. Angewandte Chemie-International Edition, 2011, 50(12): 2712-2715.
[19] Su W, Bagshaw CR, Burley GA. Addressable andunidirectional energy transfer along a DNA threeway junction programmed by pyrrole-imidazole polyamides [J]. Scienti fi c Reports, 2013, 33: 1883. doi: 10.1038/srep01883.
[20] Medintz IL, Uyeda HT, Goldman ER, et al. Quantum dot bioconjugates for imaging, labelling and sensing [J]. Nature Materials, 2005, 4(6): 435-446.
[21] Boeneman K, Prasuhn DE, Blanco-Canosa JB, et al. Self-assembled quantum dot-sensitized multivalent DNA photonic wires [J]. Journal of the American Chemical Society, 2010, 132(51): 18177-18190.
[22] Spillmann CM, Ancona MG, Buckhout-White S, et al. Achieving effective terminal exciton delivery in quantum dot antenna-sensitized multistep DNA photonic wires [J]. ACS Nano, 2013, 7(8): 7101-7118.
[23] McDermott G, Prince SM, Freer AA, et al. Crystal structure of an integral membrane light-harvesting complex from photosynthetic bacteria [J]. Nature, 1995, 374(6522): 517-521.
[24] Dutta PK, Varghese R, Nangreave J, et al. DNA-directed artificial light-harvesting antenna [J]. Journal of the American Chemical Society, 2011, 133(31): 11985-11993.
[25] Garo F, Häner R. A DNA-based light-harvesting antenna [J]. Angewandte Chemie-International Edition, 2012, 51(4): 916-919.
[26] Probst M, Langenegger SM, Häener R. A modular LHC built on the DNA three-way junction [J]. Chemical Communications, 2014, 50(2): 159-161.
[27] Benvin AL, Creeger Y, Fisher GW, et al. Fluorescent DNA nanotags: supramolecular fl uorescent labels based on intercalating dye arrays assembled on nanostructured DNA templates [J]. Journal of the American Chemical Society, 2007, 129(7): 2025-2034.
[28] Oezhalici-Uenal H, Armitage BA. Fluorescent DNA nanotags based on a self-assembled DNA tetrahedron [J]. ACS Nano, 2009, 3(2): 425-433.
[29] Woller JG, Hannestad JK, Albinsson B. Selfassembled nanoscale DNA-porphyrin complex for artificial light harvesting [J]. Journal of the American Chemical Society, 2013, 135(7): 2759-2768.
[30] King NP, Sheffler W, Sawaya MR, et al. Computational design of self-assembling protein nanomaterials with atomic level accuracy [J]. Science, 2012, 336(6085): 1171-1174.
Recent Progress in DNA-Based Assembly Photonic Arrays
WU Chunlei SU Wu
( Institute of Biomedicine and Biotechnology, Shenzhen Institutes of Advanced Technology, Chinese Academy of Sciences, Shenzhen 518055, China )
The development of nano-optoelectronic devices is especially relevant to the control of the spatial arrangement of photoactive materials with nanoscale precision. Because of its base pairing and double-helix structure, as well as easy preparation, programmable control over their shape and size, and precise spatial addressability, DNA has been used as the template to organize a variety of nanoscale elements for nanophotonics and nanomedical research. The DNA-based photonic arrays are multiple fl uorophores programmed sequentially along DNA scaffold to achieve energy transfer through multi-step fl uorescence resonance energy transfer. In this review, the discussion focused on the progrsses of DNA-based assembly photonic arrays in recent years, and insights for future directions were provided.
DNA nanotechnology; photonic arrays; fl uorescence resonance energy transfer; pyrrole-imidazole polyamides; quantum dots; light-harvesting
TN 29
A
2014-06-23
武春雷,研究助理,研究方向为多肽合成及其在功能化纳米材料中的应用;粟武(通讯作者),研究员,研究方向为 DNA 特异性识别聚酰胺的合成及其在纳米光电和生物医学等领域中的应用,E-mail:wu.su@siat.ac.cn。

