小麦SOD基因的电子克隆及生物信息学分析
孙双燕,李东方,张团,张胜利
(河南科技学院,河南新乡453003)
小麦SOD基因的电子克隆及生物信息学分析
孙双燕,李东方,张团,张胜利
(河南科技学院,河南新乡453003)
以NCBI中的EST数据库为主要数据来源,以一系列生物信息学软件为工具,进行了超氧化物歧化酶(SOD)基因的电子克隆及生物信息学分析.结果表明:通过电子克隆方法获得了SOD基因的编码区全长序列,该预测的SOD蛋白质氨基酸序列与数据库中同类基因的蛋白质氨基酸序列具有极高的相似性,并且含有完整的保守结构域;电子克隆到的SOD基因编码的不是分泌蛋白,也不是膜蛋白,而是胞质蛋白.通过某个基因的一个EST序列采用电子克隆的手段进行基因全长的电子拼接是可行的.
SOD基因;电子克隆;小麦
超氧化物歧化酶(superoxide dismutase,SOD)是一种源于生命体的活性物质,能消除生物体在新陈代谢过程中产生的过量的超氧自由基类有害物质.SOD具有防止小麦细胞膜过氧化功能,在小麦干旱、寒冷、贫瘠、病害等逆境环境下含量会增加,对小麦正常生长起保护作用.因此,SOD基因的克隆对于小麦SOD相关的逆境抗性机理研究具有重要的理论意义[1],对小麦分子育种也具有一定的参考价值.研究还发现, SOD能影响小麦花药的育性[2].对于小麦来说,其庞大的基因组(约17 G,是人类基因组的5倍多)、异源多倍体(异源六倍体)使功能基因克隆非常困难.随着高通量二代测序技术的不断成熟与广泛应用,转录组测序、EST测序等累积了海量的DNA序列数据,这为进行基因的电子克隆提供了丰富的DNA数据[3-4].本研究以NCBI中的EST数据库为主要数据来源,采用与前人类似的方法进行了小麦SOD基因的电子克隆研究.
1 材料与方法
以在GenBank中登录号为JQ230561.1的小麦SOD基因EST(长度为472 bp)为种子序列,以NCBI中的EST数据库为主要数据源[5-6],采用类似张德礼等[7]的方法进行了SOD基因的电子克隆,并利用BlastP[8]、ORF finder[9]、clustalX1.8等软件对该基因进行同源比对、开放阅读框预测等,利用ExPASy[10]生物信息学资源网站上的ProtParam、SOPMA、TMHMM、Phobius、SignalP等软件对该基因进行了蛋白质理化性质、二级结构、跨膜螺旋、信号肽等相关生物信息学分析.
2 结果与分析
2.1 SOD基因的cDNA序列电子拼接结果
经电子拼接得到的SOD基因的cDNA长为906 bp,ORF finder预测结果表明,ORF区从213 bp到890 bp,长678 bp,编码225个氨基酸(见图1).以预测的长度为225个氨基酸的蛋白为query,在CDD-42589 PSSMs蛋白保守结构域数据库中进行检索[11].结果表明:该预测的SOD蛋白质氨基酸序列与数据库中同类基因的蛋白质氨基酸序列具有极高的相似性,并且含有完整的保守结构域(见图2).因此,本研究电子拼接出来的SOD基因应该含有其编码区全长,但需要通过RT-PCR、RACE等技术进行进一步试验验证.

图1 电子克隆的SOD基因的开放阅读框Fig.1 Open reading frame of the in silicon cloning gene SOD
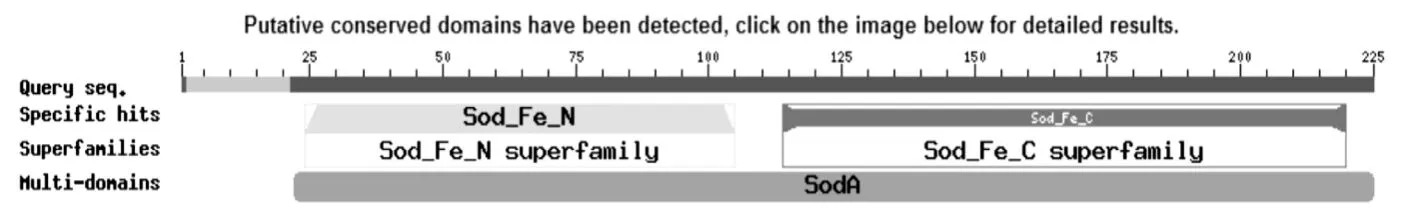
图2 电子克隆的SOD基因的保守结构域检索结果Fig.2 Conserved domain search results of in silicon cloning gene SOD
2.2 不同植物中SOD同源蛋白质序列比较分析
SOD基因蛋白序列与NCBI蛋白质数据库blastp比对结果见表1.
由表1的Blastp比对结果可知,电子克隆的SOD基因与数据库中小麦、大麦、二穗短柄草、高粱、玉米、水稻、番茄、马铃薯、拟南芥等植物中的SOD基因具有较高的同源性,蛋白序列覆盖度在98%以上,序列一致性在75%~98%之间.单子叶植物与双子叶植物中极高的序列相似性的事实表明,SOD基因是植物中普遍存在的一个逆境抗性相关基因,与不同植物对干旱、寒冷、贫瘠、病害等的抗性或适应性密切相关.电子拼接及同源分析研究结果也证明,通过某个基因的一个EST序列采用电子克隆的手段进行基因全长的电子拼接是可行的.二穗短柄草、水稻等基因组较小的模式生物中功能基因组学的研究比较深入,也克隆了大量功能基因,本研究为利用这些近缘物种的基因序列进行基因组庞大的小麦功能基因的克隆提供了重要参考.

表1 SOD基因蛋白序列与NCBI蛋白质数据库blastp比对结果Tab.1 The result of blastp between SOD amino acid and the protein database of NCBI
2.3 SOD预测蛋白的生物信息相关分析
利用ProtParam程序对SOD预测蛋白进行蛋白质理化性质分析.结果表明:该蛋白的相对分子质量为24 601.0 Da,等电点(PI)为7.14;负电荷残基(Asp+Glu)总数与正电荷残基(Arg+Lys)相同,均为24,分子式为C1119H1731N301O319S3;在哺乳动物网状细胞(体外)中的半衰期是30 h;不稳定系数(instability index)为28.59,表明该蛋白比较稳定;平均疏水性(grand average of hydropathicity)为-0.265,表明该蛋白为亲水蛋白.
SOPMA软件对SOD预测蛋白进行的二级结构分析结果表明:该蛋白质含α螺旋(Alpha helix)107个,占47.56%;含β片层(Beta sheet)39个,占17.33%;含转角(turn)16个,占7.11%;含卷曲(coil)63个,占28%.应用TMHMM预测结果表明:本研究电子拼接的SOD蛋白不是跨膜蛋白,没有跨膜螺旋.利用Phobius、SignalP等软件进行信号肽预测,证明该SOD蛋白不含有信号肽.上述预测结果暗示,本研究电子克隆到的SOD基因编码的不是分泌蛋白,也不是膜蛋白,而是胞质蛋白.
3 讨论
植物基因克隆有多种方法,常用的有图位克隆法、同源序列法、突变体筛选法、差异筛选法等[3].不同方法各有其优缺点,在克隆基因时应根据被克隆基因的特点、供体基因组大小、遗传图谱和物理图谱的完备性等选择使用.由于小麦具有巨大的基因组,对其进行基因克隆及验证相当困难,因此,从事小麦分子生物学相关研究的学者们都在积极探索小麦基因克隆的快速有效方法.随着新一代高通量测序技术的不断完善与广泛应用[12],包括EST序列在内的基因表达序列数据日益膨胀[13],这为进行基因的电子克隆提供了十分丰富的数据.前人在电子克隆方面已经做了大量的工作[4,7],为开展此类研究奠定了坚实的基础.
与RACE等其他技术相比,电子克隆具有简便﹑快速﹑节省经费等优点,如张德礼等[7]利用电子克隆结合实验验证的方法成功克隆了人类新基因C17ORF2的cDNA(GenBank登录号:AY074907和BK000260),黄新杰[4]利用此法克隆到了3个小麦条锈病相关抗性基因等.本研究采用与前人类似的方法进行了小麦SOD基因的电子克隆,克隆到了SOD基因的编码区全长序列,该预测的SOD蛋白质氨基酸序列与数据库中同类基因的蛋白质氨基酸序列具有极高的相似性,并且含有完整的保守结构域,这为进一步通过实验手段克隆该基因奠定了良好基础,也为探索小麦逆境抗耐性基因快速克隆方法提供了一定参考.
[1]李娟,杨立新,郑文寅,等.小麦FeSOD基因的克隆与序列分析[J].核农学报,2013,27(8):1111-1117.
[2]李莉,张改生,张龙雨,等.小麦生理型与遗传型雄性不育相关基因锰超氧化物歧化酶(Mn-SOD)及果糖1,6-二磷酸醛缩酶(FBA)的表达分析[J].农业生物技术学报,2012,20(3):225-234.
[3]乔纳森·佩夫斯纳.生物信息学与功能基因组学[M].孙之荣,译.北京:化学工业出版社,2006.
[4]黄新杰.三个小麦抗条锈病相关基因的全长cDNA克隆及生物信息学分析[D].杨凌:西北农林科技大学,2007.
[5]Boguski M S,Lowe T M,Tolstoshev C M.dbEST-database for“expressed sequence tags”[J].Nature Genetics,1993,4(4):332-333.
[6]dbSTS:database of“Sequence Tagged Sites”.[2014-06-10].http://www.ncbi.nlm.nih.gov/dbSTS/.
[7]张德礼,孙晓静,凌伦奖,等.人类SR蛋白超家族新成员-SFRS12(SRrp508)的基因克隆和特性分析[J].遗传学报,2002,29(5):377-383.
[8]Altschul S F,Madden T L,Sch ffer A A,et al.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucleic Acids Research,1997,25:3389-3402.
[9]ORF Finder[DB/OL].[2014-06-10].http://www.ncbi.nlm.nih.gov/gorf/gorf.html.
[10]SIB Bioinformatics Resource Portal[DB/OL].[2014-06-10].http://www.expasy.org/proteomics.
[11]Marchler-Bauer A,Lu S,Anderson J B,et al.CDD:a conserved domain database for the functional annotation of proteins[J]. Nucleic Acids Research,2011,39:225-229.
[12]Wicker T,Taudien S,Houben A,et al.A whole-genome snapshot of 454 sequences exposes the composition of the barley genome and provides evidence for parallel evolution of genome size in wheat and barley[J].Plant Journal,2009,59(5):712-722.
[13]Gunaratne P H,Coarfa C,Soibam B,et al.miRNA data analysis:next-gen sequencing[J].Methods in Molecular Biology,2012,822:273-288.
(责任编辑:邓天福)
Genes in silico cloning and bioinformatics analysis of SOD in common wheat
Sun Shuangyan,Li Dongfang,Zhang Tuan,Zhang Shengli
(Henan Institute of Science and Technology,Xinxiang 453003,China)
Taking EST database in the NCBI as the main data source,with a series of bioinformatics software as tools,the SOD gene in silico cloning and bioinformatics analysis was been done.The results showed that obtained SOD genes coding region of full-length sequence through in silico cloning.The prediction amino acid sequence of SOD protein has a very high sequence similarity with the protein amino acid of similarity genes in the protein database of NCBI,and contains a complete conservative domain structure.The encoding protein of SOD gene obtained in this study by in silico cloning is not secreted proteins,membrane protein,but the cytoplasmic protein.Gene full-length stitching of encoding region is feasible by means of in silico cloning through a relative EST sequence.
superoxide dismutase gene;in silico cloning;wheat
S512
A
1008-7516(2014)04-0007-04
10.3969/j.issn.1008-7516.2014.04.002
2014-06-16
国家级大学生创新训练项目(201310467068)
孙双燕(1990-),女,河南周口人.
张胜利(1976-),男,河北鸡泽人,博士,副教授.主要从事植物分子生物学研究.

