基于ND1基因序列的16种胡椒鲷鱼类分子系统进化关系研究
梁日深,梁志刚,方浩航,谭佳瑜,彭颖暄,薛涛
(1.仲恺农业工程学院生命科学学院,广东广州 510642)
基于ND1基因序列的16种胡椒鲷鱼类分子系统进化关系研究
梁日深1,梁志刚1,方浩航1,谭佳瑜1,彭颖暄1,薛涛1
(1.仲恺农业工程学院生命科学学院,广东广州 510642)
本研究基于线粒体DNA分子标记NADH脱氢酶亚基1基因(ND1)部分序列分析了印度-西太平洋海域的胡椒鲷属及少棘胡椒鲷属共16种胡椒鲷鱼类的系统进化关系。采用最大简约法及最大似然法构建了系统进化树。进化树上,16种胡椒鲷鱼类共形成两个形态特征截然不同的类群,其中类群一的种类体表具有各种鲜艳的颜色与多变的斑纹,主要分布在西太平洋地区;类群二的种类颜色灰暗单一,少数体表具有暗色斑点与条纹,大多数分布在印度洋地区。此外,进化树上,少棘胡椒鲷属的种类都位于胡椒鲷属的类群一内部,与斑胡椒鲷,暗点胡椒鲷形成的姐妹种有较近的亲缘关系。基于遗传距离的数据,少棘胡椒鲷属与胡椒鲷属间的平均遗传距离小于胡椒鲷属内部种间的遗传距离,显示出少棘胡椒鲷属与胡椒鲷属有非常近的亲缘关系,支持少棘胡椒鲷归类于胡椒鲷属的观点。
系统发育;仿石鲈科;胡椒鲷属;少棘胡椒鲷属;ND1
1 引言
胡椒鲷鱼类Sweetlips,包括胡椒鲷属Plectorhinchus以及少棘胡椒鲷属Diagramma的种类,隶属鲈形目Perciformes鲈亚目Percoidei仿石鲈科Haemulidae胡椒鲷亚科Plectorhynchinae,广泛分布于印度洋及太平洋西部温带,亚热带及热带沿海及岛礁海域,是重要的海洋经济食用及观赏性鱼类。胡椒鲷在世界上约有30余种,中国大约记载8~12种,主要分布在南海[1—4]。
形态上,胡椒鲷鱼类近缘物种十分相似,许多种类都具有近似的体色和斑纹,且体色与斑纹会随着个体的逐步发育发生巨大的变化,使幼鱼与成鱼的形态截然不同。如一些种类幼鱼具有横跨体表的水平条纹,而发育成鱼后条纹逐渐破裂成无数小圆点(黄点胡椒鲷Plectorhinchus flavomaculatus),另有种类幼鱼体表具不规则斑纹,而成鱼后却变为明显的水平纵带(东方胡椒鲷P.orientalis)。此外,许多种类的成鱼与其自身幼鱼形态相去甚远的同时,却与其他种类的幼鱼,半成鱼形态非常相似,如东方胡椒鲷P.orientalis成鱼与斜纹胡椒鲷P.lineatus、四带胡椒鲷P. diagrammus幼鱼,体表均为数条黑色水平纵带,各鳍为黄色,极易混淆。胡椒鲷属的形态差异多样性给传统基于外部性状的分类鉴定带来很大的困扰,许多种类的分类与命名相互混淆[1—3]。在分子水平上,国内外学者对胡椒鲷鱼类的系统进化虽有报道,但涉及胡椒鲷种类的数量都很少。朱世华等[5]利用细胞色素b基因部分序列对5种仿石鲈鱼类进行了系统进化研究,结果显示胡椒鲷P.pictus与胡椒鲷鱼类关系较远,支持胡椒鲷从胡椒鲷属Plectorhinchus划分出去的观点,但其研究只涉及两种胡椒鲷;任岗等[6]利用16S r RNA序列分析了11种仿石鲈科鱼类系统进化关系,其研究包含的4种胡椒鲷中,胡椒鲷位于胡椒鲷属的内部。Sanciangco等[7]与Tavera等[8]利用多个分子标记对北美海域的仿石鲈科鱼类的进行较为全面的系统分类研究,但他们重点放在石鲈亚科Haemulinae的种类,对胡椒鲷亚科的分析内容很少。目前关于胡椒鲷鱼类分子研究基本上都是围绕整个仿石鲈科的种类进行,涉及胡椒鲷属种类很少,且多数研究重点放在石鲈亚科,缺乏对胡椒鲷亚科种类的深入探讨,对于胡椒鲷属鱼类较为全面的分子系统进化研究至今还没见报道。
ND1是NADH脱氢酶的第一个亚基,它可以从种的水平来推断某个属的系统发育关系[9],是国内外鱼类系统分类研究广泛应用的一个分子标记,应用于鱼类不同分类阶元的系统发育研究,解决许多系统分类及进化关系问题[10—12]。本实验基于ND1基因片段序列,在分子水平上对胡椒鲷属及少棘胡椒鲷属共16种鱼类进行系统发育分析,初步揭示胡椒鲷鱼类的系统进化关系,为仿石鲈科鱼类的分类研究提供有效的分子信息。
2 材料与方法
2.1 材料来源
胡椒鲷实验样品于2009年11月—2011年6月分别采集于广东、海南等沿海城市水产市场。此外,部分样品来自于西印度洋非洲沿海的国家,以及大洋洲岛国新喀里多尼亚。主要来源于以下单位:西印度洋非洲沿海的样品主要由南非水生物多样性研究院(South African Institute for Aquatic Biodiversity,SAIAB)标本管理员Bernard Makenzie教授提供以及美国堪萨斯大学Andrew Bentley教授提供;大洋洲的样品由岛国新喀里多尼亚努美阿潟湖水族馆(Aquarium des Lagons)Jean-Lou Justine教授提供。最后共得到胡椒鲷属鱼类14种以及少棘胡椒鲷属2种。实验材料均取肌肉或尾鳍,于95%的乙醇固定,用于实验室基因组DNA提取。系统进化选取同为胡椒鲷亚科的矶鲈属中的三线矶鲈Parapristipoma trilineatum作为外群的进行分析。标本种类与来源见表1。

表1 实验材料的种类和采集地Tab.1 Species and localities of materials

续表1
2.2 总基因组DNA的提取
取0.1 g肌肉样品,加入500μl抽提液(0.3 mol/L NaCl、10 mmol/L EDTA,10 mmol/L Tris-Cl,p H 8.0)以及20 u L 10 mg/m L蛋白酶K,55℃消化3 h左右。消化产物经饱和酚-氯仿抽提,2倍体积无水乙醇沉淀,70%乙醇洗涤,最后溶解于50 uL无菌水中,电泳检测,-20℃保存备用。
2.3 PCR扩增和测序
用于扩增ND1基因片段序列引物为:ND1+5’-GTTGCMCAAACCATCTCHTAYGAA G-3’和ND1-5’-GGGGTATGGGCCCGATAGC-3’[13]。PCR反应体系总体积为50μL,其中包括10×buffer 5μL,d NTPs(各2.5 mmol/L)2μL,上下游引物各1μL,Ex Taq酶(1 U/μL)2μ。PCR反应条件为:94℃预变性5 min,94℃变性30 s,55℃退火1 min,72℃延伸1.5 min,35个循环,最后72℃延伸10 min。PCR产物用1.5%琼脂糖凝胶电泳分离,切取特异性片段纯化回收,纯化产物送到上海英骏生物技术有限公司在ABI3730自动测序仪上进行双向测序。
2.4 数据分析
测序所得序列,利用Clustal W[14]进行排序,生成序列矩阵去除两端冗余,保留同源序列进行系统发育分析。利用MEGA version 4.0[15]软件对序列的碱基组成、碱基差异百分比、变异位点、简约信息位点及转换/颠换值进行统计,分子系统进化树利用最大简约法和最大似然法进行构建。系统树分支的置信度采用重复抽样分析(Bootstrap analysis)方法,重复抽样的次数为1 000次。
3 结果分析
3.1 序列分析
测序得到16种胡椒鲷鱼类的ND1部分序列长度在698~712 bp之间,排列比对去除两端冗余后共得到一致序列为673 bp。利用Mega4.0软件分析可知,去除外群,16种胡椒鲷鱼类共有保守位点492个,约占73.2%;变异位点181个,约占26.9%;简约性信息位点141个,约21.0%。T、C、A、G碱基平均含量分别为24.9%、31.0%、27.3%和16.8%,显示G碱基相对缺乏,其中A+T的含量(52.2%)高于G+C含量(47.8%)。密码子第1位4种碱基含量相差不大,其中C-1含量比其他3种碱基含量稍微偏高,占28.3%,密码子第2位中,T-2含量高于其他3种,占35.0%,密码子第3位中A-3,C-3含量最高,分别为34.1%及34.0%,而G-3含量最少,仅有12.3%,表现出明显的反G偏倚(见图1)这一模式与其他哺乳动物相似[16]。
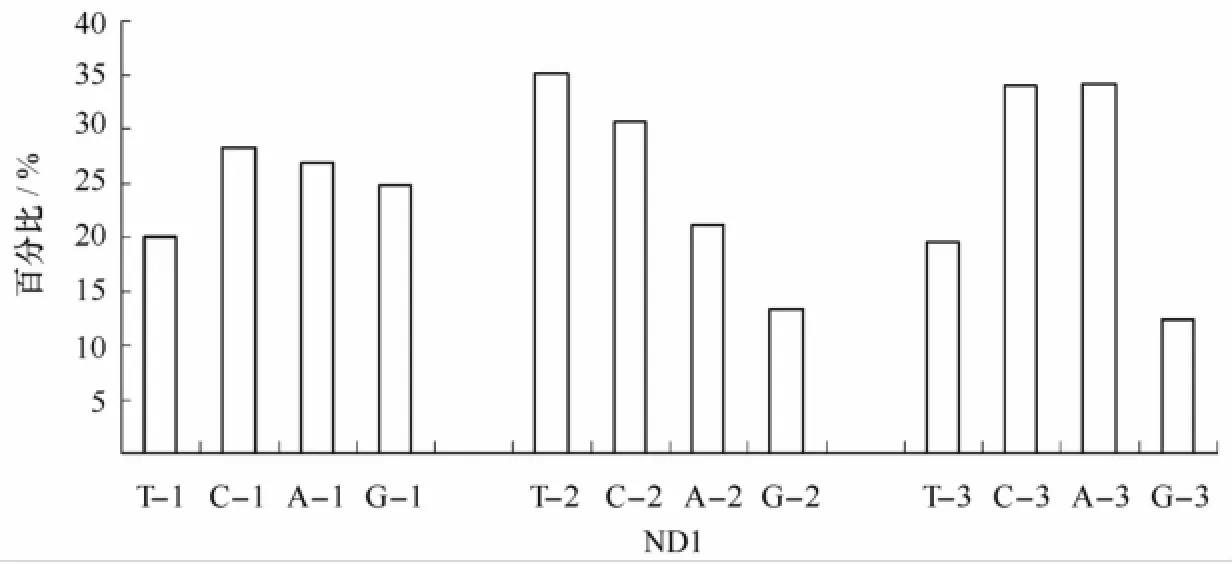
图1 NADH脱氢酶亚基1(ND1)第一、二、三位密码子碱基组成Fig.1 Base composition of the 1st,2nd,3rd codons of the dehydrogenase subunitⅠgene
序列中转换与颠换之比值为2.61(Kimura 2-parameter模型),转换明显大于颠换,显示这些位点没有突变饱和。转换和颠换主要发生在密码子的第3位,其中转换数为84.3%,而颠换为80%(表3)。这可能与密码子第3位发生的碱基替换不易造成氨基酸突变,第1,第2位点的替换容易引起氨基酸突变有关。碱基替换中转换以T-C转换形式为主,T-C转换多于A-G,颠换中A-T,A-C的颠换多于G-T,G-C(表2)。

表2 ND1序列碱基转换和颠换数Tab.2 Numbers of the transitions and tranversions on the ND1 sequences
基于Kimura 2-parameter模型计算出16种胡椒鲷遗传距离如表3所示,在内类群中,16种鱼类遗传距离范围在0.027~0.177之间。其中,两种少棘胡椒鲷遗传距离最小,序列中仅有几个碱基的差异。其次是查氏胡椒鲷与红唇胡椒鲷,为0.034;而东方胡椒鲷与肖氏胡椒鲷遗传距离最大,为0.177,其次是四带胡椒鲷与肖氏胡椒鲷,为0.173。同时,我们统计了少棘胡椒鲷属与胡椒鲷属的种类遗传距离范围在0.083~0.160之间,平均遗传距离为0.112,少于胡椒鲷属内部种间的遗传距离范围0.034~0.177,平均遗传距离0.123。

表3 基于Kimura 2-parameter模型计算16种胡椒鲷ND1序列遗传距离(下三角)以及序列间的转换/颠换数(上三角)Tab.3 Pairwise distances(below diagonal)and number of transitions/transversions(above diagonal)of 16 sweetlips for ND1 sequences based on Kimura 2-parameter model
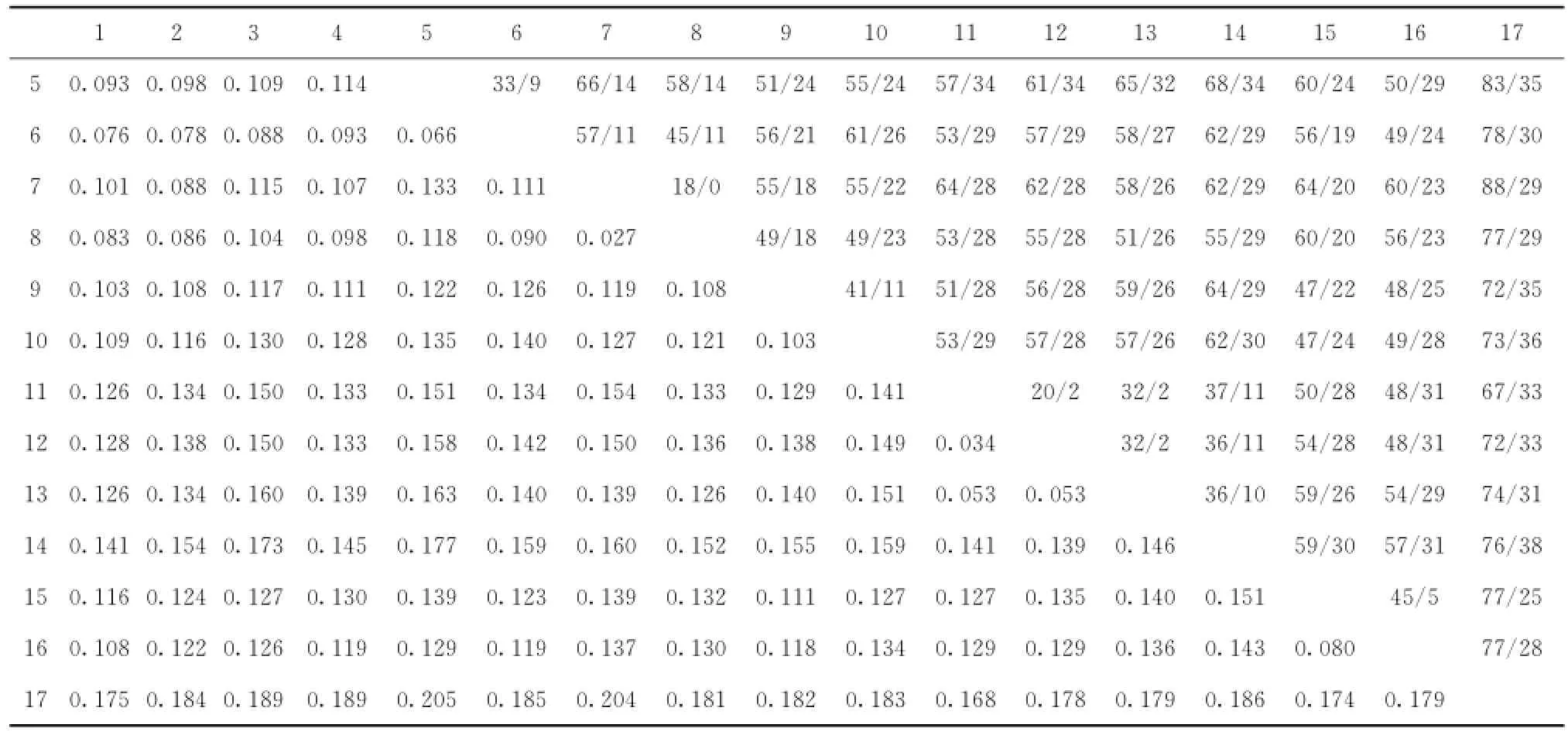
续表3
3.2 分子系统进化树的构建
基于所得的ND1部分序列,以三线矶鲈P.trilineatum作为外类群进行系统进化分析。利用最大简约法(MP)和最大似然法(ML)构建的分子系统进化树。最后构建得到MP和ML树拓扑结构几乎一致,仅在一些节点的支持率不同。其中最大简约树树长(Tree Length,TL)为507,一致性指数(Consistency Index,CI))为0.484 4,保留指数(Retention Index,RI)为0.524 7。最大似然树似然率ln L=-3 425.46。这里把MP树与ML树合并(见图2),树上各分支的数字用1 000次Bootstrap表示统计分析后对该支的支持率(MP/ML)。
由图可知,研究的16种胡椒鲷鱼类中,在系统进化树上大致可分成两个形态截然不同的类群。类群一包括斑胡椒鲷,条纹胡椒鲷等10种胡椒鲷,该类群的种类体表均有各种鲜艳的色彩和斑纹。类群二包括花尾胡椒鲷,黑胡椒鲷等6个种类,与第一类群不同的是,该类群的种类体色均暗淡单一,无鲜艳色彩与斑纹。同时,在进化树上,许多胡椒鲷都两两聚成姐妹种,而聚成姐妹种的胡椒鲷在外部形态上均有高度相似的特征。如:东方胡椒鲷与条纹胡椒鲷,体侧均有明显横跨体表的黑带,斑胡椒鲷与暗点胡椒鲷,体表分布着密集的小黑点。此外,我们发现少棘胡椒鲷属的两个种类都位于在类群一的胡椒鲷种类中,并与斑胡椒鲷,暗点胡椒鲷聚成的分支有比较近的亲缘关系。系统进化树大部分节点都拥有较高的支持率。
4 讨论
4.1 胡椒鲷属鱼类的系统进化关系
本研究共采集到14种胡椒鲷和2种少棘胡椒鲷,其中7种主要采自南海,另外9种来自西印度洋非洲海域,几乎囊括我国记录的胡椒鲷种类[1—4],部分种类甚至在我国并无分布[1—2,17—18],具有一定的代表性。基于ND1基因部分序列构建的系统发育树中,16种鱼类共形成两个形态特征截然不同的的类群,类群Ⅰ包括斑胡椒鲷等7种胡椒鲷以及2种少棘胡椒鲷,类群Ⅱ包括花尾胡椒鲷等5种胡椒鲷。形态上,类群Ⅰ的胡椒鲷种类体表均具有各种鲜艳的色彩和斑纹,色彩斑纹随着个体发育会产生变化。类群Ⅱ的胡椒鲷种类体色暗淡单一,无鲜艳色彩与斑纹,或仅有少量暗色斑纹,幼鱼与成鱼形态无明显差异。在地理分布上,类群Ⅰ的胡椒鲷种类主要分布在西太平洋地区,类群Ⅱ的种类大多数仅分布于印度洋,少数在西太平洋也有分布[17—18]。
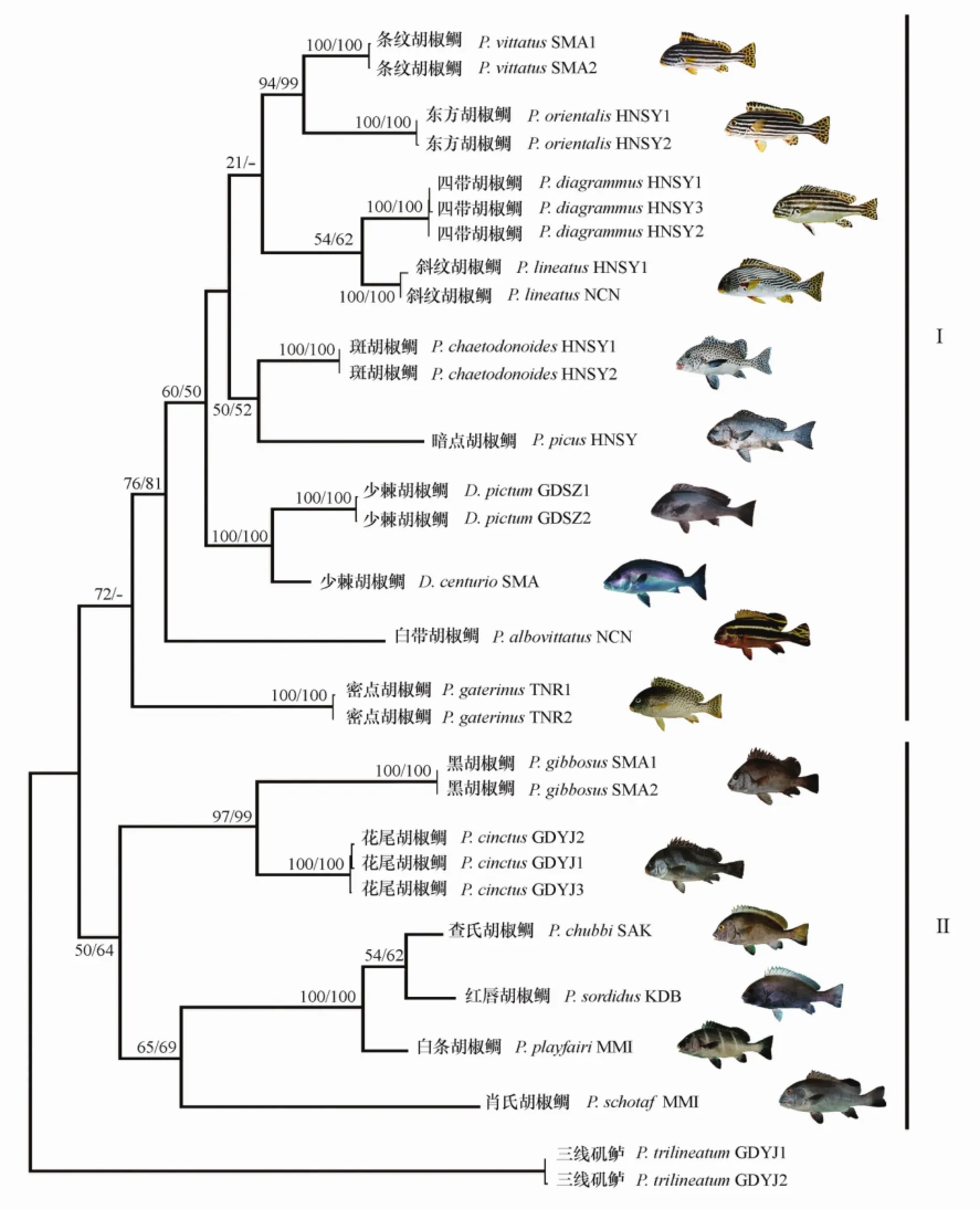
图2 16种胡椒鲷鱼类ND1基因部分序列利用最大简约法与最大似然法构建的分子系统树Fig.2 Molecular phylogenetic tree of 16 sweetlips based on partial ND1 gene sequences constructed by maximum parsimony and maximum likelihood method
形态学对于类群Ⅰ中胡椒鲷的分类存在较大争议,皆因该类群近缘种类形态相似,体色与斑纹还会随着个体发育产生巨大变化,如前言所述。这种形态差异多样性给传统形态分类鉴定带来很大的困扰,许多物种分类不清。本研究构建的聚类结果对上述胡椒鲷鱼类的分类关系有较为清晰的展现。进化树上斑胡椒鲷与暗点胡椒鲷,四带胡椒鲷与斜纹胡椒鲷,条纹胡椒鲷与东方胡椒鲷,以较高的支持率两两聚成姐妹分支。形态上,它们均拥有相似的形态特征,并且在个体发育过程中两两还具有几乎一致的变化过程[2,17—18]。如斑胡椒鲷与暗点胡椒鲷,两者在幼鱼期体表具有棕色或黑色的斑纹,而成鱼体表的斑纹逐渐断裂成无数的小黑点。四带胡椒鲷与斜纹胡椒鲷在幼鱼期体表均有数条黑色水平纵带,成鱼后,四带胡椒鲷体表保留为四条水平条带,而斜纹胡椒鲷发育为许多斜行波状条纹。对于条纹胡椒鲷与东方胡椒鲷,这两种胡椒鲷幼鱼体表都覆盖着不规则断裂状的黑色斑纹,成鱼后斑纹逐渐变成笔直的水平纵带[2,17]。当前形态分类资料对上述的胡椒鲷种类聚类关系不清,皆因各分类资料依据的分类标准不同。成庆泰和郑葆珊[1]主编的鱼类检索系统主要根据成鱼的形态进行分类,Mc Kay[17],Carpenter和Niem[18],沈世杰[2]等分类资料却结合了幼鱼形态以及个体发育形态变化过程。Mc Kay[17]认为,胡椒鲷属许多种类的颜色斑纹在个体发育中差异巨大,形态分类需要根据幼鱼,半成鱼与成鱼颜色斑纹,鳍棘与鳃耙数,才能较好的区分。本研究的系统进化树对上述胡椒鲷种类有比较清晰的分类关系。结果支持Mc Kay[17]分类观点,认为胡椒鲷鱼类的分类与亲缘关系的确定需要结合幼鱼,成鱼形态及发育过程才能真实反应其分类关系。研究为部分形态分类混乱的胡椒鲷种类提供了有效的分子水平的分类证据。
4.2 少棘胡椒鲷属的系统进化地位
我国的大部分分类资料中,少棘胡椒鲷D.pictum一直被称为胡椒鲷P.pictus,归为胡椒鲷属[1—2,19—20],而许多国外的资料却根据背鳍鳍棘条数而把它归入到少棘胡椒鲷属中,皆因其背鳍鳍棘数为9~10,而其他的胡椒鲷背鳍鳍棘都是12~14[17—18,21—23]。本研究共采集到两种少棘胡椒鲷,一种是我国广泛分布的少棘胡椒鲷D.pictum,另一种少棘胡椒鲷D.centurio采自非洲塞舌尔马埃岛。本研究结果显示两种少棘胡椒鲷紧密聚在一起,位于胡椒鲷属的内部。遗传距离数据分析上,少棘胡椒鲷属与胡椒鲷属种类之间平均遗传距离为0.112,少于胡椒鲷属内部的平均遗传距离0.123。显示少棘胡椒鲷与胡椒鲷属的种类有很近的亲缘关系。形态上,少棘胡椒鲷与其他色彩鲜艳的胡椒鲷种类一样,体色和斑纹会随着个体发育逐渐变化。两种少棘胡椒鲷的幼鱼体侧有3条黑色纵带,发育成鱼后纵带逐渐破裂成无数深色小斑点。把它归为少棘胡椒鲷属,可能仅仅是基于鳍棘数目的多少而得出的结论。但也有资料揭示少棘胡椒鲷属除了鳍棘数与胡椒鲷属不一样之外,软鳍条数目以及侧线上鳞数也与胡椒鲷属的不一致[9,18]。基于ND1序列的系统发育研究结果,在分子水平上,它们与胡椒鲷属有着很近的亲缘关系,这与当今部分分子水平的分类研究结果一致。Sanciangco等[8]与Tavera等[9]曾利用多个分子标记对北美仿石鲈科鱼类的系统分类研究也发现少棘胡椒鲷位于胡椒鲷属的内部,并没有单独形成一支。Tavera认为少棘胡椒鲷属与胡椒鲷属应该是同一个属。任岗[7]利用16SrRNA序列对12种石鲈科鱼类系统进化树也揭示胡椒鲷P.pictus与胡椒鲷属的种类聚在一起。因此,研究结果支持我国绝大多数传统的分类资料,认为少棘胡椒鲷属的种类归为胡椒鲷属比较合适。
本研究通过利用线粒体DNA分子标记ND1部分序列对胡椒鲷属鱼类进行了系统进化研究,初步阐明胡椒鲷属内种间的分类关系以及少棘胡椒鲷的分类问题,为仿石鲈科鱼类的系统进化研究提供新的分子信息。但要彻底弄清胡椒鲷鱼类的进化关系,还需以后更进一步的研究。
[1] 成庆泰,郑葆珊.中国鱼类系统检索[M].北京:科学出版社,1987:339-341.
[2] 沈世杰.台湾鱼类志[M].台北:台湾大学动物学系,1993:360-363.
[3] 黄宗国.中国海洋生物种类与分布[M].北京:海洋出版社,1994:708-709.
[4] 孟庆闻,苏锦祥,缪学祖.鱼类分类学[M].北京:中国农业出版社,1995:636-640,696-700,711-713.
[5] 朱世华,郑文娟,邹记兴,等.5种石鲈科鱼类细胞色素b基因序列及分子系统分析[J].热带海洋学报,2006,25(4):42-45.
[6] 任岗,章群,钱开诚,等.12种石鲈科鱼类线粒体16Sr RNA基因的部分序列分析[J].热带海洋学报,2007,26(3):48-52.
[7] Sanciangco M D,Rocha L A,Carpenter K E.A molecular phylogeny of the Grunts(Perciformes:Haemulidae)inferred using mitochondrial and nuclear genes[J].Zootaxa,2011,2966:37-50.
[8] Tavera J J,Acero A,Balart E F,et al.Molecular phylogeny of grunts(Teleostei,Haemulidae),with an emphasis on the ecology,evolution,and speciation history of New World species[J].BMC Evolutionary Biology,2012,12:57.
[9] Serb J M,Buhay J E,Lydeard C.Molecular systematics of the North American freshwater bivalve genus Quadrula(Unionidae:Ambleminae)based on mitochondrial ND1 sequences[J].Molecular Phylogenetics and Evolution,2003,28(1):1-11.
[10] Utevsky S Y,Utevsky A Y,Schiaparelli S,et al.Molecular phylogeny of pontobdelline leeches and their place in the descent of fish leeches(Hirudinea,Piscicolidae)[J].Zoologica Scripta,2007,36(3):271-280.
[11] Bowen B R,Kreiser B R,Mickle P F,et al.Phylogenetic relationships among North American Alosa species(Clupeidae)[J].Journal of Fish Biology,2008,72(5):1188-1201.
[12] Thacker C E,Thompson A R,Roje D M.Phylogeny and evolution of Indo-Pacific shrimp-associated gobies(Gobiiformes:Gobiidae)[J].Molecular Phylogenetics and Evolution,2011,59(1):168-176.
[13] Sorenson M D,Ast JC,Dimcheff D E,et al.Primers for a PCR-based approach to mitochondrial genome sequencing in birds and other vertebrates[J].Molecular Phylogenetics and Evolution,1999,12(2):105-114.
[14] Thompson J D,Higgins D G,Gibson T J.CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting,position-specific gap penalties and weight matrix choice[J].Nucleic Acids Research,1994,22(22):4673-4680.
[15] Tamura K,Dudley J,Nei M,et al.MEGA4:Molecular Evolutionary Genetics Analysis(MEGA)software version 4.0[J].Molecular Biology and Evolution,2007,24(8):1596-1599.
[16] Hassanin A,Lecointre G,Tiller S.The“evolutionary signal”of homoplasy in proteincoding genesequences and its consequences for a priori weighting in phylogeny[J].Comptes Rendus de l'Académie des Sciences-Series III-Sciences de la Vie,1998,321(7):611-620.
[17] Mckay R J.Haemulidae[M]//Fischer W,Bianchi G.FAOspecies identification sheets for fishery purposes.Western Indian Ocean(Fishing Area 51),1984.
[18] Carpenter K E,Niem V H.FAOSpecies Identification Guide for Fishery Purposes.The Living Marine Resources of the Western Central Pacific.Volume 5:Bony fishes part 3(Menidae to Pomacentridae)[M].Rome:FAO,2001:2961-2989.
[19] Kawamura T.Seasonal occurrence of fishes at inshore rocky reefs in Shirahama,southern Japan[J].Japanese Journal of Ichthyology,1980,27(3):243-248.
[20] Kyushin K,Amaoka K,Nakaya K,et al.Fishes of the South China Sea[M].Japan Marine Fishery Resource Research Center,Nori Otsuru,Tokyo,Japan,1982:103.
[21] Randall JE,Lim K K P.A checklist of the fishes of the South China Sea[J].The Raffles Bulletin of Zoology,2000,(Sup 8):569-667.
[22] Ramesh R,Nammalwar P.Database on coastal information of Tamilnadu[M].Institute for Ocean Management Anna University Chennal-600 025,2008:1-132.
[23] Shao K T,Ho H C,Lin P L,et al.A checklist of the fishes of southern Taiwan,Northern South China Sea[J].The Raffles Bulletin of Zoology,2008,(Sup.19):233-271.
Molecular phylogenetic relationships of 16 Sweetlips fishes based on the sequence of NADH dehydrogenase subunit I gene
Liang Rishen1,Liang Zhigang1,Fang Haohang1,Tan Jiayu1,Peng Yingxuan1,Xue Tao1
(1.Collegeof Life Science,Zhongkai University of Agriculture and Engineering,Guangzhou 510642,China)
In the present study,molecular phylogenetic relationships among 16 Sweetlips species collected from Indo-Western Pacific were analyzed based on partial DNA sequences of the molecular marker of mitochondrial NADH dehydrogenase subunit I(ND1)gene.The phylogenetic tree was constructed using maximum parsimony and maximum likelihood methods.Two morphologically different groups were formed in the tree.Group I was composed of sweetlips with wondrous coloration and diversed patterns,which were mainly found in the Western Pacific.Group II was composed of species with monotone color and uniformly dark patterns,which were mostly found in the area of Indian Ocean.Additionally,Diagramma species were all found positionally inside the Plectorhinchus tribe in group I in the phylogenetic tree.They were closely related with the sister species Plectorhinchus chaetodonoides and Plectorhinchuspicus.Based on the analysis of the genetic distance,the average value between Diagramma and Plectorhinchus was less than that among the interspecies of Plectorhinchus,indicating an extremely close relationship between Diagramma and Plectorhinchus.The result supported the opinion that Diagramma species might be classified into the genus Plectorhinchus.
phylogeny;Haemulidae;Plectorhinchus;Diagramma;ND1
Q959.46+9
A
0253-4193(2014)10-0025-08
2013-09-30;
2013-11-16。
仲恺农业工程学院大学生创新基金项目(2014A23)。
梁日深(1984—),男,广东省开平市人,博士研究生,研究方向为水生生物资源及保护利用。E-mail:cheetahliang@126.com
梁日深,梁志刚,方浩航,等.基于ND1基因序列的16种胡椒鲷鱼类分子系统进化关系研究[J].海洋学报,2014,36(10):25—32,
10.3969/j.issn.0253-4193.2014.10.003
Liang Rishen,Liang Zhigang,Fang Haohang,et al.Molecular phylogenetic relationships of 16 Sweetlips fishes based on the sequence of NADH dehydrogenase subunit I gene[J].Acta Oceanologica Sinica(in Chinese),2014,36(10):25—32,doi:10.3969/j.issn.0253-4193.2014.10.003

