北极海洋沉积物中可培养细菌及其多样性分析
王桢,李阳,车帅,林学政*
(1.国家海洋局第一海洋研究所国家海洋局海洋生物活性物质重点实验室,山东青岛 266061)
北极海洋沉积物中可培养细菌及其多样性分析
王桢1,李阳1,车帅1,林学政1*
(1.国家海洋局第一海洋研究所国家海洋局海洋生物活性物质重点实验室,山东青岛 266061)
利用Zobell 2216E培养基和涂布平板法对北极海洋沉积物中可培养细菌进行分离纯化,并利用16S rRNA基因进行分子鉴定与系统发育分析。根据菌落形态学特征,从59个站点的沉积物样品中共分离纯化获得570株细菌;基于16S r RNA基因的分子鉴定与系统发育分析表明,分离到的可培养细菌分别属于细菌域的4个门,5个纲,12个目,23个科,47个属,102个种,其中γ-Proteobactria占绝大多数;有14株菌株与模式菌株的16S r RNA基因序列相似性小于97%,为6个潜在的新种。北极海域的海洋沉积物中存在着丰富的微生物种质资源,为开发新型生物活性物质和特殊功能基因打下了基础。
北极;海洋沉积物;细菌多样性;系统发育分析
1 引言
极地具有独特的地理、环境及气候特征,其主要特点是变化极大的光照辐射、季节性的光照时间、常年极低的水温(通常在-1.8~2.0℃)和高盐度环境(海水中的盐度一般为34~35,海冰中盐囊和盐通道的盐度可达普通海水的5倍),形成了一个酷寒、强辐射和高盐度的自然环境[1]。北极地区蕴含着丰富的微生物种质资源,由于其特殊的地理位置和气候条件,生存于其中的微生物大多有其特殊的生理生化特性和代谢特征。由于数万年的沉积作用,深海沉积物成分繁复,是非常复杂的微生物栖息地。有机质是微生物易利用的物质和能源,而深海沉积物中有机质含量比海水中的要多104~105倍;同时,深海沉积物也是巨大的基因遗传变异库[2]。因此,本研究对于北极地区深海沉积物中微生物的种质及其多样性研究具有重要的意义。
北极区域的微生物多样性研究主要集中在海水-海冰生态系统中;此外,北极海水中的浮游微生物[3]和冰川中的微生物多样性研究也有报道[4],而对北极区域海底沉积物中微生物的研究则较少,并且集中在某些特定代谢类型,例如硫酸盐还原菌等[5—6]。对北极环境中的微生物进行大范围的分离培养,并在此基础上进行其微生物多样性分析仅在北极海冰样品中得到了开展[7]。
相对于北极的广阔水域和特殊气候条件,已有研究结果并不能全面反映北极海域中的微生物种质资源多样性,因此还需进一步研究调查。2012年7—9月中国第五次北极科学考察对北极海域海洋沉积物进行了大面积调查并采集了样品,本文通过原位涂布培养、分离纯化及分子鉴定与系统发育分析,分离培养并发现了大量新的细菌种质资源。这些资源对于了解北极海域海洋沉积物微生物的多样性、发现新的微生物种质资源、开发利用其特殊的功能基因和生物活性物质具有重要意义。
2 材料和方法
2.1 样品
北极海域海洋沉积物样品为2012年7—9月中国第五次北极科学考察采集,对箱式采样器和重力采样器采取的海洋沉积物样品,用无菌药匙采集后置于无菌封口袋中,于“雪龙”船现场进行可培养细菌的分离纯化并于4℃保存,回实验室后进行进一步的分离纯化培养与鉴定。
北极海域海洋沉积物采集样品主要采自于4个区域:白令海及其邻近海域、北冰洋海域太平洋扇区、北冰洋-大西洋扇及其他海域。白令海及其邻近海域采样区域为白令海和阿留申群岛周边海域在内的北太平洋北极边缘海,包括沿白令海西侧航线、阿姆奇卡特岛-努尼瓦克岛之间的断面BL,沿白令海峡南部的断面BS,在圣劳伦斯岛和普罗维杰尼亚之间的3个站点及圣劳伦斯岛以东、以南的6个站点构成的断面BM,以及白令海峡以南的BN断面。在本区域内,共对21个站位进行采样,水深从26.2~3 900 m不等,均为表层沉积物样品;除BL04站位采用重力采样器,其他20个站位均采用箱式采样器。北冰洋海域太平洋扇区采样区域为太平洋扇区的白令海峡附近及其以北的楚科奇海,包括R、C、CC和SR共4个断面。在本区域内,共对19个站位进行采样,水深从26.2~109.5 m不等,均为表层沉积物样品;均采用箱式采样器。北极海域大西洋扇区主要包括AT和BB两个断面。在本区域内,共对8个站位进行采样,水深从2 433.6~3 674.3 m不等,均为底层沉积物样品;BB04站位采用多管采样器,其余7个站位采用重力采样器。其他海域主要是对冰岛外海及冰站等11个站位进行采样,水深从491~1 597.4 m不等。
2.2 分离培养基与可培养细菌的分离纯化
海水Zobell 2216E培养基:蛋白胨5 g,酵母粉1 g,琼脂粉15 g,过滤原位海水:自来水(体积比为2∶1),1 000 m L。
无菌药匙取适量泥样,接于含5 m L无菌海水的试管中,充分混匀,经适当系列稀释后,取50μL涂布于2216E培养基平板上,于4℃低温培养箱中倒置培养15 d。根据菌落形态学特征,挑取不同的单菌落进行进一步的分离纯化。
2.3 分子鉴定与与系统发育分析
DNA模板的制备参照文献[8]进行。
16S r RNA扩增采用50μL扩增体系(1μL 27 F,1μL 1492 R,25μL Master Mix,23μL dd H2O)反应条件:95℃,5 min;95℃,1 min,55℃,30 s,72℃,1.5 min,30个循环;72℃延伸7 min。采用细菌16S rRNA扩增通用引物27 F(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R(5′-GGTTACCTTGTTACGACTT-3′)进行PCR扩增,PCR产物送至南京金斯瑞生物股份有限公司进行纯化和测序。测序获得的16S r RNA基因序列长度为1 300~1 400 bp,测序结果在NCBI进行BLAST分析。序列比对采用BioEdit的多序列比对排列(Clustalw multiple alignment),系统发育分析采用Mega 4.0的邻接法(Neighbor-joining method)。与模式菌株的16S rRNA基因相似性比较利用EzTaxon-e Database进行(http://eztaxon-e.ezbiocloud.net)[9]。
3 结果与分析
3.1 菌株的分离纯化与鉴定
经海水Zobell 2216E培养基平板4℃培养15 d,根据菌落形态学特征从59个站位的北极海洋沉积物样品中共分离并获得16S rRNA基因有效序列的细菌达570株。利用EzTaxon-e Database(http://eztaxon-e.ezbiocloud.net)检索与获得的16S r RNA基因序列相似性最高的模式菌株,结果发现,绝大多数与数据库中已有的模式菌株16S r RNA基因序列相似性达97%的可占98%,其中99%以上的可达74.6%。分子鉴定与系统发育分析表明,分离到的可培养细菌分别属于4个门,分别为变形菌门Proteobacteria、放线菌门Actinobacteria、厚壁菌门Firmicutes和拟杆菌门Bacteroidetes,5个纲;其中γ-变形菌纲γ-Proteobacteria有409株,数量最多,占总分离株数的72%;其次为黄杆菌纲Flavobacteria,共66株,占总分离株数的12%;属于α-变形菌纲。α-Proteobacteria、放线菌纲Actinobacteria_c和芽孢杆菌纲Bacilli的菌株数分别为45株、23株和13株,分别占总分离株数的8%、4%和2%(见图1)。值得指出的是,有14株菌株与已有模式菌株的16S r RNA基因序列相似性小于97%,可能为潜在的新种。
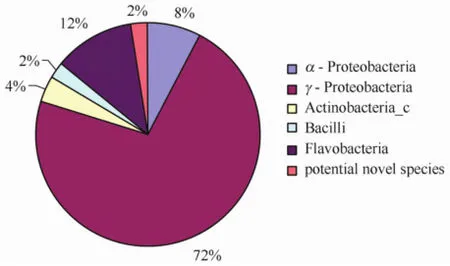
图1 北极海洋沉积物可培养细菌的组成分析Fig.1 Composition analysis of culturable bacteria isolated from Arctic marine sediment
北极海洋沉积物可培养细菌的种属、菌株数量以及分离到该菌株的站位数等基本情况见表1。可以看出,从59个站位中的北极海洋沉积物样品中分离得到的570株细菌,分属于47个属,102个种;其中γ-变形菌纲γ-Proteobacteria在细菌属水平上的多样性最为丰富,包括17个不同的属,黄杆菌纲Flavobacteria次之,包括11个属,α-变形菌纲α-Proteobacteria含有7个属,放线菌纲Actinobacteria_c和芽孢杆菌纲Bacilli各含6个属。在菌株数量上,以希瓦氏菌属Shewanella所含数量最多,共有153株,分属于8个种,占总分离菌株数的27%;且其分布也较广,共从42个站位的沉积物样品中分离到该属的菌株。其次为假交替单胞菌属Pseudoalteromonas的菌株,共有70株,分属于10个种,占总菌株数的12%;其分布也较广,从23个站位的沉积物样品中分离到该属菌株。亚硫酸盐杆菌属Sulfitobacter和科尔韦尔氏菌属Colwellia菌株的出现频率及多样性也比较高,分别包括5个种36株和5个种25株。另外,尽管有的属内菌株数量不多,如鞘氨醇单胞菌属Sphingomonas、副球菌属Paracoccus等仅含一株菌,但可反映出该海域沉积物中微生物的多样性。

表1 北极海洋沉积物可培养细菌的分离鉴定情况一览表Tab.1 Schedule of bacterial isolation and identification from Arctic marine sediment

续表1
一般认为,16S r RNA基因序列同源性小于97%,可以认为属于不同的种;同源性小于93%~95%,可以认为属于不同的属[10]。经测序与对比分析,本文发现有14个菌株序列与相似性最高的模式菌株的16S r RNA基因序列相似性小于97%,并对这14株菌株进行系统发育分析(图2)发现,可能有6株菌为潜在新种。其中有部分菌株,如菌株731与相似性最高的模式菌株Amphritea japonica JAMM 1866T的16S rRNA基因序列相似性仅有91.67%,可能为新属(表2)。
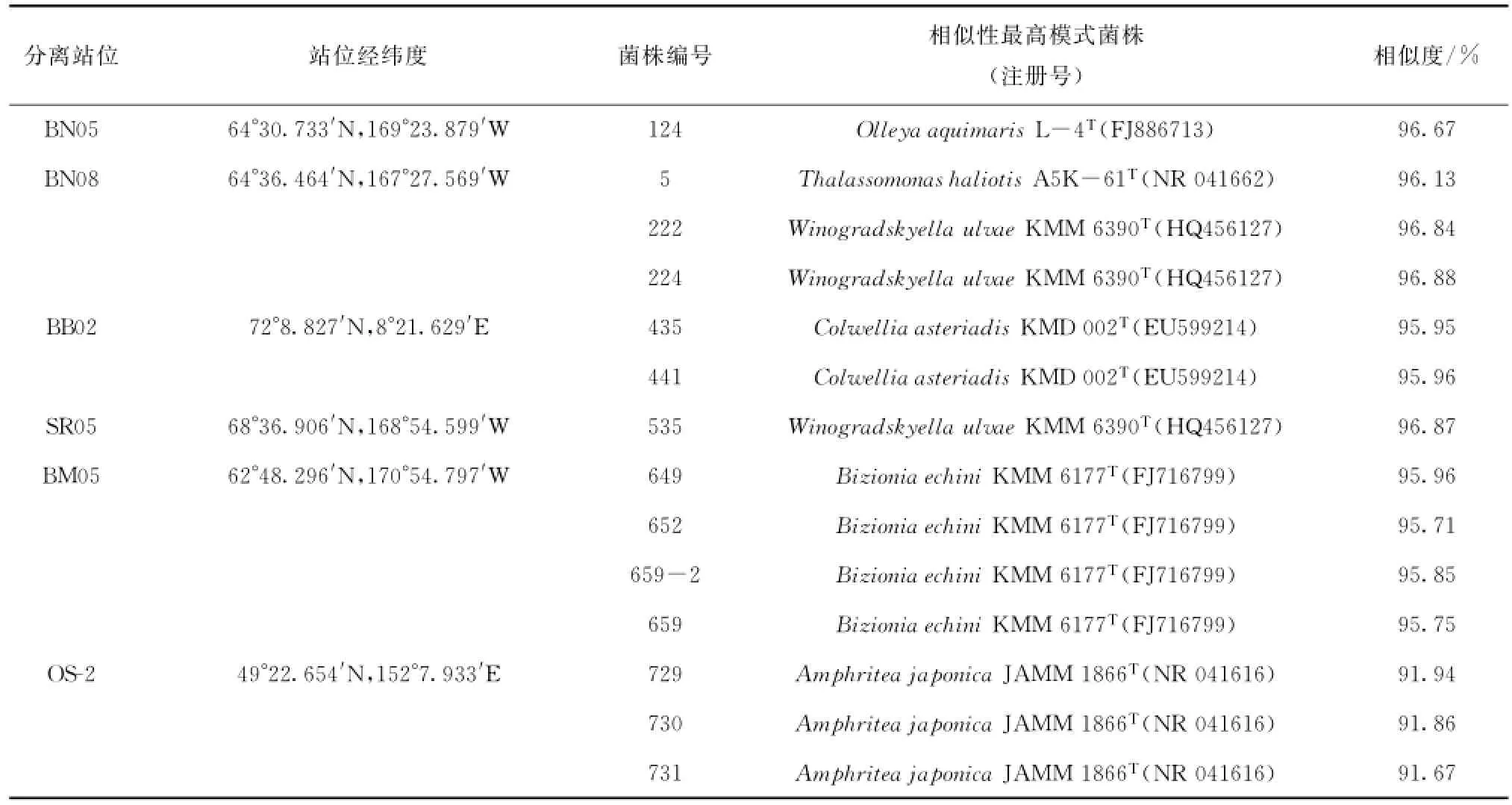
表2 疑似新种16S r DNA序列相似性比较结果Tab.2 Similarity comparison of the 16S rDNA sequences of potential novel species
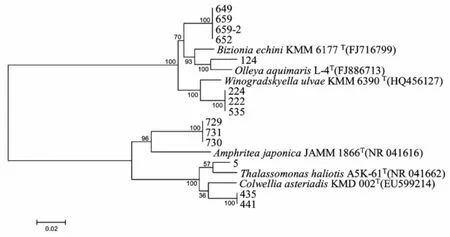
图2 北极海洋沉积物中可培养细菌中疑似新种的系统发育树Fig.2 Neighbor-Joining tree showing the phylogenetic relationships of potential novel species sequences obtained from culturable bacteria isolated from Arctic marine sediment
3.2 可培养细菌的系统发育分析
通过基于16S rRNA基因序列的系统发育分析发现,所有可培养菌聚类于5个大的类群,102个种(见图3和图4),其中,γ-Proteobacteria纲含有59个种,占据了细菌多样性的主要地位,达到了58%(59/102);其次是Flavobacteria纲细菌,达到了18%(18/102);11个序列为α-proteobacteria,占11%;8个序列为Bacilli,占8%;6个序列为Actinobacteria_c,占6%。许多可能的新种(16S rRNA序列相似性小于97%)分散于进化树的多个不同分枝,反映了北极菌种资源的丰富性和新颖性。
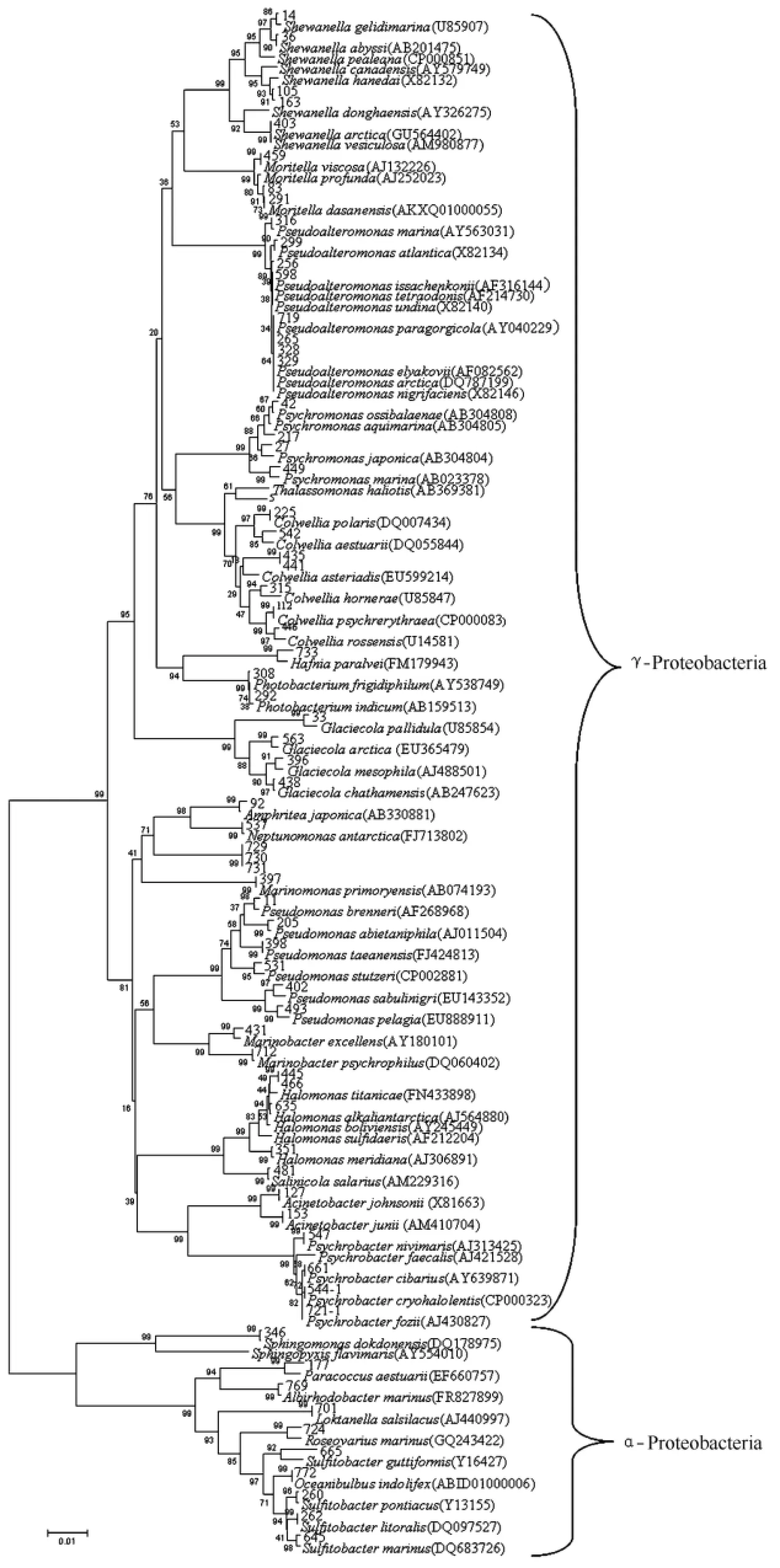
图3 北极海洋沉积物中可培养细菌中变形菌门的系统发育树Fig.3 Neighbor-Joining tree showing the phylogenetic relationships of proteobacteria sequences obtained from culturable bacteria isolated from Arctic marine sediment
在γ-Proteobacteria纲中发现多个疑似新种,如菌株435和441菌株与相似性最高的模式菌Colwellia asteriadis KMD 002T的16S rRNA相似性仅约有95.95%和95.96%;菌株729、730和731与相似性最高的模式菌Amphritea japonica JAMM 1866T16S rRNA相似性只有91.94%,91.86%和91.67%;菌株5与相似性的模式菌Thalassomonas haliotis A5K-61T的16S r RNA相似性只有96.13%。
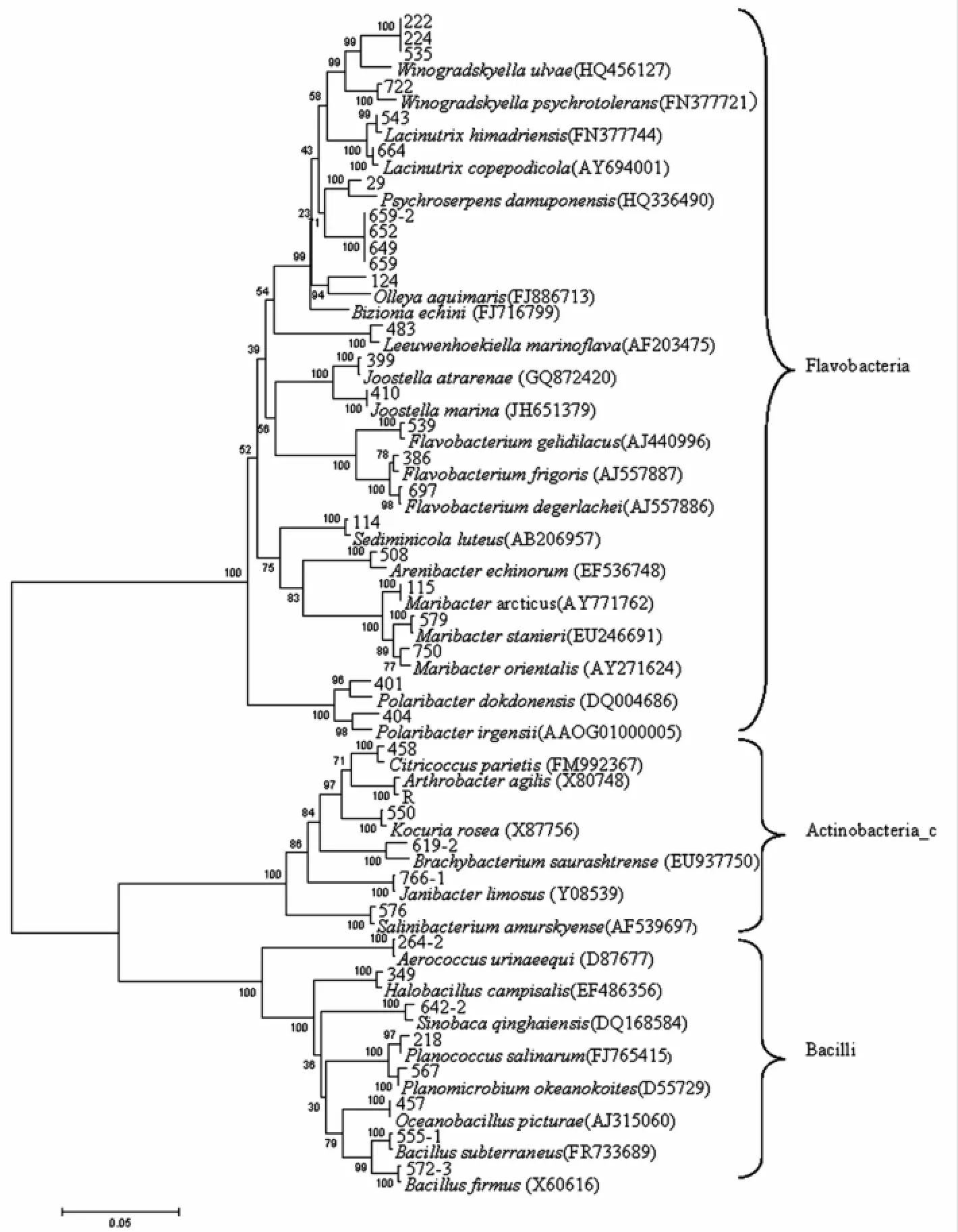
图4 北极海洋沉积物中可培养细菌中黄杆菌纲、放线菌纲、芽孢杆菌纲的系统发育树Fig.4 Neighbor-Joining tree showing the phylogenetic relationships of Flavobacteria sequences,Actinobacteria_c sequences and Bacilli sequences obtained from culturable bacteria isolated from Arctic marine sediment
Flavobacteria纲中也含有疑似新种,菌株222、224和535与最近的模式菌Winogradskyella ulvae KMM 6390T相似性只有96.84%、96.88%和96.87%。菌株649、652、659和659-2与最近的模式菌Bizionia echini KMM 6177T相似性只有95.96%、95.71%、95.75%和95.85%,菌株124与最近的模式菌Olleya aquimaris L-4T相似性只有96.67%。
4 讨论
为获得北极海域海洋沉积物中新的细菌种质资源,了解其多样性特征,本研究对来自北极海域的59个站位的海洋沉积物样品中的可培养细菌进行了分离纯化,获得了大量新的极地微生物种质资源。在测定的570株细菌中,变形菌门Proteobacteria占可培养细菌的绝大多数,其中以γ-Proteobacteria的可培养细菌数量最多,占总分离菌株数的75%,多样性也最为丰富,占细菌种类多样性的58%。这与国内外相关研究结果相一致,如林曦等[11]发现北极白令海深海沉积物中微生物的主要类群为γ-Proteobacteria,在γ-变形菌中主要的菌属为假交替单胞菌属Pseudoalteromonas,假单胞菌属Pseudomonas和希瓦氏菌属Shewanella。Srinivas等[12]对北极孔斯峡湾和斯瓦尔巴特群岛的苔原地区的海底沉积物中可培养细菌的多样性进行了研究,结果显示该区细菌分属于四大类,其中γ-Proteobacteria是该区的优势种。而Groudieva等[13]在对北极海冰可培养微生物多样性的研究中,也发现γ-Proteobacteria纲的细菌数量最多,可占总分离菌数的70%。
本研究获得的γ-Proteobacteria细菌中,属于希瓦氏菌属的细菌最多,其次为假交替单胞菌属,假单胞菌属的数量也较为丰富。假单胞菌自然界分布极广,在自然界物质转化中作用广泛且重要,能利用多种有机物,包括一些较复杂的不易被其他微生物利用的有机物[14];近来在南极土壤乃至深海沉积物中也有发现,并且与多环芳烃降解有关,或直接参与了降解过程[15—16]。希瓦氏菌属是典型的深海细菌类群之一,该菌属中绝大多数为嗜冷或耐冷的细菌[17]。本次实验还分离到芽孢杆菌纲Bacillus的细菌,该细菌一般为好氧异养细菌,它们的出现表明该环境中有机质比较丰富[18]。
近年来对环境样品中的微生物调查研究大多采用分子生物学技术,这种方法能够获取较为全面的微生物信息,而传统的人工培养方法因为受到培养基成分和培养条件等限制而较少应用。但是培养获得的微生物往往是环境微生物群落中能够反映生态环境特征的类群,可以更好地了解样品中处于活体状态的优势细菌类群,评估微生物与环境的关系[19];同时对培养获得的细菌进行生理生化及酶学研究等,可以为开发利用微生物资源打下基础[20]。
放线菌与人类关系密切,目前广泛应用的抗生素约70%是各种放线菌所产生。一些特殊的放线菌还能产生各种酶制剂、维生素和有机酸等。本实验分离获得45株放线菌纲菌株,占总分离菌株的4%。经过多年的研究,陆地放线菌的研究已经相当深入,现今逐渐转向了海洋放线菌的开发利用。北极地区地理位置、气候条件等的特异性,可能使得处于其中的放线茵也具有特殊的生理生化特性,因此对于北极地区放线菌次级代谢产物的研究具有重大的理论意义和应用价值。
北极独特的生态环境蕴藏着丰富的细菌种质资源。本研究通过优化培养基、改变培养条件将获得更多的可培养菌;而通过分子生态分析将对原位微生物多样性获得更全面的了解。北极沉积物中细菌具有极好的多样性和丰富性。本文所获结果将有助于人们深入认识和开发这一特殊海域的微生物资源。
[1] 林学政,边际,何培青.极地微生物低温适应性的分子机制[J].极地研究,2003,15(1):75-82.
[2] Zeng Y X,Zou Y,Chen B,et al.Phylogenetic diversity of sediment bacteria in the northern Bering Sea[J].Polar Biology,2011,34(6):907-919.
[3] Bano N,Hollibaugh J T.Phylogenetic composition of bacterioplankton assemblages from the Arctic Ocean[J].Applied and Environmental Microbiology,2002,68(2):505-518.
[4] Skidmore M L,Fought J M,Sharp M J.Microbial life beneath a high Arctic glacier[J].Applied and Environmental Microbiology,2000,66(8):3214-3220.
[5] Sahm K,Knoblauch C,Amann R.Phylogenetic affiliation and quantification of psychrophilic sulfate-reducingisolates in marine Arctic sediments[J].Applied and Environmental Microbiology,1999,65(9):3976-3981.
[6] Knoblauch C,Jorgensen B B,Harder J.Community size and metabolic rates of psychrophilic sulfate-reducing bacteria in Arctic marine sediments[J].Applied and Environmental Microbiology,1999,65(9):4230-4233.
[7] Junge K,Imhoff F,Staley T,et al.Phylogenetic diversity of numerically important Arctic sea-ice bacteria cultured at subzero temperature[J].Microbial Ecology,2002,43(3):315-328.
[8] 林学政,陈靠山,何培青,等.种植盐地碱蓬改良滨海盐渍土对土壤微生物区系的影响[J].生态学报,2006,26(3):801-807.
[9] Kim OS,Cho Y J,Lee K,et al.Introducing Ez Taxon-e:a prokaryotic 16Sr RNA Gene sequence database with phylotypes that represent uncultured species[J].International Journal of Systematic and Evolutionary Microbiology,2012,62(Pt 3):716-721.
[10] 李贺,林学政,何培青,等.南极抗细菌活性菌株的筛选及系统发育分析[J].微生物学通报,2011,38(2):264-269.
[11] 林曦,杨祥胜,曾润颖.北极深海沉积物中嗜冷细菌的系统发育分析[J].厦门大学学报(自然科学版),2007,46(3):411-417.
[12] Srinivas T N R,Nageswara Rao S SS,Vishnu V R P,et al.Bacterial diversity and Bioprospecting for cold-active lipases,amylases and proteases,from Culturable bacteria of Kongsfjorden and Ny-Ålesund,Svalbard,Arctic[J].Current Microbiology,2009,59(5):537-547.
[13] Groudieva T,Kambourova M,Yusef H,et al.Diversity and cold-active hydrolytic enzymes of culturable bacteria associated with Arctic sea ice,Spitzbergen[J].Extremophiles,2004,8(6):475-488.
[14] Holt J G,Krieg N R,Sneath P H A,et al.Bergey's Manual of Determinative Bacteriology(9th Edition)[M].USA:The Williams&Wilkins Company,1994:335-346.
[15] Ma YF,Wang L,Shao Z Z.Pseudomonas,the dominant poly-cyclic aromatic hydrocarbon-degrading bacteria isolated from Antarctic soils and the role of large plasmids in horizontal gene transfer[J].Environmental Microbiology,2006,8(3):455-465.
[16] Cui Z S,Lai Q L,Dong C M,et al.Biodiversity of Polycyclic Aromatic Hydrocarbon-Degrading Bacteria from Deep Sea Sediments of the Middle Atlantic Ridge[J].Environmental Microbiology,2008,10(8):2138-2149.
[17] Kato C,Nogi Y.Correlation between phylogenetic structure and function:examples from deep-sea Shewanella[J].FEMS Microbiology Ecology,2001,35(3):223-230.
[18] 孙风芹,汪保江,李光玉,等.南海南沙海域沉积物中可培养微生物及其多样性分析[J].微生物学报,2008,48(12):1578-1587.
[19] Sait M,Hugenholtz P,Janssen P H.Cultivation of globally distributed soil bacteria from phylogenetic lineages previously only detected in cultivation independent studies[J].Environmental Microbiology,2002,4(11):654-666.
[20] 王鹏,王风平.南极阿德雷岛淡水湖沉积物细菌群落研究[J].极地研究,2009,21(2):100-107.
Diversity analysis of culturable bacteria isolated from marine sediments of Arctic
Wang Zhen1,Li Yang1,Che Shuai1,Lin Xuezheng1
(1.Key Lab of Marine Bioactive Substances State Oceanic Administration,First Institute of Oceanography,State Oceanic Administration,Qingdao 266061,China)
Culturable bacteria were isolated by use of marine Zobell 2216E medium and spread plate method.Molecular identification and phylogenetic analysis were carried out on the basis of 16S r RNA gene.According to the distinct morphological character of bacterial colony on the marine Zobell 2216E medium plate,a total of 570 isolates were obtained from Arctic sediments.Molecular identification and phylogenetic analysis showed that these isolates belonged to four phylum of the bacteria domain,5 classes,47 genera,and 102 species in total.14 isolates had 16S rRNA gene similarity less than 97%with the closest type strain were grouped into 6 clusters.It indicated that these isolates might be potential novel species.A high diversity of culturable bacteria were isolated from marine sediment of Arctic in term of both species number and phylogenetic composition,which deserves further study and exploitation,such as novel bioactive substances and functional genes.
Arctic;marine sediment;bacterial diversity;phylogenetic analysis
P722.1;P727;Q939.1
A
0253-4193(2014)10-0116-08
2014-03-16;
2014-04-08。
南北极环境综合考察与评估专项(CHINARE2012-03-05,CHINARE2013-03-05)。
王桢(1989—),山东省烟台市人,主要从事极地微生物学研究。E-mail:916376659@qq.com
*通信作者:林学政,研究员,主要从事海洋微生物及其应用学研究。E-mail:linxz@fio.org.cn
王桢,李阳,车帅,等.北极海洋沉积物中可培养细菌及其多样性分析[J].海洋学报,2014,36(10):116—123,
10.3969/j.issn.0253-4193.2014.10.012
Wang Zhen,Li Yang,Che Shuai,et al.Diversity analysis of culturable bacteria isolated from marine sediments of Arctic[J].Acta Oceanologica Sinica(in Chinese),2014,36(10):116—123,doi:10.3969/j.issn.0253-4193.2014.10.012

