精子发生过程中组蛋白翻译后修饰及其作用
葛少钦赵峥辉殷会莹张慧敏综述 黄 锦审校
1. 河北大学医学部(保定 071000); 2. 北京大学第三医院; 3. 河北大学生殖医学研究所
·综 述·
精子发生过程中组蛋白翻译后修饰及其作用
葛少钦1,3赵峥辉1殷会莹1张慧敏1综述 黄 锦2*审校
1. 河北大学医学部(保定 071000); 2. 北京大学第三医院; 3. 河北大学生殖医学研究所
一、精子发生过程中组蛋白甲基化
精子发生过程中组蛋白甲基化是指发生在组蛋白H3或H4N端精氨酸或者赖氨酸残基上的甲基化,其甲基化水平受组蛋白甲基转移酶(Histone methyltransferase, HMT)和组蛋白去甲基化酶(Histone demethylase,HDM)调节。组蛋白甲基化的功能主要体现在异染色质形成,基因转录调节以及其与DNA甲基化相互作用维持特定的染色质结构等方面。不同位点不同数量的甲基化相互组合,为组蛋白甲基化在精子发生过程中发挥作用提供了巨大潜能。
(一)组蛋白甲基化与DNA甲基化
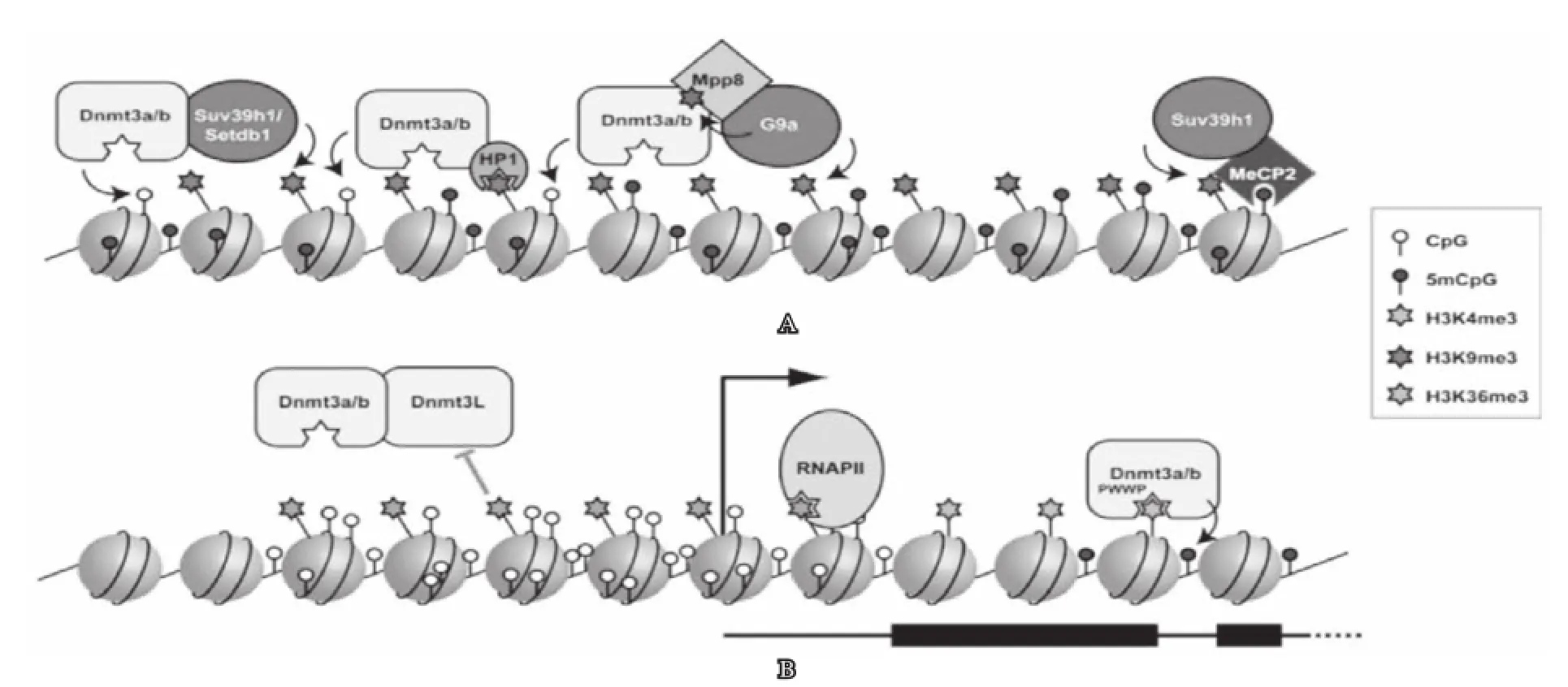
图1 DNA甲基化与组蛋白甲基化作用机制[6]
147 bp的DNA围绕着组蛋白八聚体形成核小体,组蛋白与DNA之间紧密结合,其表观遗传修饰之间可互相作用。其中,组蛋白甲基化与DNA甲基化相互作用,通过建立特定的染色质结构来调节基因表达和细胞分化等过程。Tamaru等[4]通过对粗糙脉孢菌DNA甲基化的研究发现DNA甲基化完全依赖由组蛋白甲基化酶介导的组蛋白H3K9的甲基化,Dnmt3a/b通过异染色质蛋白1(Heterochromatin protein 1,HP1)被三甲基化的H3K9募集,进而调节DNA甲基化[5,6](图1)。Spies等[7-10]研究发现基因本体中DNA甲基化与组蛋白H3K36有关,H3K36在基因本体的外显子中较丰富,其具有富集DNA甲基化的功能,H3K36甲基化与DNA甲基化协同作用调节剪接机制[11];反之,DNA甲基化也调节组蛋白甲基化。异染色质中,Dnmt3a/b通过ADD domain与Suv39h1和Setdb1作用,调节组蛋白H3K9的甲基化[12,13]。常染色质中,Dnmt3a通过染色质蛋白MPP8与组蛋白H3K9甲基转移酶G9a/GLP作用调节其甲基化水平[14]。基因本体中,Dnmt3a/b通过与H3K36me3有亲和力的PWWP domain调节H3K36甲基化[15]。因此组蛋白甲基化与DNA甲基化通过正反调节来维持平衡和协调有功能的染色质[16]。
(二)组蛋白甲基化与基因转录调控
组蛋白H3或H4赖氨酸残基甲基化是基因转录调节的一个关键因素,不同位点的组蛋白甲基化对基因转录的调节不同。精子发生过程中H3K4、H3K9和H3K27的单甲基化、二甲基化和三甲基化修饰表现为严密调控下的暂时性表达,对调节精子内DNA转录状态极其重要[17-19]。H3K4甲基化主要分布在常染色质区转录活化基因的启动子区,其通过促进临近H3K4甲基化以及H3K9去甲基化促进基因转录[3,20];H3K9甲基化通过抑制RNA聚合酶Ⅱ与染色质结合,导致基因转录抑制[3];H3K27甲基化主要存在于具有Polycomb response elements(PREs)的常染色质区, 中心粒异染色质区以及失活的X染色体上,其通过阻止RNA聚合酶的前进,导致转录抑制[3]。精子中H3K4甲基化水平较低,而H3K9和H3K27的甲基化水平则比较高,这或许是为了确保基因的沉默[21,22]。
二、精子发生过程中组蛋白磷酸化
组蛋白磷酸化指通过蛋白质磷酸激酶将ATP的磷酸基转移到组蛋白特定氨基酸上的过程,其与染色体的折叠、压缩和分离、基因转录调控以及DNA损伤修复等多种机制有关[23]。组蛋白H1、H2A、H2B、H3、H4均可发生不同程度的磷酸化,在精子发生的各个阶段发挥着重要作用。其中,H1、H2A和H2B磷酸化在DNA损伤修复以及基因转录调控过程中作用明显。
(一)H2AX磷酸化与DNA损伤修复
H2AX第139位丝氨酸的磷酸化(γ-H2AX)是DNA双链断裂(Double stranded breaks, DSBs)的早期指标,在精子发生过程中呈现不同的水平[24]。Rogakou等[25]在对CD-1小鼠细长型精子胞核DSBs位点研究时检测到了γ-H2AX的存在,Schwaba等[24]通过对雄性小鼠精子中PTIP蛋白的研究发现γ-H2AX与DNA损伤修复蛋白Rad51共同定位于DSBs部位,还有学者通过设计免疫缺陷、对放射敏感的小鼠模型(H2AX-/-mice),发现这种小鼠DNA损伤修复不完善,这些都是γ-H2AX促进胞核DSBs修复的证据[2]。此外,Elgin等[26]研究发现H4高乙酰化诱发实验可以阻止精子发生过程中DSBs,表明γ-H2AX与H4高乙酰化在DSBs损伤修复过程中具有协同作用。精子发生中任何涉及DSBs修复调控异常的因素不仅可以导致精子功能异常而且可能具有遗传性,可见γ-H2AX在调控动物生殖,甚至人类健康与生殖方面都具有非常重要的地位[27]。
从表2可以看出,树脂脱附液经Fenton氧化+Ca(OH)2预处理后,TOC和UV254均得到很好的去除,TOC去除率在53%左右,UV254去除率在44%左右。纳滤膜能截留废水中大部分的有机物,预处理后的树脂脱附液纳滤渗透液对TOC和UV254的去除率分别为97.64%、97.06%,效果均优于未预处理的树脂脱附液纳滤渗透液(TOC和UV254的去除率分别为96.08%、93.00%)。
(二)组蛋白磷酸化与基因转录调控
精子发生过程中组蛋白磷酸化与基因转录的激活与抑制息息相关。组蛋白磷酸化使携带负电荷的磷酸基团与携带正电荷的组蛋白结合,导致组蛋白与DNA之间的亲和力下降,染色质结构改变,暴露出与蛋白质作用因子结合的表面,通过与特异的蛋白质复合物相互作用,调节基因转录水平[28]。Talasz等[29]在Hela细胞的免疫荧光实验中观察到组蛋白H1亚型H1.5、H1.2磷酸化的第172位丝氨酸(Serine 172、S172)出现在有丝分裂期染色质激活转录的区域,而H1.5 N末端磷酸化的S17则出现在抑制转录的染色质区域,Lu等[30]在小鼠精子发生的不同阶段检测到了睾丸特异组蛋白TH2B的第116位苏氨酸的磷酸化,由于其在胞核的独特的区域性分布,提示其可能参与调控特殊染色质区域的基因转录[31]。此外,精子发生过程中组蛋白H2A家族中的H2AX磷酸化与ATR和BRCA1一起引发减数分裂期性染色体钝化(meiotic sex chromosome inactivation, MSCI),但是要维持整个粗线期阶段的MSCI,还需要一些包括定位于X/Y小体上H2A泛素化在内的其他表观遗传修饰协同作用[32]。因此,对精子发生过程中组蛋白磷酸化调控基因转录进行研究有助于理解组蛋白磷酸化的功能作用。
三、精子发生过程中组蛋白乙酰化
精子组蛋白乙酰化是指发生在组蛋白H3和H4 N端比较保守的赖氨酸残基上的乙酰化,其乙酰化水平由组蛋白乙酰转移酶(Histone acetyltransferase, HAT)和组蛋白去乙酰化酶(Histone deacetylase, HDAC)共同调节。组蛋白乙酰化会使染色质松弛,有利于精子发生过程中精蛋白并入DNA和基因转录。在延伸期的精细胞中,主要以H3和H4乙酰化为主,也可能出现H2A和H2B乙酰化[33]。
(一)组蛋白乙酰化与精蛋白并入DNA
以核小体为基础的染色质结构转变为以精蛋白为基础的染色质结构是人类精子形成的一大特征,这些蛋白质并入DNA的过程被紧密调节,最终形成P1:P2的比例约为1:1[33,34]。首先,标准组蛋白普遍被睾丸特异组蛋白变体替代,使核小体形成一个比较宽松的结构;然后,全基因组范围内的组蛋白开始高度乙酰化,其中组蛋白尾部乙酰化为募集Brdt提供了平台,而组蛋白折叠区域的乙酰化则是为了使乙酰基与组蛋白H4上的赖氨酸残基结合,中和赖氨酸所带正电荷,进一步使核小体解聚,易于与转录因子等转录相关蛋白结合;最后,Brdt识别乙酰化的组蛋白尾开始了组蛋白移除和精蛋白并入DNA的过程,移除的乙酰化组蛋白在精子蛋白酶体PA200/Blm10-20S的作用下降解[35-37](图2)。实际上,在组蛋白乙酰化的同时,也发生了H2AX磷酸化、H2A泛素化和组蛋白多ADP核糖基化,这些组蛋白PTMs互相作用共同调节组蛋白移除过程[38-40]。McGraw等[41]在牛细长型精子细胞中检测到了催化H3和H4的组蛋白乙酰转移酶MYST4。Steilmann等[42]在人和小鼠细长型精子细胞中发现了H3K9乙酰化。H4赖氨酸尾部乙酰化一般影响到第5、8、12、16位残基,为进一步确定延伸期组蛋白H4赖氨酸的乙酰化情况,De Vries等[43]研究发现在人类细长型精子细胞中,H4乙酰化主要发生在第8和第16位残基上。Govin等[44]观察到在小鼠伸长型精子细胞中总体乙酰化提高主要在组蛋白H4的第5、8、12位残基,而不包括第16位残基,用特异性抗体进行的免疫荧光实验表明,所有3种乙酰化赖氨酸—acK5、acK8和acK12在细长型精子细胞中与主要的卫星区域相关。此外,在哺乳动物细长型精子细胞中,观察到了不同HMT和HDM的表达,H3K4和H3K9单甲基化、二甲基化和三甲基化以及H3K27的单甲基化和三甲基化在细长型精子细胞中高度表达,这些组蛋白甲基化与乙酰化相互协调,使染色质组态更加开放[43,45]。
(二)组蛋白乙酰化与基因转录调控
精子发生过程中,组蛋白乙酰化有利于染色质解聚,以促进聚合酶Ⅱ对基因的转录[33]。乙酰化酶家族作为辅助激活因子,一般使转录激活,去乙酰化酶家族则相反[46]。HAT通过在组蛋白的N端赖氨酸残基上引入疏水的乙酰基,使DNA与组蛋白间的静电引力和空间位阻增大,两者间的相互作用减弱,DNA易于解聚,染色质呈转录活性结构,有利于转录因子与DNA模板相结合,进而募集转录共激活因子,整合转录因子传来的信息,最终决定转录的水平[47,48]。相反,HDAC使去乙酰化后带正电的组蛋白与带负电的DNA紧密结合,染色质呈致密卷曲的阻抑结构,从而抑制基因转录[48]。
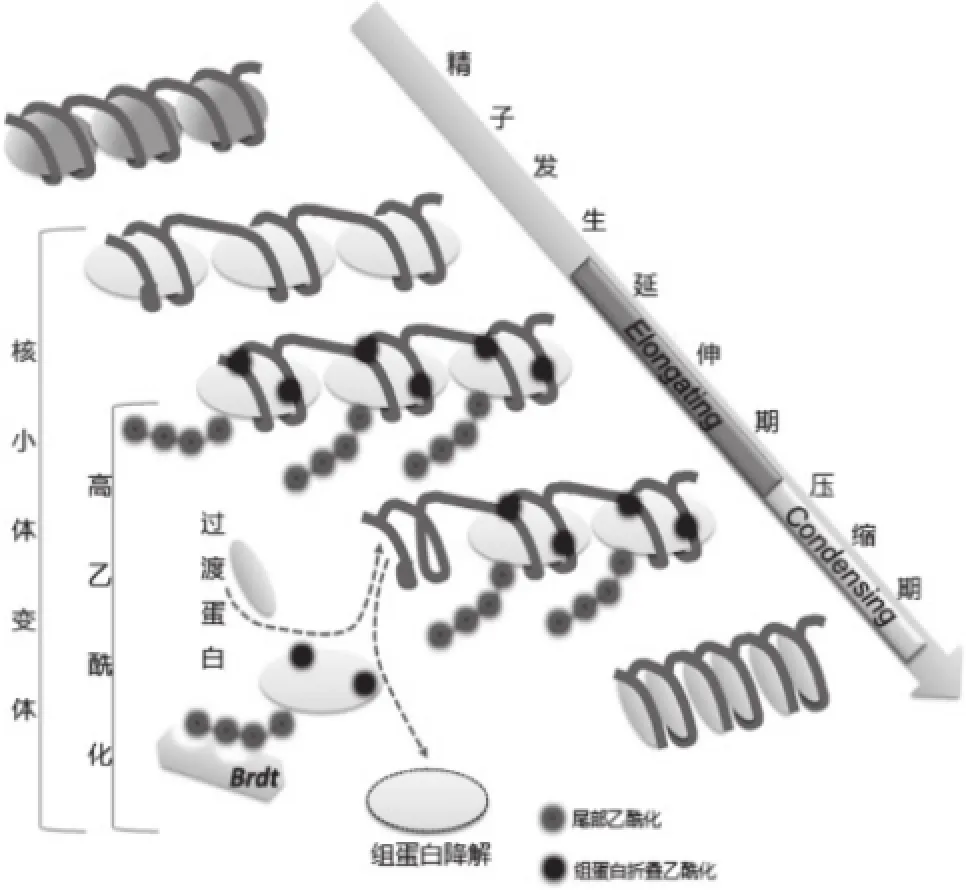
图2 精子延伸期过渡蛋白替换组蛋白模型[35]
四、精子发生过程中组蛋白泛素化
组蛋白泛素化在精子发生过程中发挥着至关重要的作用,其泛素化水平由泛素活化酶E1、泛素接合酶E2和泛素连接酶E3共同调节[30,49]。近期,蛋白质组学研究发现在4600多种人类精子蛋白中,大约有220种蛋白质是精子特有的[50]。自从泛素首次从鱼类和哺乳类动物中分离,有30多种泛素化酶被认为是精子发生过程中重要的调节因子[51-53]。据估计大约有70种E3在小鼠精子发生过程中表达,表明泛素系统有着多种重要功能[54]。哺乳动物中含有近90种去泛素化酶,他们是精子发生过程中重要的调节因子,其基因突变或DNA序列的靶向断裂会导致精子发育和生育能力的严重异常[55]。Manku等[56]研究发现在生殖母细胞和精原细胞泛素系统所表达的205个基因中有91个基因相对高表达,其中包括去泛素化酶如泛素特异蛋白酶2(ubiquitin-specif c protease 2, USP2)和USP19,表明去泛素化在生殖母细胞分化过程中发挥着重要作用。
(一)组蛋白泛素化与组蛋白替代
精子发生过程中,泛素结合蛋白的水平呈动态变化,在精原细胞和精母细胞中含量微弱,在圆形精子和长形精子中含量增加,在成熟精子中含量较低。通过蛋白免疫印迹实验发现泛素结合蛋白在精子延伸期大量出现,其可能与组蛋白的移除与降解有关[57]。组蛋白泛素化可以提高转运蛋白或精蛋白与转录调节因子的结合力有助于染色质解聚,HR6B是Rad6 E2的一种同系物,定位于常染色质区,在精子延伸期高度表达[58],Roest等[59]通过对小鼠的研究发现HR6B的破坏会造成生精细胞广泛凋亡、精子头部形态异常和不完全的组蛋白替代,表明由HR6B催化的组蛋白泛素化在组蛋白替代和核浓缩等过程中发挥着重要作用。Lu等[60]通过研究RNF8基因敲除的小鼠模型,发现在精子发生过程中组蛋白泛素化通过减少H2A-H2B二聚体释放能量来促进翻译过程,进而影响组蛋白乙酰化,RNF8 E3催化H2A泛素化的同时也会通过H4K16乙酰转移酶促进H4乙酰化,表明H2A泛素化与H4乙酰化在翻译过程中具有协同作用。此外,RNF8敲除的小鼠睾丸研究显示,组蛋白泛素化的抑制和H4K16乙酰化的去除是核小体移除过程开始的标志,可见组蛋白泛素化和乙酰化是一个高度互相作用的过程。
(二)组蛋白泛素化与基因转录调控
组蛋白泛素化在调节基因转录活性方面发挥着重要作用,对阐明泛素化与核浓缩的关系十分必要。近期研究表明,位于复制叉的Uhrf1蛋白通过其SRA domain与半甲基化位点结合,然后将其C末端的Ring domain作为E3催化H3K23泛素化,泛素化的H3K23通过与RFTS domain作用,将Dnmt1募集到复制中心,使半甲基化的CpG甲基化,进而调控基因转录[6,61]。此外,组蛋白泛素化通过作用于SCF复合体中的FBXO22,调节组蛋白去甲基化酶4A,也可影响基因转录过程[62]。组蛋白H2A泛素化会导致转录抑制[63,64],在有丝分裂的粗线期,H2A泛素化在X和Y拟常染色体联会的区域富集[65],通过与一系列特殊的组蛋白修饰如H3K9二甲基化、H4去乙酰化和H2AX磷酸化协同作用一起诱发MSCI过程使性染色体转录抑制[66,67]。此外,H3K36me2的去甲基化酶Kdm2b与H2A泛素化协同作用,前者可将PRC1复合体的变体募集到CpG岛上,后者则可抑制FACT(FACT可以促进RNA聚合酶在染色质上滑动)与染色质结合,其可能是PRC1介导基因沉默的机制之一[6,68]。组蛋白H2B泛素化一般导致转录激活[69-72],然而Sun等[73]通过对酵母菌的研究发现H2B组蛋白单泛素化会导致组蛋白H3甲基化,进而造成端粒基因的沉默。可见组蛋白泛素化调控基因转录的具体过程还有待于进一步研究。
精子发生过程中组蛋白翻译后修饰呈动态变化,各种组蛋白翻译后修饰紧密联系,通过与其他表观遗传修饰因子协同作用,精确地调节精子发生的各个过程。减数分裂时期,H2A泛素化通过影响H2B和H3高甲基化调控DNA损伤修复过程;精子形成时期,组蛋白泛素化调控与组蛋白乙酰化、甲基化、磷酸化修饰相互协调共同调控精子形成过程;此外精子表观遗传修饰如DNA去甲基化、miRNA表达以及组蛋白修饰等不仅在精子形成中发挥重要作用,在胚胎干细胞中也具有相似的功能,因此de Boer 等[74]认为,精子可能是跨代遗传不稳定和后代基因突变的源头,Carrell也发现人类精子中的组蛋白修饰主要富集在对早期胚胎发育有重要影响的Hox发育基因中,进一步证明了精子中组蛋白修饰对早期胚胎发育的重要性[75]。
五、问题与展望
随着生物学技术的迅猛发展,人们对组蛋白PTMs认识的日益深入,在组蛋白密码研究方面做出了一定成绩。人们发现在精子发生过程中,多种组蛋白PTMs可以单独或协同作用调节基因的转录水平以及参与组蛋白移除和DNA损伤修复等多种生物学过程,而且组蛋白PTMs与其他表观遗传修饰之间也可以相互作用,如组蛋白甲基化与DNA甲基化相互作用共同维持特定的染色质结构来调控基因的转录,组蛋白乙酰化与精蛋白相互作用,完成精蛋白替代组蛋白的过程。虽然不同组蛋白甲基化都能影响DNA甲基化,对基因转录起到促进或抑制作用,但这种作用的机制还不是十分明确。此外,精子发生过程中各种生物学过程均需要多种表观遗传修饰共同作用完成,只有对各种表观遗传修饰进行综合分析,才有可能对精子发生过程中染色质浓缩、基因转录调节和精蛋白替代组蛋白等过程有一个深入而全面的理解。此外,精子中各种组蛋白PTMs相互作用并与其他表观遗传修饰因子协同调控早期胚胎发育过程的具体机制将成为生殖专家研究的另一个新方向。为加强精子表观遗传研究的系统性和特异性,本实验室与美国犹他大学Andrology and IVF实验室联合开展了精子形态分类(WHO标准)基础上的表观遗传因子DNA甲基化、H3K4me2和H3K27me3的胶体金免疫定位研究,试图建立精子表观遗传质量评价标准,为进一步研究组蛋白密码的作用机制及其功能作用提供理论基础。
致谢:本课题由河北省自然科学基金资助项目(编号:H2013201259);河北省2013留学回国人员科研活动项目(编号:C2013005002)和河北大学创新训练项目(编号:201410075029,201410075073)资助
组蛋白类; 生物,基因修饰; 精子发生;转录,遗传
1 张朝晖, 康现江, 穆淑梅. 遗传 2014; 36(3): 220-227
2 Zhang Y, Reinberg D.Genes Dev2001; 15(18): 2343-2360
3 宋博研, 朱卫国. 遗传 2011; 33(4): 285-292
4 Tamaru H, Selker EU.Nature2001; 414(6861): 277-283
5 Lehnertz B, Ueda Y, Derijck AA,et al. Curr Biol2003; 13(14): 1192-1200
6 Rose NR, Klose RJ.Biochim Biophys Acta2014 pii: S1874-9399(14)00028-5
7 Spies N, Nielsen CB, Padgett RA,et al. Mol Cell2009; 36(2): 245-254
8 Kolasinska-Zwierz P, Down T, Latorre I,et al. Nat Genet2009; 41(3): 376-381
9 Andersson R, Enroth S, Rada-Iglesias A,et al. Genome Res2009; 19(10): 1732-1741
10 Huff JT, Plocik AM, Guthrie C,et al. Nat Struct Mol Biol2010; 17 (12): 1495-1499
11 Brown SJ, Stoilov P, Xing Y.Hum Mol Genet2012; 21(R1): R90-R96
12 Fuks F, Hurd PJ, Deplus R,et al.Nucleic Acids Res2003; 31(9): 2305-2312
13 Li H, Rauch T, Chen ZX,et al. J Biol Chem2006; 281(28): 19489-19500
14 Chang Y, Sun L, Kokura K,et al. Nat Commun2011; 2: 533
15 Dhayalan A, Rajavelu A, Rathert P,et al. J Biol Chem2010; 285(34): 26114-26120
16 Roudier F, Ahmed I, Berard C,et al. EMBO J2011; 30(10): 1928-1938
17 Boissonnas CC, Jouannet P, Jammes H.Fertil Steril2013; 99(3): 624-631
18 Lachner M, Jenuwein T.Curr Opin Cell Biol2002; 14(3): 286-298
19 Suganuma T, Workman JL.Cell2008; 135(4): 604-607
20 Barski A, Cuddapah S, Cui K,et al. Cell2007; 129(4): 823-837
21 Simon JA, Kingston RE.Nat Rev Mol Cell Biol2009; 10(10): 697-708
22 Gannon JR, Emery BR, Jenkins TG,et al. Adv Exp Med Biol2014; 791: 53-66
23 Khalil AM, Wahlestedt C.Epigenetics2008; 3(1): 21-28
24 Schwaba KR, Smith GD, Dressler GR.Dev Biol2013; 373(1): 64-71
25 Rogakou EP, Pilch DR, Orr AH,et al. J Biol Chem1998; 273(10): 5858-5868
26 Elgin SC, Grewal SI.Curr Biol2003; 13(23): R895-R898
27 Kimmins S, Sassone-Corsi P.Nature2005; 434(7033): 583-589
28 Ahn SH, Henderson KA. Keeney S,et al. Cell Cycle2005; 4(6): 780-783
29 Talasz H, Sarg B, Linder HH.Chromosoma2009; 118(6): 693-709
30 Lu S, Xie YM, Li X,et al.Mol Hum Reprod2009; 15(6): 373-378
31 Zalensky AO, Siino JS, Gineitis AA,et al. J Biol Chem2002; 277(45): 43474-43480
32 Carrell DT, Hammoud SS.Mol Hum Reprod2010; 16(1): 37-47
33 葛少钦, 李建忠, 张晓静. 遗传 2011; 33(9): 939-946
34 Jenkins TG, Carrell DT.Asian J Androl2011; 13(1): 76-80
35 Goudarzi A, Shiota H, Rousseaux S,et al. J Mol Biol2014; 426(20): 3342-3349
36 Kouzarides T.Cell2007; 128(4): 693-705
37 Qian MX, Pang Y, Liu CH,et al. Cell2013; 153(5): 1012-1024
38 Leduc F, Maquennehan V, Nkoma GB,et al. Biol Reprod2008; 78(2): 324-332
39 Baarends WM, Hoogerbrugge JW, Roest HP,et al. Dev Biol1999; 207(2): 322-333
40 Meyer-Ficca ML, Scherthan H, Burkle A,et al. Chromosoma2005; 114(1): 67-74
41 McGraw S, Morin G, Vigneault C,et al. BMC Dev Biol2007; 7: 123
42 Steilmann C, Paradowska A, Bartkuhn M,et al. Reprod Fertil Dev2011; 23(8): 997-1011
43 De Vries M, Ramos L, Housein Z,et al. Biol Open2012; 1(5): 446-457
44 Govin J, Escoffier E, Rousseaux S,et al. J Cell Biol2007; 176(3): 283-294
45 Godmann M, Auger V, Ferraroni-Aguiar V,et al. Biol Reprod2007; 77(5): 754-764
46 王维, 孟智启, 石放雄. 遗传 2012; 34(7): 810-818
47 卢震, 王永潮. 科学通报 1998; 43(8): 792-797
48 管晓翔, 陈龙邦. 中华肿瘤防治杂志 2007; 14(4): 307-310
49 Nobuhiro N.Cells2013; 2(4): 732-750
50 Wang G, Guo Y, Zhou T,et al. J Proteomics2013; 79: 114-122
51 Roth G, Himstedt W.Naturwissenschaften1978; 65(12): 657-658
52 Loir M, Caraty A, Lanneau M,et al. FEBS Lett1984; 169(2): 199-204
53 Hou CC, Yang WX.Mol Biol Rep2013; 40(4): 3213-3230
54 Hou X, Zhang W, Xiao Z,et al. BMC Genomics2012; 13: 495
55 Clague MJ, Barsukov I, Coulson JM,et al. Physiol Rev2013; 93(3):1289-1315
56 Manku G, Wing S, Culty M.Biol Reprod2012; 87(2): 44
57 葛少钦, 康现江, 刘桂荣, 等. 生物学杂志 2008; 25(2): 1-6
58 Koken MH, Hoogerbrugge JW, Jasper-Dekker I,et al. Dev Biol1996; 173(1): 119-132
59 Roest HP, van Klaveren J, de Wit J,et al. Cell1996; 86(5): 799-810
60 Lu LY, Wu J, Ye L,et al. Dev Cell2010; 18(3): 371-384
61 Nishiyama A, Yamaguchi L, Sharif J ,et al. Nature2013; 502(7470): 249-253
62 Labbé RM, Holowatyj A, Yang ZQ.Am J Transl Res2013; 6(1): 1-15
63 Osley MA.Brief Funct Genomics Proteomic2006; 5(3): 179-189
64 Weake VM, Workman JL.Mol Cell2008; 29(6): 653-663.
65 Baarends WM, Wassenaar E, van der Laan R.Mol Cell Biol2005; 25(3): 1041-1053
66 Turner JM, Mahadevaiah SK, Fernandez-Capetillo O,et al. Nat Genet2005; 37(1): 41-47
67 Sheng K, Liang X, Huang S,et al. Biomed Res Int2014: 870695
68 Simon JA, Kingston RE.Nat Rev Mol Cell Biol2009; 10(10): 697-708
69 Henry KW, Wyce A, Lo WS,et al. Genes Dev2003; 17(21): 2648-2663
70 Kao CF, Hillyer C, Tsukuda T,et al. Genes Dev2004; 18(2): 184-195
71 Wood A, Schneider J, Shilatifard A.Biochem Cell Biol2005; 83(4): 460-467
72 Pavri R, Zhu B, Li G,et al. Cell2006; 125(4): 703-717
73 Sun ZW, Allis CD.Nature2002; 418(6893): 104-108
74 de Boer P, Ramos L, de Vries M,et al. Mol Hum Rep2009; 16(1): 48-56
75 Carrell DT.Fertil Steril2012; 97(2): 267-274
(2014-06-25收稿)
10.3969/j.issn.1008-0848.2014.10.016
R 394.1

