猪繁殖与呼吸综合征病毒5'非翻译区一顶端茎环结构是病毒复制转录必需的顺式作用元件
高 飞,郑 浩,姜一峰,李丽薇,郑海红,周艳君,韦祖樟,童光志
(1. 中国农业科学院上海兽医研究所,上海 200241;2. 广西大学动物科学技术学院,南宁 530005)
·研究论文·
猪繁殖与呼吸综合征病毒5'非翻译区一顶端茎环结构是病毒复制转录必需的顺式作用元件
高 飞1,郑 浩1,姜一峰1,李丽薇1,郑海红1,周艳君1,韦祖樟2,童光志1
(1. 中国农业科学院上海兽医研究所,上海 200241;2. 广西大学动物科学技术学院,南宁 530005)
猪繁殖与呼吸综合征病毒(Porcine reproductive and respiratory syndrome virus,PRRSV)基因组5' 非翻译区 (untranslated region,UTR)对病毒复制转录等生物合成过程至关重要,但其作用机制尚不清楚。本实验室分别在北美型(pAPRRS) 与欧洲型PRRSV(pSHE) 感染性克隆骨架的5' UTR与ORF1之间插入碱基”at”形成Nde I位点,构建了两个突变体pTLNd4和pSHEnde。结果显示插入后的碱基未影响病毒感染性。以两株突变体为骨架将两株感染性克隆的5' UTR互换,构建了突变体克隆pTLV8和pSHSP5。结果表明pTLV8仍具有感染性,而pSHSP5株拯救出病毒。对pSHSP5和pTLV8的RNA二级结构预测结果显示,虽然嵌合突变体pTLV8的5' 端与亲本pAPRRS的二级结构差异较大,但转录调控序(transcription-regulating sequence, TRS)仍位于顶端茎环结构最突出的位置,并保证了TRS与ORF1之间的起始密码子AUG位于该茎环结构的顶端,而pSHSP5的顶端茎环结构则遭到了破坏。本研究结果表明TRS所在的顶端茎环结构在调控病毒复制转录过程中是关键的顺式作用元件之一,对其结构破坏的突变体将不能拯救出病毒。
猪繁殖与呼吸综合征病毒;5'非翻译区;嵌合突变体;转录调控序列;二级结构预测
单股正链RNA病毒基因组的5'非翻译区(5' untranslated region,5'UTR)是病毒复制的关键,在许多的生物合成过程中也发挥作用。5' UTR中的一些重要的二级结构能防止基因组RNA被核酸外切酶降解[1],例如脊髓灰质炎病毒5' 端的一个苜蓿叶状的二级结构对于基因组RNA的稳定性至关重要[2]。5' UTR对RNA合成也是决定性的,在登革热病毒中,5' 末端的一个茎环RNA结构能够作为病毒的RNA依赖的RNA聚合酶(RNA-dependent polymerase, RdRP)结合位点,通过长距离的RNA-RNA相互作用促使5' 末端与基因组的3' 端模板识别启动特定的RNA的合成[3,4]。如脊髓灰质炎病毒的苜蓿叶状结构不仅能稳定基因组,还是病毒基因组正链和负链RNA合成所必需的[5,6]。在对动脉炎病毒科的马动脉炎病毒的研究中发现,其5'末端的转录调控序列(transcriptional regulatory sequence,TRS)所在的先导序列中的顶端茎环结构在病毒亚基因组转录中发挥着启动子的作用。动脉炎病毒科中其他成员及冠状病毒的5'末端序列结构分析中也预测了同样的结构,因此其很有可能是是病毒亚基因组mRNA转录的关键性结构因子并且对TRS功能的发挥起着关键作用[7,8]。此外5' UTR还可对病毒蛋白的翻译进行调控,例如黄病毒科[9]和小RNA病毒科[10,11]的一些成员中,发现5' UTR中拥有内部核糖体进入位点(internal ribosomal entry site,IRES)这样的结构元件对蛋白翻译进行调控。
猪繁殖与呼吸综合征病毒 (Porcine reproductive and respiratory syndrome virus,PRRSV),属于套式病毒目、动脉炎病毒科、动脉炎病毒属。根据血清型分型可以将PRRSV分为欧洲型(type I)和北美型(type II)两种[12,13]。它是一种拥有单股正链RNA基因组的小囊膜病毒,病毒的基因组长约为15 kb,两端分别为5' 和3' UTR,在3' 末端还有一段poly (A)尾。基因组至少拥有9个开放阅读框 (open reading frame, ORF: ORF1a、1b、2a、2b、ORF3-7)[14]。根据序列分析,两型5' UTR区域的长度不同,序列同源性只有约50%。但是在所有株的PRRSV中,二级结构预测结果发现不少保守的茎环结构,推测它们会在病毒复制过程中发挥作用。
本研究基于反向遗传操作技术,在北美型PRRSV(type 2)和欧洲型(type 1)PRRSV的感染性克隆骨架上(pAPRRS[15,16]、pSHE[17]),对二者 5' UTR进行互换,构建相应的嵌合突变体pTLV8和pSHSP5。经过DNA转染Marc-145细胞,传代至第2代能拯救出嵌合病毒vAPLV5,而pSHSP5则是一个致死性突变体。对pSHSP5以及pTLV8进行二级结构预测发现,嵌合突变体pTLV8保留了5'端TRS所在的顶端茎环结构,而pSHSP5 的5'端TRS所在的顶端茎环结构则遭到了破坏。对另一突变体pPa2的研究中也发现突变破坏了此茎环结构。综上所述,本研究证明了TRS和ORF1的起始密码子所在的顶端茎环结构是调控病毒复制转录过程的关键顺式作用元件之一,任何对其破坏的突变都是致死性突变。
1 材料与方法
1.1 材料
1.1.1 细胞与病毒 乳仓鼠肾细胞 (BHK-21, ATCC No. CCCL10) 和非洲绿猴肾细胞 (Marc-145,No. CRL-12219,Manassas,VA)由中国农业科学院上海兽医研究所猪病实验室(以下简称本实验室)保存;vAPRRS(GenBank 登录号:GQ330474)在本实验室中作为亲本病毒对照与其他嵌合病毒和突变病毒作比较,同时它也作为与vSHE(GenBank 登录号:GQ461593)5' UTR替换的骨架。
1.1.2 试剂 突变PCR聚合酶为Pfu Turbo®Hotstart DNA Polymerase,购自Strategene公司;限制性内切酶购自NEB和TaKaRa公司;TOP10感受态细胞购自TIANGEN公司;化学转染试剂FuGENE®HD购自Roche公司;Lipofectamine LTX/PLUS试剂、OPTI-MEM购自Invitrogen公司;RT-PCR反转录酶为AMV,购自TaKaRa公司;Northern杂交试剂购自Ambion公司。
1.1.3 主要仪器 超净工作台,上海三超净化设备有限公司;PCR仪,DNA Thermal Cycler 480,美国PERKIN ELMER公司生产;恒温培养摇床,型号THZ-C,江苏太仓实验设备厂;恒温培养箱,上海医疗器械七厂;水平电泳仪,型号DY-a,上海康达电子仪器厂生产;荧光倒置显微镜,Olympas公司;离心机,型号5415D,Eppendorf公司。
1.2 方法
1.2.1 突变体构建 type 1 PRRSV和type 2 PRRSV全长感染性克隆pSHE 和 pAPRRS以及type 1 5' UTR type 2骨架嵌合克隆pTLV8由本研究室构建[16]。在pSHE的5' UTR与ORF1之间插入“at”形成Nde I位点,由此形成的全长突变体是pSHEnde,在pSHEnde的基础上,将pAPRRS的5' UTR替换入pSHE中,构建了嵌合突变体pSHSP5。所有突变质粒均经过突变位点测序及Sma I酶切鉴定。以pAPRRS为骨架,利用Afl II和Spe I酶切使其缺失1693~13 116 nt片段,构建了复制功能缺失的质粒pAS作为检测复制的阴性对照[15,17]。所用到的引物见表1。

表1 文中所用的引物和探针Table1 Primers and probes for this study
1.2.2 病毒拯救及鉴定 待六孔板的BHK-21细胞长到密度大约为60%~80%时,用Lipofectamine LTX & PLUS试剂(Invitrogen,Carlsbad,CA) 以等量(3.75 μg) 全长突变质粒转染BHK-21细胞,在5% CO2、37℃温箱中培养,转染72 h后,收集上清,并接种于Marc-145细胞,每天观察细胞病变(cytopathic effect,CPE)。当出现80%的病变时,收集上清并保存于-70℃,作为初次拯救的病毒,标记为P0。P0代病毒液稀释1000倍后,接种于新鲜的MARC-145细胞,感染后d4收集病毒上清,标记为P1,以相同的方式收集P2~P5代病毒。
传代的病毒上清用QIAamp Viral RNA Mini Kit(QIAgen,Hilden,Germany)提取病毒RNA,参考说明书进行操作。以提取好的RNA为模板,使用TaKaRa公司的AMV反转录酶进行病毒RNA反转录,引物Qst(序列见表1),得到相应的cDNA,利用该模板及相应的引物得到突变位点所在的PCR片段,PCR产物经1%的琼脂糖凝胶电泳鉴定及纯化后,送Invitrogen公司进行测序,测序结果用DNAStar软件与vAPRRS和vSHE基因进行比对。
1.2.3 负链基因组复制及亚基因组mRNA转录水平分析 以提取的细胞总RNA为模板,用引物Qst进行反转录,共20 μL体系,接着42℃水浴1 h,最后冰浴2 min,接着进行PCR扩增。
首先进行β-actin内参扩增,PCR上下游引物为F-actin和 R-actin。共25 μL体系,PCR反应参数为:95℃预变性3 min;95℃变性30 s,55℃退火30 s,72℃延伸20 s,共进行30个循环;72℃延伸10 min,然后取5 μL PCR产物在1.5%琼脂糖凝胶电泳中鉴定结果。
以反转录的cDNA为模板PCR扩增sg mRNA 7,扩增sg mRNA 7的4对引物:SF12/SR15284、SFLV1/ SR15284、SFLV1/SRLV14835、SF12/SRLV14835。共25 μL体系,PCR反应参数:95℃预变性3 min;95℃变性30 s,63℃(sg mRNA 7)退火30 s,72℃延伸30 s,共进行40个循环;72℃延伸10 min。取25 μL PCR产物在1.5%琼脂糖凝胶电泳中鉴定结果。
用DNA-free™ kit中的DNAase I(Ambion) 处理提取的细胞总RNA中残余的质粒DNA,具体处理方法参见产品说明书。再以处理好的细胞总RNA为模板,用引物SF12/SFLV101进行反转录,共20 μL体系,42℃恒温水浴1 h,最后冰浴静置2 min。
为了消除cDNA中残留RNA对后续试验的影响,每10 μL cDNA中加入1 μL RNAase A,置于37℃水浴30 min,随后95℃水浴灭活RNase A 10 min。
以处理好的cDNA为模板PCR扩增负链基因组((-) gRNA),第一轮PCR反应所用的4对引物:SF12/SR683、SFLV101/SR683、SFLV101/ SRLV572、SF12/SRLV572。将第一轮PCR的产物进行1:1000稀释,以此为模板进行巢式PCR反应,所用到的4对引物:SF12/SR343、SFLV101/SR343、SFLV101/SRLV378、SF12/SRLV378。
1.2.4 间接免疫荧光(immunofluorescence analysis,IFA) BHK-21细胞转染72 h后,弃去维持液培养基。用冰甲醇固定10 min,1% BSA室温封闭30 min,用1:600的PRRSV N蛋白的特异性单抗(美国South Dakota State University的Dr. Ying Fang惠赠)37℃孵育2 h,再加入Alexa Red标记羊抗鼠的二抗1: 800,37℃孵育1 h,PBS洗5遍后,在倒置荧光显微镜下观察结果,并拍照保存。
1.2.5 拯救病毒的生物学特性分析
1.2.5.1 空斑形态学分析 将本研究中所有的第3代(P3)拯救病毒用DMEM系列稀释后,取300 μL感染六孔板中的Marc-145细胞,37℃孵育1 h后,弃掉病毒液,加入等比例的2%低熔点琼脂糖与2×MEM,再加入4% FBS(终浓度为含2%FBS的MEM,含1%的琼脂糖),5 mL/孔铺到细胞板中。室温凝固后,于37℃培养箱倒置培养4~5 d,观察,待出现空斑后,在孔中加入2 mL 4%的甲醛溶液固定1 h,甩掉凝胶,用5%结晶紫溶液(溶于20%乙醇溶液)染色。
1.2.5.2 第一轮(first cycle)拯救病毒滴度测定 将本研究中所有的全长突变体按照1.2.2的转染方法转染80%密度的六孔板BHK-21细胞。待转染48 h后,收集细胞培养上清在Marc-145细胞上进行第一轮病毒TCID50[18]滴定。具体操作步骤如下:90%密度的Marc-145细胞在96孔板上培养,用2% FBS的EMEM进行10倍比稀释,每孔100 μL上清及上清倍比稀释液覆盖细胞单层,每个稀释度重复4孔,感染5 d后,根据每孔产生CPE的情况,按照计算公式,计算病毒滴度,用GraphPad软件绘出病毒滴度的柱状图。
1.2.5.3 多步生长曲线的绘制 将300 μL系列传代的P5代病毒上清(0.01MOI)接种于Marc-145细胞,37℃孵育1 h后,弃掉病毒液,每孔补充3 mL含有2%FBS的MEM培养基,置于37℃、5%CO2培养箱中,并在不同时间点(6、12、24、36、48、60、72、84、96、108、120 h)收集200 μL细胞上清,同时补充200 μL新的含2%FBS的MEM培养基。测定每个时间点的病毒滴度并以TCID50[18]表示,并根据计算出的病毒滴度用GraphPad软件绘制病毒的多步生长曲线。
1.2.6 RNA二级结构预测 利用mfold 网络服务器V2.3对PRRSV 5′UTR比对后形成的保守序列进行RNA二级结构的预测,服务器地址为 http:// frontend.bioinfo.rpi.edu/applications/mfold/cgi-bin/ rna-form1-2.3.cgi。使用服务器默认的参数(37℃, 1 mol/L Nacl,no divalent ions, and no limit on distance between paired bases),预测得到的二级结构用RNAviz 2.0 (http://rnaviz.sourceforge.net/)进行编辑。
2 结果
2.1 两型5'UTR互换嵌合突变全长克隆的构建与感染性检测基于北美型全长感染性克隆pAPRRS,在其ORF1的起始密码子和TRS之间通过PCR-based mutagenesis插入“at”形成Nde I以及“ttaa”形成Pac I构建的突变体pTLNd4以及pPa2,并在pTLNd4的基础上将5'UTR与欧洲型进行替换构建pTLV8突变体。同样的,在欧洲型全长感染性克隆pSHE的基础上,通过SOE PCR构建一个中间质粒pBzgSHEnde,由此中间质粒构建全长pSHEnde,最后将欧洲型5'UTR替换为北美型5'UTR,构建成嵌合中间克隆pBzgSPnde,最后再通过与pSHE的Xba I和Kas I的各自双酶切产物的连接构建成全长嵌合克隆pSHSP5。质粒酶切图谱见图1。对得到的全部全长突变体进行测序和Sma I酶切鉴定确定均构建成功,见图1。将所有突变体克隆及亲本全长克隆直接DNA转染BHK-21细胞,在转染24 h后收集上清传代于新鲜的Marc-145细胞,每天观察CPE情况。pTLNd4、pSHEnde、pTLV8及亲本克隆都能够产生CPE,值得注意的是,pTLV8是在Marc-145细胞上传代2次之后才产生CPE的;pPa2 和pSHSP5均无法拯救出活病毒,无法产生CPE,结果见图2。
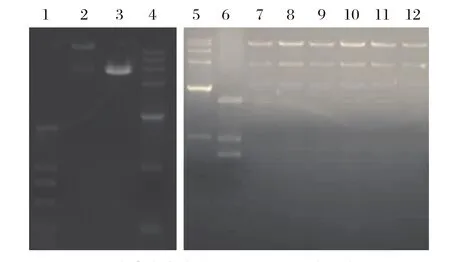
图1 突变嵌合克隆的构建及Sma I 酶切鉴定图谱Fig.1 Construction of chimeric mutants and identifi cation of full-length cDNA clone by Sma I digestion
1: DNA分子量标准(DL2000); 2: pSHE Xba I 和Kas I双酶切产物; 3: pBzgSPnde Xba I 和Kas I双酶切产物; 4: DNA分子量标准(DL15000); 5: DNA 分子量标准(DL15000);
6: DNA 分子量标准(DL2000); 7~12: pAPRRS、pTLNd4、pTLV8、pSHE、pSHEnde、pSHSP5 Sma I 酶切产物
1: DNA Marker(DL2000); 2: Product of pSHE digested by Xba I and Kas I; 3: Product of pBzgSPnde digested by Xba I and Kas I; 4: DNA Marker (DL15000); 5: DNA Marker (DL15000);
6: DNA Marker (DL2000); 7-12: Product of pAPRRS, pTLNd4, pTLV8, pSHE, pSHEnde, pSHSP5 digested by Sma I , respectively
2.2 突变对病毒结构蛋白表达的影响将等量上述突变病毒与亲本毒转染BHK-21细胞,用间接免疫荧光的方法检测核衣壳蛋白(Nucleocapsid protein, N)的表达。发现除pSHSP5之外所有的全长克隆转染孔中均能产生抗N蛋白的特异性荧光。pPa2转染孔视野中荧光量较少,如图3。将各自的BHK-21转染上清分别转移至Marc-145细胞中,发现pPa2和pSHSP5传代孔中无法检测到抗N蛋白的特异性荧光,其余病毒传代孔中都出现了大范围的抗N蛋白的特异性荧光。
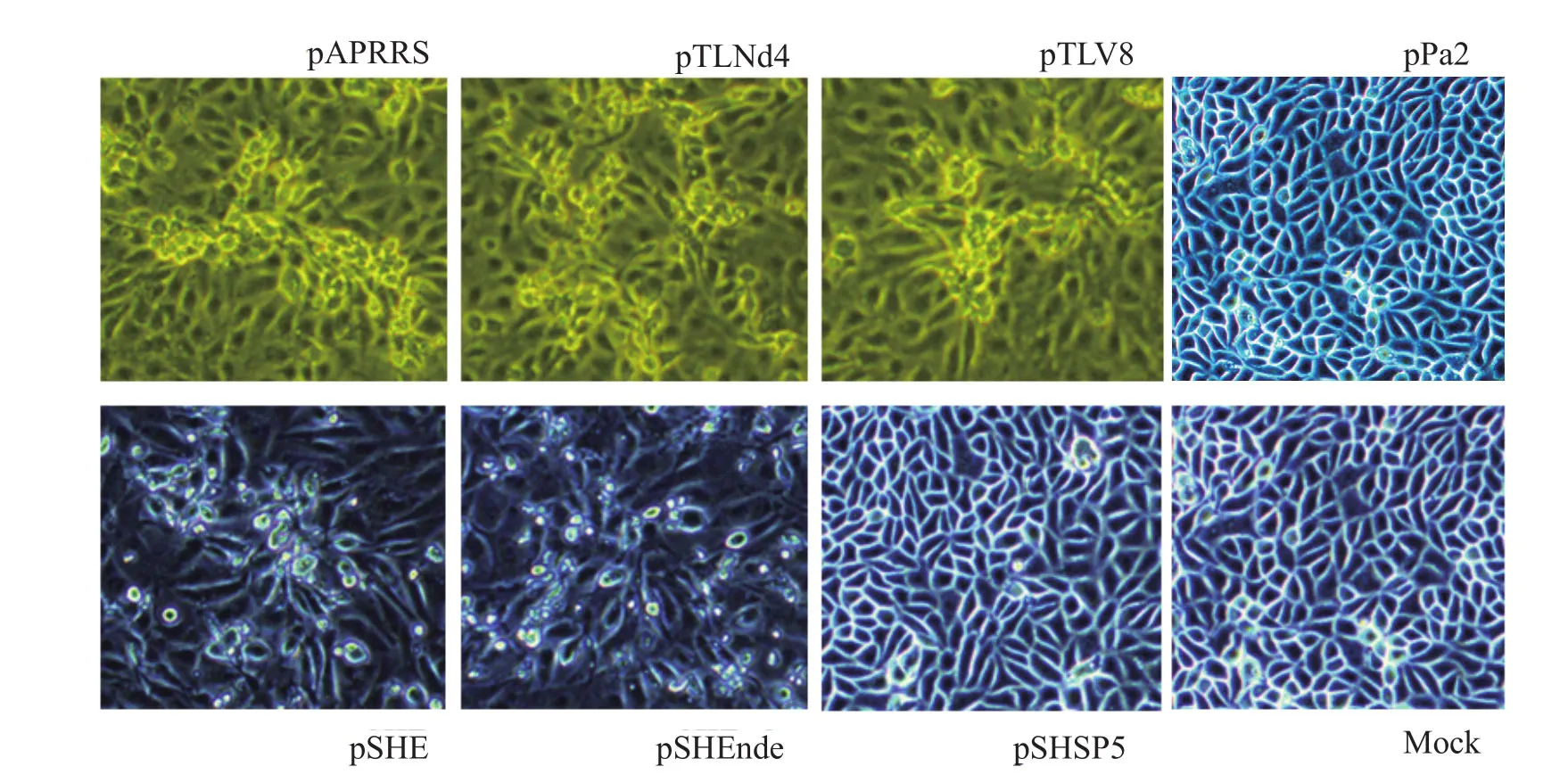
图2 突变全长cDNA克隆的感染性检测Fig.2 The infectious identifi cation for the mutant viruses and the parental viruses
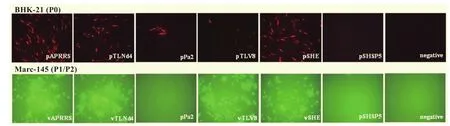
图3 嵌合突变病毒及其亲本病毒的间接免疫荧光(N蛋白特异性抗体)检测Fig.3 IFA detection (N-protein McAb) of the mutant viruses and the parental virus on BHK-21 cells and Marc-145 cells
2.3 突变对病毒基因组复制与亚基因组转录的影响分析将所有全长克隆转染BHK-21细胞,24 h后提取细胞总RNA,RT-PCR检测亚基因组及负链基因组,引物见表1,pAS为复制酶 (1693-13116) 缺失的阴性对照质粒。结果显示,所有的突变克隆均能检测到负链基因组的复制,其中嵌合克隆pTLV8和pSHSP5的负链合成水平很低;而所有的全长突变克隆除pSHSP5之外均能检测到sg mRNA 7的合成,但是pTLV8嵌合突变体sg mRNA 7的含量明显少于其他突变体,如图4所示。结果表明嵌合突变体较亲本野毒会降低或阻断亚基因组的有效合成;异源5' UTR的替换不会阻断负链基因组合成,但会使负链合成水平下降。
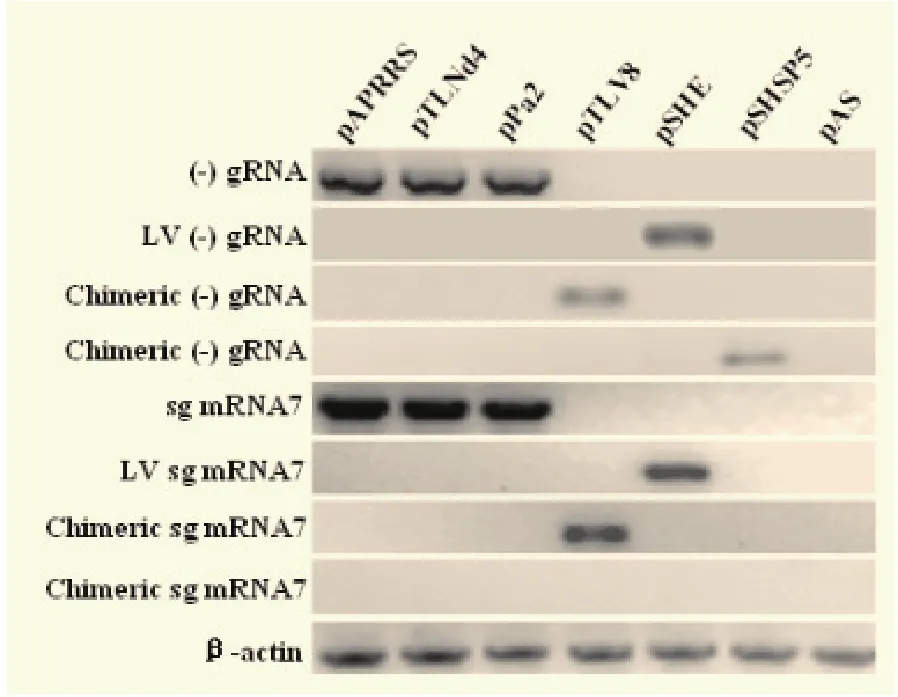
图4 突变对病毒的基因组复制与亚基因组转录的影响分析Fig.4 Analysis of genomic RNA replication and sg mRNA transcription for mutant viruses and WT
2.4 突变病毒与野毒在病毒生物学水平的比较对突变病毒和野毒进行第一轮拯救病毒的滴定,结果如图5所示,从中可见在BHK-21细胞上转染的第一轮亲本毒vAPRRS是1×103.67TCID50/mL ,而vTLV8在P1代不产生CPE,无法拯救出病毒,因此滴度为0。另外一组vSHE的病毒滴度为1×104TCID50/mL, vMSHEnde的病毒滴度与亲本比较差异不显著,为1×103.67TCID50/mL。同时对这些病毒(选取P5代毒)进行空斑形态学比较(图6),结果表明这些突变病毒在空斑形态、数目上均与亲本病毒无显著差异。结合多步生长曲线结果(图7)证明了突变嵌合病毒与亲本病毒在病毒学水平差异不显著。
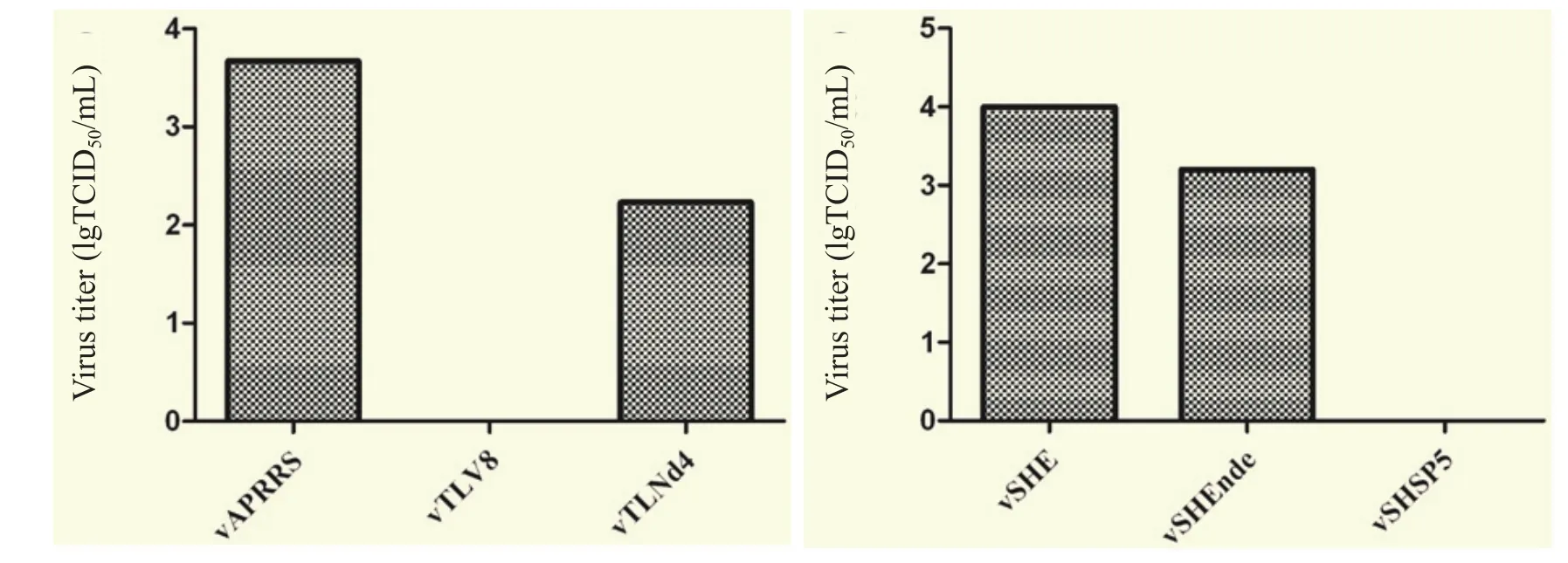
图5 突变病毒的第一轮拯救病毒浓度滴定Fig.5 The fi rst cycle viral titration the mutant viruses
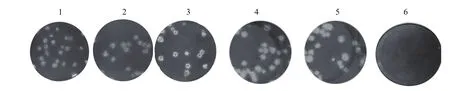
图6 突变病毒的空斑形态学比较Fig.6 Plaque morphology comparison for the mutant viruses1: vAPRRS; 2: vTLNd4; 3: vTLV8; 4: vSHE; 5: vSHEnde; 6: Mock1: vAPRRS; 2: vTLNd4; 3: vTLV8; 4: vSHE; 5: vSHEnde; 6: Mock
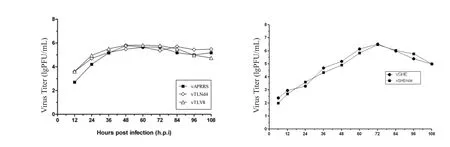
图7 突变病毒与亲本病毒的多步生长曲线Fig.7 The multi-step growth curves of the mutant viruses and the parental viruses
2.5 嵌合突变体二级结构预测对pSHSP5以及pTLV8的二级结构预测中发现(图8),嵌合突变体pTLV8的5' 端虽然与亲本pAPRRS的二级结构差异很大,但保留了TRS所在的顶端茎环(C-SL5)结构,且TRS与ORF1的AUG均位于C-SL5上。而pSHSP5的顶端C-SL5则遭到了破坏,TRS不再位于相应环上。由此推测TRS和ORF1的起始密码子“AUG”所在的顶端茎环结构是调控病毒复制转录过程的关键顺式作用元件之一,对其破坏的突变都是致死性突变。
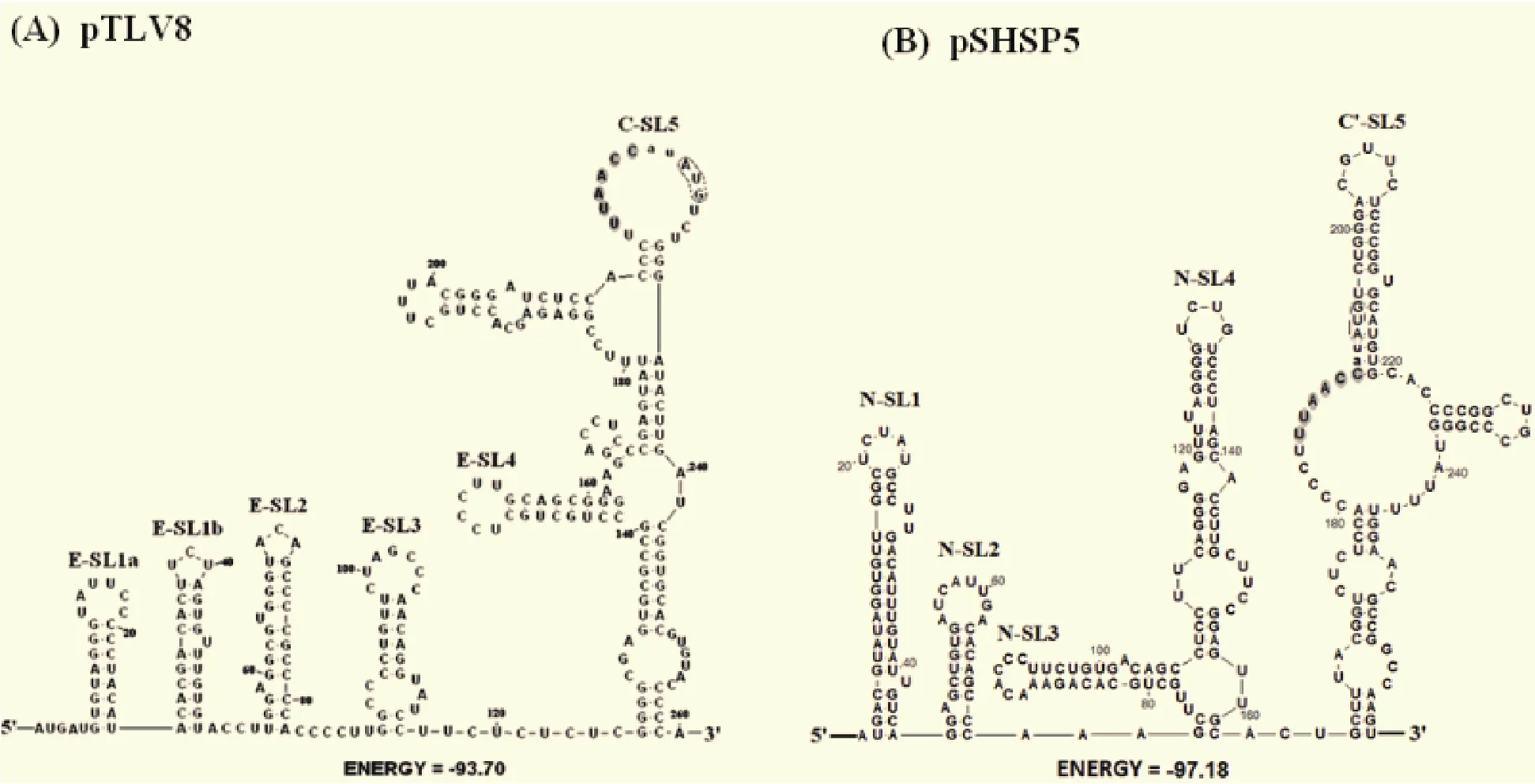
图8 嵌合全长克隆的二级结构预测Fig.8 The secondary structure prediction for the 5' proximal sequences of chimeric full length cDNA clones
3 讨论
PRRSV有两个血清型,欧洲型(1型)和北美型(2型),二者序列同源性只有约60%,而其基因组成是一致的[12]。目前PRRSV仍处于通过随机点突变及基因型之间的RNA重组快速进化过程中。
目前已证实许多单股正链RNA病毒的5' UTR包含了病毒基因组复制及亚基因组mRNA转录所需要的顺式作用元件[2-4,6]。在马动脉炎病毒(Equinearteritisvirus,EAV)亚基因组mRNA转录过程中,位于5' UTR 中的顶端茎环结构上的Leader-TRS(转录调控序列)以及位于下游基因组中Body-TRS的互补配对介导了此转录过程[7]。这个茎环结构同时发现于其他动脉炎病毒基因组的5' 端,也存在于许多冠状病毒中[8]。在本研究中,利用Mfold RNA二级结构软件,基于能量最低原则,对两种血清型PRRSV 5'端序列的二级结构进行预测分析发现,虽然其一级序列同源性只有60%,但是茎环结构呈现相当程度的相似性,尤其是顶端茎环结构。与动脉炎病毒的原型病毒EAV的相应结构比较发现,EAV的5' 端茎环结构较多,单链区较PRRSV的更多,但是顶端的茎环结构在PRRSV中则更突出。在病毒RNA合成的模板交换(template switching)过程中,顶端茎环结构被认为能够为Leader TRS作为一个接头提供便利,EAV此结构较PRRSV大。虽然二级结构预测并不能保证有100%的正确性,但是有研究已经证明用酶法和化学法检测的真实的RNA结构与mfold软件预测的二级结构有极高的一致性。
在本研究中,对嵌合PRRSV 5' 端260 bp的序列进行二级结构预测,发现pTLV8和pSHSP5之间以及其与各自母本相比差异都很大,但是pTLV8的Leader TRS仍然位于顶端的茎环结构上,pSHSP5的顶端茎环结构则完全遭到了破坏。对突变嵌合病毒的感染性试验等发现pTLV8在传代2次之后能够产生CPE,而pSHSP5则是一个致死性突变体。而为了便于构建两型5'UTR嵌合的全长突变体,在TRS与ORF1的起始密码子“AUG”之间插入“at”形成Nde I酶切位点的两个突变体pSHEnde和pTLNd4,二者顶端茎环结构没有改变,转染后具有感染性。综上所述,PRRSV 5' 末端顶端的茎环结构在病毒生命活动中发挥着关键的调控作用。本研究证明Leader TRS所在的茎环结构以及TRS和ORF1起始密码子AUG的相对位置是调控病毒复制转录过程的关键顺式作用元件之一,任何对其结构破坏的突变都会成为致死性突变。
本研究首次报道了PPRSV 1型5'UTR在2型的基因组骨架上能完全发挥调控作用,在功能上可以替换,并对Leader TRS所在的茎环结构进行初步解析,为进一步剖析PRRSV 5' UTR的结构与功能的关系,及病毒致病机理等提供了科学依据。
[1] Gallie D R. A tale of two termini: a functional interaction between the termini of an mRNA is a prerequisite for efficient translation initiation[J]. Gene, 1998, 216(1): 1-11.
[2] Barton D J, O'Donnell B J, Flanegan J B. 5' cloverleaf in poliovirus RNA is a cis-acting replication element required for negative-strand synthesis[J]. Embo J, 2001, 20(6): 1439-1448.
[3] Filomatori C V, Lodeiro M F, Alvarez D E, et al. A 5' RNA element promotes dengue virus RNA synthesis on a circular genome[J]. Genes Dev, 2006, 20(16): 2238-2249.
[4] Lodeiro M F, Filomatori C V, Gamarnik A V. Structural and functional studies of the promoter element for dengue virus RNA replication[J]. J Virol, 2009,83(2): 993-1008.
[5] Andino R, Rieckhof G E, Baltimore D. A functional ribonucleoprotein complex forms around the 5' end of poliovirus RNA[J]. Cell, 1990, 63(2): 369-380.
[6] Herold J, Andino R. Poliovirus requires a precise 5' end for efficient positive-strand RNA synthesis[J]. J Virol, 2000, 74(14): 6394-6400.
[7] Van Den Born E, Gultyaev A P, Snijder E J. Secondary structure and function of the 5'-proximal region of the equine arteritis virus RNA genome[J]. RNA, 2004, 10(3): 424-437.
[8] Van Den Born E, Posthuma C C, Gultyaev A P, et al. Discontinuous subgenomic RNA synthesis in arteriviruses is guided by an RNA hairpin structure located in the genomic leader region[J]. J Virol, 2005, 79(10): 6312-6324.
[9] Dumas E, Staedel C, Colombat M, et al. A promoter activity is present in the DNA sequence corresponding to the hepatitis C virus 5' UTR[J]. Nucleic Acids Res, 2003, 31(4): 1275-1281.
[10] Rijnbrand R, Abell G, Lemon S M. Mutational analysis of the GB virus B internal ribosome entry site[J]. J Virol, 2000, 74(2): 773-783.
[11] Rijnbrand R, van der Straaten T, van Rijn P A, et al. Internal entry of ribosomes is directed by the 5' noncoding region of classical swine fever virus and is dependent on the presence of an RNA pseudoknot upstream of the initiation codon[J]. J Virol, 1997, 71(1): 451-457.
[12] Nelsen C J, Murtaugh M P, Faaberg K S. Porcine reproductive and respiratory syndrome virus comparison: divergent evolution on two continents[J]. J Virol, 1999, 73(1): 270-280.
[13] Wang Y, Liang Y, Han J, et al. Attenuation of porcine reproductive and respiratory syndrome virus strain MN184 using chimeric construction with vaccine sequence[J]. Virology, 2008, 371(2): 418-429.
[14] Cho J G, Dee S A. Porcine reproductive and respiratory syndrome virus[J]. Theriogenology, 2006, 66(3): 655-662.
[15] Zhou Y, Zheng H, Gao F, et al. Mutational analysis of the SDD sequence motif of a PRRSV RNA-dependent RNA polymerase[J]. Sci China Life Sci, 2011, 54(9): 870-879.
[16] Yuan S, Wei Z. Construction of infectious cDNA clones of PRRSV: separation of coding regions for nonstructural and structural proteins[J]. Sci China C Life Sci, 2008, 51(3): 271-279.
[17] 庄金山, 袁世山, 张建武. 欧洲型PRRSV解毒株全长基因cDNA克隆的构建与分析[J]. 中国兽医科学, 2008, (8): 658-664.
[18] Zheng H, Sun Z, Zhu X Q, et al. Recombinant PRRSV expressing porcine circovirus sequence reveals novel aspect of transcriptional control of porcine arterivirus[J].Virus Res, 2010, 148(1-2): 8-16.
[19] Pizzi M. Sampling variation of the fifty percent endpoint, determined by the Reed-Muench (Behrens) method[J]. Hum Biol, 1950, 22(3): 151-190.
[20] Warter L, Cohen L, Benureau Y, et al. A cooperative interaction between nontranslated RNA sequences and NS5A protein promotes in vivo fitness of a chimeric hepatitis C/GB virus B[J]. PLoS One, 2009, 4(2): e4419.
[21] Kang H, Feng M, Schroeder M E, et al. Putative cisacting stem-loops in the 5' untranslated region of the severe acute respiratory syndrome coronavirus can substitute for their mouse hepatitis virus counterparts[J]. J Virol, 2006, 80(21): 10600-10614.
A PROMINENT STEM-LOOP STRUCTURE IN 5' UNTRANSLATED REGION OF PORCINE REPRODUCTIVE AND RESPIRATORY SYNDROME VIRUS IS ESSENTIAL FOR VIRAL REPLICATION AND TRANSCRIPTION
GAO Fei1, ZHENG Hao1, JIANG Yi-feng1, LI Li-wei1, ZHENG Hai-hong1, ZHOU Yan-jun1, WEI Zu-zhang2, TONG Guang-zhi1
(1. Shanghai Veterinary Research Institute, CAAS, Shanghai 200241, China; 2. College of Animal Science and Technology, Guangxi University, Nanning 530005, China )
The 5' untranslated region (UTR) of the genomic RNA is believed to be vital for the replication and transcription of Porcine reproductive and respiratory syndrome virus (PRRSV), yet its functional mechanism remains unclear. Based on infectious full-length cDNA clones pAPRRS and pSHE of type 1 and type 2 PRRSV, exogenous “at” was inserted between 5' UTR and ORF1 to form Nde I digestion site, then pTLNd4 and pSHEnde were obtained. After transfection and passage on cell monolayers, the infectiousness of pTLNd4 and pSHEnde was demonstrated. Afterwards, the 5' UTR chimeric mutants pTLV8 and pSHSP5 were obtained. Interestingly,pTLV8 developed cytopathic effect (CPE) on MARC-145 cells while pSHSP5 was a lethal mutant. Comparison of the secondary structures of 5' proximal sequences revealed that a signifi cant change occurred in pSHSP5 but not in pTLV8. The prominent stem-loop structure remained functional in pTLV8 as TRS and AUG of ORF1 were located in the top loop. Taken together, the prominent stem-loop structure where TRS is located is one of the crucial cis-acting elements in regulating viral replication and transcription. Therefore, any changes that alter the stem-loop structure would be lethal for mutant viruses.
Porcine reproductive and respiratory syndrome virus (PRRSV); 5' untranslated region (5'UTR); chimeric virus; transcriptionregulating sequence (TRS); secondary structure
S852.659.6
A
1674-6422(2014)04-0007-10
2014-03-24
国家自然科学基金(31300140);863计划(2011AA10A208-2);科研院所基本科研业务费(2013JB01,2013JB02)
高飞,女,博士,助理研究员,主要从事分子病毒学研究
童光志,E-mail: gztong@shvri.ac.cn

