云南胡椒花叶病病原检测及亚组鉴定
刘向阳, 刘爱勤, 周 琳
(1.河南农业大学植物保护学院,河南 郑州 450002; 2.河南省新型农药创制与应用重点实验室,河南 郑州 450002; 3.中国热带农业科学院香料饮料研究所,海南 万宁 571533)
胡椒(PipernigrumL.)原产印度,现广泛栽培于热带国家和地区[1].中国于1947年对胡椒进行引种,目前种植面积和胡椒产量世界排名分别为第6位和第5位[2].对于包括中国在内的很多东南亚国家来说,胡椒是用来赚取外汇的最重要的香辛料之一[3].胡椒的种植面积虽然在近些年来持续增长,但其产量和质量常受到生物或非生物因素的影响而大幅降低.在影响胡椒生产的生物因素中,胡椒病毒病作为胡椒重要病害之一,危害日益突出,严重地区发病率高达80%以上[4].胡椒病毒病在世界许多胡椒产区都有发生,包括中国云南、海南、广东、广西及福建等产区.侵染胡椒的病毒主要有2种,分别为胡椒黄斑病毒(Piperyellow mottle virus, PYMV)和黄瓜花叶病毒(Cucumber mosaic virus, CMV)[5, 6].PYMV和CMV是不同属的病毒,但二者侵染胡椒后引起的胡椒感病症状极为相似,均表现为花叶症状,即感病胡椒叶脉清亮透明,叶片出现黄色斑点、变小、扭曲等[7~9].因此,仅从感病症状难以辨别胡椒花叶病的病原.牛立霞等[6]通过巢式RT-PCR方法鉴定了海南胡椒花叶病的病原为CMV,中国的其他产区胡椒花叶病的病原还有待鉴定.云南是中国胡椒主产区之一,该产区内胡椒花叶病时有发生.本研究以云南感染花叶病的胡椒为对象,以海南感病胡椒为参照,利用双抗夹心酶联免疫吸附法(DAS-ELISA)对致病病毒进行鉴定,利用反转录-聚合酶链式反应(RT-PCR)对CMV外壳蛋白(Coat protein, CP)基因进行扩增和克隆,并依据CMVcp核酸序列对CMV进行亚组鉴定.该研究旨在为胡椒病毒病的诊断和综合防治以及转基因抗病毒胡椒品系的选育奠定基础.
1 材料与方法
1.1感病材料
典型感病的胡椒叶片(叶色斑驳,形似马赛克,叶片变小、皱缩、卷曲、畸形)分别采自云南德宏和海南万宁的胡椒种植园,置于-20 ℃冰箱备用.
1.2方法
1.2.1 DAS-ELISA检测 样品分为4类,分别为样品1(无病健康胡椒叶片,阴性对照)、样品2(感病黄瓜叶片,阳性对照)、样品3(海南胡椒病叶,阳性对照)和样品4(云南胡椒病叶).样品用洗涤缓冲液(PBST,pH 7.4,NaCl 8.0 g,Na2HPO4·12H2O 2.9 g,KH2PO40.2 g,KCl 0.2 g)+2%聚乙烯基吡咯烷酮(PVP)溶液(体积比为1∶5)研磨.参照Agdia公司CMV检测试剂盒(CMV DAS-ELISA Kit)操作说明书进行DAS-ELISA检测.
利用样本与阴性对照吸光度的比值作为判断标准:阳性:I/H≥ 2;阴性:I/H< 2;I/H=(样品的平均吸光值-空白对照平均吸光值)/(阴性对照的平均吸光值-空白对照平均吸光值)
1.2.2 胡椒叶片总RNA的提取 分别称取100 mg采自云南和海南的胡椒病叶,用液氮研磨成粉,转入1.5 mLRNase-free离心管中,然后按照E.Z.N.A.®植物总RNA提取试剂盒(E.Z.N.A.®Plant RNA Kit)操作说明书完成后续步骤,提取的胡椒病叶总RNA置-20 ℃冰箱备用.
1.2.3 CMVcp单链cDNA的合成 以上述提取的胡椒病叶总RNA为模板,参照TaKaRa公司的AMV逆转录酶操作说明,合成CMVcp第1链cDNA.
1.2.4 CMVcp基因的克隆和测序 根据CMV亚组I外壳蛋白基因序列,经Primer Premier 6.0软件(加拿大Premier公司)设计1对引物(Cp-F和Cp-R),具体序列见表1.

表1 试验所用引物
分别取10×Buffer 5 μL、Cp-F和Cp-R引物(10 mmol·L-1)各1 μL,dNTPs(10 mmol·L-1)1 μL,Taq聚合酶1 μL,cDNA模板0.4 μL于200 μL离心管中,用双蒸水补至50 μL.设定PCR程序为:94 ℃预变性1 min,94 ℃变性45 s,55 ℃退火45 s,72 ℃延伸1 min,35个循环,72 ℃保温10 min.PCR产物经琼脂糖凝胶验证后,用E.Z.N.A.TM胶回收试剂盒(E.Z.N.A.TMGel Extraction Kit)进行回收.
将纯化回收的PCR产物与pMD18-T simple载体于4 ℃过夜连接,转化大肠杆菌DH5α,涂布含氨苄青霉素(100 g·L-1)的LB固体培养基平板上,37 ℃过夜培养,挑取白色单菌落接种于含氨苄青霉素的LB液体培养基中,37 ℃震荡培养(250 r·min-1)14 h,取少量菌液提质粒,质粒酶切鉴定后委托上海英骏(Invitrogen)生物技术有限公司测序.
1.2.5 CMVcp基因的序列分析 序列的分子量用DNAstar V5.0.6预测.同源序列搜寻在NCBI网站(http://www.ncbi.nlm.nih.gov)通过BLAST程序进行.用Clustal X 2.0[10]对搜索到的CMVcp基因同源序列(表2)进行双重和多重比对,随后用MEGA 4[11]以N-J法构建进化树,进行1 000次重复,分析序列间可能的亲缘关系.

表2 用作比对的CMV cp基因序列
2 结果与分析
2.1胡椒花叶病病原检测
DAS-ELISA检测结果表明,采自海南的胡椒病叶(样品3)和采自云南的胡椒病叶(样品4)提取液与CMV抗血清反应均呈阳性,二者的I/H值分别为3.04和3.48(表3),均大于2.0,因此可以认定,与海南胡椒花叶病病原一样,云南胡椒花叶病的病原也为CMV.将云南和海南两地侵染胡椒的CMV分别命名为云南胡椒分离物(CMV isolated from Yunnan provinces, CMV-YNP)和海南胡椒分离物(CMV isolated from Hainan provinces, CMV-HNP).
2.2CMVcp基因RT-PCR扩增及克隆
分别以Cp-F和Cp-R为正反引物,扩增CMV-YNPcp和CMV-HNPcp基因.0.8%琼脂糖凝胶电泳检测结果显示,两地胡椒病样的扩增产物均在约660 bp处出现1条明显的DNA条带,大小与已知CMVcp基因相对分子质量相当(图1),初步认定二者为CMVcp基因.分别将CMV-YNPcp和CMV-HNPcp基因条带回收,连接到pMD 18-T simple载体,命名为pT-CPYNP和pT-CPHNP.
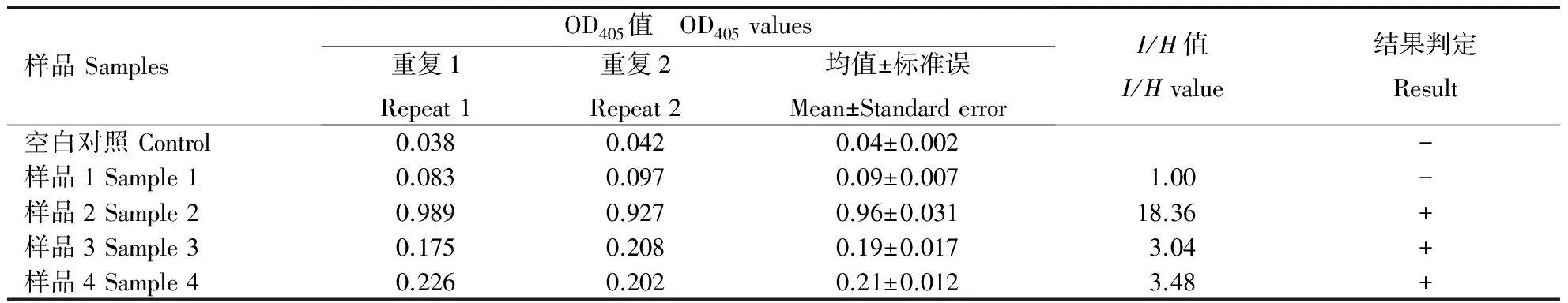
表3 DAS-ELISA法测定结果

M.DNA质量分子标准;1, 3.健康样品(阴性对照);2.海南染病样品;4.云南染病样品.
2.3CMVcp基因的序列分析
分别取少量经酶切验证的pT-CPYNP和pT-CPHNP菌液送上海英骏生物技术有限公司测序,测序结果经NCBI网站Blast(http://blast.ncbi.nlm.nih.gov/)在线分析证实二者确为CMVcp基因.序列一致性分析表明,CMV-YNPcp与从云南省南瓜感病植株中分离的CMV-YNcp基因(GenBank登录号:DQ459482)核酸序列相似性最高,为97%;CMV-HNPcp则分别与从海南省感病胡椒中分离的CMV-Chinacp基因(GenBank登录号:EU926956)和从海南省感病鸡蛋果中分离的CMV-Pfcp基因(GenBank登录号:AF368192)核酸序列相似性最高,均为98%.CMV-YNP与CMV-HNP虽然寄主同为胡椒,但二者的cp基因核酸序列相似性并不高,仅为93%.CMV-HNPcp和与从印度卡纳塔克邦感病胡椒中分离的CMVcp基因(GenBank登录号:AY545924)核算序列相似性为92%,CMV-YNPcp与该序列的相似性稍高,但也仅为94%.CMV-YNPcp和CMV-HNPcp基因全长657 bp,编码218个氨基酸(图2和图3).将CMV-YNPcp和CMV-HNPcp基因核酸序列提交到NCBI,获得GenBank登录号分别为JQ692129和JQ895557.
BLAST搜索结果表明,与CMV-YNP和CMV-HNP序列相似性程度高的CMV多来自中国,其次是距离中国较近的日本.为了确定CMV-YNP和CMV-HNP的亚组归属,以具有代表性的CMVcp基因(表2)作为参照,选择N-J法构建了29个CMV株系或分离物的进化树(图4).系统进化树显示,CMV株系或分离物明显分为2个亚组,即亚组I和亚组II,而亚组I又进一步分为亚组IA和IB.CMV-HNP(图4,星号所指)、CMV-YNP(图4,箭头所指)与从印度卡纳塔克邦感病胡椒中分离的CMVcp基因(In-Pn,图4)均归属于CMV亚组IB,但是这3个侵染胡椒的CMV在系统进化树中并没有聚在一起.CMV-YNP与其它6个CMV分离物聚为1簇,在这6个与CMV-YNP进化关系最接近的CMV分离物中,4个来自中国,另外2个分别来自韩国和保加利亚.同为从胡椒寄主中分离的CMV-HNP没能和其它28个CMV株系或分离物中的任何1个聚为1簇,而是独自形成1个分支(图4).

图2 CMV-YNP cp核酸和氨基酸序列

图3 CMV-HNP cp核酸和氨基酸序列
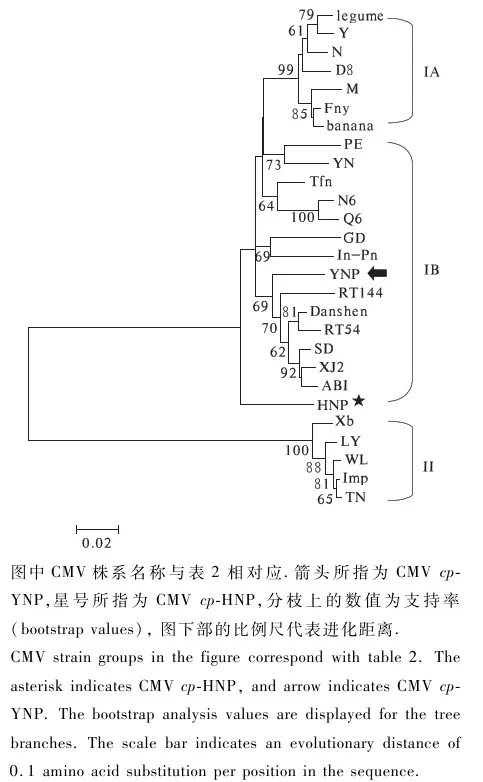
图中CMV株系名称与表2相对应.箭头所指为CMV cp-YNP,星号所指为CMV cp-HNP,分枝上的数值为支持率(bootstrap values), 图下部的比例尺代表进化距离.
3 讨论
CMV和PYMV亲缘关系较远,本是不同属的病毒,但二者侵染胡椒后,均可以引起感病胡椒表现出花叶症状,仅从感病胡椒表现的症状难以确定病原,因此为胡椒花叶病的防治带来了困难.CMV是直径为27~30 nm的球形RNA病毒,PYMV是无包膜的杆状病毒(30 nm×120 nm),具有双链DNA[5, 8],在电镜下较容易将二者区分开来.本研究首先采用DAS-ELISA法,检测到云南和海南感病胡椒叶片中的病毒为CMV,随后根据CMVcp基因末端保守序列设计引物,通过PCR从病叶总RNA中克隆出cp基因,并依据测得的CMVcp基因序列对侵染胡椒的CMV进行亚组鉴定.
本研究通过双抗夹心酶联免疫吸附法鉴定了云南胡椒花叶病的病原为CMV,为胡椒花叶病的防治提供了依据.CMV具有典型的三分体(RNA 1,RNA 2和RNA 3)单链正义RNA病毒的特点,其中RNA 3编码2个蛋白质,即3a运动蛋白和外壳蛋白.根据血清学特征及核酸序列的相似性,CMV可分为2个亚组,即亚组I和亚组II,随着研究的深入,CMV亚组I又进一步分为亚组IA和IB[12].作为RNA病毒,CMV较易发生变异,特别是RNA 3,最易发生重组变异[13].CMVcp基因序列比较表明,CMV-YNP与CMV-HNP虽然寄主同为胡椒,但二者的cp基因核酸序列差异较大,一致性仅为93%;CMV-YNPcp与从印度卡纳塔克邦感病胡椒中分离的CMVcp基因的相似性稍高,但也仅为94%,同源性较低.该部分试验从分子水平上证实了CMV-YNP与上述2个CMV胡椒分离物属于不同的株系.
从寄主上看,CMV-YNP、CMV-HNP和In-Pn 3个CMV株系均是从胡椒中分离,即便CMV-HNP、CMV-YNP和In-Pn在系统进化树中同属CMV IB亚组,但三者并没有聚在一簇,而是各自分开,特别是CMV-HNP,独自形成一个小的分支(图4),这或许表明寄主并非CMV发生、进化、变异的唯一决定因素.序列比对显示,与CMV-YNPcp基因核酸序列相似性最高的CMV株系并不是分离于同样寄主的胡椒,而是分离于相近地域(云南昆明)感病南瓜,这或许预示CMV株系分化、变异具有地域性,而感染胡椒的CMV或许来自于当地的其它感病物种.
胡椒花叶病的危害日趋严重,但目前仍然没有理想药剂能对其进行直接有效地防治.此外,国内外尚未筛选出对胡椒花叶病具有抗性的胡椒品系,所以对胡椒花叶病的防治措施仅限于清除感病植株和加强检疫以防止其扩散.CMV不仅是胡椒花叶病的病原,而且是番茄病毒病的主要病原之一.已有报道表明,将CMVcp基因导入番茄基因组中进行表达,可以有效防治CMV对番茄的侵染[14].本研究在鉴定了胡椒花叶病的病原为CMV的基础上,进行了cp基因的克隆,从而为获得抗CMV的转cp基因胡椒植株奠定了基础.
参考文献:
[1]LIM T K. Edible medicinal and non-medicinal plants [M]. Netherland: Springer Press, 2012.
[2]杨建峰, 邬华松, 孙 燕, 等. 我国胡椒产业现状及发展对策[J]. 热带农业科学, 2010, 30(3): 52-55.
[3]SIJU S, MADHUBALA R, BHAT A I. Sodium sulphite enhances RNA isolation and sensitivity of cucumber mosaic virus detection by RT-PCR in black pepper[J]. Journal of Virological Methods, 2007, 141(1): 107-110.
[4]王 赟, 李晓娜, 李增平, 等. 胡椒主要病害及其防治方法[J]. 河北农业科学, 2009, 13(7): 30-32.
[5]LOCKHART B E L, KIRATIYA-ANGUL K, JONES P, et al. Identification of Piper yellow mottle virus, a mealybug-transmitted badnavirus infectingPiperspp. in Southeast Asia[J]. European Journal of Plant Pathology, 1997, 103: 303-311.
[6]牛立霞, 王健华, 冯团诚, 等.海南胡椒中黄瓜花叶病毒分离物的分子鉴定[J]. 热带作物学报, 2008, 29(4): 510-513.
[7]BHAT A I, SIJU S. Development of a single-tube multiplex RT-PCR for the simultaneous detection ofPiperyellow mottle virus and cucumber mosaic virus associated with stunt disease of black pepper[J]. Current Science, 2007, 93(7): 973-976.
[8]SILVA D P P, JONES P, SHAW M W. Identification and transmission ofPiperyellow mottle virus and cucumber mosaic virus infecting black pepper (Pipernigrum) in Sri Lanka[J]. Plant Pathology, 2002, 51(5): 537-545.
[9]SARMA Y R, KIRANMAI G, SREENIVASULU P, et al. Partial characterization and identification of a virus associated with stunt disease of black pepper (Pipernigrum) in south India[J]. Currrent Science, 2001, 80(3): 459-462.
[10]THOMPSON J D, GIBSON T J, PLEWNIAK F, et al. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 1997, 25(24): 4876-4882.
[11]TAMURA K, DUDLEY J, NEI M, et al. MEGA4: Molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24(8): 1596-1599.
[12]PALUKAITIS P, GARCA-ARENAL F. Cucumo viruses[J]. Advances in Virus Research, 2003, 62: 241-323.
[13]BONNET J, FRAILE A, SACRISTN S, et al. Role of recombination in the evolution of natural populations of cucumber mosaic virus, a tripartite RNA plant virus[J]. Virology, 2005, 332(1): 359-368.
[14]LIN C H, SHEU F, LIN H T, et al. Allergenicity assessment of genetically modified cucumber mosaic virus (CMV) resistant tomato (Solanumlycopersicon)[J]. Journal of Agricultural and Food Chemistry, 2010, 58(4): 2302-2306.

