广州地区2012年1、2型登革病毒基因型特征研究
白志军,曹毅敏,曹 庆,杨智聪,狄 飚
登革病毒(dengue virus, DENV)是流行于热带和亚热带地区的一种虫媒病毒,通过埃及伊蚊和白纹伊蚊传播。自然界存在的登革病毒有1、2、3、4血清型(DENV 1、DENV 2、DENV 3、DENV 4), 可引起登革热(dengue fever,DF)、登革出血热(dengue hemorrhagic fever,DHF)和登革休克综合征(dengue shock syndrome, DSS)等不同类型的急性传染病[1-5]。 DENV为单股正链RNA病毒,E基因是病毒的主要包膜蛋白,E蛋白在病毒与宿主细胞相互作用过程中发挥着重要的功能,E基因也常用于病毒基因进化分析研究中[6-7]。
本文对2012年广州地区分离获得DENV 1、DENV 2型8株,RT-PCR扩增毒株的E基因,同时针对E基因序列,与基因库中已经公布的序列进行比对和进化分析,以期了解其序列特征与可能的传播来源。
1 材料与方法
1.1试剂 RNA 提取试剂盒、DNA凝胶回收试剂盒均购自德国Qiagen公司、一步法RT-PCR试剂盒购自大连宝生物科技有限公司。
1.2引物设计与合成 参照方美玉等[8]设计的序列合成DENV 1、DENV 2特异性引物;参考DENV l新加坡S275/90(GenBank登录号:M87512)株基因组序列,DENV 2牙买加1409(GenBank登录号:M20558)株基因组序列,用Premier Primer5.0引物设计软件分别设计合成DENV 1和DENV 2的E基因扩增引物。引物由上海英骏生物技术有限公司合成。
1.3病毒培养及RNA提取 收集478例2012年广州市登革病毒抗体监测阳性患者的急性期血清,用RPMI 1640培养基1∶10 稀释后,取100 μL接种到25 cm2细胞瓶培养的单层C6/36白蚊伊蚊传代细胞中, 吸附2 h后弃上清液,加入含体积分数为0.1的小牛血清的RPMI 1640培养基,29 ℃培养箱中培养,7 d后传代,连续传代3次,取细胞培养上清液140 μL,用RNA 提取试剂盒提取RNA。以出现细胞病变,或细胞培养上清液经登革特异性引物PCR扩增,有扩增产物者则判断为阳性,连续传代3次均无细胞病变且经过登革特异性引物PCR扩增,无扩增产物者,则判断为阴性。
1.4RT-PCR扩增 用一步法RT-PCR试剂盒进行RT-PCR,反应条件,50 ℃ 30 min;94 ℃ 2 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 3 min,35个循环;72 ℃ 7 min。用Tris-硼酸(TBE)缓冲液配制含0.5 mg/L溴化乙锭的质量浓度为0.015 kg/L的琼脂糖凝胶,取5 μL PCR产物,与1 μL上样缓冲液混和,在TBE缓冲液中以80伏恒压电泳30 min,在透射紫外分析仪上观察,以出现相应的特异性条带为阳性结果,用DNA凝胶回收试剂盒切胶回收。
1.5序列测定和拼接 纯化后的PCR产物由上海英骏生物技术有限公司进行序列测定。测定后的各片段序列采用Seqman程序( DNAstar 分析软件之一)进行拼接并组装成完整的E基因序列,经过核对校正后上传至GenBank。
1.6基因序列的同源性和进化分析 应用MegAlign程序( DNAstar分析软件之一)将分离的2012年广州DENV 1、DENV 2流行株E基因序列与GenBank中我国近30年来本土流行DENV 1和DENV 2的毒株进行序列碱基和氨基酸的同源性比对;应用DANMAN version 6基因分析软件,将以上流行株序列与其他作为参考的各亚型病毒株E基因(均源自GenBank)进行序列多重比对(multiple alignment),采用Kimura校正模型的领接法(neighbor-joining,NJ)构建系统发育树。
2 结 果
2.1C6/36细胞分离培养病毒 478份血清标本,用C6/36细胞进行分离培养,其中49份标本均出现细胞聚堆等细胞病变现象,见图1。

图1C6/36细胞病变鉴定(100×)
Fig.1IdentificationofC6/36cellcytopathiceffect(Originalmagnification:100×)
2.2登革病毒E基因的RT-PCR扩增 细胞病变阳性的培养上清液,提取病毒RNA,用DENV 1、DENV 2型特异性引物进行RT-PCR, 8份培养上清液可扩增特异性产物。继以扩增E基因,发现8份培养上清液均可扩增出特异性的产物,各片段长度均与预期相符,对E基因PCR产物进行纯化回收供测序使用。8株病毒感染病例资料见表2。
2.3序列测定、基因特征 所有片段经双向测序反应均可获得一致的碱基序列,应用Seqman程序进行拼接并组装成完整的E基因序列。6株DENV 1和2株DENV 2的E基因均由1485个碱基组成,编码495个氨基酸,序列经分析均已上传至GenBank。
2.4E基因序列的同源性分析 DENV 1和DENV 2碱基(氨基酸)序列同源性分别为90.2%~99.9%(95.8%~100.0%)和91.3%~99.5%(95.4%~99.8%),见表3,4。

表1 DENV 1和DENV 2PCR引物序列
Note: * T letters for DENV 1 and DENV 2 virus specific primers in primers; E letters for the primer of E gene; F letters for the forward primer; R letters for the reverse primer.
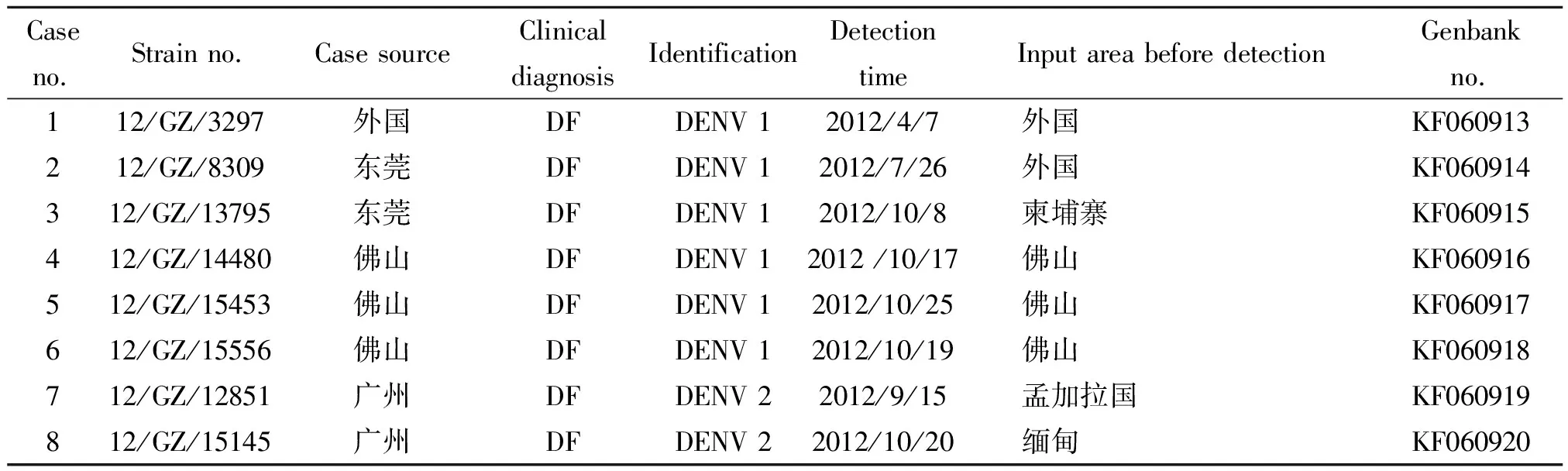
表2 8株DEN感染病例流行病学资料
2012年的6株DENV 1E基因碱基或氨基酸分析显示,其中12/GZ/3297(D1-3297)、12/GZ/8309(D1-8309)和12/GZ/13795(D1-13795)毒株氨基酸序列同源性较高99.8~100.0%;12/GZ/14480(D1-14480)、12/GZ/15453(D1-15453)和12/GZ/15556(D1-15556)碱基序列同源性较高均为99.9%。我们将国内近30年来9次不同年份的DENV 1流行分离株与2012年6株DENV 1的E基因进行同源性比较,结果显示2012年所有DENV 1与2011、2006和2004年份的11/GZ/03、GZ061707、FJ231/04、ZJ 01/2004等4株国内流行株氨基酸序列同源性较高在98.8%~99.8%之间,尤其与2006年的GZ061707氨基酸同源性最高分别为99.8%、99.6%、99.8%、99.8%、99.8%、99.6%,而与其他年份国内流行株氨基酸相对较低在96.2%~98.4%之间。
我们将国内近30年来9次不同年份的DENV 2流行分离株与2012年2株DENV 2,即12/GZ/12851(D2-12851)和12/GZ/15145(D2-15145)的E基因进行同源性比较,结果显示2012年的2株DENV 2与国内流行株之间碱基(氨基酸)同源性均相对较低在91.3%~96.8%(96.2%~98.8%)之间。
2.5基因序列的进化分析 参照Rico-Hesse等[9]基因分型原则。本研究中的6株DENV 1均在同一基因型,即亚洲型,系统发育树观察D1-3297、D1-8309和D1-137953株与2011年广州分离的柬埔寨输入病例毒株11/GZ/02进化距离最近,碱基同源性均在99.8%以上,尤其D1-3297与11/GZ/02完全一致无差异,碱基完全相同。D1-14480、D1-15453和D1-15556则与我国福建2004年流行株FJ231/04进化距离较近,碱基同源性分别为98.7%、98.8%和98.7%。

表3 广州DENV 1 E基因核苷酸及推导氨基酸的同源性比较(%)
Note: Base (lower left) and the deduced amino acids (upper right).
11/GZ/03 (Guangzhou, China, 2011); GZ061707 (Guangzhou,China,2006);
FJ231/04(Fujian, China, 2004); ZJ01/2004 (Zhejiang, China, 2004);
GZ/218/2002 (Guangzhou, China, 2002); GD99/99 (Chaozhou, China, 1999);
GD01/97 (Chaozhou, China, 1997); GD95/95 (Zhongshan, China,1995);
GD03/91 (Guangzhou, China, 1991); GZ/80 (Guangzhou, China, 1980).

表4 广州DENV 1 E基因核苷酸及推导氨基酸的同源性比较(%)
Note: Base (lower left) and the deduced amino acids (upper right).
10/GZ/11864 (Guangzhou, China, 2010); 1008DHF (Taiwan, China, 2004);
GD19/2001 (Jiangmeng, China, 2001); FJ-10 (Fujian, China, 2000);
GD05/98(Nanhai, China, 1998); GD09/93 (Fushan, China, 1993);
44 (Hainan, China, 1989); 43 (Guangxi, China, 1987); 04 (Hainan, China, 1985)
2株DENV 2属于两个不同的亚型, D2-12851属于马来西亚/印度次大陆型, D2-15145属于东南亚型(见图3),系统发育树显示D2-12851与2010年新加坡分离株SG(EHI)D2/34820Y10,D2-15145与2006年台湾分离株0606aTw进化距离较近,碱基同源性分别为99.5%和99.3%。
3 讨 论
广州市地处亚热带,年平均气温高,降雨量大,气候条件适合登革热传播媒介白蚊伊蚊的生长,利于登革热疫情的发生。广州市自1978 年以来曾发生9次登革热流行,并且登革病毒1~4四个血清型都曾有过流行。其中1995、2002、2006和2011年是DENV 1型,1985、1986和1988年是DENV 2型[8,10-11]。2012年广州市疾病预防控制中心在广州地区登革病毒抗体全年监测中筛出IgM/IgG抗体阳性患者478例,其血清经C6/36细胞分离培养获得49株病毒, PCR鉴定并测序,同年中DENV 1~4型均存在,发现其中6例是DENV 1型,2例是DENV 2型,15例DENV 3,26例DENV 4,DENV 3和DENV 4我们已经做过相关分析[12-13], 本研究我们针对DENV 1和DENV 2型E基因序列进行生物信息学的分析。
Rico-Hesse等[9]利用全序列的DENV 1、DENV 2型进行系统发育分析,确认了登革病毒血清型亚型的存在,并将登革病毒基因型明确定义为“一类基因序列差异不超过6%的登革病毒” 碱基序列差异小于6%为高同源,反之低同源。按照地理分布分别命名为,DENV 1有森林/马来西亚型、美洲/非洲型、南太平洋型、亚洲型和泰国型;DENV 2有森林/西非型、美洲型、马来西亚/印度次大陆型、东南亚型。
8株登革病毒E基因序列未见序列插入或缺失。将这些DENV中的DENV 1、DENV 2,分别与GenBank中我国近30年来本土流行的10株DENV 1和9株DENV 2进行E基因序列碱基和氨基酸的同源性比对(见表3和4),与GenBank中多株全球不同地区的DENV 1型和DENV 2E基因代表序列绘制系统发育树(见图2和3),目的以探索其可能的来源以及是否存在本土隔年传播的可能。
6株DENV 1的数据分析结果表明D1-3297、D1-8309、D1-13795、D1-14480、D1-15453和D1-15556均属于亚洲型。经病例资料分析,D1-3297、D1-8309和D1-13795在发病前均有境外活动史,且患者进入广州后发病,D1-13795可确定其是柬埔寨输入,尚不能确定D1-3297和D1-8309来源于境外何处,但通过E基因同源分析和系统发育树中可发现,这3株与2011年我们在广州人群分离的DENV 1柬埔寨输入株11/GZ/02高度同源且进化距离相当接近, 3株DENV 1在流行过程中,病毒的E基因核酸序列发生了一定程度的突变,但这些突变绝大部分是“同义突变”,即不影响其编码的氨基酸序列,因此可以推测它们均为同一来源的柬埔寨流行株。
而D1-14480、D1-15453和D1-15556均为佛山居民,来广州区域活动时发病而被监测,3株病毒E基因碱基同源性均为99.9%。同样,它们的碱基突变部分是“同义突变”。据报道在2012年6月以来佛山人群暴发登革热疫情[14],广州与佛山地理位置邻近,因此可以判断此3株病毒是同一来源的2012年佛山流行株。以上6株DENV 1与以往本土流行株传播链不存在直接的关系,均为输入性毒株。
2株DENV 2数据分析结果表明D2-12851和D2-15145分别属于马来西亚/印度次大陆型和东南亚型。基因同源性分析,此2株病毒与我国的DENV 2流行株差异较大,没有发现它们之间属同一毒株进化而来。经病例资料分析,他们发病前分别来自孟加拉国和缅甸,同样,这两株DENV 2与以往本土流行株传播链也不存在直接的关系,可推断病毒均为境外输入株。
早在20世纪70年代,就有学者发现异型登革病毒二次感染会造成抗体依赖性感染增强作用(ADE),从而易引发登革出血热,进而提出了抗体依赖性感染增强学说[11,15-16]。经过流行病学调查2012年广州地区记录的登革热病例均未曾有登革热感染史,目前广州地区还未有DHF和DSS的报道,ADE现象还不明确。但随着近年登革热在广州呈现同年不同型别、不同型别不同年交替流行的状态,且2010[17-18]和2012年DENV 1~4型均出现在广州,让我们一直担心广州人群中是否也将会出现重症的DHF和DSS,这提醒广州防疫部门及时发现和控制传染源,以防登革热重症病例的出现。
参考文献:
[1]Rico-Hesse R. Molecular evolution and distribution of dengue viruses type 1 and 2 in nature[J]. Virology, 1990, 174(2): 479-493.
[2]Weaver SC, Vasilakis N. Molecular evolution of dengue viruses: contributions of phylogenetics to understanding the history and epidemiology of the preeminent arboviral disease[J]. Infect Genet Evol, 2009, 9 (4): 523-540. DOI: 10.1016/j.meegid.2009.02.003
[3]Vasilakis N, Weaver SC. The history and evolution of human dengue emergence[J]. Adv Virus Res, 2008, 72: 1-76.
[4]World Health Organization. Dengue/dengue haemorrhagic fever[EB/OL]. [2013-01-17]. http://www.who.int/csr/disease/dengue/en/
[5]World Health Organization. Dengue and severe dengue[EB/OL]. [2013-01-29]http://www.who.int/mediacentre/factsheets/fs117/en/index.html
[6]Poornima P, Patrick C, Yolanda T, et al. Genome-wide patterns of intrahuman dengue virus diversity reveal associations with viral phylogenetic clade and interhost diversity[J]. J Virol, 2012, 86(16): 8546-8558. DOI: 10.1128/JVI.00736-12
[7]Clarke T. Dengue virus: break-bone fever[J]. Nature, 2002, 416(6882): 672. DOI: 10.1038/416672a
[8]Fang MY, Lin LH, Liu JW. Arthropod-borne infectious diseases[M]. Beijing: Military Medicine Scienea Press, 2005: 20-122. (in Chinese)
方美玉,林立辉,刘建伟. 虫媒传染病[M]. 北京:军事医学科学出版社, 2005:20-122.
[9]Rico-Hesse R. Microevolution and virulence of dengue viruses[J]. ADEN Virus Res, 2003, 59: 315-341.
[10]Jiang LY, Cao YM, Xu Y, et al. Epidemiological situation and the E gene evolution of dengue virus in Guangzhou, 2011[J]. Chin J Epidemiol, 2012, 33(12): 1273-1275. DOI: 10.3760/cma. jissn.0254-6450.2012.12:017 (in Chinese)
蒋力云, 曹毅敏, 许杨, 等. 广州市2011年登革病毒流行状况及E基因进化特征分析[J]. 中华流行病学杂志, 2012,33(12)1273-1275. DOI: 10.3760/cma. jissn. 0254-6450. 2012. 12:017
[11]Qin ED, Qin CF, Jiang T. Dengue virus and dengue virus disease[M]. Beijing: Science Press,2008: 20-53. (in Chinese)
秦鄂德,秦成峰,姜涛. 登革病毒和登革病毒病[M]. 北京:科学出版社,2008:20-53.
[12]Bai Z, Liu LC, Jiang LY, et al. Complete genome sequence of dengue virus serotype 3 from Guangzhou[J]. Genome Announc, 2013, 1(2): e00208- e00212. DOI: 10.1128/genomeA.00208-12
[13]Bai Z, Liu Q, Jiang LY, et al. Complete genome sequence of dengue virus serotype 4 from Guangzhou[J]. Genome Announc, 2013, 1(3): e00299-00313. DOI: 10.1128/genomeA.00299-13
[14]Guangdong Provincial Health Office of Public Affairs in 2012. The positive prevention and control of dengue fever in Guangdong[EB/OL][2012-09-18]. http://www.gdwst.gov.cn/a/zwxw/2012091810050.html
广东省卫生厅政务公开2012年. 我省积极防控登革热疫情[EB/OL].[2012-09-18]. http://www.gdwst.gov.cn/a/zwxw/2012091810050.html
[15]Klik SC, Nisalak A, Brandt WE, et al. Antibody-dependent enhancement of dengue virus growth in human monocytes as a risk factor for dengue hemorrhagic fever[J]. Am J Trop Med Hyg, 1989, 40: 444-451.
[16]Hughes HR, Crill WD, Chang GJ. Manipulation of immunodominant dengue virus E protein epitopes reduces potential antibody-dependent enhancement[J]. Virol J, 2012, 9: 115. DOI: 10.1186/1743-422X-9-115
[17]Bai ZJ, He P, Di B, et al. Envelope gene evolution analysis on type 1, 2, 3 dengue virus in Guangzhou in 2010[J]. Chin J Infect Dis, 2012, 3(30): 152-156. DOI: 10.3760/cma.i.issn.1000-6680.2012.03.006 (in Chinese)
白志军,和鹏,狄飚,等.广州市2010年1、2、3型登革病毒E基因进化分析[J].中华传染病杂志,2012,3(30):152-156. DOI: 10.3760/cma. i. issn. 1000-6680. 201 2. 03. 006
[18]He P, Bai ZJ, Di B. Isolation and E gene evolutional analysis of new emerged type 4 dengue virus from the outbreak of Guangzhou in 2010[J]. Chin J Epidemiol, 2011, 32(10): 1051-1053. DOI: 10.3760/cma.j.issn.0254-6450.2011.10.000 (in Chinese)
和鹏,白志军,狄飚,等. 广州市2010年登革4型病毒的分离及其E基因进化分析[J].中华流行病学杂志,2011 ,32(10):1051-1053. DOI: 10.3760/cma.j.issn.0254-6450.2011.10.000

