苹果微型反向重复转座元件(MITE)的分离与鉴定
孙海悦,张志宏
(1 吉林农业大学 园艺学院,吉林 长春 130118;2 沈阳农业大学 园艺学院,辽宁 沈阳 110866)
转座元件也称“跳跃基因”,在基因组中能够从一个基因位点转移到另一个位点。根据其结构和转座机制分为2大类:类型1转座元件称为反转录转座子(retrotransposon),以RNA为中间媒介,采用“copy-and-paste”方式转座;传统的类型2转座元件称为DNA转座子(DNA transposon),采用“cut-and-paste”机制来实现,一般不增加基因组的大小。DNA转座子具有短的末端反向重复(terminal inverted repeat,TIR)序列和编码转座酶(transposase,TPase)的单一基因,可分为自主元件(autonomous element)、非自主元件(non-autonomous element)和微型反向重复转座元件(miniature inverted-repeat transposable element,MITE)3种亚类。此外,还有一类新的类型2转座元件——Helitrons,它以单链DNA(single strand DNA,ssDNA)为中间媒介,采用“rolling-circle”的方式进行转座[1]。
MITEs最初发现于植物中[2],之后在多种真核生物包括动物、人类和真菌中都有发现[3]。MITEs在结构上与非自主DNA转座子相似[4],长度短(<500 bp),具有靶位点重复(target site duplication,TSD)和TIR序列,缺乏基因编码能力,一般富含A/T。在某些情况下,TIR可以形成稳定的茎环结构[4]。MITE的高拷贝数、特征靶位点和家族内序列的一致性,使其明显不同于其他非自主DNA转座子。与其他主要的转座元件(TE)家族不同,MITEs更易插入基因或基因附近[5-7],通过提供新的剪切位点、转录起始位点、新的外显子和poly(A)位点来影响基因表达[8-10]。此外,MITEs可以产生小干扰RNA(small interfering RNA,siRNA)基因来调控与其邻近的非必需基因[7,11-12]。通常,植物基因组含有103到105个拷贝的MITE[13],MITEs的高拷贝和经常与基因相联的特性,使其对基因调控起重要作用,并可能在基因组进化及生物多样性形成中扮演重要角色[4]。
苹果(Malus×domestic)是重要的世界性温带果树,目前生产上使用的大多数品种来自于芽变选种,如富士、元帅、嘎拉等芽变品种系。在苹果和葡萄上已经发现转座元件插入基因可以导致芽变[14-15],但对芽变产生的分子机理尚未有深刻解析。因此,本研究从分离苹果DNA转座子PIF/Harbinger家族的TPase入手,通过寻找TSD和TIR特异位点来分离苹果MITE,以期阐明MITEs在苹果基因组进化过程中的重要作用,为苹果优异芽变品种的选育提供科学依据。
1 材料与方法
1.1 材 料
1.1.1 植物材料 富士苹果(Malus×domesticacv.Fuji)的新鲜幼嫩叶片取自沈阳农业大学果树基地,植物组织经液氮处理后于-80 ℃超低温冰箱中保存。
1.1.2 菌株、质粒与试剂 大肠杆菌EscherichiacoliJM109、克隆载体pMD18-T购自宝生物工程(大连)有限公司;TaqDNA聚合酶、LongTaqDNA聚合酶,分别购自普洛麦格(北京)生物技术有限公司和天根生化科技(北京)有限公司;限制性内切酶DraⅠ、EcoRⅤ、PvuⅡ、StuⅠ购自NEB(北京)有限公司;胶回收试剂盒、质粒提取试剂盒,购自宝生物工程(大连)有限公司;染色体步移试剂盒Genome Walker Universal Kit,购自BD Biosciences Clontech公司。引物合成与测序服务由生工生物工程(上海)股份有限公司提供。
1.2 方 法
1.2.1 苹果基因组DNA的提取 用改良CTAB法提取苹果基因组DNA[16]。
1.2.2 苹果PIF TPase基因部分序列的扩增 使用PIF TPase简并引物[17]扩增苹果PIF TPase基因的部分序列。引物序列为PIF1:5′-GGIGCHHTIGATGGHACWCA-3′(编码氨基酸GALDTH)和PIF2:5′-ATGICKMIRRTRAACAAYTC-3′(编码氨基酸ELFNPRH)。PCR扩增体系为50 μL,包括50~100 ng 基因组DNA,7.5 μL 10×PCR Buffer,5 μL 25 mmol/L MgCl2,1 μL 2.5 mmol/L dNTPs(TaKaRa),15 pmol PIF1和PIF2引物,2.5 UTaqDNA聚合酶(Promega)。PCR反应程序为:94 ℃预变性3 min;94 ℃变性30 s,50 ℃退火30 s,72 ℃延伸1 min,共36个循环;72 ℃延伸5 min。
1.2.3 苹果PIF TPase未知侧翼序列的获得 采用说明书推荐的4种平端限制性内切酶DraⅠ、EcoRⅤ、PvuⅡ和StuⅠ,分别对苹果基因组DNA进行酶切并构建染色体步移文库,酶切反应体系参照厂家提供的说明。将酶切后的DNA与接头(Adaptor和Adaptor plus)于16 ℃连接过夜,并将这些与接头连接的产物标记为染色体步移库(DL1、DL2、DL3和DL4),作为PCR反应的模板。根据已知的PIF TPase部分序列——PIF-Malus8,使用引物设计软件Primer Premier 5.0设计特异引物GSP引物,引物设计要求为:2个巢式引物长度在26~30 bp,Tm值在67~72 ℃,2个引物不重叠,内外巢式引物距离尽量大于50 bp。用于扩增PIF TPase未知侧翼序列的GSP引物序列见表1。

表 1 采用染色体步移技术扩增苹果PIF TPase未知侧翼序列所用的PCR引物
初始PCR(Primary-PCR)是以adapter primer(AP1)和GSP-out为引物,分别从4个文库中取 0.5 μL作为模板进行PCR反应。PCR反应体系如下:0.5 μL稀释10倍的连接产物,2.5 μL BufferⅡ,2 μL 2.5 mmol/L dNTP(TaKaRa),0.5 μL 10 μmol/L AP1引物,0.5 μL 10 μmol/L GSP-out引物, 5 U LongTaqDNA聚合酶(天根),总体积25 μL。PCR反应条件为:94 ℃ 25 s,72 ℃ 3 min,7个循环;94 ℃ 25 s,67 ℃ 3 min,32个循环;67 ℃延伸7 min;4 ℃保温。初始PCR反应结束后,将初始PCR产物稀释50倍,取0.5 μL作模板,以AP2和GSP-in为引物进行第2轮巢式PCR(nest-PCR),其他PCR反应成分及用量同初始PCR。反应程序为:94 ℃ 25 s,72 ℃ 3 min,5个循环;94 ℃ 25 s,67 ℃ 3 min,20个循环;67 ℃延伸7 min;4 ℃保温。结束反应后取8 μL在10 g/L琼脂糖凝胶中检测,对主要的第2轮产物进行回收和测序。
1.2.4 苹果MITE的扩增 根据染色体步移所获得的TSD和TIR特征序列设计单向特异引物TIRF1,引物序列为:5′-ATTAGGGGGGTGTATTCAATTAGGA-3′。PCR 反应程序为:94 ℃预变性2 min;94 ℃变性1 min,66.8 ℃退火1 min,72 ℃延伸1 min,共35个循环;72 ℃延伸10 min。PCR扩增体系为50 μL,包括50~100 ng 基因组DNA,5 μL 10×PCR Buffer, 3 μL 25 mmol/L MgCl2,1 μL 2.5 mmol/L dNTPs(TaKaRa),15 pmol TIRF1引物,2.5 UTaqDNA聚合酶(Promega)。
1.3 苹果相关基因序列的分析方法
利用NCBI的BLAST程序对测序结果进行基因注释,使用Contig Express软件对序列进行拼接,使用EMBOSS的Eiverted软件查找TIR序列,使用Genedoc软件进行序列同源比对,使用NetGene分析选择性剪切情况。
2 结果与分析
2.1 苹果PIF TPase基因部分序列的分离及其特性
利用PIF TPase简并引物从苹果基因组中扩增出长度约450 bp的片段(图1),将目标片段进行回收、纯化、克隆,挑取18个单克隆进行测序。序列分析结果表明,该片段具有PIF TPase的典型基序5′-GAL(M)DGTH 和3′-ELFNP(S)R(S)H,并与GenBank上的已知PIF TPase具有较高的同源性,因此,将这些序列注释为苹果PIF TPase基因的部分序列,命名为PIF-Malus,PIF代表PIF TPase,Malus代表苹果,GenBank登录号为KF999492~KF999509。

图 1 苹果PIF转座酶部分序列的PCR扩增结果
由表2可知,这些序列长度介于427~482 bp,有较大差异。通过BLASTX和选择性剪切分析发现,PIF-Malus1等9条序列(50%)具有终止子和移码的双重突变情况;PIF-Malus5等4条序列(22%)的长度为438 bp,具有未打断的ORF;PIF-Malus3等其余5条序列(28%)的编码区存在内含子,内含子长度很短,平均86 bp,2个外显子区域均有未打断的开放阅读框(open reading frame, ORF)。
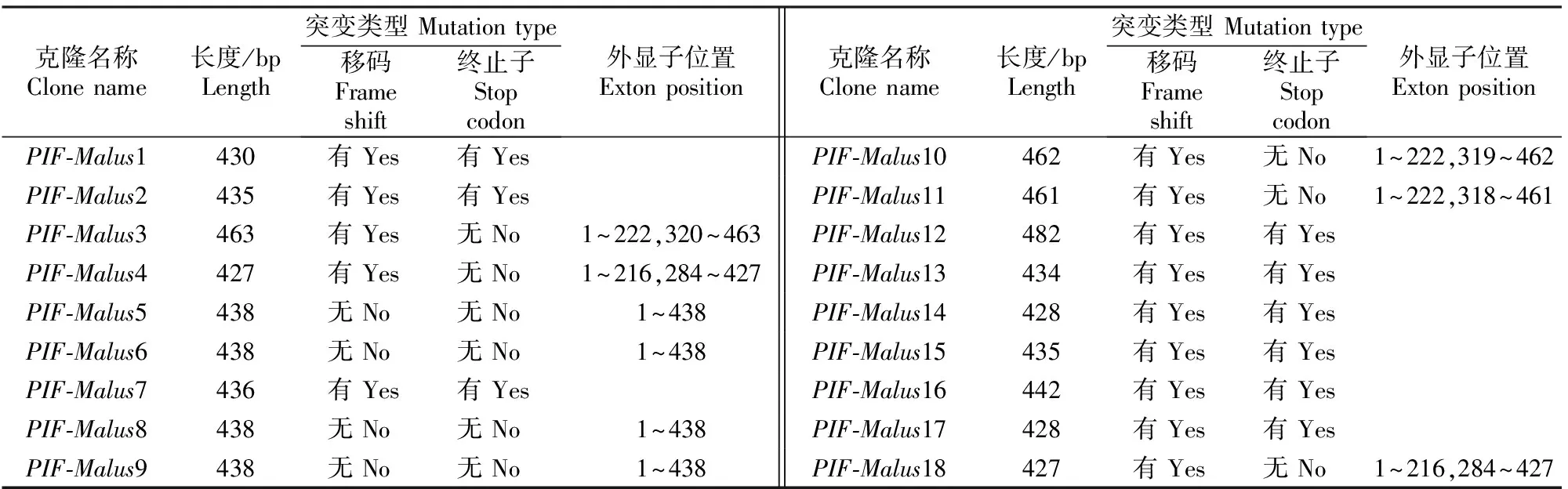
表 2 苹果PIF TPases部分序列的特性
为了研究PIF TPase区域序列的系统进化关系,对具有未打断ORF的PIF-Malus序列进行进一步分析,先去除内含子,再翻译成氨基酸序列,采用距离法(1 000次重复)建立系统进化树。结果(图2)显示,这9条功能序列存在一定的异质性,序列同源性为80.8%,聚类成3个家族(F1~F3)。F1家族具有完全的开放阅读框,不含内含子,F2家族具有67 bp的内含子,F3家族具有98 bp的内含子。F1和F2家族的苹果PIF TPase序列与拟南芥的Transposase_11具有较高的同源性,被聚类在同一小组,F3家族的苹果PIF TPase序列与葡萄的Transposase_11具有较高的同源性。
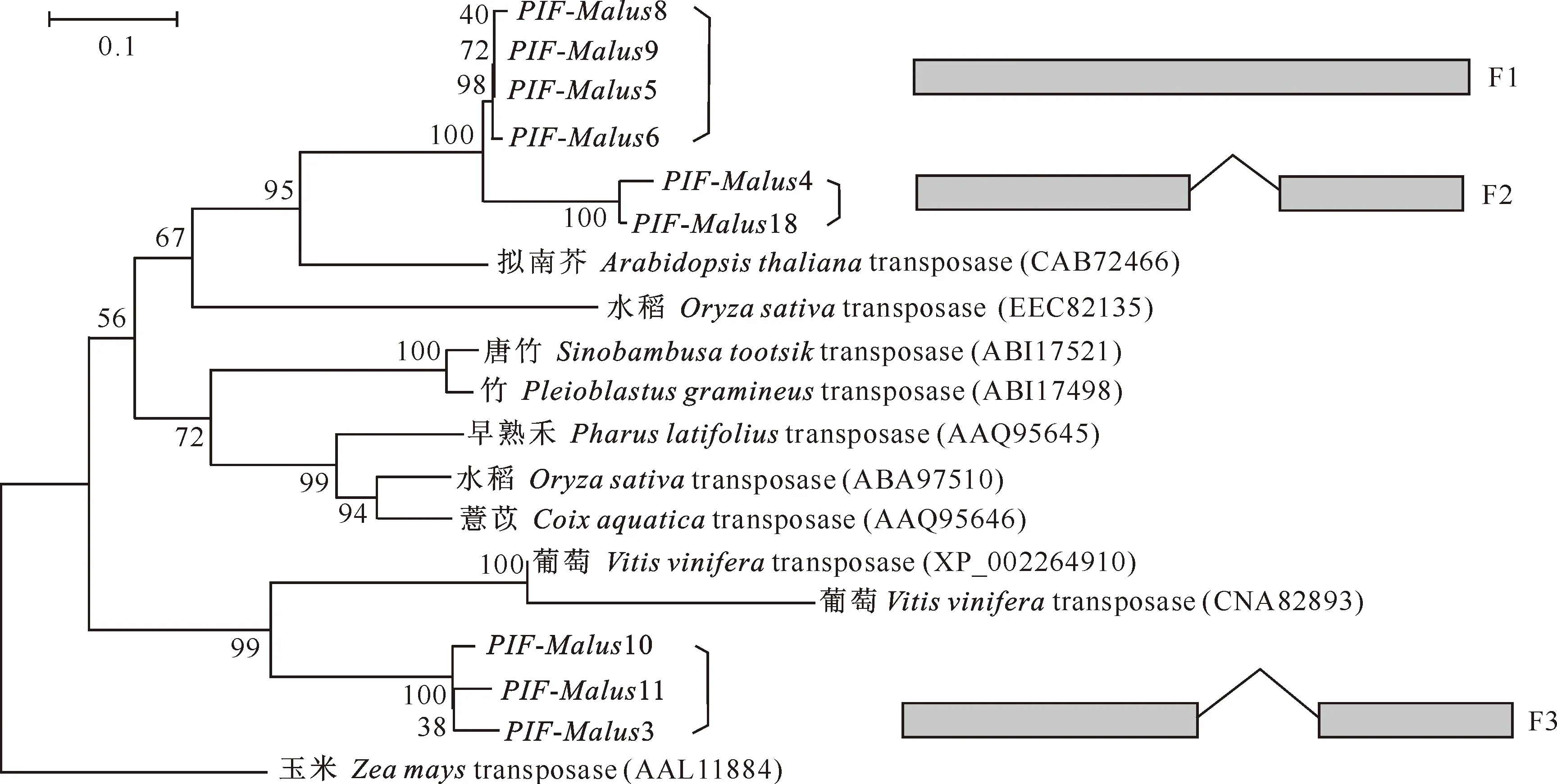
图 2 基于距离邻接法构建的苹果PIF转座子转座酶的系统进化树
2.2 苹果PIF非自主DNA转座子的查找
在进行染色体步移过程中,通过Eiverted软件查找到了TIR,发现其具有TouristMITE的TSD特征序列(TTA和TAA),含有43 bp的保守TIR(图3),中间区域883~1 642编码转座酶,与拟南芥Transposase_11(GenBank 登录号:CAB72466)具有58%的同源性,但这段区域发生移码突变且包含终止子,不具有转座功能,属于非自主DNA转座子,因此,将这段长2 704 bp的序列命名为PIFdefectiveDNAtransposonMalus(PIFdMalus),GenBank登录号为KJ004499。

图 3 苹果PIF非自主DNA转座子与MITE的关系示意图
2.3 苹果MITE的特点
在苹果PIF非自主DNA转座子PIFdMalus的TIR区域设计特异的单向引物(TIRF1),通过PCR扩增获得了大小为400 bp左右的片段,将目标片段进行回收、纯化、克隆,挑取4个单克隆进行测序。结果(图4)显示:(1)它们具有TouristMITE的TSD特征序列(TTA);(2)具有21 bp的保守TIR,即5′-GGGGGGTGTATTCAATTAGGA~TCCTAATTGAATACACCCCCCTAA-3′(图3);(3)这些序列长度很小,并且富含A+T,其含量在69.2%~72.5%(表3);(4)它们之间具有较高的同源性,序列同源性在73.6%~99.8%(表4);(5)它们具有较低的自由能(ΔG)值(-201.9~-216.0 kJ/mol,表3),容易形成二级结构(图5)。这些特点表明,这4条序列具有MITE的典型特征,可以认定为苹果基因组中一个属于Tourist家族的新MITE,命名为MITEMalus1~4,GenBank登录号为KJ004500~KJ004503。

图 4 苹果MITE的核苷酸序列

表 3 植物中不同MITE家族的特性

表 4 苹果MITE核苷酸序列的同源性
3 讨 论
PIF TPase基因广泛存在于植物基因组中,Zhang等[17]采用PIF TPase简并引物,从20种禾本科植物中扩增出约360 bp和450 bp的2种片段;Remigereau等[18]从珍珠稷中也扩增到约350 bp和450 bp的2种片段。本研究采用相同引物从苹果基因组中分离出约450 bp 的片段,与前人结果不同,但测序后发现这18条苹果PIF TPase基因的部分序列长度不等,序列之间也有差异。这可能是在基因组长期进化中形成的,基因组会在转座激活和转座抑制之间保持动态平衡[19],一些TPases会不断地累积突变,最终失去转座功能,如本研究中50%的苹果PIF TPase是移码或终止子突变产生的假基因,另一些TPase可能对基因组有利,保留了可翻译的ORF,可能具有转座活性。因此,推测苹果PIF TPase仍可能是活跃的,可以通过识别TIR位点来驱动MITE转座。
MITE的起源和形成机制是自主DNA转座子在基因组进化过程中发生内部缺失,产生不同长度的非自主DNA转座子,这些非自主DNA转座子的TPase不具备转座驱动能力,但是,其中一个或几个长度较小的非自主DNA转座子在相应的自主DNA转座子的TPase顺式驱动下发生复制,激增至高拷贝,最终形成MITE[13,20]。研究表明,PIF/Harbinger和Tc1/marineDNA转座子分别调控MITE的Tourist和Stowaway元件,此后陆续证明大多数DNA转座子家族都有相应的MITE[3,21-23]。本研究利用苹果非自主DNA转座子的典型TSD和TIR序列,通过TIR引物成功扩增到苹果MITE,验证了苹果PIF家族DNA转座子与MITEMalus的相关性,但苹果MITEMalus与PIF DNA 转座子的相似关系仅限于TPase调控的TIR序列,内部不含有相似序列,这一结果与拟南芥、水稻、玉米、葡萄、酵母等的MITE结果相似[24-32]。
在植物基因组中,MITEs的长度很小,相对于较大的DNA转座子大部分被沉默的结果,MITEs更加适应于宿主基因组的环境,在整个进化过程中更容易保持转座活性,MITEs经常与基因相连,可以对基因表达产生较大影响,并且它们的高拷贝数也对基因组有很大影响[20]。本研究分离得到的苹果MITEMalus与李的ACC氧化酶基因(ACO)的启动子和5′非翻译区(5′-untranslated region,5′UTR)具有较高的相似性,同源性为82%~93%,并且在这个基因内形成了非常显著的茎环结构。MITEs比较倾向于插入基因的启动子、UTRs、内含子、外显子区域[3-4],这种插入特异性可能是在基因组的长期进化选择压力下形成的,这样MITEs可以采用多种方式来调解mRNA,包括稳定性、进程和通过转录后水平基因沉默(Post-transcriptional gene silencing,PTGS)来降解mRNA[6,10,12,33-35]。目前,已经在水稻、马铃薯、花生、葡萄中发现了有转座活性的MITE家族[21,36-39],为了进一步研究苹果MITEs的转座活性,以这4条苹果MITE作为比对序列对NCBI的除人类之外的EST数据库进行BLASTN搜索,结果显示,苹果MITEMalus在EST数据库中的匹配序列高达50多条植物EST,有一定的表达量,而且其中大部分EST是来自苹果基因组的,推测本研究分离的苹果MITEMalus家族在苹果基因组中有表达,可能是一个活跃的转座元件。
本研究分离得到的苹果Tourist家族MITE,起源于苹果PIF转座子,可能具有转座活性,因此将进一步研究其在苹果基因组中的拷贝数、转座活性和分布,以揭示MITEMalus在苹果基因组和基因进化中的作用。
[参考文献]
[1] Kapitonov V V,Jurka J.Helitrons on a roll: Eukaryotic rolling-circle transposons [J].Trends Genet,2007,23(10):521-529.
[2] Bureau T E,Wessler S R.Tourist:A large family of small inverted repeat elements frequently associated with maize genes [J].The Plant Cell,1992,4(10):1283-1294.
[3] Jiang N,Feschotte C,Zhang X,et al.Using rice to understand the origin and amplification of miniature inverted repeat transposable elements (MITEs) [J].Current Opinion In Plant Biology,2004,7(2):115-119.
[4] Wessler S R,Bureau T E,White S E.LTR-retrotransposons and MITEs:Important players in the evolution of plant genomes [J].Current Opinion In Genetics & Development,1995,5(6):814-821.
[5] Mao L,Wood T C,Yu Y,et al.Rice transposable elements:A survey of 73 000 sequence-tagged-connectors [J].Genome Research,2000,10(7):982-990.
[6] Yang G,Lee Y H,Jiang Y,et al.A two-edged role for the transposable element Kiddo in the rice ubiquitin 2 promoter [J].The Plant Cell,2005,17(5):1559-1568.
[7] Piriyapongsa J,Jordan I K.A family of human microRNA genes from miniature inverted-repeat transposable elements [J].PLoS One,2007,2(2):e203.
[8] Santiago N,Herraiz C,Goni J R,et al.Genome-wide analysis of the Emigrant family of MITEs ofArabidopsisthaliana[J].Mol Biol Evol,2002,19(12):2285-2293.
[9] Ohmori Y,Abiko M,Horibata A,et al.A transposon,Ping,is integrated into intron 4 of the DROOPING LEAF gene of rice,weakly reducing its expression and causing a mild drooping leaf phenotype [J].Plant Cell Physiol,2008,49(8):1176-1184.
[10] Kuang H,Padmanabhan C,Li F,et al.Identification of miniature inverted-repeat transposable elements(MITEs)and biogenesis of their siRNAs in the Solanaceae:New functional implications for MITEs [J].Genome Res,2009,19(1):42-56.
[11] Piriyapongsa J,Marino-Ramirez L,Jordan I K.Origin and evolution of human microRNAs from transposable elements [J].Genetics,2007,176(2):1323-1337.
[12] Piriyapongsa J,Jordan I K.Dual coding of siRNAs and miRNAs by plant transposable elements [J].RNA,2008,14(5):814-821.
[13] Feschotte C,Jiang N,Wessler S R.Plant transposable elements:Where genetics meets genomics [J].Nat Rev Genet,2002,3(5):329-341.
[14] Yao J,Dong Y,Morris B A.Parthenocarpic apple fruit production conferred by transposon insertion mutations in a MADS-box transcription factor [J].Proc Natl Acad Sci USA,2001,98(3):1306-1311.
[15] Kobayashi S,Goto-Yamamoto N,Hirochika H.Retrotransposon-induced mutations in grape skin color [J].Science,2004,304(5673):982.
[16] Lodhi M A,Ye G N,Weeden N F,et al.A simple and efficient method for DNA extraction from grapevine cultivarsVitisspecies andAmpelopsis[J].Plant Mol Biol Rep,1994,12:6-13.
[17] Zhang X,Jiang N,Feschotte C,et al.PIF- and Pong-like tran-sposable elements:Distribution,evolution and relationship with Tourist-like miniature inverted-repeat transposable elements [J].Genetics,2004,166(2):971-986.
[18] Remigereau M S,Robin O,Siljak-Yakovlev S,et al.Tuareg,a novel miniature-inverted repeat family of pearl millet (Pennisetumglaucum) related to the PIF superfamily of maize [J].Genetica,2006,128(1/2/3):205-216.
[19] Oliver K R,McComb J A,Greene W K.Transposable elemen-ts:Powerful contributors to angiosperm evolution and diversity [J].Genome Biol Evol,2013,5(10):1886-1901.
[20] Casacuberta J M,Santiago N.Plant LTR-retrotransposons and MITEs:Control of transposition and impact on the evolution of plant genes and genomes [J].Gene,2003,311:1-11.
[21] Jiang N,Bao Z,Zhang X,et al.An active DNA transposon family in rice [J].Nature,2003,421(6919):163-167.
[22] Yang G,Zhang F,Hancock C N,et al.Transposition of the rice miniature inverted repeat transposable element mPing inArabidopsisthaliana[J].Proc Natl Acad Sci USA,2007,104(26):10962-10967.
[23] Yang G,Nagel D H,Feschotte C,et al.Tuned for transposition:Molecular determinants underlying the hyperactivity of a Stowaway MITE [J].Science,2009,325(5946):1391-1394.
[24] Casacuberta E,Casacuberta J M,Puigdomenech P,et al.Presence of miniature inverted-repeat transposable elements (MITEs) in the genome ofArabidopsisthaliana:Characterisation of the Emigrant family of elements [J].Plant J,1998,16(1):79-85.
[25] Surzycki S A,Belknap W R.Characterization of repetitive DNA elements inArabidopsis[J].J Mol Evol,1999,48(6):684-691.
[26] Feschotte C,Mouches C.Evidence that a family of miniature inverted-repeat transposable elements (MITEs) from theArabidopsisthalianagenome has arisen from a pogo-like DNA transposon [J].Molecular Biology and Evolution,2000,17(5):730-737.
[27] Turcotte K,Srinivasan S,Bureau T.Survey of transposable elements from rice genomic sequences [J].Plant J,2001,25(2):169-179.
[28] Zhang X,Feschotte C,Zhang Q,et al.P instability factor: An active maize transposon system associated with the amplification of Tourist-like MITEs and a new superfamily of transposases [J].Proc Natl Acad Sci USA,2001,98(22):12572-12577.
[29] Yang G,Hall T C.MDM-1 and MDM-2:Two mutator-derived MITE families in rice [J].J Mol Evol,2003,56(3):255-264.
[30] Loot C,Santiago N,Sanz A,et al.The proteins encoded by the pogo-like Lemi1 element bind the TIRs and subterminal repeated motifs of theArabidopsisEmigrant MITE:Consequences for the transposition mechanism of MITEs [J].Nucleic Acids Res,2006,34(18):5238-5246.
[31] Hancock C N,Zhang F,Wessler S R.Transposition of the Tourist-MITE mPing in yeast:An assay that retains key features of catalysis by the class 2 PIF/Harbinger superfamily [J].Mob DNA,2010,1(1):5.
[32] Benjak A,Boué S,Forneck A,et al.Recent amplification and impact of MITEs on the genome of grapevine (VitisviniferaL.) [J].Genome Biol Evol,2009,1:75-84.
[33] Lu C,Chen J,Zhang Y,et al.Miniature inverted-repeat transposable elements (MITEs) have been accumulated through amplification bursts and play important roles in gene expression and species diversity inOryzasativa[J].Mol Biol Evol,2012,29(3):1005-1017.
[34] Mo Y J,Kim K Y,Shin W C,et al.Characterization of Imcrop,a Mutator-like MITE family in the rice genome [J].Genes Genomics,2012,34(2):189-198.
[35] Piriyapongsa J,Jordan I K.Dual coding of siRNAs and miRNAs by plant transposable elements [J].RNA,2008,14(5):814-821.
[36] Kikuchi K,Terauchi K,Wada M,et al.The plant MITE mPing is mobilized in anther culture [J].Nature,2003,421(6919):167-170.
[37] Nakazaki T,Okumoto Y,Horibata A,et al.Mobilization of a transposon in the rice genome [J].Nature,2003,421(6919):170-172.
[38] Momose M,Abe Y,Ozeki Y.Miniature inverted-repeat transposable elements of Stowaway are active in potato [J].Genetics,2010,186(1):59-66.
[39] Shirasawa K,Hirakawa H,Tabata S,et al.Characterization of active miniature inverted-repeat transposable elements in the peanut genome [J].Theor Appl Genet,2012,124(8):1429-1438.

