系统比较氨基酸描述子在多肽定量构效关系中的应用
许 航, 田菲菲, 吴 坚
(西南交通大学 生命科学与工程学院, 成都 610031)
生命科学发展迅速,该领域的研究文献也正在以难以计量的速度巨增。关于氨基酸描述子在多肽定量构效关系中的研究文献也成倍的增长,目前已有的氨基酸描述子已有近30种。如何在众多的氨基酸描述子建立的定量构效模型中寻求到最好的表达方式、解释、归类、列举知识成为了人们需要解决的重大课题之一。
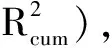
氨基酸描述子[2]是氨基酸的拓扑性质、物理化学性质、三维结构性质的量化数值,用于氨基酸的结构与性质的定量描述,把氨基酸序列转换成结构描述符矩阵的一类向量。目前在建立多肽的各种定量构效关系(quantitative sequence activity relationship, QSAR)模型中,普遍采用的方法是通过收集20种氨基酸的多种物理化学性质参数,然后采用主成分分析(principal component analysis, PCA)提取能表征原始参数数据绝大部分信息的几个性质。用这些主成分得分表征肽序列的各个氨基酸,最后将表征得到的参数与实验观测活性建立起肽QSAR模型。我们把经过主成分分析得到的氨基酸的主成分得分称为氨基酸描述子,并且利用氨基酸描述子表征多肽是当前多肽定量构效关系研究的主要方法。
Kidera等人[3]从已有文献中提取出描述20种氨基酸的188种性质参数,并运用因子分析方法,得到10个正交因子,对氨基酸的性质进行定量描述。随后,Hellerg等人在此基础上,提出氨基酸描述子Z-scales。Collantes等人提出3D氨基酸侧链描述子侧链表面积(isotropic surface area, ISA),电荷指数(electronic charge index, ECI)。田菲菲对收集到的天然氨基酸的23种静电性质,37种立体性质、54种疏水性质和5种氢键性质进行信息压缩滤噪处理,得到10主成分得分,提出了新型氨基酸描述子氨基酸理化性质分类得分(divided physicochemical property scores, DPPS)[4],T-scale[5]等、梅虎等[6]通过采用主成分分析(principal component analysis, PCA)分别处理收集到的天然氨基酸的25种拓扑化学信息和50种物理化学性质,得到两组新氨基酸描述子,即氨基酸结构与拓扑性质得分(principal component scores vector of structural and topological variables, VSTV)和疏水性立体结构带电性得分(principal component score vector of hydrophilicity, steric, and electronic properties,VHSE)[7]。梁桂兆等用多元分析方法分析挑选20种天然氨基酸的149个疏水性质参数,提出氨基酸广义疏水标度矢量GH-scale[8],它们对肽均表现出较好的表征能力。

收集的27种氨基酸描述子都未考虑所表征肽链内部的氨基酸残基之间的相互影响,如氨基酸之间相互带电性,氢键,疏水性之间的相互影响。同时传统的氨基酸描述子对肽的表征都是建立在真空模型基础之上,没有考虑肽与周围环境之间的影响,如人体的液体环境,肽配基与相应的靶点蛋白相互结合后的构效变化等因素。我们认为,氨基酸描述子有其自身的局限,过于复杂,种类繁多,干扰因素太多,难以解释,不易操作。常规的方法已经难以在建模效果的质上取得飞跃,需要从方法学上改变氨基酸QSAR模型的性能。
1 原理与方法
1.1 氨基酸描述子
本文收集已有的氨基酸描述子,通过“descriptor of amino acids”或“氨基酸描述子”为检索策略,在springer、SDOS、NCBI、CNKI等数据库中进行文献检索。通过对比筛选,去除重复的氨基酸描述子,共收集到27种不同种类的氨基酸描述子,并从中提取各氨基酸描述子的数值(27种氨基酸描述子的数值见表1)。
目前主要有3大类型描述子:1)物理化学参数, 它是一种以氨基酸分子式为基础的描述子,如氨基酸的整体与部分基团带电性,疏水性,立体结构,氢键贡献等参数,代表描述子有VHSE、HSEHPCSV 等。2)三维结构参数和量子化学参数,通过这些参数可以了解整个氨基酸分子的立体几何构型与特性,如描述子VSGETAWAY、SVEEVA等。3)拓扑指数,它通过图论方法以数量来表征分子的结构,计算分子的整体连接情况,常见的拓扑指数有如Wiener指数,Randic指数,balaban指数等,代表描述子有VSTV、T-scales等。4)其它类型描述子,如广义疏水描述子,氨基酸疏水性指标及残基溶解状态参数,代表描述子GH-scale;量子能量参数描述子,通过量子化学理论获得的,通常分为电子结构参数和空间几何参数,代表描述子t-scales、SVEEVA;OD-3D结构信息描述子,氨基酸0D-3D的结构信息,代表描述子SZOTT。

表1 27种氨基酸描述子的类型
1.2 经典肽数据集

表2 经典肽数据集
氨基酸描述子的提出是为了表征肽序列,实现肽序列的结构性质参数化,将肽结构转化成氨基酸描述子矩阵中的一个向量,用于测试氨基酸描述子的有效性。肽的类似物可以成为一个集合,可以从实验或相关文献中得到相应的活性数值(8组肽集的活性数值见表2),又称为肽数据集,目前研究最多的肽类有血管紧张素转化酶抑制剂、苦味活性二肽、后叶催产素、血管舒缓激肽促进剂等。
本文选取8组肽数据集,每组肽数据集包含的多肽数量不同,最少的肽集合后叶催产素只有21个,最多的三肽集合有162个,同时这8组肽序列长短不同,最短的肽血管紧张素转化酶抑制剂、苦味活性二肽是二肽,最长的肽CAMELs(抗菌肽)为十五肽,便于后续对比分析。这些肽数据集在许多有关多肽的定量构效关系的研究文献中反复被人们使用,且利用它们作为肽数据集取得了比较好的建模效果,是作为多肽定量构效关系研究中的常用材料。
其中以血管紧张素转化酶抑制剂、苦味活性二肽作为氨基酸描述子表征肽序列的文献已多次报道,如Hellberg 提出描述子Z-scales、Cocchi提出的描述子t-scales、Collantes提出的侧链描述子ISA-ECI都将58个血管紧张素转化酶抑制剂、48个苦味活性二肽作为多肽定量构效关系(QSAR)样本集。随后梅虎[6]提出的拓扑描述子VSTV,丁俊杰[2]提出的三维结构描述子C-scales,仝建波[19-21]提出的三维氨基酸描述子SVG、SVTD,田菲菲[31]提出的拓扑描述子T-scale都是以58个血管紧张素转化酶(ACE)抑制剂作为多肽表征样本集。Zaliani[25]提出三维结构描述子MS-WHIM scores,梁桂兆[30]提出的0D-3D结构信息描述子SZOTT,舒茂[12]提出的物理化学性质描述子HSEHPCSV,仝建波[21]提出的三维氨基酸描述子VSGETAWAY,彭剑秋提出的物理化学性质描述子SVHEHS都是以48个苦味活性二肽作为多肽表征样本集。
1.3 建模与验证
1.3.1 偏最小二乘法(partial lest square regression, PLS)建模
1983年,最先由Wold和Albano等人提出偏最小二乘法,它能有效的处理高维度、强噪音、复共线性、甚至数据缺失的线性回归建模问题。主要适用于多自变量对多因变量的线性回归建模,特别适合当多肽的结构描述符多于样本数目的情况下进行建模。偏最小二乘法首先需要对自变量矩阵X和因变量矩阵Y进行双线性分解:
X=TPT+E
(1)
Y=UQT+F
(2)
潜因变量T和U要求最大限度的包含各自数据矩阵中的变异信息,并且两者之间的重叠或相关性要达到最大。因此可得公式(3):
U=CT+e
(3)
在公式3中,C为系数,e为残差矢量,根据上述要求潜因变量T和U应最大限度的代表数据矩阵X和数据矩阵Y中的信息。本文所有建模都采用偏最小二乘法(PLS),由Simcap10.0 完成,交互检验为留一法(leave one out),其它均为软件默认设置。
1.3.2 模型验证
模型的质量评价是定量构效关系中十分重要的一个环节。模型验证采用常规的内部留一法(LOO)交叉验证以及外部验证。


本文将收集到的27种氨基酸描述子分别表征8组肽集合,随后每组被表征的肽序列都要进行随机划分,共需随机划分216次,全手动完成。随机划分样本的2/3为训练集,剩余的1/3为测试集,进行外部验证。
1.3.3 建模结果统计量
评价模型的优劣主要涉及到4个统计量[31]。
(4)
(5)
(6)
(7)
2 结果与讨论
2.1 建模统计量数值分析
8组肽集分别经27种氨基酸描述子表征后,利用偏最小二乘法得到8组27种描述子的建模统计数值(见表3),横坐标为27种氨基酸描述子,且每种描述子对应5个建模统计量,纵坐标为5个建模统计量的数值,可得建模柱形图,如图1所示。
以苦味二肽为例,从图中可以看出除氨基酸描述子VSGETAW没有数值,建模效果最差,其它26种描述子均有数值,其中15种氨基酸描述子ISA-ECI、MS-WHIM scores、SFED、VHSE、GH-scales、SZOTT、T-scale、SVRDF、HSEHPCSV、SVG、SVTD、HESH 、T-scale、G-scale的主成分数(A)为1,9种氨基酸描述子Z-scales、t-scales、VSTV、C-scales、VHSEH、VSW、V、SVEEVA、SVHEHS的主成分数(A)为2,2种氨基酸描述子DPPS、SVRG的主成分数(A)为3。
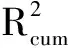

3种氨基酸描述子SVEEVA、DPPS、V的RMSEE数值在0.1~0.2之间,13种氨基酸描述子GH-scales、T-scale、ISA-ECI、VSW、VSTV、HSEHPCSV、C-scales、t-scales、HESH、Z-scales、SVHEHS、VHSEH、SVRG的RMSEE数值在0.2~0.3之间,8种氨基酸描述子SFED、MS-WHIM score、SVRDF、SZOTT、G-scale、ST-scale、FASGAI、VHSE的RMSEE数值在0.3~0.4之间,2种氨基酸描述子SVG、SVTD的RMSEE数值在0.4以上。
6种氨基酸描述子VHSE、HESH、V、VHSEH、DPPS、ISA-ECI的RMSEP数值在0.2~0.3之间,13种氨基酸描述子VSTV、G-scale、G-scale、T-scale、SVEEVA、MS-WHIM scores、ST-scale、VSW、HSEHPCSV、SFED、C-scales、SVHEHS、FASGAI、t-scales的RMSEP数值在0.3~0.4之间,7种氨基酸描述子SVTD、SVRDF、GH-scales、SVG、SZOTT、SVRG、Z-scales的RMSEP数值在 0.4以上。
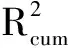
综合比较8组肽集用27种氨基酸描述子表征后得到的5个建模统计数值,按建模效果从优到劣的顺序依次为:苦味二肽>ACE抑制剂>舒缓肽>后叶催产素>抗原肽>CAMEL>三肽>激肽拮抗剂。总体上肽序列越短,建模效果越好,肽序列越长,则影响因素越多,建模效果越差。
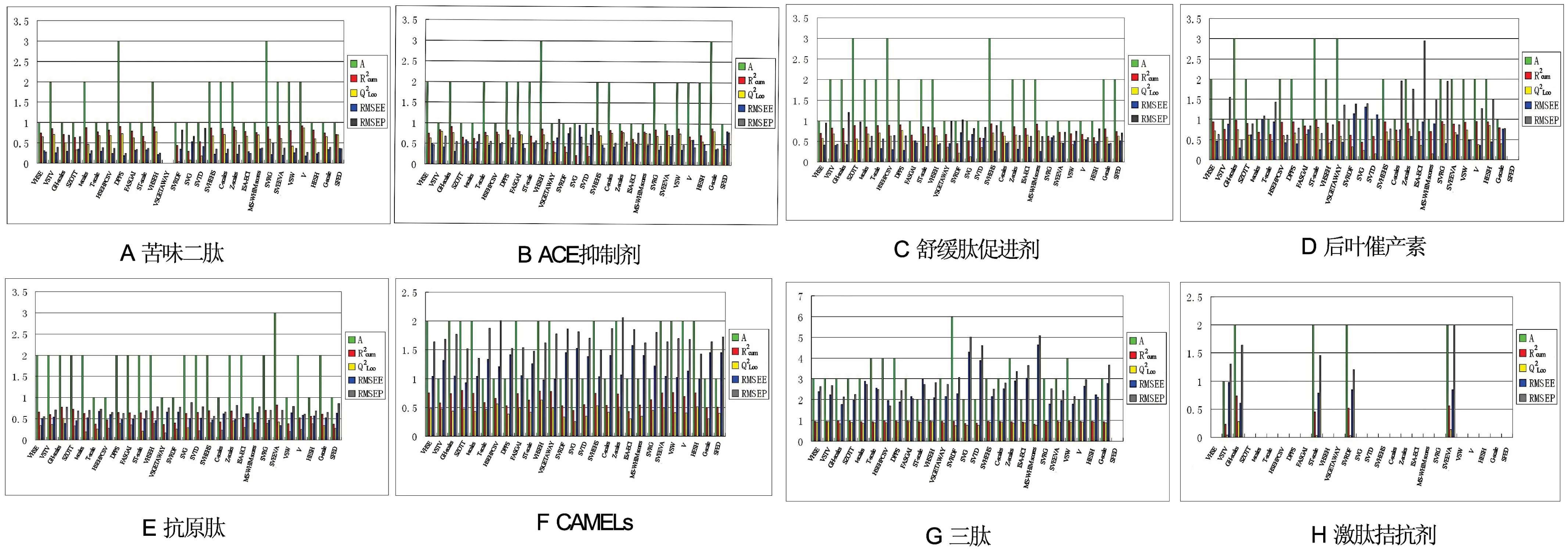

图1 8组肽集用27种氨基酸描述子表征后建模柱形图
Fig 1 The column chart of eight sets of peptide with 27 amino acid descriptors set of modeling
2.2 不同类型描述子性能比较
在收集的氨基酸描述子中,主要有3大类描述子,其中有11个物理化学描述子VHSE、HSEHPCSV、DPPS、FASGAI、VHSEH、SVHEHS、Z-scales、V、HESH、G-scale、SFED,9个三维结构描述子VSGETAWAY、SVRDF、SVG、SVTD、C-scales、ISA-ECI、 MS-WHIM scores、SVRG、VSW,3个拓扑描述子VSTV、T-scale、ST-scale。
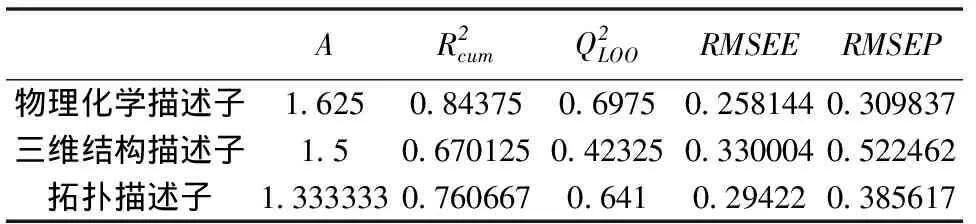
表3 苦味二肽用3类描述子表征后建模统计量平均值
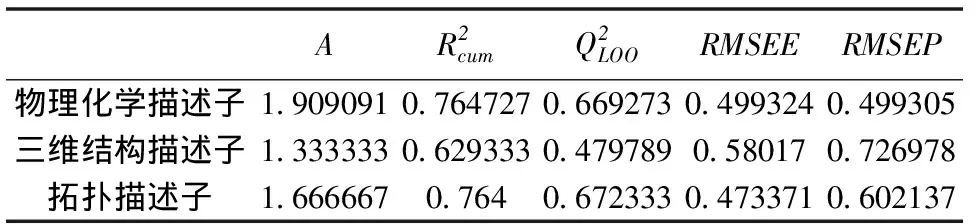
表4 ACE抑制剂用3类描述子表征后建模统计量平均值
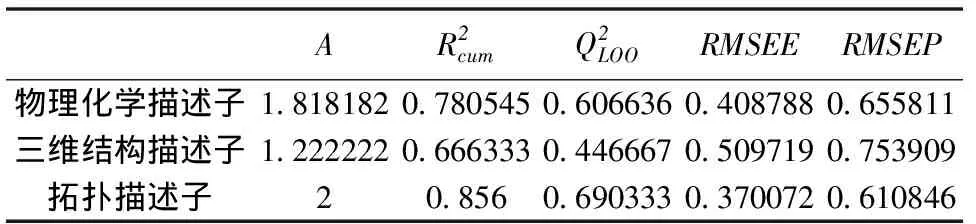
表5 舒缓肽促进剂用3类描述子表征后建模统计量平均值

表6 后叶催产素用3类描述子表征后建模统计量平均值

表7 抗原肽用3类描述子表征后建模统计量平均值
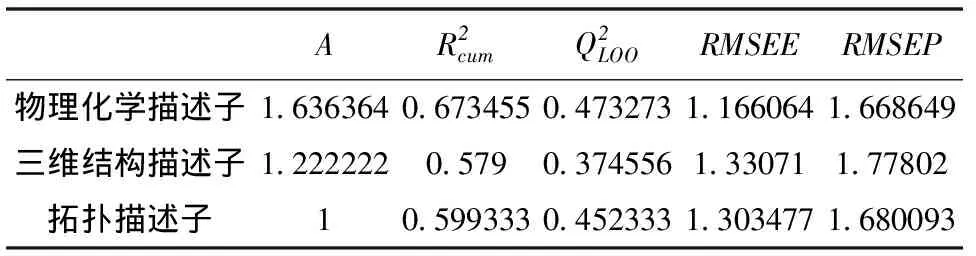
表8 CAMELs用3类描述子表征后建模统计量平均值
2.3 不同年代描述子性能比较
7种肽集用27种氨基酸描述子表征得到建模统计量折线图,如图2所示。横坐标为按年代依次发表的氨基酸描述子,纵坐标为每个描述子对应的5个统计量的数值。可以看出随着描述子发表的年代的推进,后发表的描述子并不一定比先发表的描述子有质的进步,它们的建模效果是呈现一个波动状态的。

表9 三肽用3类描述子表征后建模统计量平均值
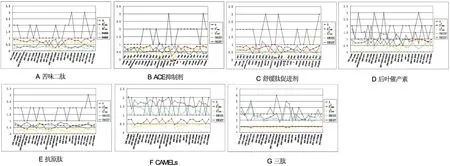
图2 7种肽集用27种描述子表征所得建模统计量折线图
Fig 2 The statistics of line graph of seven peptides sets with 27 descriptors to characterize

表10 7组肽集各建模统计量的平均值
3 结论
本文通过利用已经发表的27种描述子表征不同肽集,得出不同种类氨基酸描述子的建模统计量数值。通过比较分析这些建模统计量,可以看出不同年代的描述子的建模效果并没有实质性的进步,物理化学描述子的建模效果优于拓扑描述子,拓扑描述子的建模效果优于三维结构描述子。我们认为氨基酸描述子自身固有的问题限制了它们对多肽QSAR模拟效果的实质性提高,传统的方法并未考虑肽链内部氨基酸之间的交互效应,表征都是建立在真空模型基础之上,未考虑肽与周围环境之间的影响,如肽配基与相应的靶点蛋白相互结合后的构效变化等因素,本课题也将对此展开后续研究。
参考文献:
[1]覃礼唐, 刘树森, 肖乾芬, 等. QSAR模型内部与外部实验方法综述[J]. 环境化学, 2013, 32(7): 1026-1211.
[2]丁俊杰, 丁晓琴, 赵立锋, 等. 多肽定量构效关系与分子设计[J]. 化学进展, 2005, 17(1): 131-136.
[3]车 挺.新型氨基酸描述子及其在肽QSAR中的应用[D]. 西安: 陕西科技大学, 2012.
[4]Tian F F, Yang L, Lv F L. In silico quantitative prediction of peptides binding affinity to human MHC molecule: an intuitive quantitative structure-activity relationship approach[J]. Amino Acid, 2009, 36, 535-554.
[5]Tian F F, Zhou P, Li Z L J. T-scale as a novel vector of topological descriptors for amino acids and its application in QSARs of peptides[J].Mol Struct, 2007, 830: 106-115.
[6]梅 虎, 周 原, 孙立力, 等. 一种新的氨基酸描述子及其在肽QSAR中的应用[J]. 物理化学学报, 2004, 20(8): 821-825.
[7]Mei H, Liao Z H, Zhou Y, et al. A new set of amino acid descriptors and its application in peptide QSARs[J]. Pept Sci, 2005, 80: 775-786.
[8]梁桂兆, 李志良, 周 原, 等. 氨基酸广义疏水标度(GH-scale)用于HLA-A*0201限制性CTL表位定量预测[J]. 科学通报, 2006, 51(11): 1259-1263.
[9]Yang L, Shu M, Ma K W. ST-scale as a novel amino acid descriptor and its application in QSAM of peptides and analogues[J].Amino Acid, 2010, 38: 805-816.
[10]Shu M, Huo D Q, Mei H. New descriptors of amino acids and its application to peptide quantitative structure-activity relationship[J]. Chinese J Struct Chem, 2008, 27(11): 1375-1383.
[11]Liang G Z, Yang L, Kang L F. et al. Aset of new aminoacid descriptors applied in prediction of MHC class I binding peptides[J]. Eur J Med Chem, 2009, 44: 1144-1154.
[12]杨善彬, 夏之宁, 舒 茂, 等. 氨基酸描述子VHSEH用于多肽定量序效建模研究[J].高等学校化学学报, 2008, 29(11): 2213-2217.
[13]剑 秋, 刘 静, 管 骁. 一种新的氨基酸描述符SVHEHS在生物活性肽QSAR中的应用研究[J].食品科学, 2012, (337): 26-31.
[14]Hellberg S, Eriksson L, Jonsson J. Minimum analogue peptidesets(MAPS) for auantitative structure-activity relationships. Int[J]. Protein Res. 1991, 37, 414-424.
[15]Lin Z H, Long H X, Bo Z, et al. New descriptors of amino acids and their application to peptide QSAR study[J].Pept. 2008, 29: 1798 1805.
[16]Hu M, Mei H, Yang S B, et al. Structural parameter characterization and bioactivity simulation based on peptide sequence[J].QSAR Comb. Sci. 2009, 28: 27-35.
[17]Wang X Y, Wang J, Lin Y, et al. QSAR study on angiotensin-converting enzyme inhibitor oligopeptides based on a novel set of sequence information descriptors[J]. Mol Model, 2011, 17: 1599-1606.
[18]Kim J, Nam K Y, Cho K H, et al. Theoretial study on hydrophobicity of amino acid by the salvation free energy density model[J].Bull Krorean Chem Soc, 2003, 24(12): 1742-1750.
[19]仝建波, 张生万. 一种新的三维氨基酸描述子及其在肽类药物QSAR中的应用[J].物理化学学报, 2007, 23(1): 37-43.
[20]仝建波, 张生万, 成素丽, 等. 三维氨基酸结构描述子矢量SVRDF及其在肽QSAR中的应用[J].药学学报, 2007, 42(1): 40-46.
[21]仝建波, 刘淑玲, 刘玉婷, 等.氨基酸描述子SVG及其在肽序列QSAR中的应用[J].精细化工, 2008, 25(7): 655-659.
[22]仝建波, 刘淑玲, 来水利, 等.一种新三维氨基酸描述子SVTD及在肽QSAR的应用[J].分析科学学报, 2008, 24(5):522-526.
[23]丁俊杰, 丁晓琴, 赵立峰, 等. 新型三维氨基酸结构描述符的研究及其在多肽QSAR中的应用[J].药学学报, 2005, 40(4): 340-346.
[24]Collantcs E R, Donn W J S J.Amino acid side chain descriptors for quantitative structure-activity relationship studies of peptide analogues[J]. Med Chem, 1995, 38: 2705-2713.
[25]Zaliani A, Gancia E. MS-WHIM scores for amino acids: a new 3D-Description for peptide QSAR and QSPR studies[J]. Chem Inf Comput Sci, 1999, 39: 525-533.
[26]Tong J B, Che T, Li Y F, et al. A descriptor of amino acids: SVRG and its application to peptide quantitative structure activity relationship. SAR and QSAR in Environmental Research. 2011, 22(5-6): 611-620.
[27]Tong J, Liu S, Zhou P, et al. A novel descriptor of amino acids and its application in peptide QSAR[J]. Theor Biol, 2008, 253: 90-97.
[28]Cocchi M, Johansson E. Amino acids characterization by GRID and multivariate data analysis[J]. Quant Struct Act Relat, 1993, 12: 1-8.
[29]Tong J B, Che T, Liu S L, et al. Arch Pharm SVEEVA descriptor application to peptide QSAR[J]. Chem Life Sci, 2011, 344: 719 725.
[30]梁桂兆, 李志良, 周 原, 等.一组新氨基酸描述子用于肽定量构效关系研究[J]. 化学学报, 2006, 64(5): 393-396.
[31]田菲菲. 肽结构表征及统计建模方法学研究与应用[D]. 重庆: 重庆大学, 2011.

