辽宁满族人群19个STR基因座的遗传多态性及其与28个群体的遗传关系研究
祁荣,刘鹏,郭丽,李晓亮,郑瑞,刘萍,王栾,刘健
(1.沈阳医学院中心实验室,沈阳110034;2.沈阳市沈北新区黄家锡伯族九年一贯制学校,沈阳110121;3.北京武警总队第三医院,北京100141)
辽宁满族人群19个STR基因座的遗传多态性及其与28个群体的遗传关系研究
祁荣1,刘鹏2,郭丽1,李晓亮3,郑瑞1,刘萍1,王栾1,刘健1
(1.沈阳医学院中心实验室,沈阳110034;2.沈阳市沈北新区黄家锡伯族九年一贯制学校,沈阳110121;3.北京武警总队第三医院,北京100141)
目的调查辽宁地区满族无关个体的19个短串联重复序列(STR)基因座多态性,选择具有高度遗传多态性与稳定性的13个STR位点进行辽宁满族与来自28个不同国家和地区的人群之间的群体遗传关系分析,研究其在法医学个体识别、亲子鉴定和人类学研究中的应用价值。方法应用GoldeneyeTM20A五色荧光标记系统对采集的150例口腔黏膜细胞样本进行19个基因座的复合扩增,应用ABI3130XL全自动测序仪对扩增产物进行检测,采用GeneMapper v3.2软件对其进行基因分型。用Neighbor-Joining法构建系统发生树,并结合有关资料分析他们之间的遗传关系。结果GoldeneyeTM20A系统的19个STR基因座在辽宁地区满族人群的累积个体识别能力(TDP)为0.999 999 999 999 999 999 994 2,累积非父排除率(CPE)为0.999 999 996 777。结论辽宁满族的19个STR位点具有高度遗传多态性,可用于辽宁地区满族人群法医学个体识别、亲权鉴定及人类学研究。
STR基因座;基因频率;遗传多态性;满族
满族是中国最古老的民族之一,是继元朝蒙古族后,在全国范围内建立起统一王朝——清朝的又一个少数民族。满族的最早祖先是先秦时期生活在东北广大地区的肃慎,然后经历挹娄、勿吉、靺鞨、女真等不同时期。1635年,皇太极正式将女真改称为满族[1]。2010年人口普查时,满族人口为1 038万。在中国55个少数民族中仅次于壮族和回族位居第3位。
对于辽宁满族的15个短串联重复序列(short tandem repeat,STR)位点的遗传多态性曾有报道[2],但本文采用北京基点认知公司GoldeneyeTM20A荧光标记复合扩增系统在15个STR位点的基础上,又增加了4个STR位点,即对满族人群的19个STR位点(D8S1179,D21S11,D7S820,CSFIPO,D3S1358,TH01,D13S317,D16S539,D2S1338,D19S433,vWA,TPOX,D18S51,D5S818,FGA,PentaD,PentaE,D6S1043,D12S391)进行基因频率调查,获得该地区满族群体遗传多态性参数,为法医学个体识别、亲权鉴定以及群体遗传学研究等提供重要的理论依据。
1 材料与方法
1.1 材料收集
根据“知情同意”的原则,随机选取辽宁省北镇市满族中学的150名无关健康知情个体,保证其3代以上均为满族。采集受试者口腔黏膜细胞,Chelex100法提取DNA分子[3]。
1.2 PCR扩增
采用北京基点认知公司GoldeneyeTM20A PCR扩增试剂盒[4],对D8S1179,D21S11,D7S820,CSF1PO,D3S1358,TH01,D13S317,D16S539,D2S1338,D19S433,vWA,TPOX,D18S51,D5S818,FGA,Penta D,Penta E,D6S1043和D12S391基因座进行复合扩增,PCR反应在ABI9700型扩增仪上进行。PCR反应总体系为10 μL,其中PCR反应混合物3.84 μL,引物2 μL,Taq聚合酶0.16 μL(5 U/μL),模板DNA适量,加去离子水补足至10 μL。反应条件见试剂盒说明。
1.3 扩增产物电泳及检测
扩增产物经甲酰胺变性后,ABI3130XL型遗传分析仪上进行电泳,使用Data Collection 2.0软件进行数据收集,GeneMapper ID v3.2软件进行基因型分型。
1.4 统计学处理
应用PowerStats V1.2软件[5]计算每个基因座的基因频率、个人识别率(power of discrimination,PD)、多态信息总量(polymorphism information content,PIC)、非父排除率(probability of exclusion,PE)、个人匹配概率(matching probability,PM)和实际杂合度(observed heterozygosity,Ho)。应用公式计算期望杂合度(expected heterozygosity,He),累积非父排除能力(cumulative probability of exclusion,CPE)和累积个人识别率(cumulative power of discrimination,TDP)。应用HWE 1.20软件进行Hardy-Weinberg遗传平衡检验。应用Poptree2软件[6]构建系统发生树。
2 结果
2.1 受试者的等位基因频率分布
辽宁满族群体19个STR位点的等位基因及基因频率分布见表1。对此19个STR基因座的基因型频率进行了Hardy-Weinberg遗传平衡检验,检验结果为除了D13S317位点,其他18个位点的基因型分布均符合Hardy-Weinberg平衡定律。

表1 辽宁地区满族19个STR基因座基因频率(n=150)Tab.1 Allele frequencies of the 19 autosomal STR loci of the Manchu population in China(n=150)
2.2 受试者19个STR位点的法医学应用参数
在辽宁满族群体中19个STR基因座的Ho,He,PD,PIC,PE及PM见表2。
2.3 29个群体间的系统发生树
用Neighbor-Joint法对来自29个不同国家和地区的群体构建系统发生树(图1),发现来自亚洲的
22个群体聚为一大类,而来自非洲、欧洲、拉丁美洲和大洋洲的7个群体聚为另一大类[7~13]。

续表1
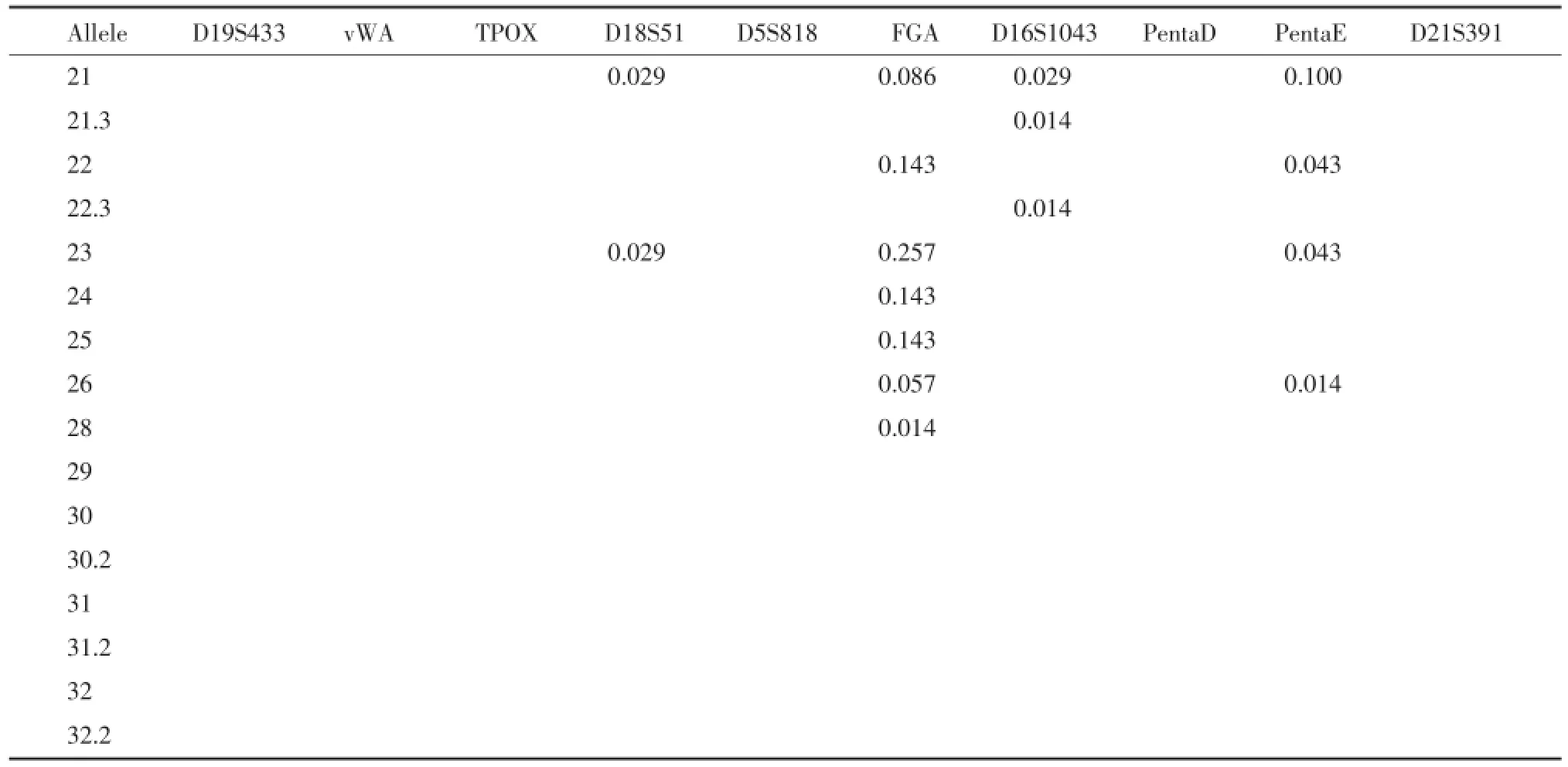
续表1
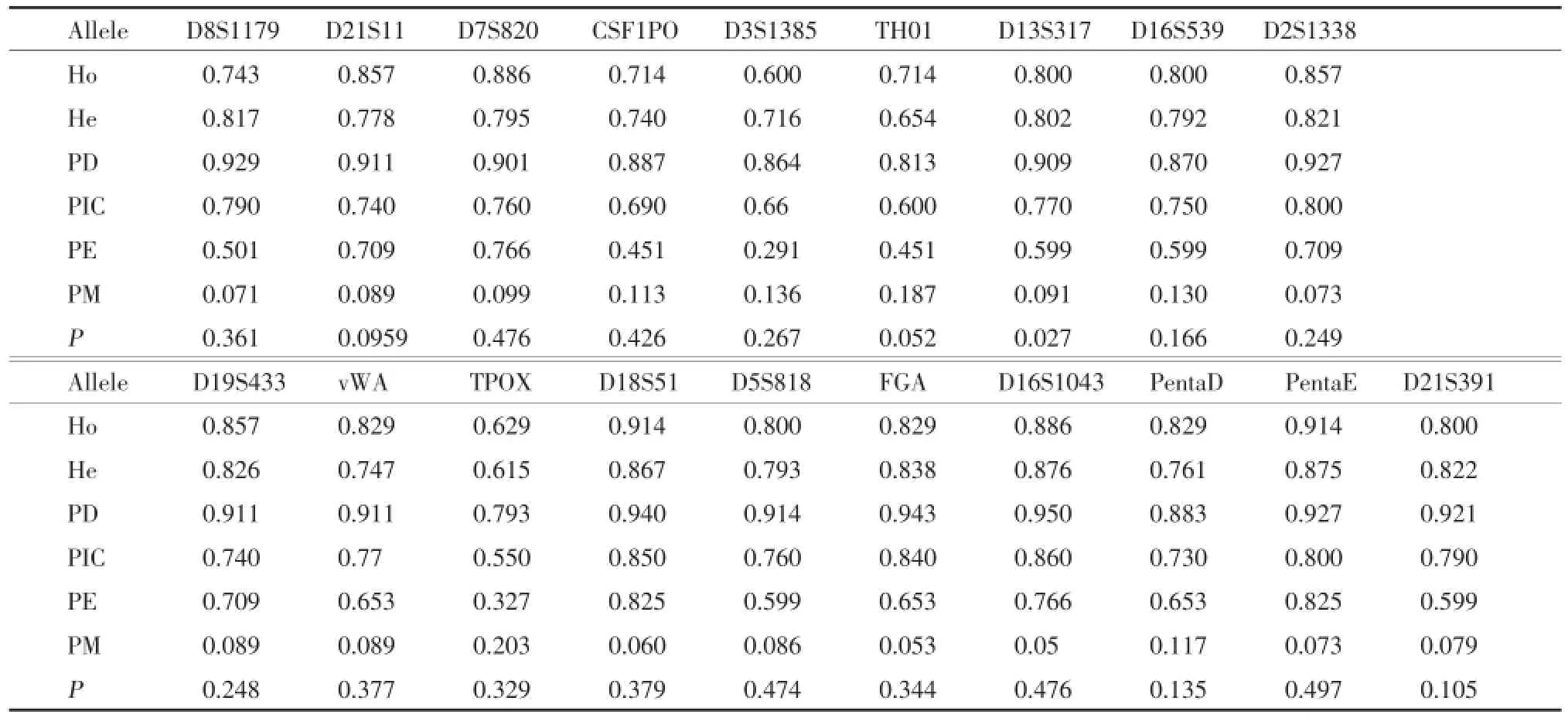
表2 辽宁满族群体19个STR基因座遗传学指标(n=150)Tab.2 The statistical parameters of the 19 autosomal STR loci of the Manchu population in China(n=150)
3 讨论
3.1 辽宁满族各遗传学指标
本研究共发现154个等位基因,相应的等位基因频率0.010~0.529。辽宁满族群体的各基因座Ho值0.6~0.914,均值为0.803;He值0.615~0.876,均值为0.786;该检测系统TDP值为0.999 999 999 999 999 999 994 2,CPE值达0.999 999 996 777,符合人类STR位点高识别能力的标准。因此,辽宁满族的调查结果在满族人群法医学个人识别及亲权鉴定中具有很高的应用价值。
3.2 群体间遗传学关系力
语言是民族的特征之一,民族之间的贸易往来、文化交流、移民杂居、战争征服都离不开语言的参与。满族人所使用的语言属阿尔泰语系满—通古斯语族满语支。满族人所使用的文字是16世纪末参照蒙古文字母创制的。29个群体构成的系统发生树的结果显示辽宁满族与蒙古族、日本民族、韩国民族、鄂伦春民族及鄂温克民族聚为一类。这些民族所使用的语言都属于阿尔泰语系。据史料记载满族是以满族为主体,在长期共同生活中吸收和融合一部汉族人,蒙古族人和朝鲜族人而组成的新的民族共同体[1]。从本文所构建的进化系统树发现满族、蒙古族和朝鲜族聚为一类,说明他们之间确实具有较近的亲缘关系,从而证实了这一史实。而对于满族、鄂伦春族及鄂温克族的语言同属于阿尔泰语系、通古斯语族,而且,有一种观点认为满族、鄂伦春族及鄂温克族源自共同祖先。本文所构建的遗传进化系统树表明此3个民族确实具有较近的亲缘关系,使这一观点得到进一步证实。另外,广西境内7个少数民族聚为一类,这7个少数民族所使用的语言同属于汉藏语系,同时还可能是广西少数民族长期杂居在一起,各族之间存在广泛的基因交流,致使他们之间遗传距离变小。来自摩洛哥和非洲裔的美国人首先聚为一类然后与来自非洲的突尼斯人聚类,从所使用的语言上,他们都使用阿拉伯语,属于闪含语系闪语族,为全世界穆斯林的宗教语言;从地理位置上他们都来自非洲,虽然非洲裔的美国人现住在美国,但是他们的祖先属于非洲人,所以他们与来自非洲的两个群体间有较近的亲缘关系。从本研究结果可以发现遗传上的分化与语言分化具有很高的一致性。进一步验证了在不同的人群进化过程中,地理、语言和宗教都会对群体的遗传结构产生影响。
[1]张新科,杜文玉,白玉林.清史解读[M].云南:教育出版社,2011:1-2.
[2]徐国昌,朱艳,任甫,等.辽宁满族15个短串联重复序列基因座遗传多态性[J].解剖学杂志,2010,33(6):825-827.
[3]Walsh PS,Metzger DA,Higuchi R.Chelex 100 as a medium for simple extraction of DNA for PCR-based typing from forensic material[J].Biotechniques,1991,10(4):506-513.
[4]Jiang X,Guo F,Jia F,et al.Development of a 20-locus fluorescent multiplex system as a valuable tool for national DNA database[J]. Forensic Sci Int Genet,2013,7(2):279-289.
[5]PowerStats,A computer program for the analysis of population statistics;1999.(free program distributed by the authors over the internet from http://www.promega.com/geneticidtools/)
[6]Takezaki N,Nei M.Genetic distances and reconstruction of phylogenetic trees from microsatellite DNA[J].Genetics,1996,144(1):389-399.
[7]Rerkamnuaychoke B,Rinthachai T,Shotivaranon J,et al.Thai population dat a on 15 tetrameric STR loci D8S1177S820,D21S11,D7S820,CSF1PO,D3S1358,THO1,D13S317,D16S539,D2S1338,D19S433,VWA,TPOX,D18S51,D5S818 and FGA[J].Fore Sci Inter,2006,158:234-237.
[8]Tie J,Wang X,Oxida S.Genetics polymorphisms of 15 STR loci in a Japanese population[J].J Forensic Sci,2006,51(1):188-189.
[9]Butler JM,Schoske R,Vallone PM,et al.Allele frequencies for 15 autosomal STR loci on U.S.Caucasian,African American,and Hispanic populations[J].J Forensic Sci,2003,48(4):908-911.
[10]Rebała K,Wysocka J,Kapińska E,Belarusian population genetic database for 15 autosomal STR loci[J].Forensic Sci Inter,2007,173:235-237.
[11]Cherni L,Loueslati Yaâcoubi B,Pereira L,et al.Data for ausosomal STR markers from two Tunisian populations:Kesra(Berber)and Zriba(Arab)[J].Forensic Sci Inter,2005,147(1):101-106.
[12]Abdin L,Shimada I,Brinkmann B,et al.Analysis of 15 short tandem repeats reveals significant differences between the Arabian populations from Morocco and Syria[J].Legal Med,2003,5:150-155.
[13]Qiu GR,Qiu GB,Gong LG,et al.Genetic Analysis of 17 STR Loci in Chinese Han Population from Liaoning[J].Chin J Forensic Med,2011,26(3):225-227.
[14]Shriver MD,Jin L,Boerwinkle E,et al.A novel measure of genetic distance for highly polymorphic tandem repeat loci[J].Mol Biol Evol,1995,12(5):914-920.
[15]Gill P,Urquhart A,Millican E,et al.A new method of STR interpretation using inferential logic development of a crimininal intelligence database[J].Int J Leg Med,1996,109(1):14-22.
(编辑 裘孝琦)
Genetic Polymorphism of19 STRLociin Manchu Nationality in Liaoning and Their Genetic Relationship with Other 28 Populations
QIRong1,LIU Peng2,GUOLi1,LIXiao-liang3,ZHENGRui1,LIUPing1,WANG Luan1,LIUJian1
(1.Central Laboratory,Shenyang Medical College,Shenyang 110034,China;2.Shenyang Xinchengzi District Huangjia Xibe Middle School,Shenyang 110121,China;3.Third HospitalofBeijing Armed Police Force,Beijing 100141,China)
ObjectiveTo investigate the genetic polymorphism of 19 short tandem repeat(STR)loci in Liaoning Manchu population with GoldeneyeTM20A multiplex amplification system,and to determine the molecular genetic relationship of Liaoning Manchu with other 28 populations around the world.MethodsDNA samples were obtained from 150 unrelated individuals.PCR products of the 19 loci were analyzed by ABI3130XL sequencer.The genetic distances among 29 differentpopulationswere calculated by Nei’smethod.The phylogenetic tree was constructed using Neighbor-Joining method.ResultsThese loci were highly polymorphic.The combined power of discrimination is 0.999 999 999 999 999 999 994 2 and the combined paternity of exclusion is 0.999 999 996 777.ConclusionThe 19 STR loci of Manchu nationality in Liaoning possess the characteristics of high genetic diversity,which can be used for anthropological and other comparative studies of populations,and is also useful for forensic and paternity testing ofthe Manchu population in China.
STR locus;gene frequency;genetic polymorphism;Manchu nationality
R394
A
0258-4646(2014)06-0542-05
祁荣(1973-),女,高级实验师,本科.
刘健,E-mail:liujian910709@163.com
2014-01-07
网络出版时间:

