重叠延伸PCR法克隆蓖麻蚕β-FFase同源基因
张蕾 李静 戴伟宏 孟艳
(安徽农业大学生命科学学院 230036)
蓖麻蚕(Samia cynthia ricini)是一种与家蚕亲缘关系较近的野蚕,食性广,但不吃桑叶。桑叶乳汁中含有大量DNJ、D-AB1等生物碱,对S.c.ricini等非食桑的昆虫具有强烈的毒性,阻碍其生长以致死亡[1,2]。生物碱主要抑制昆虫肠道的蔗糖水解酶的活性,从而达到阻碍昆虫正常生长发育的毒性效果[3,4]。
β-呋喃果糖苷酶(β- fructofuranosidase,β-FFase)是一种蔗糖水解酶,在植物和微生物中广泛存在,家蚕BmSUC1是第一个被分子克隆和功能鉴定的动物β-FFase[5]。家蚕BmSuc1在家蚕幼虫中肠的杯状细胞大量表达,其编码的酶蛋白具有典型的蔗糖水解酶功能,且几乎不受 DNJ的抑制作用[4]。BmSUC1的发现和功能鉴定,对于解读家蚕为何能够巧妙地回避桑树生物碱毒害作用的酶学适应机制具有重要的科学意义。作为家蚕的近缘物种,S.c.ricini不以桑叶为饲料,桑叶生物碱对其生长和发育具有较强的阻碍作用[5],而我们发现S.c.ricini的基因组中同样存在β-FFase同源基因,并已克隆获得可拼接为全长cDNA的各个片段。Sc-Suc1和ScSuc2对于 S.c.ricini而言是否具有功能,其生物学意义如何,值得研究。
重叠延伸PCR(gene splicing by overlap extension PCR,简称SOE PCR)技术是采用具有互补末端的引物,使PCR产物形成重叠链,从而在随后的扩增反应中通过重叠链的延伸,将不同来源的扩增片段重叠拼接起来的技术[6]。该技术操作简便,并且可以获得内切酶消化和连接酶处理的方法难以得到的产物[8]。如今SOE PCR已经成为分子生物学研究中的常用方法,应用非常广泛,如扩增长片段DNA,基因定点诱变,构建融合基因等[7,8]。
笔者利用SOE PCR的方法,对S.c.ricini的β-FFase基因 ScSuc1 和 ScSuc2 的 5'末端、3'末端以及中间片段分别进行了扩增和拼接,以期获得S.c.riciniβ-FFase同源基因的完整开放阅读框(open reading frame,ORF),为将来进行基因的体外表达及其功能研究提供基础。
1 材料与方法
1.1 材料
连接有ScSuc1和ScSuc2 cDNA各个片段的pGEM-T Easy重组载体为本实验室前期所构建。大肠杆菌DH5α感受态细胞购自北京全式金公司。DNA Marker、克隆载体pMD-19T试剂盒、PCR反应试剂盒购自TAKARA公司。DNA纯化回收试剂盒购自北京艾德来公司。琼脂糖购自Invitrogen公司。氨苄青霉素(ampicillin)、LB培养基购自上海生工公司。
1.2 方法
1.2.1 引物设计
为了拼接得到ScSuc1和ScSuc2全长ORF,我们根据实验室前期克隆的ScSuc1-5’(上游片段)、ScSuc1-dgPCR(中间片段)、ScSuc1-3’(下游片段)和 ScSuc2-5’(上游片段),ScSuc2- dgPCR(中间片段),ScSuc2-3’(下游片段)的序列,设计了6对PCR扩增引物(表1)。

表1 PCR引物序列及扩增目的
1.2.2 PCR 扩增各模板 DNA 片段
以含有ScSuc1或ScSuc2cDNA各个片段的pGEM-TEasy重组质粒DNA为模板,用表1所示的引物分别扩增ScSuc1和ScSuc2的上游、中间和下游片段,使得拼接片段的重叠部分的碱基个数合适。50uLPCR反应体系中包含5×PrimSTARBuffer 10uL,1.5UPrimSTARHSDNA聚合酶,0.2 ~0.5 μg模板 DNA,终浓度为10umol/L的dNTPMix和0.2umol上下游引物(F和R),最后加 ddH2O补齐至50μL。PCR反应程序为:94℃预变性5min后接30个循环,每个循环包括98℃变性10s、50℃退火10s和72℃延伸40s,循环结束后72℃反应10min。
1.2.3 连接、转化与测序
PCR反应结束后,将扩增得到的PCR产物经1.0%的琼脂糖凝胶电泳检测,并用DNA纯化回收试剂盒回收目的片段。按TAKARA公司T-载体试剂盒说明书将纯化的PCR产物连接到pMD-19T载体,然后用连接产物转化大肠杆菌DH5α感受态细胞,用含100mg/LAmp的LB平板培养细胞,并筛选阳性克隆。挑取单菌落由Invitrogen公司测序,进行序列确认。
1.2.4 SOEPCR
获得重叠部分碱基个数合适且序列无误的各片段后,利用SOEPCR法将各片段进行拼接。首先以上游和中间片段1:1混合作为模板进行第一轮拼接,将拼接后得到的PCR产物经切胶回收纯化后,按1.2.3所述方法进行克隆和测序确认,序列正确后再与下游片段混合作为模板,进行第二轮拼接,最终得到完整的ORF。
SOEPCR的反应体系为50μL,包含5×Prim-STARBuffer10uL,1.5UPrimSTARHSDNA 聚合酶,两个模板 DNA 各 0.1 ~ 0.3μg,终浓度为10umol/L 的 dNTPMix和 0.2μmol上下游引物(F和R),最后加ddH2O补齐至50μL。PCR反应程序为:94℃预变性5min后接30个循环,每个循环包括98℃变性 10s、46℃退火(第一轮。第二轮为55℃)10s和72℃延伸1min,循环结束后72℃反应10min。第二轮PCR反应结束后,再次按1.2.3所述方法对最终获得的PCR产物进行克隆和测序,确认基因全长ORF序列信息。
2 结果和分析
2.1 用于SOEPCR的DNA模板的扩增
为了获得SOEPCR拼接用的模板DNA,我们根据实验室前期克隆所得到S.c.riciniβ-FFase同源基因ScSuc1和ScSuc2的cDNA序列设计了6对引物(表1),以包含各个基因片段的pGEM-TEasy重组质粒DNA为模板,用常规PCR的方法分别对每个基因的上游、中间和下游片段进行了扩增反应。拼接前各个基因片段的预期大小如表2所示。PCR产物经琼脂糖凝胶电泳分析,所得片段长度与预期完全相符(图1,图2,泳道1~3)。对各片段进行切胶回收,并与T载体连接测序,结果表明各片段的碱基序列正确,并且在上游片段(5’)末端、中间片段(dgPCR)前端以及中间片段末端、下游片段(3’)前端均有重叠个数合适的碱基。

表2 拼接前后各基因片段的预期大小(bp)
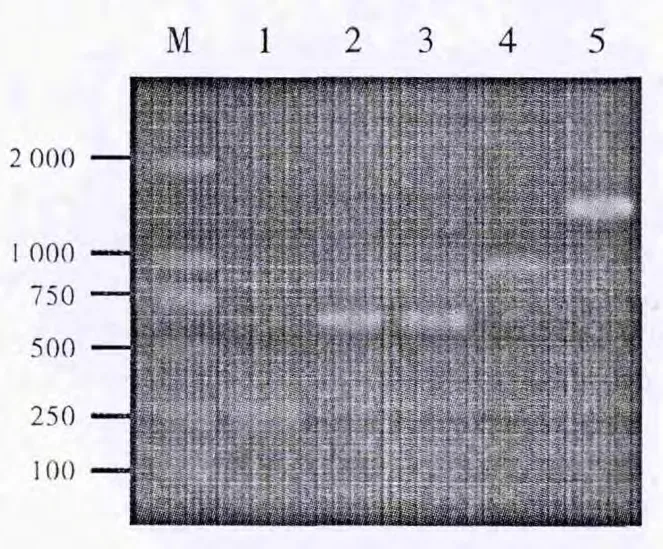
图1 ScSuc1的扩增和拼接
2.2 SOEPCR对基因ORF的拼接
如图1所示的拼接流程,我们首先对ScSuc1的各片段进行拼接。以ScSuc1上游和中间片段为混合模板,5’F和dgPCRR为上下游引物进行第一轮拼接,预计拼接片段大小为868bp(表2)。SOE PCR产物的琼脂糖凝胶电泳结果显示,所得片段长度与ScSuc15’-dgPCR的长度一致(图1,泳道4)。经切胶回收和克隆测序分析表明,碱基的序列及大小均无误。然后以ScSuc15’-dgPCR和ScSuc1下游片段为混合模板,5’F和3’R为上下游引物进行第二轮SOEPCR拼接,片段大小预期为1464bp(表2)。PCR产物的琼脂糖凝胶电泳结果显示,所得片段长度与ScSuc15’-3’长度一致(图1,泳道5)。经切胶回收和克隆测序分析表明,我们成功地获得了ScSuc1的完整ORF。按照相同的方法和步骤,我们得到了ScSuc2的完整ORF全长片段。如图2所示,泳道4为第一轮SOEPCR的拼接产物ScSuc25’-dgPCR,泳道5为第二轮拼接产物Sc-Suc25’-3’。
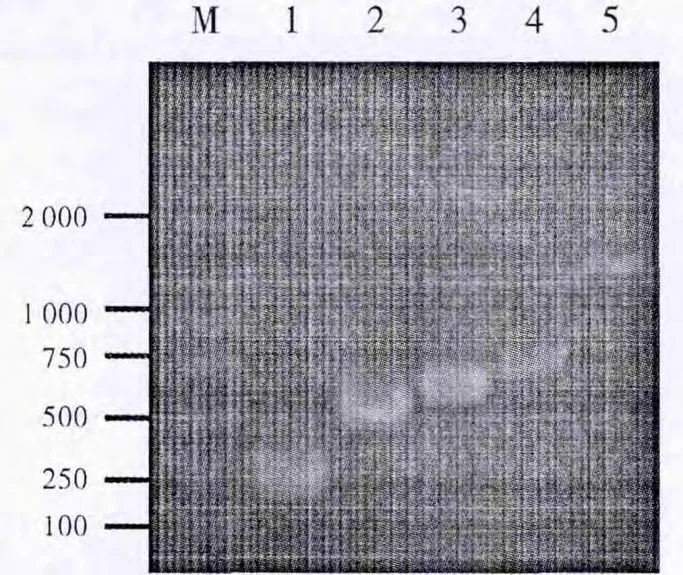
图2 ScSuc2的扩增和拼接
3 讨论
蓖麻蚕为非食桑昆虫,我们的前期研究发现虽然在S.c.ricini的基因组中存在β-FFase同源基因ScSuc1和ScSuc2,但2个基因在体内的转录水平都很低,难以通过反转录PCR的方法直接得到全长cDNA。本研究利用SOEPCR的方法,把前期得到的ScSuc1和ScSuc2的5'端、中间片段以及3'端各个cDNA片段成功拼接起来,分别获得了两个基因的完整ORF片段(图1,图2),很好地解决了因转录水平低而带来的直接克隆基因的困难,为类似问题提供方法借鉴。另外,本研究为今后开展基因的体外表达及研究S.c.ricini中β-FFase基因是否具有蔗糖水解酶的活性功能以及酶的活力特性分析等研究奠定了必要的实验基础。
[1]Konno K,Ono H,Nakamura M,et al.Mulberry latex rich in antidiabetic sugar-mimic alkaloids forces dieting on caterpillars.Proc.Natl.Acad.Sci.U.S.A.2006,103:1337 ~1341.
[2] Hirayama C,Konno K,Wasano N,et al.Differential effects of sugar-mimic alkaloids inmulberry latex on sugarmetabolism and disaccharidases of Eriand domesticated silkworms:enzymatic adaptation of Bombyxmori tomulberry defense.Insect Biochem.Mol.Biol.2007,37:1348 ~1358.
[3]Asano N,Yamashita T,Yasuda K,etal.Polyhydroxylated alkaloids isolated from mulberry trees(Morus alba L.)and silkworms(Bombyx mori L.).J.Agric.Food Chem.2001,49:4208~4213.
[4]Kite G C,Scofield A M,Lees D C,et al.Alkaloidal glycosidase inhibitors and digestive glycosidase inhibition in specialist and generalistherbivores of Omphalea diandra.J.Chem.Ecol.1997,23:119 ~135.
[5]Daimon T,Taguchi T,Meng Y,et al.Beta-fructofuranosidase genes of the silkworm,Bombyx mori:Insight into enzymatic adaptation of B.mori to toxic alkaloids in mulberry latex.J.Biol.Chem.2008,283:15271~15279.
[6]Horton R M,Cai Z L,Ho SN,et al.Gene splicing by overlap extension:tailor-made genes using the polymerase chain reaction,BioTechniques,1990,8:528~535.
[7]徐芳,姚泉洪,熊爱生,等.重叠延伸PCR技术及其在基因工程上的应用[J].分子植物育种,2006(4):747~750.
[8]李守宇.重叠延伸PCR技术及其在生物学中的应用[J].生物学教学,2013(4):65~66.

