随机扩增多态性法分析西藏牦牛奶酪中乳酸菌的遗传多样性
蒋厚阳,赵国华,2,杨吉霞,2,*
(1.西南大学食品科学学院,重庆 400715;2.重庆市农产品加工技术重点实验室,重庆 400715)
随机扩增多态性法分析西藏牦牛奶酪中乳酸菌的遗传多样性
蒋厚阳1,赵国华1,2,杨吉霞1,2,*
(1.西南大学食品科学学院,重庆 400715;2.重庆市农产品加工技术重点实验室,重庆 400715)
目的:利用随机扩增多态性(randomly amplified polymorphic DNA,RAPD)法对西藏地区牦牛奶酪中27 株乳酸菌基因型进行同源性分析。方法:利用5 个随机引物对Mg2+浓度、dNTP用量、退火温度、模版用量/引物用量(ng/pmol)4 个条件做单因素梯度试验,建立最佳反应条件,筛选最佳引物,然后对27 株乳酸菌和4 株乳酸菌标准菌株进行随机扩增,用NTsys 2.10e软件对扩增条带进行聚类和遗传相似性系数分析,分析结果与16S rRNA测序得到的菌种鉴定结果进行对比。结果:31 株菌 遗传相似系数在0.72~1.00之间,当相似性系数在0.82时,菌株被分成了8 组,菌株按照不同种属聚类,聚类结果同16S rRNA测序结果基本一致,同时成功将Lactobacillus casei和Lactobacillus paracasei两个亚种区 分开。结论:RAPD技术可以较好地应用于西藏地区牦牛奶酪中乳酸菌亲缘性关系分析。
西藏;牦牛奶酪;乳酸菌;随机扩增多态性;聚类分析
西藏牦牛素有“高原之舟”的美称,是我国的重要畜种。在高原地区牦牛乳及其制品是当地居民的重要食品。目前对于牦牛乳及其成分的报道日渐增多,但是对于牦牛奶酪中乳酸菌的报道却很少。
乳酸菌作为奶酪发酵过程中的关键菌种,对其进行分类鉴定有着极其重要的意义。
随机扩增多态性(randomly amplified polymorphic DNA,RAPD)技术最初是由美国科学家Williams[1]和Welsh[2]等运用于基因多态性分析中。它应用短的随机引物,通过聚合酶链式反应(polymerase chain reaction,PCR)随机扩增后产生特异性的DNA图谱,具有多态性高、操作简单,引物没有严格限制,可以实现高通量测定等优点。扩增图谱重现性不高是RAPD面临的一个问题,但是通过PCR反应程序和体系的优化,引物的筛选可以解决图谱重现性这个问题[3-4]。目前很多国外学者将RAPD技术应用于乳酸菌基因型的研究中,如乳酸球菌属[5](Lactococcus)、明串珠菌属[6](Leuconostoc)、乳杆菌属[7](Lactobacillus)、肠球菌属[8](Enterococcus),很好地区别开亲缘性较近的菌种。但是国内只有很少文献报道了RAPD在乳酸菌基因分型中的应用。如高大威等[9]利用RAPD技术对鲜牛奶、西红柿和酸菜中分离出的乳酸菌进行聚类分析,确定了乳酸菌间亲缘性关系。秦丹[10]利用RAPD方法对四川腊肉和香肠在加工和贮藏过程中的乳酸菌进行分析,并将结果与传统微生物培养法进行比较,两种方法得到的结果不完全相同。
本研究通过筛选出随机引物M13,单因素试验获得较好的图谱重现性后对27 株分离纯化后的乳酸菌和4 株乳酸菌标准菌株进行RAPD扩增,用NTsys 2.10e软件对扩增后图谱进行聚类分析,将聚类结果与各个菌株产地进行归类对比,同时与16S rRNA序列分析后的菌种鉴定结果结果进行对比,从而探讨RAPD技术在天然乳酸菌菌种分类鉴定方面的应用价值。
1 材料与方法
1.1 菌种、培养基与试剂
供试菌株分离自西藏牦牛奶酪,由西藏农牧学院提供(表1)。标准菌株:Lactobacillus casei ATCC 393,Lactobacillus paracasei ATCC BAA-52,Lactobacillus plantarum ATCC 8014,Lactobacillus rhamnosus ATCC 7469,购于青岛海博生物技术有限公司。

表1 供试菌株Table1 Strains isolated from yak milk cheeses

续表1
MRS培养基的配制参照参考文献[11]。
Lambda DNA/Hind Ⅲ Marker 北京鼎国生物科技有限公司;Taq酶(5 U/μL)、dNTPs(10 mmol/L)、10×酶缓冲液、MgCl2(25 mmol/L) 宝生物工程(大连)有限公司;1 kb DNA Ladder、溶菌酶、乙二胺四乙酸钠、Tris-base、异硫氰酸胍、焦碳酸二乙酯 加拿大BBI公司;琼脂糖 西班牙Agarose公司;蛋白酶K 美国Sigma公司;Gelred核酸染料 美国Biotium公司;细菌基因组提取试剂盒 天根生化科技(北京)有限公司;实验引物100 bp DNA Ladder 生工生物工程(上海)股份有限公司。
1.2 仪器与设备
HB031金属恒温加热器 上海博彩生物技术有限公司;SIM-F140AY65型制冰机 三洋国际贸易有限公司;1-15PK小型台式离心机 德国Sigma实验室离心机公司;Biospec-mini型岛津DNA/RNA/蛋白质分析装置日本岛津制作所;S1000高性能PCR仪、164-5050伯乐基础电源电泳仪、Syngene GeneGenius 凝胶成像系统美国Syngene有限公司;GL-88B涡旋混合器 海门市其林贝尔仪器制造有限公司。
1.3 方法
1.3.1 乳酸菌DNA基因组提取
用试剂盒提取乳酸菌基因组DNA,提取DNA保存于-20 ℃冰箱中。测定提取DNA的OD260nm、OD280nm及OD260nm/OD280nm值,DNA质量浓度ρ/(μg/mL)按下式计算[12]。
ρ= OD260nm×50×n
式中:50为比例系数;n为稀释倍数。
1.3.2 乳酸菌16S rRNA PCR
采用细菌16S rDNA基因的通用引物[13]:正向引物为27f(5’-AGA GTT TGA TCM TGG CTC AG-3’),反向引物为1 492r(5’- GGT TAC CTT GTT ACG ACT T-3’)对27 株菌提取的DNA进行扩增,扩增产物用2.0%琼脂糖进行凝胶电泳。将扩增产物送上海生物工程有限公司测序。
1.3.3 16S rRNA序列同源性分析
登陆NCBI(http://www.ncbi.nlm.nih.gov)数据库,用BLAST软件将16S rRNA测序结果与已知序列进行相似性分析[11],同时对27 个菌株的测序结果采用Unweighted Pair Group Method with Arithmetic Mean(UPGMA)法进行聚类分析,并构建各菌株的遗传距离聚类图。
1.3.4 RAPD随机引物的筛选及PCR反应体系和条件的优化
随机引物的筛选:通过对5 条不同随机扩增引物(表2)进行PCR反应,根据条带的多态性和亮度选择出最适引物,进行后续单因素试验。
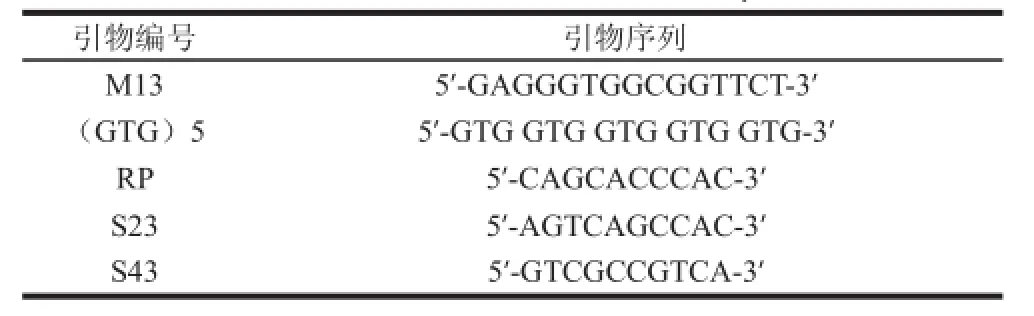
表2 随机引物编号及序列Table2 Primers of RAPD and their sequences
PCR反应体系优化:预设PCR反应体系[15]为5 U/μL Taq酶0.2 μL,10 μmol/L引物0.5 μL,模版DNA/引物用量=20,10×Taq酶缓冲液(无Mg2+)2.5 μL,25 mmol/L MgCl22.5 μL,2.5 mmol/L dNTPs 0.6 μL。
Mg2+浓度优化:预设PCR反应体系中Mg2+浓度分别设为1.0、1.5、2.0、2.5、3.0 mmol/L,其余试剂浓度保持不变,选择最佳Mg2+浓度。
模版DNA/引物用量比优化:预设PCR反应体系中模版DNA/引物用量的比例分别设为5、10、20、40、80,其余试剂浓度保持不变,选择最佳模版DNA/引物用量比例。
dNTP用量优化:预设PCR反应体系中dNTP用量分别设为30、60、90、120、150 μmol/L;其余试剂浓度保持不变,选择最佳dNTP用量。
PCR反应条件优化:预设PCR反应程序[15]为94 ℃预变性3 min,94 ℃变性1 min;50 ℃退火1 min;72 ℃延伸2 min;35 个循环,然后72 ℃延伸10 min。
退火温度的优化:预设PCR反应条件中退火温度分别设为46、47、48、49、50℃,其余条件保持不变,选择最佳退火温度。
PCR扩增产物通过1.0%琼脂糖凝胶电泳(70 V,1 h),Gelred染色后凝胶成像。通过电泳条带的数量、亮度、重复性和特异性条带来确定最佳反应条件。
1.3.5 RAPD扩增产物检测
扩增产物经1.5%琼脂糖凝胶电泳分离。取10 μL扩增产物与2 μL Loading Buffer混匀,点样于凝胶孔,用1 kb DNA Ladder作为分子质量标尺,电泳缓冲液为1×TAE,70 V恒压电泳60 min,Gelred染色后通过凝胶成像系统拍照。
1.3.6 RAPD 谱带分析
各菌株的扩增图谱上特定位点无条带的赋值为0,有条带的赋值为1,用NTsys 2.10e制作为“0,1”文件,采用UPGMA法进行聚类分析,并构建各菌株的遗传距离聚类图。
2 结果与分析
2.1 DNA提取和PCR扩增电泳图及16S rRNA序列同源性分析结果
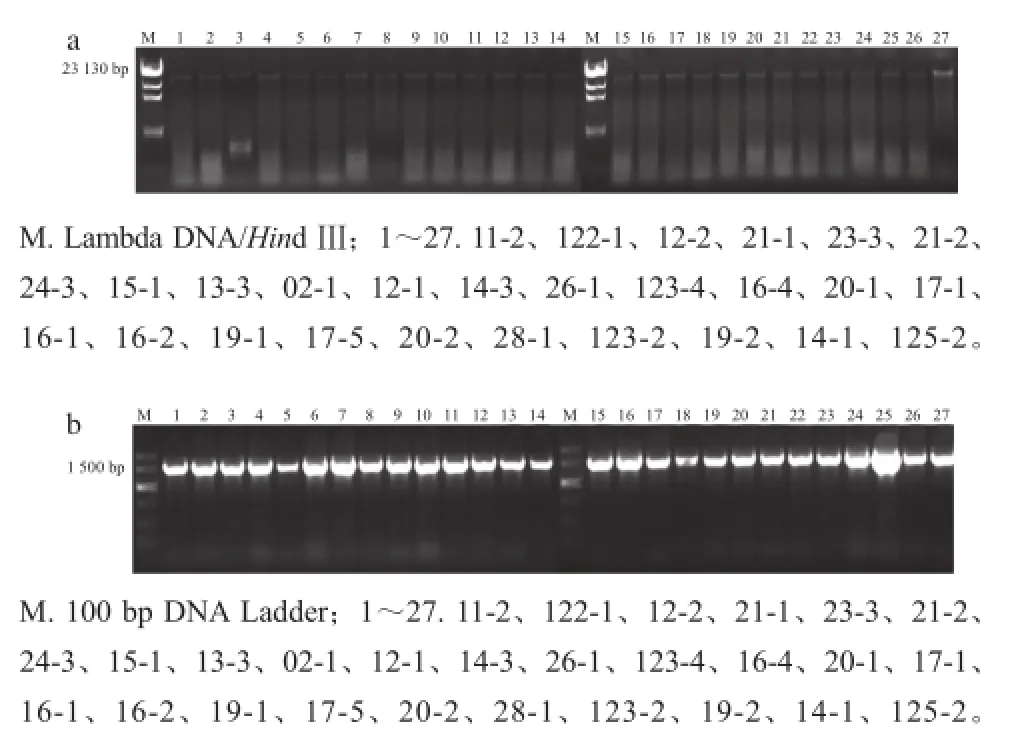
图1 DNA电泳图(a)和16S rRNA片段扩增图(b)Fig.1 Electrophoresis of genomic DNA (a) and 16S rRNA amplification products (b)
图1a显示了27 株菌的DNA提取结果,OD260nm/ OD280nm的范围在1.8~2.2之间,在23 000 bp处有明显的单一条带。图1b显示了27 株菌16S rRNA的PCR扩增结果,在1 500 bp处有明显的单一条带。说明各菌株基因组DNA均被成功提取出,同时成功扩增出目标片段。

图2 27株菌的16S rRNA系统发育树Fig.2 Phylogenetic tree of 27 strains based on 16S rRNA
由图2可知,27 株菌分为6 个大的分支,其中21-1、16-4、17-1、26-1、15-1、21-2、123-4、12-2、122-1、20-1、13-3、23-3、11-2、24-3、12-1、02-1、14-3、19-2位于一个分支,属于Lactobacillus casei或者Lactobacillus paracasei。125-2处于一个分支,属于Lactobacillus parabuchneri。16-1、16-2、19-1位于一个分支,属于Lactobacillus diolivorans。17-5、28-1、20-2位于一个分支,属于Lactobacillus plantarum。14-1位于一个分支,属于Lactobacillus helveticus。123-2位于一个分支,属于Leuconostoc mesenteroides。图中未能将Lactobacillus casei和Lactobacillus paracasei分开。
2.2 单因素试验结果
2.2.1 退火温度
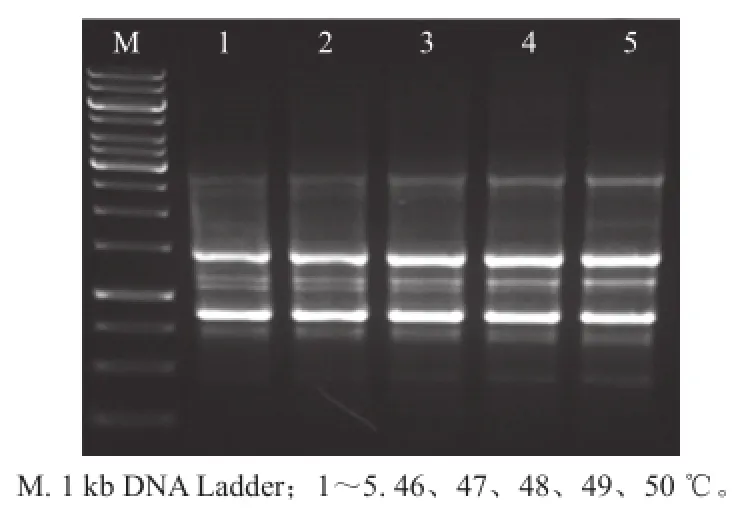
图3 不同退火温度下菌株23-3的RAPD扩增图谱Fig.3 Influence of PCR annealing temperature on RAPD profiles of strains 23-3
由图3可知,退火温度由46 ℃升到48 ℃过程中,条带的亮度逐渐增加,温度由48 ℃升到50 ℃的过程中,条带的亮度和数量保持不变,同时条带整体重现性较好,考虑到退火温度较低时引物与模版结合更好,所以PCR反应中退火温度选用48 ℃。
2.2.2 模版DNA/引物用量比
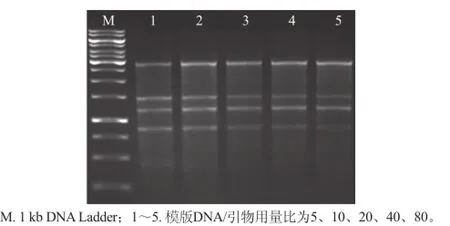
图4 不同模版DNA/引物用量对菌株23-3 RAPD扩增图谱的影响Fig.4 Influence of DNA template/primer ratio on RAPD profiles of strains 23-3
由图4可知,电泳条带的整体重现性很好,在模板DNA/引物用量比为5和10的情况下出现5 个条带,同时考虑到在用量比为10情况下条带亮度更佳,所以25 μL PCR反应体系模版DNA/引物用量比选用10。
2.2.3 Mg2+浓度

图5 不同Mg2+浓度对菌株23-3 RAPD扩增图谱的影响Fig.5 Influence of Mg2+concentration on RAPD profiles of strains 23-3
由图5可知,电泳条带整体重现性很好,随着Mg2+浓度的升高,条带的亮度逐渐增加,在Mg2+浓度为3.0 mmol/L时效果最佳,所以25 μL PCR反应体系中Mg2+的浓度选用3.0 mmol/L。
2.2.4 dNTP用量
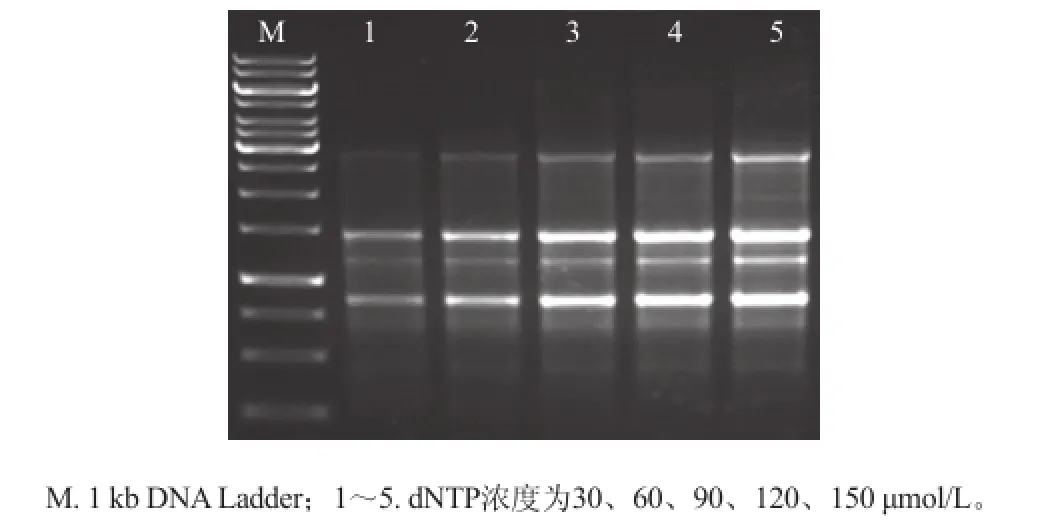
图6 不同dNTP用量对菌株23-3 RAPD扩增图谱的影响Fig.6 Influence of dNTP amount on RAPD profiles of strains 23-3
由图6可知,随着dNTP用量的增加条带的亮度显著增加,在150 μmol/L时达到最亮,同时条带整体重现性较好,在150 μmol/L条件下达到了7 条,所以25 μL PCR反应体系中dNTP用量选用150 μmol/L。经过单因素试验,得到优化的RAPD扩增程序为:94 ℃预变性3 min,94 ℃变性1 min;48 ℃退火1 min;72 ℃延伸2 min;35 个循环,然后72 ℃延伸10 min。优化的PCR反应体系为:25 μL总反应体积,模版DNA/引物用量=10,10×Taq酶缓冲液(无Mg2+)2.5 μL,25 mmol/L MgCl23.0 μL,2.5 mmol/L dNTPs 1.5 μL,10 μmol/L引物0.5 μL,5 U/μL Taq酶0.2 μL,灭菌纯水补至25 μL。对4 株乳酸菌标准菌株和27 株分离乳酸菌进行RAPD扩增。
2.3 整体乳酸菌RAPD图谱
由图7可知,27 株西藏牦牛奶酪分离乳酸菌和4 株乳酸菌标准菌株用M13引物扩增并电泳后,所有菌株均扩增出明显的条带,同时16S rRNA鉴定出相同属的菌株之间可以看出明显的相似性,各个菌株之间也存在特异性,扩增出的条带数目在3~10 条之间,所获得的片段分子质量大小在250~3 000 bp之间。

图7 牦牛奶酪中31株乳酸菌的RAPD扩增图谱Fig.7 RAPD profiles of 31 strains isolated from yak milk cheeses
2.4 聚类分析结果
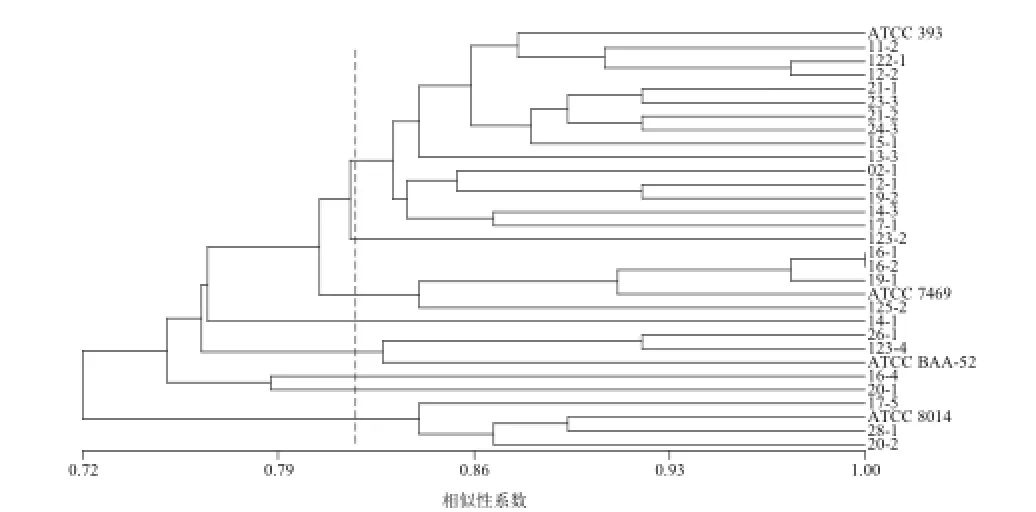
图8 31 株乳酸菌亲缘关系聚类树状图Fig.8 Cluster dendrogram generated from similarity coefficient matrix of 31 lactic acid bacteria
用NTsys2.10e软件,按照UPMGA法,对供试菌株之间的遗传相似系数矩阵进行聚类分析,得到聚类分析图。由图8可知,当相似性系数为0.82时,分离自牦牛奶酪的27 株乳酸菌和4 株乳酸菌标准菌株被分成了8 个组。第一组包括11-2、122-1、12-2、21-1、23-3、21-2、24-3、15-1、13-3、02-1、12-1、19-2、14-3、17-1和casei标准菌株,它们都是Lactobacillus casei。第二组只有一支菌123-2,为Leuconostoc mesenteroides。第三组中16-1、16-2、19-1聚为第一分支,它们是Lactobacillus diolivorans。另一分支为125-2,是Lactobacillus parabuchneri,Lactobacillus rhamnosus单独成为一个分支。第四组只有一支菌14-1,为Lactobacillus helveticus。第五组中paracasei标准菌株单独成为一分支,26-1和123-4聚为另一分支,为Lactobacillus paracasei。第六组只有一支菌16-4,为Lactobacillus casei。第七组只有一支菌20-1,为Lactobacillus casei。第八组包括17-5、28-1、20-2和plantarum标准菌株,它们都是Lactobacillus plantarum。通过聚类分析图和16S rRNA测序结果之间的相互比较可以发现RAPD聚类分析结果同16S rRNA测序结果基本一致,但是16S rRNA 进化树图并不能够将Lactobacillus paracasei和Lactobacillus casei两个亚种区分开,而利用RAPD聚类分析结果可以将其区分开。由此可以得出在乳酸菌基因型分类鉴定中,RAPD对于亲缘型很近菌株的鉴定效果要优于16S rRNA同源性分析,更适合于西藏牦牛奶酪中乳酸菌亲缘性关系分析。
3 讨 论
西藏地区居民主要采用传统手工方法制作奶酪,由于产地、乳酸菌种、制作工艺的不同,导致奶酪风味和质地的多样化。乳酸菌又是奶酪制作过程中的核心,是奶酪品质好坏的关键。建立系统的鉴定方法对奶酪中乳酸菌进行分析,会对当地奶酪制品质量的提高起到很大的借鉴作用。
目前对于细菌的鉴定主要有表型鉴定和基因型鉴定两种方法。表型鉴定不仅在操作上具有一定的复杂性,同时在鉴定的水平上只能达到属的水平,难以达到种水平的鉴定[16]。而RAPD技术运用随机引物对不同种群的基因组DNA进行扩增,不仅可以在分子水平上对菌株之间进行分类和鉴定,还可以整体基因组DNA进行多态性检测,同时操作简易,准确性高。许多国外学者已经成功地将RAPD技术应用于乳酸菌的分离鉴定。如Gevers等[17]运用随机引物(GTG)5成功地完成对干香肠中乳杆菌亚种的分类和鉴定。Corroler等[18]运用RAPD技术对不同季节采集的牛奶中L. lactis subsp. lacti和L. lactis subsp. cremoris两个亚种进行分类和鉴定。Cocconcelli等[19]运用RAPD技术区分开嗜酸乳杆菌(L. acidophilus)、瑞士乳杆菌(L. helveticus)、干酪乳杆菌(L. casei)、植物乳杆菌(L. plantarum)、粪肠球菌(Ent. faecalis)、屎肠球菌(Ent. faecium)、嗜热链球菌(Streptococcus thermophilus)等菌株。Nigatu等[20]利用RAPD技术对45株乳杆菌进行区分,所有菌株均扩增出了不同的多态性条带,并且相似度在72%以上,对乳杆菌属进行了最集中和广泛的研究。
根据决策对象、决策指标、聚类结果建立决策表,令决策表DT=(U,P∪D),子集D={d}为决策属性集,子集P={aj|j=1,2,…,m}为条件属性集,U={i|i=1,2,…,n}为论域,aij表示决策对象i在条件属性aj上的值。
本研究利用16S rRNA和RAPD技术对西藏牦牛奶酪中乳酸菌的基因型和遗传多样性进行了分析,用NTsys 2.10e软件计算出了31 株乳酸菌菌株之间的遗传相似系数矩阵,并用UPMGA法作聚类分析图,与16S rRNA序列分析结果相对比。在与16S rRNA序列分析结果的比较中,两种基因型方法得出的结果基本是一致的。同时在RAPD的聚类图中将L. casei和L. paracasei这两个亚种有效地区分开来,这是16S rRNA技术在进行同源性分析时做不到的,同时相关文献中也无类似报道。综合比较结果考虑,RAPD技术在乳酸菌分类鉴定中有一定的应用价值,同时它有操作简单快速、种间和种内区分效率高、费用低的优势,与其他表型和基因型方法相结合,可提高菌种鉴定效率和准确性。
参考文献:
[1] WILLIAMS J G K, KUBELIK A R, LIVAK K J, et al. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J]. Nucleic Acids Research, 1990, 18(22): 6531-6535.
[2] WELSH J, MCCLELLAND M. Fingerprinting genomes using PCR with arbitrary primers[J]. Nucleic Acids Research, 1990, 18(24): 7213-7218.
[3] TYLER K D, WANG G, TYLER S D, et al. Factors affecting reliability and reproducibility of amplification-based DNA fingerprinting of representative bacterial pathogens[J]. Journal of Clinical Microbiology, 1997, 35(2): 339-346.
[4] MICHELI M R, BOVA R, PASCALE E, et al. Reproducible DNA fingerprinting with the random amplified polymorphic DNA (RAPD) method[J]. Nucleic A cid Research, 1994, 22(10): 1921-1922.
[5] PSONI L, KOTZAMANIDIS C, YIANGOU M, et al. Genotypic and phenotypic diversity of Lactococcus lactis isolates from Batzos, a Greek PDO raw goat milk cheese[J]. International Journal of Food Microbiology, 2007, 114(2): 211-220.
[7] LPPEZ I, TORRES C, RUIZ-LARREA F. Genetic typification by pulsed-field gel electrophoresis (PFGE) and randomly amplified polymorphic DNA (RAPD) of wild Lactobacillus plantarum and Oenococcus oeni wine strains[J]. European Food Research and Technology, 2008, 227(2): 547-555.
[8] HOSSEINI S V, ARLINDO S, BOHME K, et al. Molecular and probiotic characterization of bacteriocin-producing Enterococcus faecium strains isolated from nonfermented animal foods[J]. Journal of Applied Microbiology, 2009, 107(4): 1392-1403.
[9] 高大威, 朱光华, 王立欣, 等. 三种食物中乳酸菌的亲缘性关系及抗氧化活性研究[J]. 燕山大学学报, 2012, 36(1): 84-88.
[10] 秦丹. 四川腊肉和香肠加工贮藏过程中微生物系统的RAPD分析[D].雅安: 四川农业大学, 2011: 21-53.
[11] 李平兰, 贺稚非. 食品微生物学实验原理与技术[M]. 北京: 中国农业出版社, 2005: 264.
[12] 林万明. 细菌分子遗传学分类鉴定法[M]. 上海: 上海科学技术出版社, 1990: 48-83.
[13] AO X, ZHANG X, ZHANG X, et al. Identification of lactic acid bacteria in traditional fermented yak milk and evaluation of their application in fermented milk products[J]. Journal of Dairy Science, 2012, 95(3): 1073-1084.
[14] WARD L J H, TIMMINS M J. Differentiation of Lactobacillus casei, Lactobacillus paracasei and Lactobacillus rhamnosus by polymerase chain reaction[J]. Letters in Applied Microbiology, 1999, 29(2): 90-92.
[15] 朱红梅, 廖万清, 戴建新, 等. 用微小卫星引物PCR鉴定红色毛癣菌和须癣毛癣菌[J]. 第二军医大学学报, 2001, 22(3): 252-254.
[16] 王庭柱. 乳源球菌菌株的多相分类鉴定[D]. 哈尔滨: 东北农业大学, 2006: 3-7.
[17] GEVERS D, HUYS G, SWINGS J. Applicability of rep-PCR fingerprinting for identification of Lactobacillus species[J]. FEMS Microbiology Letters, 2001, 205(1): 31-36.
[18] CORROLER D, MANGIN I, DESMASURES N, et al. An ecological study of lactococci isolated from raw milk in the Camembert cheese registered designation of origin area[J]. Applied and Environmental Microbiology, 1998, 64(12): 4729-4735.
[19] COCCONCELLI P S, PORRO D, GALANDINI S, et al. Development of RAPD protocol for typing of strains of lactic-acid bacteria and enterococci[J]. Letters in Applied Microbiology, 1995, 21(6): 376-379.
[20] NIGATU A, AHRNÉ S, MOLIN G. Randomly amplified polymorphic DNA (RAPD) profiles for the distinction of Lactobacillus species[J]. Antonie van Leeuwenhoek, 2001, 79(1): 1-6.
Randomly Amplified Polymorphic DNA Analysis of Lactic Acid Bacteria from Yak Milk from Tibet Area
JIANG Hou-yang1, ZHAO Guo-hua1,2, YANG Ji-xia1,2,*
(1. College of Food Science, Southwest University, Chongqing 400715, China; 2. Chongqing Key Laborat ory of Agricultural Product Processing, Chongqing 400715, China)
Objective: This study analyzed the homology of 27 strains of lactic acid bacterial genotype from Tibetan yak cheese by randomly amplif ed polymorphic DNA (RAPD) method. Methods: To establish a reliable RAPD protocol with 5 primers, the optimal primers and PCR conditions for the quality and reproducibility of RAPD prof les were investigated, including Mg2+concentration, dNTP amount, annealing temperature, and DNA template/primer ratio. The optimal PCR procedure and reaction reagent concentration were employed for RAPD amplif cation of 27 isolates from yak milk cheeses and 4 standard strains of lactic acid bacteria. Electrophoretic profiles were analyzed by NTsys 2.10e software, genetic similarity coeff cients were calculated and a cluster dendrogram was plotted. Results: Genetic similarity coeff cients of 31 strains ranged from 0.72 to 1.00. The cluster dendrogram showed that 31 isolates were classif ed into 8 groups using the similarity coeff cient of 0.82 as a cut-off point. The group discrimination corresponded well with the species assignment by 16S rRNA sequence analysis, and Lactobacillus casei and Lactobacillus paracasei were separated successfully. Conclusion: The RAPD technique was useful for relationship analysis of lactic acid bacteria isolated from Tibetan yak milk cheeses.
Tibet; yak milk cheese; lactic acid bacteria; randomly amplif ed polymorphic DNA (RAPD); cluster analysis
TS252.54
A
1002-6630(2014)23-0215-06
10.7506/spkx1002-6630-201423042
2013-12-19
西南大学博士启动基金项目(SWU113035);中央高校基本科研业务费专项资金项目(XDJK2009C039)
蒋厚阳(1989—),男,硕士研究生,研究方向为食品安全与质量控制。E-mail:rxxlcool@126.com
*通信作者:杨吉霞(1978—),女,讲师,博士,研究方向为食品生物技术。E-mail:yangjx@188.com

